Os adenovírus desempenham um papel importante na etiologia da infecção aguda grave do trato respiratório inferior, especialmente entre crianças. O objetivo do estudo foi avaliar a detecção do adenovírus humano (HAdV) por diferentes métodos (imunofluorescência direta – DFA e reação em cadeia da polimerase nested – nested PCR) em amostras coletadas de diferentes populações de pacientes pediátricos.
MétodosO material foi coletado de crianças portadoras de doença cardíaca congênita (DCC – 123 aspirados nasais coletados em 2005, 2007 e 2008) e de crianças da comunidade (CC – 165 aspirados nasais coletados em 2008). As crianças eram consideradas elegíveis se apresentassem infecção respiratória aguda (IRA) de provável etiologia viral, com até sete dias de início dos sintomas. Todas as amostras coletadas no estudo foram avaliadas por meio de DFA e nested PCR.
ResultadosDe 209 amostras incluídas, 43 (14,8%) foram positivas em pelo menos um dos testes feitos: 17/165 (10,3%) das crianças da comunidade e 26/125 (20,8%) das crianças cardiopatas. As taxas de detecção por nested PCR foram 15/165 (9,1%) em crianças da comunidade e 24/125 (19,2%) em crianças cardiopatas. O método molecular mostrou maiores taxas de detecção quando comparado com a DFA (p<0,001). A análise univariada mostrou que as crianças portadoras de cardiopatia congênita apresentaram chance significativamente maior de adquirir HAdV (odds ratio 2,3; IC 95%: 1,18‐4,43).
ConclusõesBaseado nos resultados obtidos na presente avaliação, recomenda‐se a vigilância de rotina em pacientes de risco (DCC) por métodos moleculares, que melhora o fluxo diagnóstico e a eficiência da detecção.
Adenoviruses play an important role in the etiology of severe acute lower respiratory infection, especially in young children. The aim of the present study was to evaluate the Human Adenovirus (HAdV) detection by different methods (Direct Fluorescence Assay – DFA and Nested Polymerase Chain Reaction – nested PCR), among samples collected from different groups of pediatric patients.
MethodsCollection of samples was made in children with congenital heart disease (CHD – 123 nasal aspirates collected in the years of 2005, 2007 and 2008) and in community children (CC – 165 nasal aspirates collected in 2008). Children were eligible if they presented acute respiratory infection (ARI) of probable viral etiology, within up to 7 days of symptoms’ onset. All studied samples were evaluated by DFA and nested PCR assay.
ResultsOf the 290 samples included during the study period, 43 (14.8%) were positive on at least one test: 17/165 (10.3%) of the CC and 26/125 (20.8%) of the CHD children. The nested PCR detection rates in the community children were 15/165 (9.1%), and for children with CHD, 24/125 (19.2%). Molecular method showed higher detection rates when compared to the DFA test (p<0.001). Univariate analysis showed that children with congenital heart disease presented a significantly higher chance for acquiring the HAdV (Odds Ratio 2.3; 95% CI: 1.18‐4.43).
ConclusionsBased on data obtained in the present evaluation, we suggest that a routine surveillance should be performed in high risk patients by molecular methods, thus improving diagnostic flow and efficiency.
Os adenovírus desempenham um papel importante na etiologia da infecção aguda grave do trato respiratório inferior, especialmente em crianças menores.1 Os adenovírus humanos (HAdV) disseminam‐se rapidamente em ambientes fechados, o que muitas vezes resulta em doença epidêmica em comunidades populosas. Além disso, a infecção por adenovírus é clinicamente difícil de distinguir de outras infecções respiratórias virais ou bacterianas.2 Essa combinação de fatores indica que métodos microbiológicos clínicos mais eficazes são necessários para a detecção da doença respiratória aguda causada por adenovírus.
Antes do advento da reação em cadeia de polimerase (PCR), a imunofluorescência direta (DFA) e o isolamento viral eram os métodos mais sensíveis disponíveis para a detecção dos vírus respiratórios.3,4 Entretanto, um número significativo de amostras de pacientes com infecção respiratória viral clinicamente compatível era incorretamente classificado como negativo pela DFA e pela cultura viral, o que implica falha na identificação do vírus causador em uma percentagem significativa de casos.5‐7 Os métodos moleculares demonstram maior sensibilidade quando comparados com ensaios convencionais para detecção de adenovírus em amostras respiratórias.8,9
Na população pediátrica, o HAdV é o segundo patógeno mais frequentemente detectado10 e o vírus é responsável por 5% a 15% das doenças respiratórias.10,11 Amostras clínicas de crianças geralmente apresentam cargas virais maiores quando comparadas com as dos pacientes adultos.12 Os estudos brasileiros descrevem uma frequência que varia de 3% a 7,1%, de acordo com a técnica usada.13‐16 Outro grupo de risco para infecção respiratória adquirida por HAdV são as crianças com cardiopatias congênitas, embora não haja publicação que comprove esse fato.
Nesse contexto, o objetivo do presente estudo foi avaliar a ocorrência de HAdV por meio de diferentes métodos (imunofluorescência direta – DFA e reação em cadeia da polimerase nested – nested PCR) em amostras coletadas de crianças de diferentes populações.
MétodoEste estudo transversal incluiu duas populações distintas avaliadas de 2005 a 2008.
1) Crianças com doença cardíaca congênita (DCC): durante 2005, 2007 e 2008, 123 pacientes ambulatoriais com doença cardíaca congênita atendidos no Serviço Pediátrico de Doenças Cardíacas Congênitas foram incluídos. Durante 2006, a ala de cardiopatia congênita passou por uma reforma estrutural e administrativa, o que impossibilitou a coleta das amostras. Dos pacientes incluídos, 49,7% eram do sexo masculino, com idade média de 3,9 anos (mediana de 2,9 anos), variação de um ano e seis meses a 11 anos.
2) Crianças da comunidade (CC): em 2008, foram incluídos 165 pacientes ambulatoriais atendidos por um pediatra em uma unidade básica de saúde. Entre esses pacientes, 47,2% eram do sexo masculino e a idade média era de 3,9 anos (mediana de três), variação de sete meses a 11 anos.
Para ambos os grupos, as crianças eram elegíveis se apresentassem infecção respiratória aguda (IRA) de provável etiologia viral, de preferência com intervalo de até sete dias do início dos sintomas até a coleta das amostras. No grupo de crianças da comunidade, os pacientes foram incluídos quando os pais ou responsáveis procuraram atendimento médico em consequência de uma infecção respiratória aguda. No grupo DCC, a inclusão de pacientes foi definida por um médico durante uma consulta de rotina previamente agendada. A população DCC foi considerada em risco para complicações de infecção respiratória viral; portanto, foram coletadas amostras de crianças com mais de sete dias do início dos sintomas (variação de um‐60 dias). Os sintomas respiratórios avaliados foram: coriza, tosse, dor de garganta ou congestão nasal; os sintomas sistêmicos foram: febre, dor de cabeça, mal‐estar, calafrios ou fadiga. Foi obtido termo de consentimento livre e esclarecido de todos os adultos responsáveis por cada paciente antes da inclusão.
Todas as amostras nasais foram mantidas a 4°C e imediatamente transportadas para o laboratório. Uma alíquota (1mL) foi separada para análise molecular e armazenada a –70°C. O volume da amostra restante foi avaliado no mesmo dia por DFA, de acordo com estudo anterior.17 Em seguida, uma alíquota de cada amostra armazenada teve o DNA extraído com o kit de extração QIamp DNA Blood extraction kit (Qiagen, EUA), de acordo com as instruções do fabricante, seguido por nested PCR para a detecção de todos os serótipos do adenovírus, como anteriormente descrito.18
Todas as amostras estudadas foram avaliadas pela técnica de DFA. Os testes foram feitos com o kit Simulfluor Respiratory Screen and Panel (Chemicon Int., EUA), de acordo com as instruções do fabricante.
Os primers usados para a detecção tiveram como alvo a região do gene Hexon do HAdV. O par de primers exteriores, hex1deg (5’‐GCC SCA RTG GKC WTA CAT GCA CAT C‐3’) e hex2deg (5’‐CAG CAC SCC ICG RAT GTC AAA‐3’) resultaram em um produto de 301bp. O segundo par de primers (nested primers), nehex3deg (5’‐GCC CGY GCM ACI GAI ACS TAC TTC‐3’) e nehex4deg (5’‐CCY ACR GCC AGI GTR WAI CGM RCY TTG TA‐3’), produziu um amplicon de 171bp.
Para a primeira reação de amplificação, 5μL de DNA extraído foram colocados em um tubo com uma mistura de reação com 2,5μL de 10× buffer (200mM Tris‐HCl, pH 8,4, 500mM de KCl), 3,5μM de MgCl2, 0,5μM de primers Hex1deg e Hex2deg, 1μL de mistura de dNTPs com 20mM de cada nucleotídeo, 2,5U de Platinum® TaqDNA polimerase (Invitrogen, Brasil) e água MilliQ autoclavada, em um volume final de 25μL.
Para a segunda reação de amplificação, 2μL do amplicon da primeira reação foram colocados em um tubo com uma mistura de reação constituída por 2,5μL de 10× buffer (200mM Tris‐HCl, pH 8,4, 500mM KCl), 3,5μM de MgCl2, 0,5μM de primers Nehex3deg e Nehex4deg, 1μL de mistura de dNTPs, com 20mM de cada nucleotídeo, 2,5U de Platinum® TaqDNA polimerase (Invitrogen, Brasil) e água MilliQ autoclavada, em um volume final de 25μL. Controles positivos (Adenovírus Sorotipo 3) e controle redundante negativo (água MilliQ autoclavada) e foram incluídos em cada série. Foram considerados resultados positivos quando o amplicon foi visualizado após eletroforese em gel de agarose a 2%. A sensibilidade da reação foi padronizada e apresentou um limite de detecção de 10‐4TCID50/mL.
A associação entre diferentes variáveis categóricas (variável resposta: presença ou ausência de HAdV; variável independente: crianças da comunidade e crianças com doença cardíaca congênita) foi avaliada pelo teste do qui‐quadrado e a razão de probabilidade univariada (SPSS, versão 11.5). Valor de p<0,05 foi considerado estatisticamente significativo. Para comparar os métodos usados, o teste Kappa foi feito (R software versão 2.11.1) e a interpretação dos coeficientes foi feita com o uso da classificação de Altman.19
O presente estudo foi aprovado pelo Comitê de Ética em Pesquisa do Hospital São Paulo e da Universidade Federal de São Paulo (1654/09).
ResultadosAs amostras de 290 pacientes sintomáticos foram analisadas [48,6% (141/290) do sexo feminino e 51,4% (149/290) do sexo masculino], 56,9% (165/290) de crianças da comunidade e 43,1% (125/290) de crianças com DCC. O tempo de início dos sintomas até o momento da coleta variou de um a 60 dias.
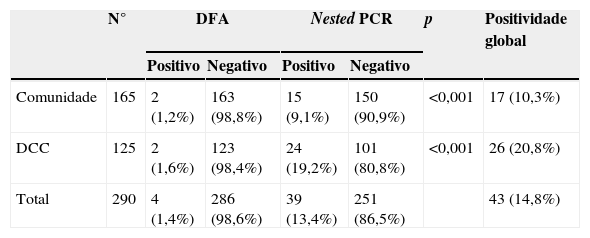
Entre as 290 amostras de pacientes sintomáticos incluídas durante o período do estudo, 41 (14,1%) foram positivas em pelo menos um teste: 17/165 (10,3%) crianças da comunidade, 26/125 (20,8%) crianças com DCC. A tabela 1 mostra a detecção diferencial entre os ensaios de acordo com as diferentes populações estudadas. No geral, 4/290 (0,7%) das amostras foram DFA positivas e 39/290 (13,40%) das amostras foram nested PCR positivas. O método da nested PCR apresentou uma maior taxa de detecção, estatisticamente significativa, entre todas as populações estudadas quando comparada com o DFA (p<0,001). Comparando‐se os grupos de crianças por métodos moleculares, as do grupo DCC apresentaram maior taxa de detecção (p=0,02).
Detecção de HAdV por DFA e nested PCR entre as diferentes populações estudadas
| N° | DFA | Nested PCR | p | Positividade global | |||
|---|---|---|---|---|---|---|---|
| Positivo | Negativo | Positivo | Negativo | ||||
| Comunidade | 165 | 2 (1,2%) | 163 (98,8%) | 15 (9,1%) | 150 (90,9%) | <0,001 | 17 (10,3%) |
| DCC | 125 | 2 (1,6%) | 123 (98,4%) | 24 (19,2%) | 101 (80,8%) | <0,001 | 26 (20,8%) |
| Total | 290 | 4 (1,4%) | 286 (98,6%) | 39 (13,4%) | 251 (86,5%) | 43 (14,8%) | |
DCC, crianças com doença cardíaca congênita; DFA, imunofluorescência direta; PCR, reação em cadeia de polimerase.
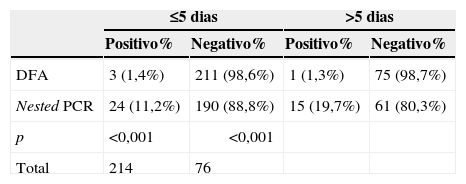
A análise do tempo decorrido desde o início dos sintomas até o dia da coleta mostrou uma diferença estatística entre os métodos DFA e nested PCR (tabela 2). Entre as crianças da comunidade, a média±desvio padrão, a mediana e o intervalo desde o início dos sintomas foram respectivamente 3,2±2,72; 3; 1‐20 dias; e entre as crianças do grupo DCC 6,5±7,1; 5; 1‐60 dias. O método nested PCR, em comparação com a DFA, detectou maior número de casos, tanto para amostras coletadas com ≤5 dias desde o início dos sintomas como para amostras com >5 dias desde o início dos sintomas.
Tempo do início dos sintomas de acordo com o teste usado
| ≤5 dias | >5 dias | |||
|---|---|---|---|---|
| Positivo% | Negativo% | Positivo% | Negativo% | |
| DFA | 3 (1,4%) | 211 (98,6%) | 1 (1,3%) | 75 (98,7%) |
| Nested PCR | 24 (11,2%) | 190 (88,8%) | 15 (19,7%) | 61 (80,3%) |
| p | <0,001 | <0,001 | ||
| Total | 214 | 76 | ||
DFA, imunofluorescência direta; PCR, reação em cadeia de polimerase.
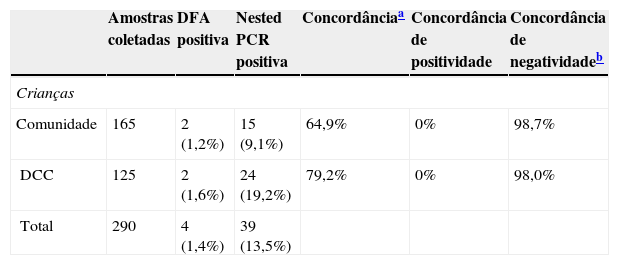
A tabela 3 mostra a concordância entre os testes avaliados. A concordância geral foi baixa, principalmente entre as crianças da comunidade. A concordância relativa aos resultados negativos foi elevada para ambas as populações estudadas.
Avaliação de concordância entre os testes usados
| Amostras coletadas | DFA positiva | Nested PCR positiva | Concordânciaa | Concordância de positividade | Concordância de negatividadeb | |
|---|---|---|---|---|---|---|
| Crianças | ||||||
| Comunidade | 165 | 2 (1,2%) | 15 (9,1%) | 64,9% | 0% | 98,7% |
| DCC | 125 | 2 (1,6%) | 24 (19,2%) | 79,2% | 0% | 98,0% |
| Total | 290 | 4 (1,4%) | 39 (13,5%) | |||
DCC, crianças com doença cardíaca congênita; DFA, imunofluorescência direta; PCR, reação em cadeia de polimerase.
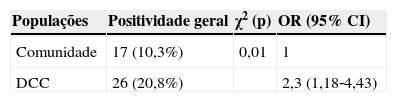
A tabela 4 mostra o resultado da análise da razão de probabilidade univariada de ocorrência do HAdV entre as diferentes populações estudadas. A variável resposta foi a presença ou ausência de HAdV e a variável independente foi o grupo estudado: as crianças da comunidade e as crianças com DCC. As crianças com DCC apresentaram aproximadamente duas vezes mais chance de infecção adquirida por HAdV quando comparadas com as crianças da comunidade.
Análise univariada da razão de chances (odds ratio – OR) da ocorrência de HAdV entre as populações estudadas
| Populações | Positividade geral | χ2 (p) | OR (95% CI) |
|---|---|---|---|
| Comunidade | 17 (10,3%) | 0,01 | 1 |
| DCC | 26 (20,8%) | 2,3 (1,18‐4,43) |
DCC, crianças com doença cardíaca congênita; OR, odds ratio; IC95%, Intervalo de Confiança de 95%.
Entre as crianças da comunidade, a taxa de detecção de adenovírus foi semelhante à descrita na literatura nacional, variação de 6% a 7,1%.13‐16 Um estudo brasileiro recente avaliou 1.121 amostras coletadas de crianças hospitalizadas e encontrou ocorrência de adenovírus em 15,8%, o que corrobora os resultados do presente estudo.10 Estudos internacionais relatam a ocorrência do HAdV variando de 2% a 3,8% em populações pediátricas, o que é ligeiramente menor quando comparado com os nossos resultados.20‐22 Estudos com crianças com cardiopatia congênita demonstram a circulação dos vírus respiratórios, com taxas relativamente altas de ocorrência, e mostram que esses agentes devem ser considerados na prática clínica e diagnóstica.22 O presente estudo é o primeiro a descrever a infecção respiratória por HAdV em crianças brasileiras com DCC. A taxa de detecção de HAdV nos cardiopatas foi maior (20,8%) quando comparada com a de crianças da comunidade (10,3%). Vale ressaltar que, entre as crianças com cardiopatia congênita, a coleta de amostras foi feita durante a consulta de rotina. Se o paciente apresentava infecção respiratória durante a consulta agendada, a amostra era colhida. Portanto, a taxa encontrada pode estar subestimada, porque prováveis casos podem ter sido perdidos por conta da falta de demanda dos pacientes. Uma hipótese para a maior detecção pode estar relacionada com a presença de comorbidades entre essas crianças.23
Ao avaliar os dois testes usados neste estudo, algumas questões importantes para clínicos e administradores hospitalares são levantadas: Qual é o melhor teste? Até qual dia é o período ideal para coletar a amostra? Há necessidade de repetir o teste? Qual é o número ideal de amostras para o fluxo de rotina do laboratório? Qual é o impacto de testes negativos e positivos em pacientes de alto risco? Alguns desses aspectos puderam ser avaliados no presente estudo.
A análise da associação entre o início dos sintomas no paciente e os resultados dos testes laboratoriais mostrou que o método molecular foi mais sensível do que o DFA e essa diferença foi mais evidente após o quinto dia do início dos sintomas (p<0,001). A menor detecção por DFA estava relacionada com a baixa carga viral nas amostras respiratórias.24 As taxas de detecção mais elevadas entre os pacientes com mais de cinco dias do início dos sintomas eram esperadas por conta da maior sensibilidade do teste nested PCR.25,26 Para o sucesso do teste DFA são necessários alguns fatores‐chave, como a boa coleta de amostras e pessoal de laboratório experiente,10 enquanto o nested PCR, baseado na amplificação de ácidos nucleicos, pode detectar um número muito baixo de partículas virais.26,10
A análise univariada mostrou que crianças com cardiopatia congênita têm maior risco de adquirir o HAdV (OR: 2,29) quando comparadas com as crianças da comunidade. Utokaparch (2011)27 descreveu que pacientes pediátricos com infecção respiratória do trato inferior apresentavam carga viral mais elevada quando comparados com pacientes com infecção do trato respiratório superior. Echavarria et al.28 descreveram que crianças têm 2 a 3,5 vezes mais chances de ser infectadas por HAdV do que os adultos. Esses dados corroboram os resultados do presente estudo.
Uma das limitações do estudo foi a ausência de pesquisa de outros vírus respiratórios. Entre as amostras negativas para HAdV, outros vírus poderiam estar presentes. Como resultado da alta sensibilidade dos métodos moleculares, as infecções virais poderiam ser detectadas semanas após o término dos sintomas,29,30 mas, no presente estudo, as amostras incluídas foram coletadas de crianças com infecção respiratória aguda, o que diminui a chance de detecção de uma infecção anterior. Outra limitação do estudo foi a falta de coleta de amostras durante 2006; no entanto, a ideia principal do estudo foi pesquisar a ocorrência do vírus na comunidade (crianças da comunidade) e em uma população com um risco elevado de complicações após uma infecção respiratória viral (crianças com DCC). Alguns estudos descrevem uma ocorrência de adenovírus durante diferentes anos e ao longo do ano,31,32 com padrões de ocorrência semelhantes; portanto, a coleta de amostras durante anos diversos aparentemente não comprometeu o objetivo do estudo.
Três parâmetros podem indicar a validade da escolha do teste diagnóstico: o custo por amostra, o tempo de resposta (tempo necessário para fornecer o resultado) e a confiabilidade do teste. Em um estudo publicado em 2009, Mahony et al.33 compararam o custo do DFA com o da técnica molecular para o diagnóstico de infecção pelo vírus respiratório e descreveram que o teste molecular tinha um custo ligeiramente maior, mas, ao mesmo tempo, apresentava um custo‐benefício excelente por conta da redução do tempo de hospitalização. Diferentes estudos descreveram o tempo de resposta do DFA e do PCR. Ambos os testes apresentaram a capacidade de fornecer resultados em um dia.34,35 Gharabaghi et al.34 compararam a sensibilidade e especificidade do DFA e do ensaio molecular comercial. Esse estudo mostrou uma baixa sensibilidade do teste DFA em comparação com o teste molecular para todos os vírus pesquisados. O adenovírus apresentou a menor sensibilidade (38,1%).34
Sugerimos que a vigilância de rotina seja feita em pacientes de alto risco por métodos moleculares, o que melhora o fluxo de diagnóstico e a eficiência. Nossos resultados também demonstraram que, em amostras com mais de cinco dias do início dos sintomas, o método nested PCR pode ser a melhor opção.
FinanciamentoConselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq), Brasil por meio de bolsa de estudo para DP.
Conflitos de interesseOs autores declaram não haver conflitos de interesse.