: El adenocarcinoma es actualmente el tipo más común de cáncer de pulmón. Las mutaciones en EGFR son predictivas de respuesta a TKI y son mutuamente excluyentes con mutaciones en KRAS. El análisis molecular del DNA libre circulante (cfDNA) ha prometido ser una herramienta útil para realizar estas determinaciones. Este estudio compara tres diferentes técnicas para determinar mutaciones en KRAS y EGFR en muestras de biopsia y sangre de pacientes con adenocarcinoma de pulmón.
Pacientes y Método: Se incluyeron pacientes con sospecha de cáncer de pulmón y se almacenaron muestras de plasma y suero. En los pacientes con diagnóstico confirmado de adenocarcinoma se analizó el estado mutacional de EGFR y KRAS en biopsia de tejido, plasma y suero mediante COBAS® KRAS/EGFR Mutation Tests y SSCP (Single Stranded Conformational Polymorphism) confirmado por secuenciación Sanger.
ResultadosSiete pacientes de un total de 19 tenían mutaciones detectadas en biopsia por COBAS® y SSCP: 3 en EGFR y 4 en KRAS. Las mutaciones de EGFR fueron detectadas en 2 pacientes en etapa I y 1 en etapa IV. Las de KRAS, 1 en adenocarcinoma in situ, 2 etapa I y 1 etapa IV. Sólo una mutación fue detectada en cfDNA obtenido a partir de plasma (KRAS, etapa IV), siendo la correlación entre la biopsia y cfDNA 14% (1/7). Discusión: La técnica COBAS® permitió detectar mutaciones en cfDNA de plasma. Las mutaciones observadas en muestras de tejido fueron alternativamente detectadas tanto por SSCP y secuenciación Sanger como COBAS®. El cfDNA puede ser una alternativa para detectar mutaciones en adenocarcinoma pulmonar.
Adenocarcinoma is currently the most common type of lung cancer. Mutations in EGFR are predictive of TKIresponseandmutuallyexclusivewithmutationsin KRAS. Molecularanalysisoffreecirculating DNA (cfDNA) has promised to be a useful tool for making these determinations. This study compares two different techniques for determining mutations in KRAS and EGFR in biopsy and blood samples from patients with lung adenocarcinoma.
Patients and Method: Patients with suspected lung cancer were included and plasma and serum samples were obtained. When the diagnosis of adenocarcinoma was confirmed, the mutational status of EGFR and KRAS in biopsies, plasma and serum were analyzed by COBAS® KRAS/EGFR Mutation Tests and SSCP (Single Stranded Conformational Polymorphism) and confirmed by Sanger's sequencing. Results: Seven patients out of 19 had mutations detected on COBAS® and SSCP biopsy: 3 in EGFR and 4 in KRAS. EGFR mutations were detected in 2 patients in stage I and 1 in stage IV. Those of KRAS 1 in adenocarcinoma in situ, 2 in stage I and 1 in stage IV. Only one mutation was detected in plasma-derived cfDNA (KRAS, stage IV), observing a correlation of 14% between biopsy and cfDNA (1/7). Discussion: The COBAS® technique allowed to detect mutations in plasma cfDNA. Both SSCP and Sanger sequencing and COBAS® techniques allowed the detection of mutations in biopsies. cfDNA could be used to detect mutations lung adenocarcinomas.
El cáncer de pulmón sigue siendo la principal causa de muerte por cáncer, con un estimado de 1,8 millones de muertes al año1. En Chile el cáncer pulmonar es la primera causa de muerte por cáncer tanto en hombres como en mujeres2. Dentro del cáncer pulmonar, el cáncer pulmonar de células no pequeñas (NSCLC en inglés) es el más frecuente correspondiendo aproximadamente al 85% de los cánceres pulmonares. En este grupo el adenocarcinoma ha ido aumentando progresivamente su incidencia hasta convertirse en el subtipo más frecuente, representando el 40-50% de los NSCLC.
Es en los adenocarcinomas pulmonares donde se describieron inicialmente alteraciones genéticas con valor pronóstico y predictivo3–5 que han llevado a una verdadera revolución en el enfrentamiento de estas neoplasias. Las mutaciones del receptor del factor de crecimiento epidérmico (EGFR, por su sigla en inglés) son predictivas de respuesta a la terapia con inhibidores de tirosina quinasa (TKI) y son mutuamente excluyentes con las mutaciones presentes en el oncogén KRAS (homólogo al oncogén del virus Kirsten de sarcoma de rata). Aunque el número de pacientes con mutaciones accionables es pequeño, se ha visto que la sobrevida libre de progresión está significativamente aumentada en pacientes con terapia dirigida comparado con pacientes que reciben quimioterapia. El rango de la tasa de respuesta es del 50 al 80% en pacientes que presentan mutaciones en EGFR, BRAF, ALK y que reciben terapia dirigida, incrementándose la sobrevida total de 18 a 38,6 meses6–8. Hoy en día el estudio de mutaciones en EGFR y de la translocación EML4-ALK (gen que resulta de la fusión entre los genes de la proteína análoga a microtúbulos de equinodermo tipo 4 (EML4) humana y la proteína quinasa de linfoma anaplásico (ALK)) en pacientes con adenocarcinoma pulmonar, es considerado estándar de tratamiento, tanto en la biopsia inicial como en nuevas biopsias, en el caso que el paciente desarrolle resistencia a la terapia con TKI3–11.
La identificación de estas mutaciones en DNA libre circulante (cfDNA, por su sigla en inglés), parece ser un método más asequible para llevar a cabo este tipo de estudios.
El cfDNA fue descrito por primera vez hace más de 50 años12, desde entonces se han observado niveles elevados de cfDNA en distintos tipos de cánceres, principalmente en estadios avanzados13. El análisis molecular del cfDNA está siendo estudiado extensa-mente para determinar las alteraciones moleculares en diferentes tipos de tumores. Estas “biopsias líquidas” tendrían la ventaja de ser un método más rápido, sencillo y seguro; permitiendo evaluar las diferentes mutaciones susceptibles de terapia previo al inicio de cualquier tratamiento y siendo fácilmente repetibles frente a cualquier cambio en la biología del tumor14,15.
En el caso particular del cáncer pulmonar, en cfDNA aislado en suero y plasma se han identificado mutaciones presentes en los genes de EGFR, KRAS, ALK, HER2 (receptor 2 de factor de crecimiento epidérmico humano), entre otros, los que se han correlacionado con las mutaciones observadas en el tumor primario16,17. El propósito de este estudio fue evaluar la detección de mutaciones en los genes EGFR y KRAS en muestras líquidas de suero y plasma y correlacionarlas con la detección en muestra de tejido tumoral de pacientes con adenocarcinoma pulmonar. Para ello se compararon tres estrategias diferentes para determinar las mutaciones en estos genes: PCR en tiempo real mediante el kit de COBAS®, las técnicas de SSCP y secuenciación automática de Sanger.
2Pacientes y método2.1PacientesEstudio observacional transversal. Se reclutaron en forma consecutiva los pacientes con sospecha de cáncer pulmonar que ingresaron a nuestro centro para ser sometidos a cirugía resectiva o biopsia quirúrgica, entre octubre de 2012 y marzo de 2014. Veintiún pacientes aceptaron ser parte del estudio, dos pacientes fueron posteriormente excluidos debido a que el informe final de patología mostró hiperplasia atípica, quedando conformado el grupo de estudio por 19 pacientes con adenocarcinoma pulmonar. Todos los procedimientos respetaron las normas éticas concordantes con la Declaración de Helsinki, el estudio fue aprobado por el comité de ética de la investigación de nuestra institución y todos los pacientes firmaron un consentimiento informado autorizando ser incluidos en el estudio.
2.2Obtención de suero y plasmaA cada paciente, previo a la cirugía, se extrajeron 20ml de sangre a partir de los cuales se aisló suero y plasma. Para aislar suero, 10ml de sangre fueron tomados en tubos BD Vacutainer sin coagulante. Se dejó en reposo a temperatura ambiente por 45min. y luego se centrifugó a 1.300g por 20min. a 4°C. El suero se guardó en alícuotas de 1ml a -80°C hasta ser procesado. Para aislar plasma, se recolectaron 10ml de sangre en tubos BD Vacutainer con K2EDTA 10,8mg y se centrifugaron inmediatamente a 1.300g por 20min. a 4°C. El plasma se guardó en alícuotas de 1ml a -80°C hasta ser procesado.
2.3Extracción de DNAEl DNA tumoral se extrajo desde muestras quirúrgicas fijadas en formalina e incluidas en parafina (FFPE). Los cortes teñidos con hematoxilina-eosina fueron analizados por el patólogo del grupo de investigación, quien seleccionó zonas enriquecidas con más de 80% de células tumorales. Se extrajo el DNA tumoral de cortes seriados de 8μm de espesor no teñidos, mediante el Sistema QIAmp DNA FFPE Tissue extraction kit (QIAGEN, Hilden, Alemania), según las instrucciones del fabricante. La integridad del DNA se analizó mediante la amplificación por PCR multiplex de 2 amplicones de GAPDH, 1 amplicón de β-globina y 1 amplicón de β-actina.
El DNA libre circulante (cfDNA) se aisló desde suero y plasma utilizando el kit de ácido nucleico circulante QIAamp® Circulating Nucleic Acid Kit (QIAGEN, Hilden, Alemania) de acuerdo a las instrucciones del fabricante.
2.4Análisis de mutacionesLas mutaciones de EGFR (exones 18, 19, 20 y 21) y KRAS (codones 12, 13 y 61) se analizaron mediante el equipo COBAS utilizando los kits COBAS® KRAS y EGFR Mutation Tests (Roche. Mannheim, Alemania) a partir de DNA aislado desde muestras de tejido y cfDNA. Además, se amplificó por PCR fragmentos que contienen las mutaciones para EGFR (exones 18 y 21) y KRAS (exón 1) usando partidores específicos propios a partir de 50ng de DNA tumoral y 10μl de cfDNA. Estos productos de PCR se analizaron por la presencia de conformómeros de simple hebra mediante geles de acrilamida (SSCP, Single Stranded Conformational Polymorphism)18. Finalmente, los mismos productos de PCR obtenidos desde tejido y cfDNA fueron secuenciados mediante secuenciación automática de Sanger. Todos los datos fueron analizados en doble ciego.
3Resultados3.1Características de los pacientes incluidos en el estudio y sus respectivas muestras de tumorSe analizaron finalmente muestras de 19 pacientes: 14 casos eran tumor primario pulmonar, 2 casos eran metástasis pleural, 2 casos de metástasis linfonodal y 1 caso de metástasis cerebral.
En la Tabla 1 se muestran las características clínicas y patológicas generales de todos los pacientes estudiados. Siete pacientes, presentaron mutaciones en las biopsias de tejido detectadas por COBAS®, 3 en el gen de EGFR y 4 en el de KRAS.
Características clínico-patológicas de los pacientes con mutaciones
| Pacientes con Mutaciones | |||||||
|---|---|---|---|---|---|---|---|
| Género | n (7) | Edad Promedio [intervalo] | Mutación | Estadio | |||
| EGFR | KRAS | In Situ | I-II | III-IV | |||
| Hombres | 3 | 71,6 [65,66,67,68,69,70,71,72,73,74,75] | 2 | 1 | 1 | 2 | 0 |
| Mujeres | 4 | 61,3 [54,55,56,57,58,59,60,61,62,63,64,65,66,67,68,69] | 1 | 3 | 0 | 2 | 2 |
Cuatro de nueve pacientes de sexo femenino presentaron mutaciones (1 en EGFR y 3 en KRAS), en hombres tres de diez presentaron mutaciones (2 en EGFR y 1 en KRAS). En pacientes varones con mutaciones, la edad de diagnóstico fue más tardía y se presentaron en estadios más tempranos. Las mutaciones de EGFR se presentaron en 2 pacientes en estadio I y 1 en estadio IV y las mutaciones en KRAS en 1 paciente con adenocarcinoma in situ, 2 en etapa I y 1 en etapa IV (Tabla 2).
Características clínico-patológicas de los pacientes sin mutaciones
| Pacientes sin Mutaciones | |||||||
|---|---|---|---|---|---|---|---|
| Género | n (12) | Edad Promedio [intervalo] | Mutación | Estadio | |||
| EGFR | KRAS | In Situ | I-II | III-IV | |||
| Hombres | 7 | 63,3 [49,50,51,52,53,54,55,56,57,58,59,60,61,62,63,64,65,66,67,68,69,70,71,72,73,74,75,76,77,78,79] | 0 | 0 | 1 | 4 | 2 |
| Mujeres | 5 | 71,4 [64,65,66,67,68,69,70,71,72,73,74,75,76,77,78] | 0 | 0 | 0 | 3 | 2 |
En las biopsias líquidas, mediante la técnica COBAS® sólo se detectó una mutación en plasma (mutación KRAS en paciente en etapa IV). No se detectaron mutaciones en el cfDNA obtenido a partir de suero. La correlación entre la detección de mutaciones en biopsia de tejido y biopsia líquida fue por tanto de 14% (1/7) (Tabla 2 y Fig. 1).
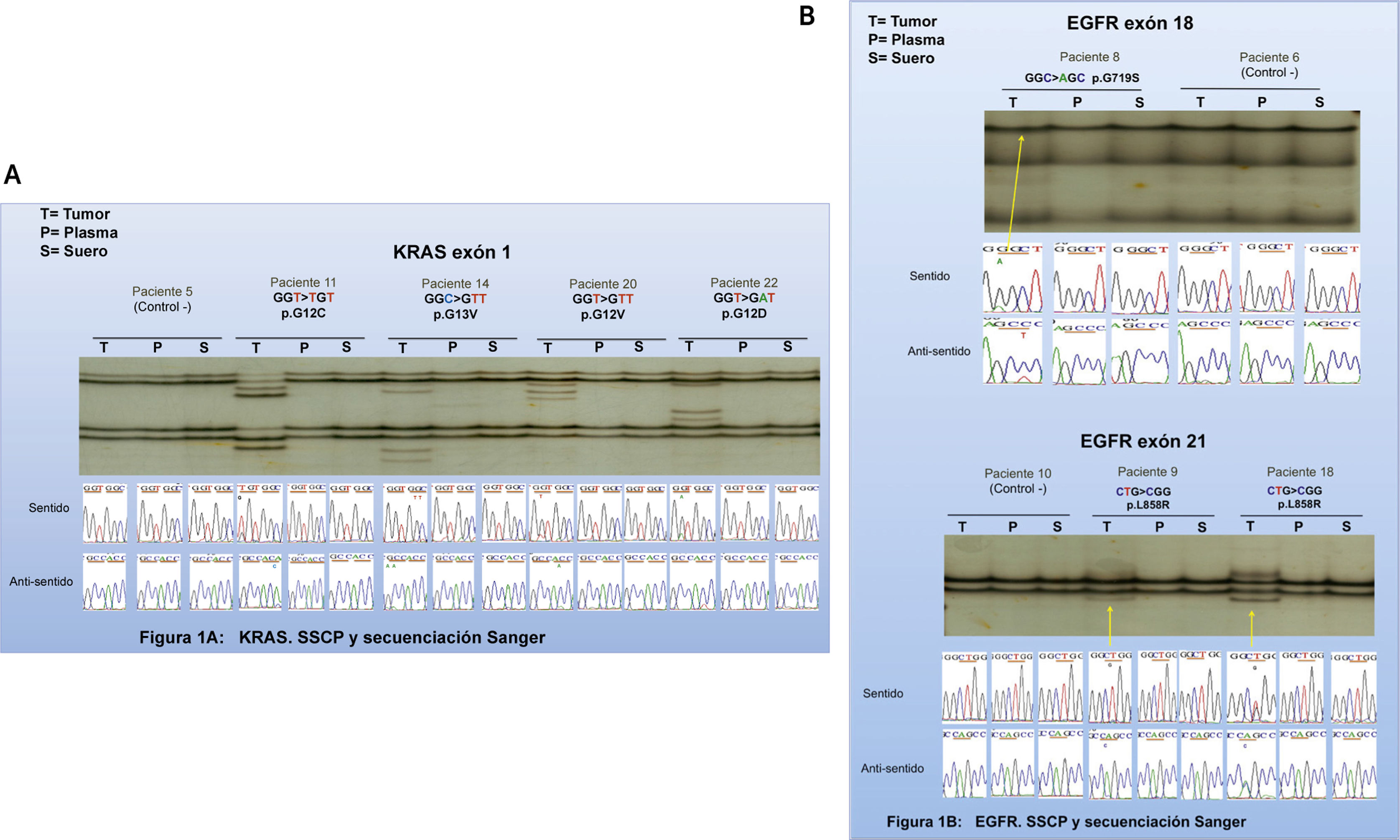
Se evaluó finalmente la detección de mutaciones de EGFR y KRAS en biopsia de tejido y en biopsia líquida mediante SSCP, corroboradas posteriormente mediante secuenciación automática de Sanger. Todas las mutaciones de KRAS detectadas por COBAS® en muestras de tejido fueron también detectadas por SSCP. En la Figura 1 se presentan los patrones de migración de los 4 pacientes con mutaciones en KRAS en la pieza tumoral (P11, P14, P20 y P22) los que resultaron claramente diferentes a los de pacientes sin mutación ejemplificados por el P5.
No se observaron diferencias en el patrón de migración en las biopsias líquidas, tanto en plasma como en suero.
Mediante la secuenciación de Sanger de los pacientes con mutación de KRAS detectada por SSCP, se identificó que el paciente 11 tenía la mutación G12C; el paciente 14, la mutación G13D; el paciente 20, la mutación G13V y el paciente 22, la mutación G12D, todas ubicadas en el exón 1 del gen KRAS (Fig. 1A).
En muestras de tejido tumoral se observó por tanto una completa correlación entre las mutaciones de KRAS detectadas por PCR en tiempo real mediante el kit de COBAS®, SSCP y secuenciación automática de Sanger (Tabla 2 y Fig. 1).
En relación al gen EGFR, todas las mutaciones detectadas por COBAS® en muestras de tejido también fueron detectadas por SSCP y secuenciación automática de Sanger. En la Fig. 1B se muestra el resultado de SSCP y su correspondiente secuenciación Sanger en pacientes con mutación en EGFR. Mediante secuenciación Sanger se determinó que el paciente 18 presentaba la mutación G719S en el exón 18 y que los pacientes 9 y 18 presentaban la misma mutación L858R en el exón 21 del gen EGFR (Fig. 1B). Todas las mutaciones encontradas se presentan en forma heterocigota y están descritas en las bases de datos de mutaciones somáticas.
4DiscusiónEn este estudio, se logró detectar la mutación en el gen KRAS en la muestra líquida de plasma en un paciente y correlacionarla con la detección en muestra de tejido tumoral. De las tres técnicas utilizadas, se demostró que el kit de COBAS® que solo estaba validado para muestras obtenidas desde tejido embebido en parafina y fijado en formalina al momento de la realización de este estudio, fue capaz de detectar también una mutación presente en cfDNA obtenido a partir de plasma, pero en una baja proporción en relación a las mutaciones detectadas en tejido tumoral (14%).
Una de las limitaciones de este estudio es la baja sensibilidad observada en las biopsias líquidas, la que podría deberse a que se extrajo una baja proporción de DNA tumoral circulante (ctDNA) del total de DNA libre circulante (cfDNA). Diferentes autores han denominado a la porción del cfDNA que contiene las alteraciones tumorales específicas, DNA tumoral circulante (ctDNA). El ctDNA es variable y representa sólo una pequeña fracción del cfDNA total19,20. Está constituido en parte importante por fracciones de bajo peso molecular, lo que hace que su extracción sea poco predecible. Su proporción en el cfDNA total es por tanto muy variable, lo que dificulta la interpretación de los resultados y la comparación entre diferentes estudios21,22. Para enriquecer la extracción de ctDNA, algunos estudios han sugerido incluir en la técnica de obtención del plasma o suero, una segunda centrifugación a alta velocidad entre 3000 y 16000g190,23. Esta centrifugación permitiría remover la contaminación celular residual y generar una muestra más limpia y enriquecida, para la posterior extracción de ctDNA mediante el protocolo regular del kit QIAmp. Este estudio fue realizado con anterioridad a la publicación del trabajo, por lo que no se realizó esta segunda centrifugación. Si se hubiese incluido este paso, la proporción de mutaciones detectadas en las biopsias líquidas podría haber sido mejor. Por lo que se recomienda incorporar este procedimiento en futuros estudios con cfDNA.
Una segunda explicación es la alta proporción de pacientes con etapas iniciales del cáncer incluidos en nuestro estudio. En los estudios realizados en diferentes tipos de cánceres, se ha logrado extraer cfDNA principalmente en estadios avanzados, por lo que en este grupo de pacientes las biopsias líquidas tendrían mayor utilidad24,25. Por el diseño de nuestro estudio, que incluyó principalmente a pacientes potencialmente candidatos a cirugía, la mayoría se presentó en etapas I y II (13/19), y sólo 6 en etapas avanzadas. La única mutación detectada mediante biopsia líquida fue en muestra de plasma en paciente en etapa IV. En los pacientes de este grupo la correlación fue del 50% (1/2) pero el tamaño muestral no permite mayores conclusiones. Es importante destacar que la única mutación detectada está en KRAS, mientras el otro paciente en estadio IV la presenta en EGFR. No sabemos si mutaciones en estos genes determinan que las células liberen DNA circulante en mayor proporción dependiendo del gen mutado en cuestión. Probablemente el rendimiento de las biopsias líquidas debería ser mayor en etapas avanzadas del cáncer, donde se asume que la cantidad de ctDNA es mayor, lo que deberá evaluarse en futuros estudios24.
En nuestro estudio sólo detectamos una mutación en cfDNA a partir de plasma, pero no en suero. La obtención de muestras de suero como de plasma fueron realizadas en paralelo y con el mismo procedimiento, a excepción de la etapa inicial donde se espera la formación del coágulo para aislar el suero. La presencia de nucleasas durante el proceso de formación del coágulo, podrían haber alterado el cfDNA, lo que explicaría que sólo se detectaran mutaciones en las muestras de plasma. Esto deberá ser evaluado en futuros estudios con mayor número de pacientes.
Tanto la técnica de COBAS® como SSCP y secuenciación automática de Sanger detectaron las mutaciones en muestras de tejido con una correlación de un 100%. El número de pacientes con mutaciones en el gen de EGFR (3/19, 16%) fue menor al esperado en la población chilena (22%), lo que podría deberse al reducido tamaño de la muestra26.
En biopsias líquidas sólo la técnica de COBAS® permitió la detección de mutaciones.
El análisis por SSCP y secuenciación Sanger, requiere de una amplificación previo de la muestra de DNA. En muestras de tejido el DNA tumoral se amplifica a partir de células tumorales aisladas desde la biopsia. Como se mencionó previamente en biopsias líquidas la cantidad de ctDNA es variable y el DNA puede estar fragmentado. Condiciones que no permiten una adecuada amplificación, y que podrían explicar que no se detectaran mutaciones en plasma ni en suero en nuestro estudio.
Sin embargo, la secuenciación de Sanger permite una mejor identificación específica de las mutaciones. Por ejemplo, la mutación en el codón G719 en el equipo COBAS® interpretó la mutación del aminoácido glicina en el codón 719 por otro aminoácido no sinónimo, pero sin especificar cuál. La secuenciación Sanger nos permitió discriminar que el aminoácido X corresponde a una serina. El kit de COBAS® tampoco discrimina si la mutación en el gen KRAS está en el codón 12 o en el codón 13 y a qué cambio aminoacídico corresponde, otra ventaja que entrega la secuenciación Sanger y la técnica de SSCP (Fig. 1). Esta información pudiese eventualmente ser relevante como factor pronóstico para el paciente lo que representaría una ventaja sobre la técnica de COBAS®. Futuros estudios con mayor proporción de ctDNA deberán evaluar la utilidad de estas técnicas en biopsias líquidas.
La secuenciación masiva es actualmente la técnica más promisoria para el estudio de mutaciones en biopsias líquidas. Esta técnica tiene las ventajas de ser rápida, fácilmente repetible y permitir el análisis de varias mutaciones en forma simultánea. Ha sido validada recientemente en cáncer pulmonar para la detección de mutaciones de EGFR tanto al momento del diagnóstico como en caso de recidiva. Sin embargo, aún existen limitaciones para su uso, la correlación con las biopsias de tejido no es perfecta y no hay claridad en el panel de genes que debería estudiarse especialmente en el seguimiento de pacientes en tratamiento24.
Futuros estudios, con adecuada representación de ctDNA, deberán evaluar la utilidad clínica de las biopsias líquidas y la mejor técnica a utilizar para determinar mutaciones, tanto en el diagnóstico como en el seguimiento de pacientes con cáncer pulmonar.
En nuestro estudio detectamos la presencia de mutaciones en adenocarcinomas pulmonares en 7 de los 19 pacientes estudiados.
Finalmente, podemos concluir que las mutaciones en muestras de tejido tumoral fueron detectadas por las 3 técnicas utilizadas: PCR en tiempo real mediante kit de COBAS®, SSCP y secuenciación automática de Sanger con una correlación de un 100%.
Aunque, sólo mediante la técnica de COBAS® se logró determinar una mutación de KRAS en el cfDNA obtenido a partir del plasma en un paciente.
En futuros estudios se podría lograr una mayor detección de mutaciones, y una mejor correlación con las muestras de tejido, aumentando la proporción de ctDNA del total cfDNA mediante una segunda centrifugación a alta velocidad y dirigiendo esta técnica a pacientes con mayor posibilidad de tener altos niveles de ctDNA circulante (etapas avanzadas), y considerar el uso de secuenciación masiva, la cual posee aún mayor sensibilidad que la PCR en tiempo real y se perfila como una herramienta promisoria para su uso en biopsias líquidas.
5AgradecimientosEste trabajo fue financiado por la Dirección Académica de Clínica Las Condes. Proyecto Interno: PI2012_DA014.
Declaración de conflicto de interésLos autores declaran no tener conflictos de intereses.