El cáncer es una patología con prevalencia en alza y cuyo origen se encuentra en el DNA, más específicamente en las alteraciones que lo afectan, y que finalmente se traducen en la desregulación del ciclo celular y de los mecanismos celulares reparadores del DNA. La desregulación del ciclo celular se traduce finalmente en la formación de tumores, que pueden invadir tejidos u órganos distantes, provocando la muerte del paciente. Conocer y entender las alteraciones y/o daños que puede sufrir el DNA permite comprender el proceso de carcinogénesis a escala molecular, pero también a escala humana. Esta revisión pretende actualizar y/o clarificar algunos conceptos moleculares básicos del cáncer, cómo éstos pueden estudiarse en un laboratorio de biología molecular y cómo pueden ayudar a mejorar la práctica clínica con un paciente oncológico.
Cancer prevalence is rising worldwide. The main pathogenic events of this devastating disease are alterations in the DNA, which eventually result in the dysregulation of both the cell cycle and DNA repair mechanisms. This dysregulation of the cell cycle results in the formation of a tumor, from which cancer cells disseminate to distant organs, finally bringing about patient death. Knowledge and understanding of the nature of the DNA alterations is essential to the comprehension of the carcinogenic process at both a molecular and human scale. This review aims to update and clarify some basic molecular concepts in cancer, focusing principally on how alterations can be studied within the molecular biology lab, and how this discipline can improve clinical cancer practice.
La alta prevalencia del cáncer a nivel mundial y la gravedad de sus implicancias lo han convierten la más icónica de las patologías humanas. Es así como prácticamente cualquier persona tiene una noción más o menos acertada de qué es el cáncer. La Organización Mundial de la Salud define al cáncer como “un proceso de crecimiento y diseminación incontrolado de células que puede aparecer prácticamente en cualquier lugar del cuerpo. El tumor suele invadir el tejido circundante y puede provocar metástasis en puntos distantes del organismo”. Sin embargo, lo que mayoritariamente no se sabe es que el cáncer podría también definirse como una enfermedad del DNA, y que como consecuencia del daño en el DNA se afecta la proliferación celular con todas las consecuencias frecuentemente conocidas.
El DNA es otro ícono de las ciencias biológicas, con una estructura tridimensional de doble hélice fácilmente reconocible e instalada en el subconsciente colectivo. Destaca además por ser la molécula codificadora de las instrucciones genéticas usadas en el desarrollo y funcionamiento de todos los organismos vivos conocidos y algunos virus. Además, es la molécula responsable de la transmisión hereditaria. Sin embargo, poco se sabe sobre las alteraciones que puede sufrir el DNA, de sus repercusiones, o de los mecanismos que utiliza el organismo para defenderse o reparar los daños a los que estamos habitualmente expuestos. Entendiendo los tipos de alteraciones y/o daños que puede sufrir el DNA es posible comprender el proceso de carcinogénesis desde un punto de vista molecular, pero a la vez amplio, y aplicable en el entorno clínico diario. Esta revisión pretende, en términos muy simples, actualizar y/o clarificar algunos conceptos moleculares básicos del cáncer y cómo éstos pueden estudiarse en un laboratorio de biología molecular.
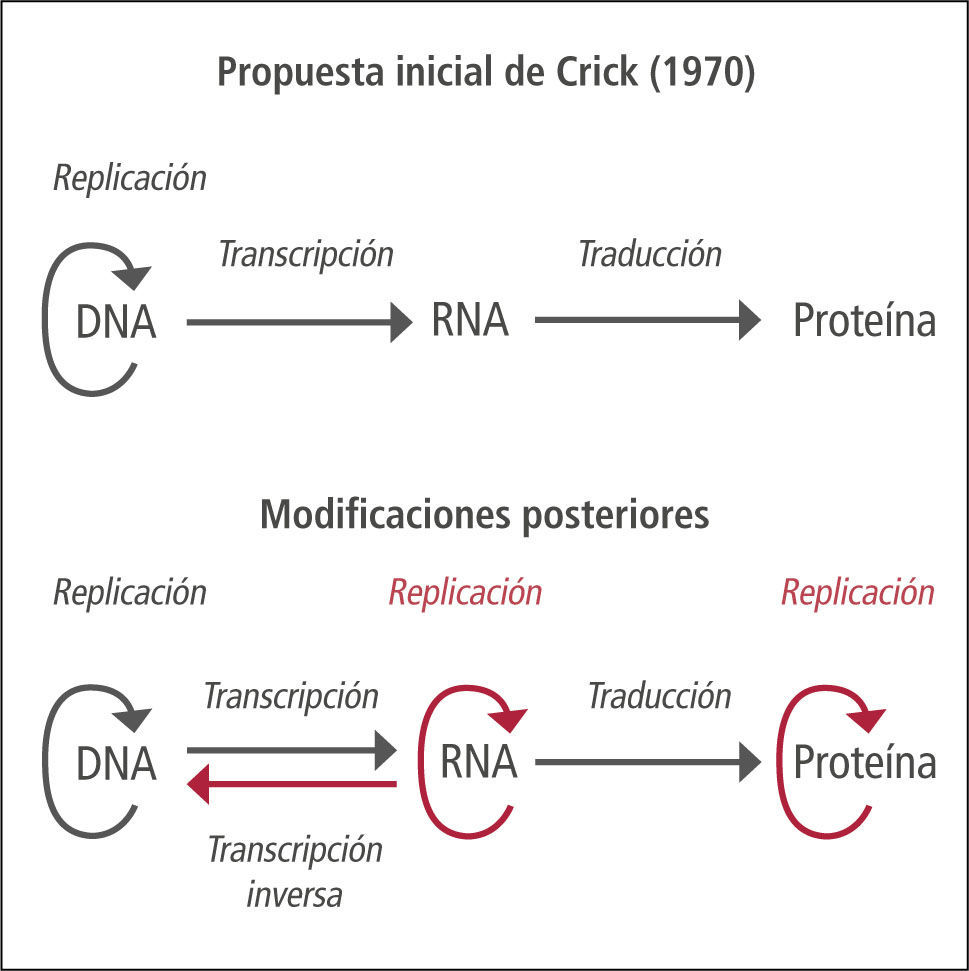
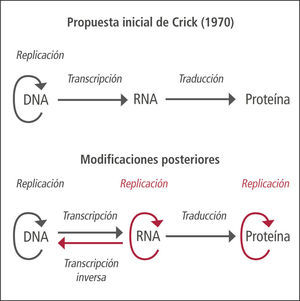
Nuevo dogma de la biología molecularEl dogma central de la biología molecular es un concepto que ilustra el mecanismo de transmisión de la herencia genética y de su expresión, formulado por Francis Crick en 1970 (1). Inicialmente se proponía un sentido unidireccional en el flujo de la información genética, comenzando por el DNA, seguido por el proceso de transcripción1 a RNA y finalmente por la traducción2 a una proteína. Actualmente se sabe que el proceso de flujo de la información genética es bidireccional, con procesos como la transcripción inversa o paso de RNA a DNA, fenómeno utilizado por retrovirus para multiplicarse en las células infectadas. El DNA puede también duplicarse, generando una nueva copia de DNA. Además, algunas proteínas son capaces de duplicarse y proliferar en ausencia de DNA, como en el caso de los priones (Figura 1).
Dogma central de la biología molecular
Concepto propuesto inicialmente en 1958 por Francis Crick, postulándose nuevamente en un artículo de la revista Nature publicado en 1970, que ilustra un mecanismo unidireccional de transmisión y expresión de la herencia, desde el DNA hasta las proteínas, pasando por el RNA. El dogma inicial postulaba que sólo el ADN puede duplicarse y, por tanto, reproducirse y transmitir la información genética a la descendencia. Actualmente se ha “ampliado” el dogma debido al descubrimiento de la transcriptasa inversa, enzima capaz de transformar el RNA a DNA, presente en retrovirus, o los priones, proteínas capaces de replicarse en ausencia de DNA, o de las ribozimas, RNAs con propiedades autocatalíticas capaces de modificarse y duplicarse a sí mismos en ausencia de proteína y ADN. Otra situación que rompe con la secuencia definida por el dogma es la posibilidad de obtener proteína in vitro, en un sistema libre de células y en ausencia de ARN, por lectura directa del ADN mediante ribosomas, en presencia de neomicina.
Actualmente se define como gen a un segmento de DNA que codifica para cualquier molécula con actividad biológica3, no sólo proteínas. Así, encontramos genes que codifican para ribozimas (fragmentos de RNA que cortan y destruyen a otro RNA), RNA de transferencia (tRNA) necesarios para la síntesis de proteínas, micro-RNA (miRNA) o RNA con actividad reguladora de la expresión génica, entre otras, siendo las proteínas el producto principal (en adelante generalizaremos como proteínas al producto de la expresión génica). Funcionalmente, los genes están divididos en 2 regiones: (i) la región codificante y (ii) la región promotora. Se llama región codificante al segmento de DNA que contiene la información que dará origen a una proteína. Por otro lado, la región promotora (o promotor) es un segmento de DNA localizado inmediatamente adelante o “rio arriba” de la región codificante, y que posee una función reguladora de la expresión génica. El promotor es el responsable de que no todos los genes se expresen en todas las células del cuerpo. Así, la insulina se expresa sólo en las células beta del páncreas y no en neuronas o cardiomiocitos. En otras palabras, puede visualizarse al promotor como un interruptor genético que determina la expresión específica de una proteína en un tejido particular, o en un ciclo circadiano día/noche, o en respuesta a un estímulo específico.
La región codificante a su vez esta dividida funcionalmente en 2 tipos de segmentos; (i) los exones y (ii) los intrones. Los exones e intrones se intercalan ordenadamente como vagones de un ferrocarril. Los exones contienen la información genética cifrada mientras que los intrones no. Los intrones deben ser removidos, dejando únicamente a los exones alineados y ordenados. Este proceso se conoce como corte y empalme o “splicing” y genera una secuencia de RNA específica, más corta que su contraparte de DNA.
El código genético y los tipos de alteraciones genéticasEl DNA está compuesto por los nucleótidos A, T, C y G. La combinación de estas cuatro “letras” en triadas llamadas codones, dan origen a una biblioteca genética con aproximadamente 27000 “libros” o genes, todo esto dentro de cada una de nuestras células nucleadas. Las distintas combinaciones de nucleótidos en diferentes codones dan finalmente origen a 23 aminoácidos. Algunas combinaciones tales como T-A-G no codifican para un aminoácido sino que son señales de término de la traducción, es decir, donde se detiene la síntesis de la cadena proteica. Una característica importante del código genético es que más de un codón puede codificar para un mismo aminoácido. Por ejemplo, las combinaciones G-G-G y G-G-A codifican para el aminoácido glicina. Se agrega así un sistema de seguridad frente a alteraciones en el DNA para que, aunque cambie G por A, no se produzca un cambio en la proteína ya que ambas combinaciones dan origen al mismo aminoácido, glicina. El orden de importancia de cada letra de un codón aumenta de derecha a izquierda, siendo el primer nucleótido fundamental en la codificación de cada aminoácido. Un cambio en la primera letra del codón siempre cambiará el aminoácido codificado.
Tipos de alteraciones genéticasNuestro organismo y nuestro DNA está sometido constantemente a la acción de agentes nocivos con capacidad de introducir alteraciones en la secuencia de nucleótidos; radiación U V, tabaco, polución ambiental, etc. Una secuencia de DNA puede sufrir tres tipos de alteraciones principales:
- 1.
Sustituciones nucleotídicas.
- 2.
Inserciones nucleotídicas.
- 3.
Deleciones nucleotídicas.
Las sustituciones son remplazos de un nucléotido por otro. El ejemplo anterior grafica una sustitución de G por A en el codón que codifica para glicina. Algunas sustituciones pueden (o no) cambiar el aminoácido. Aquellas sustituciones que no cambian el aminoácido se denominan sustituciones sinónimas. Por otro lado, las inserciones y deleciones, como sus nombres indican, agregan o eliminan letras del código, generando un corrimiento en el marco de lectura genética. En otras palabras, desde el lugar de inserción (o deleción) nucleotídica en adelante se cambia el sentido del código, generándose una cadena peptídica completamente diferente a la originalmente codificada y/o generando una proteína más corta o truncada debido a la aparición aleatoria de un codón de término temprano.
Existen otros tipos de alteraciones que implican grandes reordenamientos o rearreglos genético o genómicos, los que pueden incluir pérdida (o duplicaciones) de fragmentos de genes, genes completos, fragmentos de cromosomas e incluso cromosomas completos, con un muy amplio espectro de repercusiones.
Alteraciones nucleotídicas en regiones intrónicas también pueden afectar la naturaleza o actividad de la proteína (recordemos que los intrones no contienen secuencias codificantes). La región intrónica inmediatamente colindante al exón contiene señales para el correcto splicing del mRNA. Alteraciones en esta región, aunque no alteran directamente la codificación de aminoácidos, pueden producir la retención total o parcial de un intrón, o la pérdida total o parcial de un exón, cambiando la naturaleza de la proteína sintetizada.
Mutaciones versus polimorfismosCuando se piensa en alteraciones en el DNA es casi inevitable asociar el término “mutación”. El término mutación describe aquellas alteraciones nucleotídicas que producen un cambio drástico en la función de la proteína. Este cambio puede incluir pérdida total o parcial de la función de la proteína. Podemos poner como ejemplo una mutación que altere la estructura de una enzima, haciéndola permanentemente inactiva. Por otro lado, una mutación puede ser responsable de la activación permanente de un receptor de membrana, gatillando una respuesta aún en ausencia de la unión del ligando específico. Las mutaciones pueden entonces producir alteraciones tanto por pérdida como por ganancia de función. Una mutación puede ser la responsable de la generación de una proteína truncada sin función, o alterar levemente la estructura tridimensional de la proteína, pudiendo afectar fuertemente la interacción con otras proteínas de un complejo proteico mayor. Es decir, es muy difícil predecir el efecto de una alteración genética por el solo hecho de saber su ubicación en el genoma, y confirmar el efecto de una mutación requiere de estudios funcionales de la proteína en estudios in vitro.
No todos los cambios nucleotídicos producen alteraciones drásticas en la función de la proteína. Algunas alteraciones nucleotídicas están presentes en más del 1% de la población sin afectar la salud del individuo que la porta. Incluso, es posible que dichas alteraciones confieran una ventaja adaptativa frente al individuo que no la porta. Estas alteraciones se denominan polimorfismos y son responsables de la diversidad genética entre distintos individuos y/o étnicas entre distintas poblaciones. Un ejemplo clásico es un polimorfismo muy frecuente en la población japonesa que afecta la actividad de la enzima metabolizadora de alcohol Acetaldehído deshidrogenasa tipo 2 (ALDH2). Los portadores de este polimorfismo no son capaces de metabolizar el acetaldehído producido por la degradación del etanol. Aunque son perfectamente sanos, los portadores acumulan acetaldehído en la circulación con efectos disfóricos como náuseas y enrojecimiento facial, protegiéndolos contra el alcoholismo, problema de salud publica muy importante en Chile y Latinoamérica.
Finalmente, se denominan variantes alélicas a aquellas alteraciones nucleotídicas con un efecto incierto o desconocido. Las variantes alélicas pueden explicarse por cambios nucleotídicos sinónimos donde el cambio de nucleótido no cambia el aminoácido (como el mencionado anteriormente), cambios nucleotídicos silentes o aquellos que caen en intrones o regiones no codificantes ni regulatorias. Finalmente puede ocurrir que el cambio nucleotídico efectivamente cambie el aminoácido, pero que este cambio no afecte de forma conocida la función de la proteína.
Mutaciones somáticas y mutaciones de línea germinalAquellas mutaciones que se localizan sólo en el DNA de las células tumorales se denominan mutaciones somáticas. Por otro lado, una mutación puede originarse (aleatoriamente) en células de la línea germinal de un individuo y será por lo tanto heredada a su descendencia en una proporción mendeliana. Así, esta mutación heredada estará presente en todas las células del cuerpo del individuo. Este tipo de mutaciones se denominan mutaciones de línea germinal. Las mutaciones somáticas pueden asociarse con la agresividad tumoral o con respuesta a terapias con anticuerpos monoclonales. La detección de mutaciones somáticas puede ofrecer una alternativa terapéutica a pacientes que serán buenos respondedores de acuerdo a perfil mutacional tumoral que posea. De lo contrario, la detección de un paciente portador de una mutación somática que lo haga mal respondedor a una terapia dada podría evitar efectos adversos severos sin un beneficio terapéutico. Este es el caso de la detección de mutaciones en el gen KRAS para pacientes con cáncer colorectal. La detección mutaciones en el gen KRAS que activan permanentemente a la proteína KRAS permite distinguir aquellos pacientes que no responderán a terapias con anticuerpos monoclonales dirigidos al receptor de EGFR.
Las mutaciones de línea germinal pueden predisponer a un individuo portador al desarrollo de cáncer. En otros casos, la presencia (o ausencia) de una determinada mutación puede confirmar (o descartar) un diagnóstico basado solamente en los hallazgos clínicos. Podemos mencionar mutaciones en genes como RET, PTEN, APC, BRCA1 y BRCA2, entre muchos otros. Las mutaciones en estos genes pueden activar o inhibir constitutivamente a la proteína, con efectos diversos según cada caso. Así, un niño que haya heredado una mutación activante en el codón 918 del gen RET tiene un 99% de probabilidades de desarrollar cáncer medular de tiroides antes del 1er año de vida. Una mujer portadora de una mutación inactivante en el gen BRCA1 tiene un riesgo aumentado de desarrollar cáncer de mama y/o ovario a edad temprana. En cada caso, el conocimiento de la presencia de una mutación en la línea germinal entrega información valiosa que permitirá ofrecer una tiroidectomía profiláctica en el caso del gen RET, o una vigilancia activa en el caso de una mujer portadora de una mutación en BRCA1.
Laboratorio de biología molecular en oncología: ¿cómo se detectan las mutaciones?La solicitud de búsqueda de mutaciones de línea germinal se realiza previa sospecha clínica y de acuerdo a criterios de inclusión bien definidos. La entrevista previa con el paciente en la que se indaga por antecedentes familiares relevantes, construyéndose una genealogía lo más completa posible cobra una gran relevancia. De esta forma es posible descartar la realización de un estudio genético en casos en que no hay reales antecedentes de predisposición familiar al desarrollo de cáncer. En el caso de haber antecedentes fundados que avalen la realización de un estudio genético, éste debe realizarse en el paciente afectado con cáncer. Basta con la toma de una muestra de sangre (en tubo con anticoagulante) para extraer el DNA de leucocitos. En el caso de las mutaciones somáticas, se requiere una muestra tumoral para extraer el DNA de las células cancerosas. Normalmente se utilizan los tacos de tejido tumoral fijado en formalina y embebido en parafina. Un patólogo debe seleccionar la zona del tejido con la mayor proporción de células tumorales. Cabe recordar que una muestra de tejido tumoral es un tejido heterogéneo, compuesto tanto por células tumorales como por células normales. Debido a que la mutación está presente sólo en ciertas células tumorales, es fundamental el trabajo del patólogo en la identificación de las células tumorales para la extracción del DNA. De lo contrario, las células tumorales podrían estar sub-representadas en un contexto de células normales, arrojando un posible resultado falso negativo.
PCR: reacción en cadena de la polimerasaLa tarea de encontrar una mutación en un gen del genoma humano equivale a hallar una falta ortográfica en una página de un libro de una biblioteca con 27000 ejemplares. Esa biblioteca es el genoma humano con aproximadamente 27000 genes, cuyas secuencias se conocen completamente gracias a los dos proyectos simultáneos de secuenciación del genoma humano, uno financiado con fondos públicos y dirigido por el Instituto Nacional de Salud Norteamericano NIH (2) y otro financiado con fondos privados y llevado a cabo por la compañía Celera Genomics dirigida por Craig Venter (3). Con esto en mente, la estrategia de búsqueda de una mutación puede realizarse de forma dirigida a un nucleótido específico, en un exón específico de un gen de interés, en caso de tratarse de una mutación conocida (por ejemplo, se sabe que la mutación V600E que cambia el aminoácido 600 de la proteína de valina a ácido glutámico, se ubica en el nucleótido 1799 del gen BRAF, en el que se sustituyó T por A). En otras palabras, es equivalente a ir directamente a la página 1000 de “El Quijote de la Mancha” porque sabemos que en esa particular edición del libro existe una falta ortográfica. Por otro lado, si no se conoce la mutación específica, se debe examinar la secuencia completa del gen, letra por letra, en busca de alguna diferencia con respecto a la secuencia de referencia conocida. En otras palabras, se debe leer completamente el libro en busca de una eventual falta ortográfica. Estas tareas se han facilitado gracias a la técnica de PCR, acrónimo del inglés “Polymerase Chain Reaction” o reacción en cadena de la polimerasa. La técnica de PCR fue desarrollada por Kary Mullis (4) en 1983 y su propósito es amplificar un fragmento de ADN particular, partiendo de un mínimo; en teoría basta partir de una única copia de ese fragmento original, o molde. Volviendo a la analogía literaria inicial, la técnica de PCR puede imaginarse como una fotocopiadora génica que permite sacar múltiples copias de la página 1000 de “El Quijote de la Mancha” o de cualquier otro de los 27000 “libros” o genes. Tras la amplificación se obtiene una gran cantidad de DNA “fotocopiado” lo que facilita mucho la identificación de cualquier mutación, por baja que sea la abundancia relativa del DNA mutado respecto al DNA normal. Esta técnica tiene un sinnúmero de otras aplicaciones como detección de virus o bacterias, identificación de personas (cadáveres) o hacer investigación científica sobre el ADN amplificado. Con el tiempo, la técnica de PCR ha derivado en una serie de otras técnicas más específicas, sofisticadas y sensibles, pero que comparten un principio fundamental muy simple; las dos hebras que componen la doble hélice del DNA pueden separarse o volver a unirse específicamente, como una cremallera, con el solo hecho de aumentar o bajar la temperatura.
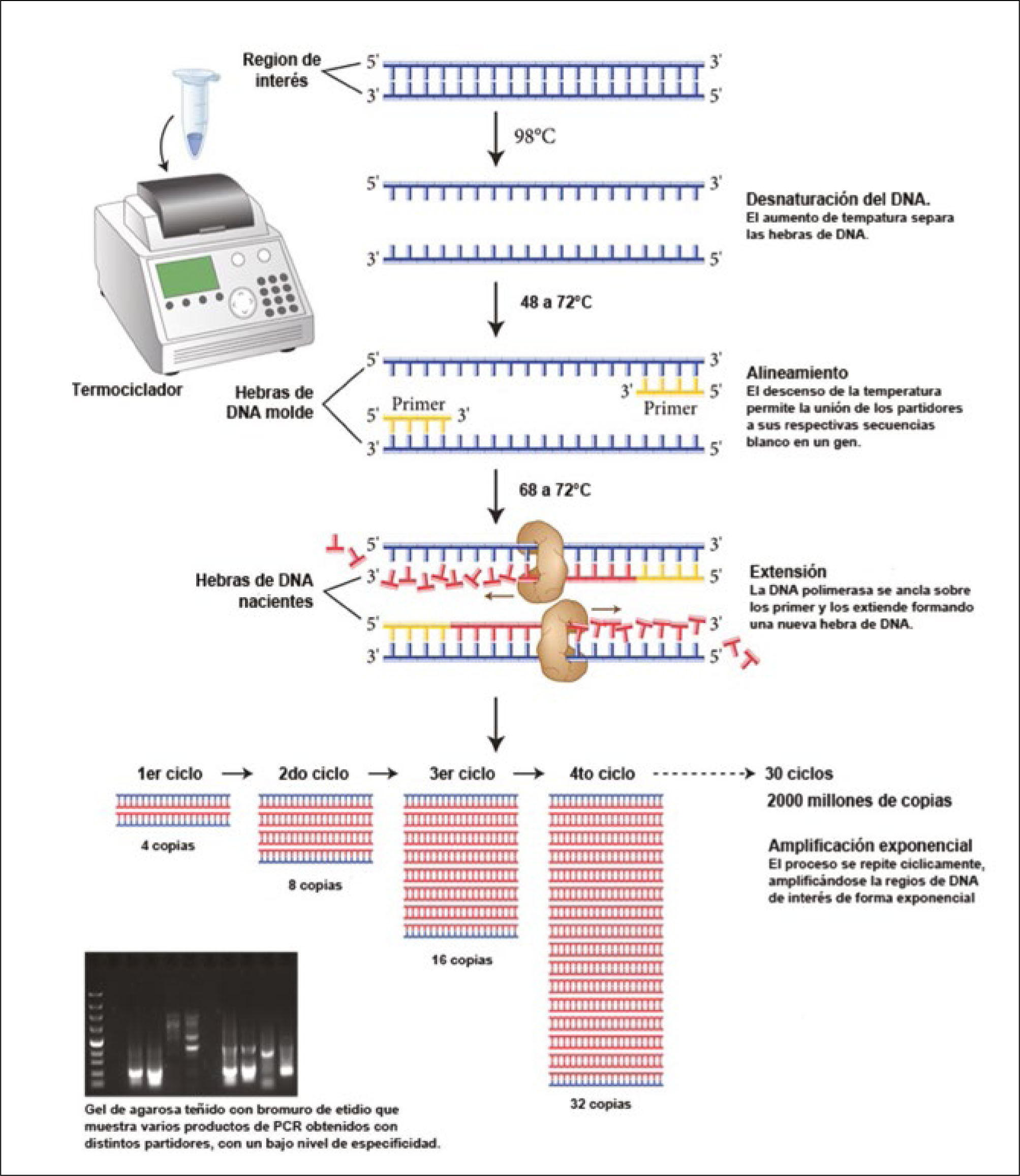
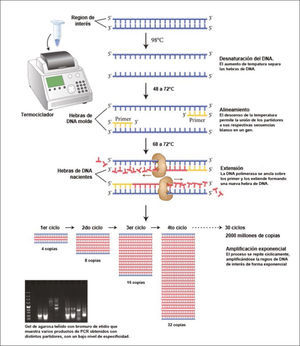
La técnica de PCR descansa en la propiedad natural de la enzima DNA polimerasa para replicar hebras de DNA. Para ello se emplean ciclos alternados de altas y bajas temperaturas, elevando la temperatura de reacción para separar las dobles hebras de DNA, permitir la replicación de cada una de las hebras simples, dejando que posteriormente vuelvan a unirse al bajar la temperatura, formando más y nuevas dobles hebras tras cada ciclo de replicación. De esta forma se logra una amplificación exponencial de cada hebra de DNA molde original. Debido a que la temperatura necesaria para la separación de las hebras de DNA (fase de desnaturalización del DNA) alcanza los 95°C se emplean ADN polimerasas termoestables, extraídas de microrganismos, generalmente arqueas, como Thermus aquaticus o Pyrococcus furiosus, adaptados a vivir a altas temperaturas (ej. géiseres).
Técnicamente, el proceso de PCR consiste simplemente en una serie de 20 a 35 cambios repetidos de temperatura llamados ciclos. Cada ciclo suele consistir a su vez en 3 etapas a diferentes temperaturas (Figura 2). Las temperaturas usadas y el tiempo aplicado en cada ciclo dependen de gran variedad de parámetros tales como la enzima usada para la síntesis de DNA, la concentración de iones y de los nucleótidos libres necesarios para la síntesis de la nueva hebra de DNA, y la temperatura de unión de los partidores5, así como la longitud o tamaño del DNA que se desea amplificar. Hoy, el proceso es parcialmente automatizado mediante un aparato llamado termociclador, que permite calentar y enfriar rápidamente los tubos de reacción, controlando finamente la temperatura necesaria en cada etapa de la reacción6. En la práctica, los “ingredientes” de una PCR son (i) una mínima cantidad del DNA molde que se desea amplificar (como el DNA extraído de un tumor), (ii) los partidores específicos para el fragmento de DNA que deseamos amplificar, (iii) la enzima DNA polimerasa termoestable, (iv) un exceso de nucleótidos libres A, T, C y G que formarán parte de las nuevas hebras de DNA que se sintetizarán en cada ciclo y finalmente (v) agua, sales y iones necesarios para ofrecer condiciones de pH óptimas, todo en un tubo de ensayo de un volumen menor a 1 mL. El tubo se somete a tres distintas temperaturas en forma cíclica, obteniéndose finalmente un tubo con gran cantidad de copias del DNA molde original.
Reacción en cadena de la polimerasa o PCR
Esquema del proceso cíclico de amplificación de un fragmento de DNA específico, por acción de la DNA polimerasa. Al subirse la temperatura de la reacción se logra separar las 2 hebras del DNA. Bajando la temperatura se logra la unión específica al DNA blanco de unos pequeños fragmentos de DNA, llamados partidores o primers, diseñados in vitro para ser compatibles con regiones conocidas del gen que se desea estudiar. Esta unión permite el posicionamiento correcto de la polimerasa, la que comienza a replicar la hebra molde usando nucleótidos libres agregados a la mezcla de reacción. Al cabo de 30 ciclos se habrá logrado la generación de más de 2 mil millones de copias del fragmento original, permitiendo tener una gran cantidad de DNA de interés replicado para la realización de diversos estudios, como la secuenciación en busca de mutaciones. Esquema adaptado de New England Biolabs.
¿Cómo puedo amplificar específicamente una región de un gen en la que sospecho puede existir una mutación? La especificidad de la reacción de PCR está dada por los partidores o “primers”. Como se mencionó, se conoce completamente la secuencia del genoma humano. Esto es equivalente a decir que sabemos exactamente lo que dice cada uno de los 27000 libros de nuestra biblioteca génica. La probabilidad de que más de un gen o “libro” contenga una página que comience y finalice con exactamente la misma combinación de palabras es muy baja. Entonces, conociendo las secuencias nucloeotídicas o “palabras” con que comienza y termina nuestra página de interés, podemos especificar nuestro “fotocopiado génico” exactamente a nuestra página deseada. En otras palabras, es como utilizar en nuestro computador un procesador de texto para que busque una página que comience y termine con una combinación específica de palabras. Las palabras iniciales y finales corresponden a las secuencias hacia las cuales se dirigen los partidores para alinearse y unirse por complementariedad de bases, dando así inicio al proceso de amplificación. La enzima DNA polimerasa “lee” una secuencia de DNA de hebra simple y sintetiza la hebra complementaria inmediatamente a medida que avanza en la lectura, como un escáner genético; si lee una A, introduce una T, si lee una G, introduce una C a la nueva cadena de DNA en formación. Sin embargo, para poder comenzar a sintetizar las copias de DNA, la DNA polimerasa necesita instalarse sobre una pequeña región de DNA de doble hebra. Esta región esta dada por la unión del partidor diseñado in vitro a la secuencia blanco en el genoma. Así se da inicio específicamente al copiado del DNA en la región escogida previamente.
Secuenciación génica y otras técnicas de diagnóstico molecularUna vez amplificado un gen mediante PCR, el fragmento de DNA amplificado o amplicón puede secuenciarse y leerse completamente, letra por letra, nucleótido por nucleótido. La secuencia resultante se entrega como un gráfico llamado electroforetograma (Figura 3) con cientos de peaks de cuatro colores que representan los cuatro nucleótidos. La ausencia de un peak, o un peak extra, o un doble peak, son señales que pueden dar cuenta de la presencia de una mutación. El análisis profesional y acucioso de las secuencias permite discriminar entre una mutación real y posible falsos positivos producto de artefactos propios de la técnica de secuenciación.
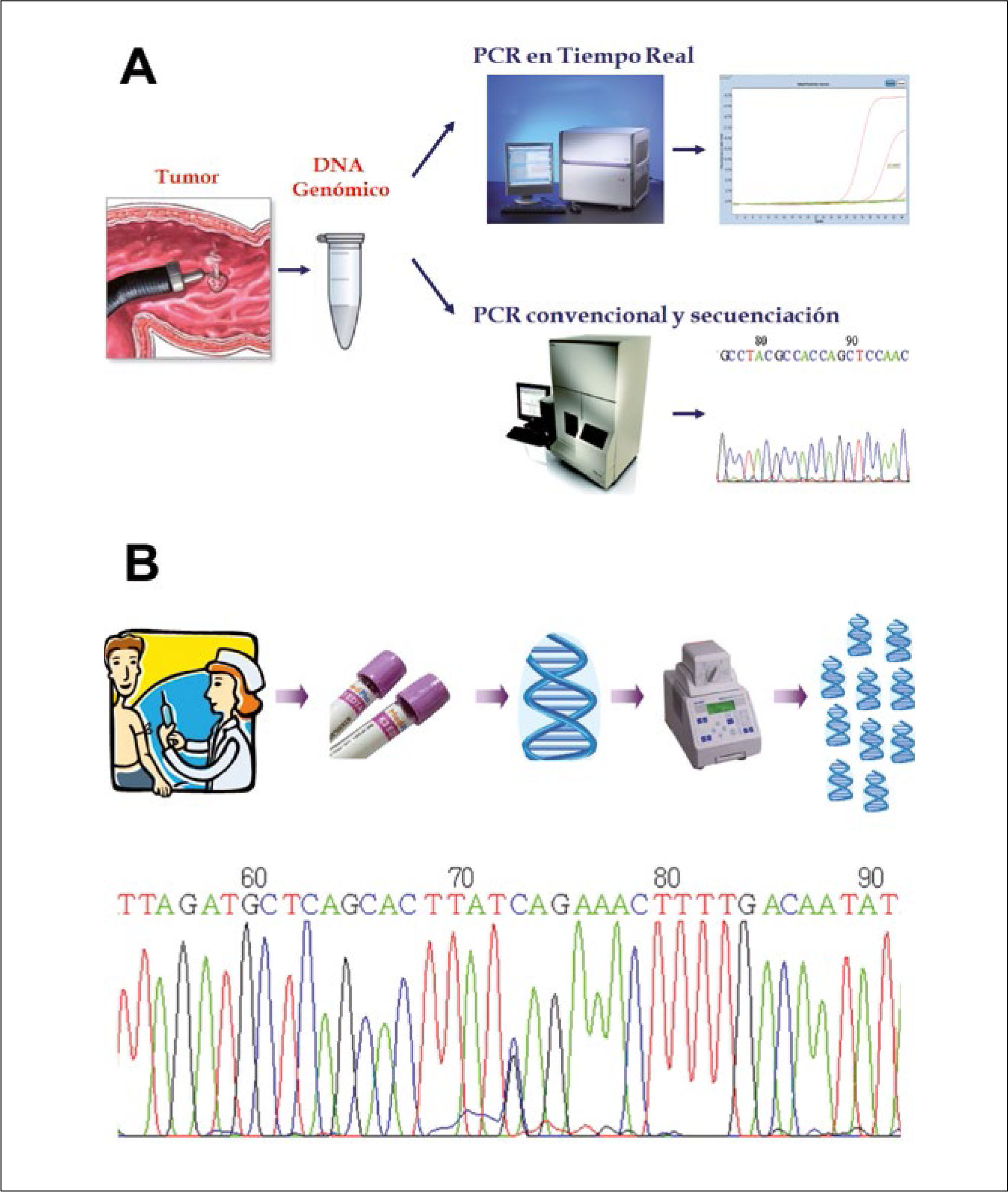
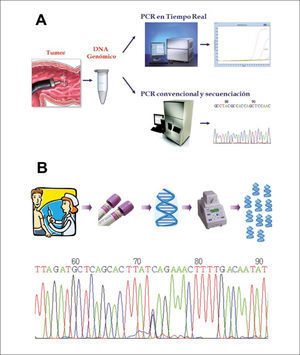
Esquema general de las estrategias para estudio de mutaciones
(A) El estudio de mutaciones somáticas se realiza a partir del DNA extraído de una pieza tumoral. Es amplificado mediante PCR y estudiado por secuenciación o PCR en tiempo real, técnica que incorpora partidores fluorescentes que permiten aumentar enormemente la sensibilidad y disminuir los tiempos de trabajo. (B) El estudio de mutaciones de línea germinal se realiza a partir del DNA extraído de una muestra de sangre del paciente. Se amplifica mediante PCR y se secuencia en busca de mutaciones, las que se visualizan en un electroforetograma como el doble peak correspondiente al nucleótido 73 de la secuencia mostrada.
Existen otras técnicas moleculares utilizadas en el diagnóstico molecular de una mutación o alteración genética. Podemos destacar algunas como (i) hibridación in situ de DNA fluorescente o FISH (del inglés Fluorescence in situ hybridization), que permite realizar estudios de citogenética molecular, detectando anomalías cromosómicas grandes como aneuploidias o reordenamientos cromosómicos, lo que puede implicar la pérdida o sobre-representación de aquellos genes que se encuentren en fragmento cromosómico estudiado. En la técnica de FISH se utiliza un fragmento de DNA de hebra simple llamado sonda que es complementario a la región cromosómica de interés, y que esta marcado con una molécula fluorescente. La sonda fluorescente permite la visualización microscópica de los fragmentos cromosómicos duplicados y su cuantificación. (ii) La técnica de MLPA o Multiplex ligationdependent probe amplification permite detectar rearreglos genéticos grandes, como pérdidas o duplicaciones de exones de un gen, lo que puede implicar la obtención de una proteína truncada y sin función o una enzima inactiva. Esta técnica también se basa en la unión de una sonda fluorescente específica para distintas regiones de un gen específico. A diferencia de la técnica de FISH que permite visualizar rearreglos en un cromosoma, la técnica de MLPA permite visualizar rearreglos en un gen. Los resultados se visualizan en un equipo de PCR adaptado para detectar la señal fluorescente de la sonda y cuantificarla. (iii) Los microarreglos de DNA o DNA microarray permiten ver el patrón de expresión de miles de genes a la vez. Utilizando el mismo principio que las técnicas de FISH y MLPA donde sondas marcas fluorescentemente, permiten visualizar la presencia, ausencia y/o cantidad relativa de miles de genes (o de mRNA) a la vez. Sistemas robotizados logran ordenar sobre una pequeña superficie como un portaobjeto de vidrio miles de pequeñísimas gotas, cada una conteniendo sondas o fragmentos de distintos genes conocidos. El portaobjeto con las sondas adheridas se incuba con una muestra de DNA (ej. DNA total extraído de un tumor), su resultado se compara con el resultado obtenido de una muestra control (ej. tejido sano). Así es posible visualizar el patrón de expresión génica, de forma amplia, pero específica, y con mucha sensibilidad. Cabe mencionar que gran parte de las patologías con un componente genético tienen más de un gen alterado, por lo que la visualización a través de DNA microarray de la expresión génica permite detectar conjuntos de genes potencialmente alterados e involucrados en algun cáncer. Detección de DNA libre circulante de células tumorales circulantes se presentan como alternativas diagnósticas y pronósticas a corto plazo. En Clínica Las Condes ya se trabaja en un banco de suero y plasma de pacientes con cáncer para la detección de DNA tumor libre en la circulación que permita a futuro la implementación de una técnica de pronóstico o seguimiento que sólo requiera de una muestra de sangre por parte del paciente.
Consejería genéticaLos avances científicos y tecnológicos han permitido el advenimiento de sistemas de detección de diversos tipos de aberraciones genéticas y genómicas que permiten obtener resultados con una sensibilidad extraordinaria, a una velocidad inimaginada sólo algunos años atrás (ej. sistemas de secuenciación masiva, PCR en tiempo real, etc). Sin embargo, la cantidad y complejidad de los resultados obtenidos puede ser abrumadora e incomprensible incluso para el cuerpo médico y, más importante aún, para el paciente con cáncer. Entender que una mutación puede predisponer al desarrollo de cáncer, que pudo ser heredada (o no) de uno de sus padres y que por lo tanto puede ser transmitida a su descendencia, es fundamental para desarrollar estrategias de prevención y/o de vigilancia activa, y de apoyo emocional. La consejería genética cobra un rol fundamental en el acercamiento de la brecha que existe entre el mundo de la ciencia básica y el paciente. Profesionales capacitados en consejería genética son fundamentales para aclarar dudas y conceptos en un paciente que ya debe lidiar con una carga emocional fuerte relacionada a su enfermedad o a la de un familiar. Entender conceptos básicos de biología molecular del cáncer ayuda a aclarar estos conceptos que parecen oscuros y complejos, pero que en el fondo son simples y disponer de ellos puede hacer una diferencia en el tratamiento integral de un paciente oncológico.
El autor declara no tener conflictos de interés, con relación a este artículo.
Se llama transcripción al proceso de paso de DNA a RNA. Tanto el DNA como el RNA están compuestos por las mismas unidades básicas, los nucleótidos adenina (A), timina (T), guanina (G) y citosina (C). Haciendo una analogía literaria es posible imaginar que el DNA y RNA “hablan” un mismo idioma, el idioma de los nucleótidos. Cobra sentido entonces el hecho que el nombre del proceso de paso de DNA a RNA se llame transcripción, ya que solo se esta transcribiendo o copiando un mensaje en el mismo idioma.
Se llama traducción al proceso de paso de RNA mensajero (o mRNA) a una proteína. A diferencia del DNA y RNA, las proteínas están compuestas por unidades básicas distintas a los nucleótidos, los aminoácidos. Continuando con la analogía anterior, en el paso del RNA a proteínas existe un “cambio de idioma”, traduciendo el idioma de los nucleótidos al idioma de los aminoácidos.
Inicialmente se aceptaba el axioma de “Un gen, una proteína” haciendo referencia a que cada proteína de nuestro cuerpo estaba codificada por su único y exclusivo gen. El genoma humano esta compuesto por aprox. 27000 genes. Sin embargo, poseemos mucho más de 27000 proteínas distintas. En la actualidad se sabe que un gen puede dar origen a más de una proteína mediante un proceso llamado “corte y empalme alternativo” o “splicing alternativo”.
La vía de señalización de EGFR esta comúnmente activada en varios tipos de cáncer por la sobreexpresión del receptor de EGF. Normalmente la vía se activa por unión de un ligando al receptor de EGF, el cual se dimeriza y activa, transmitiendo y amplificando la señal hacia el interior de la célula hasta el núcleo donde se iniciará la expresión de un sinnúmero de genes que orquestarán una respuesta celular coordinada y regulada. Esta señal se transmite al núcleo celular a través de una serie de proteínas intermediarias que traspasan una señal química (ej. fosforilación) de una a otra, como una posta de relevos en atletismo o un efecto dominó. Cuando una de las proteínas intermediarias (como KRAS) esta mutada y constantemente activada, la señal de activación se envía al núcleo celular pese a no haber estimulo alguno en el receptor de EGF. Pacientes con mutaciones en KRAS no serán buenos respondedores a terapias que busque bloquear el receptor de EGF ya que la vía seguirá activa por acción de la proteína KRAS mutada.
Los partidores son fragmentos cortos de DNA de hebra simple de aproximadamente 15 a 25 nucleótidos de longitud, diseñados específicamente para ser complementarios a la secuencia génica de interés según las reglas de Watson y Crick donde A se une a T y C se une a G.
El sistema original usaba grandes fuentes con agua a distintas temperaturas en las cuales se sumergían manualmente los tubos de ensayo con la mezcla de reacción, durante varios ciclos.