El tratamiento para cáncer de próstata localizado (prostatectomía radical o radioterapia) ofrece unas altas tasas de curación; sin embargo, del 20 al 30% de los casos desarrollan recurrencia bioquímica. Actualmente, existen factores clínicos y patológicos que ayudan a predecir recurrencia; no obstante tanto el carácter heterogéneo de estos tumores, las diferencias en los tiempos de progresión de cáncer localizado a metastásico como la resistencia al tratamiento han dado lugar a imprecisiones en la predicción del pronóstico y a tratamientos insuficientes o excesivos. Debido a esto se han estudiado biomarcadores con el fin de estratificar más acertadamente el riesgo y mejorar las decisiones de tratamiento de una manera adecuada y oportuna. Este manuscrito presenta una revisión de marcadores moleculares de pronóstico que se han propuesto en los pacientes con cáncer de próstata localizado, lo que podría permitir establecer con mayor precisión el riesgo de recurrencia de la enfermedad.
Treatment for localised prostate cancer (radical prostatectomy or radiotherapy), offers high cure rates; nevertheless, 20% to 30% of the cases develop biochemical recurrence. There are clinical and pathological features that are currently being used to predict recurrence of the disease. However, tumour heterogeneity in prostate cancer, along with differences in time of progression to metastasis and treatment resistance, have led to inaccuracies in predicting the risk of biochemical relapse, and therefore, misleading in treatment decisions. Because of this, many genetic markers have been studied in order to refine risk stratification and improve treatment decisions in a suitable and opportune manner. This paper presents a review of molecular prognostic markers that have been proposed in patients with localised prostate cancer, which potentially could allow establishing the risk of recurrence of the disease more accurately.
A nivel mundial el cáncer de próstata (PC, por sus siglas en inglés) es el segundo cáncer más incidente y la quinta causa de muerte por cáncer en hombres1. En Colombia, es el tipo de cáncer más incidente con cerca de 9.000 casos nuevos al año por cada 100.000 habitantes, que ocasionan 2.400 muertes, y lo constituye en la segunda causa de mortalidad por cáncer en hombres2.
A partir de la implementación de la prueba del antígeno prostático específico (PSA, por sus siglas en inglés), aprobada en 1994 como prueba de tamización, se registró un aumento en el número de casos con PC, principalmente en estadios tempranos3,4. Algunos de estos casos corresponden a enfermedad latente, y deben estar sujetos a seguimiento para detectar una posible progresión; mientras que otros casos son sometidos a tratamientos con intención curativa, como prostatectomía radical (PR) o radioterapia, a pesar de sus efectos secundarios. Dentro de estos últimos, pueden existir algunos pacientes mal clasificados que realmente no requerían tratamiento sino seguimiento y que tuvieron un deterioro en su calidad de vida sin un beneficio significativo en la supervivencia5.
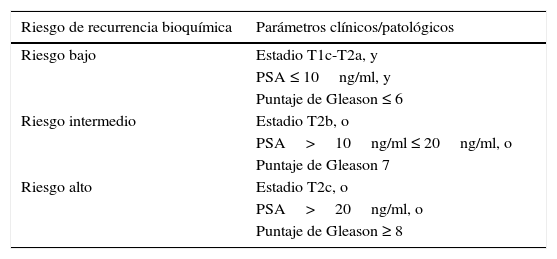
Con el fin de mejorar la clasificación de los pacientes, actualmente se usan herramientas clínicas y patológicas, como el nivel de PSA inicial, la estadificación clínica y el puntaje de Gleason en la biopsia, para ayudar a predecir el pronóstico del PC al momento del diagnóstico y determinar el tratamiento (tabla 1); sin embargo, estas aún no son suficientes. Por ejemplo, se ha reportado que cerca del 15% al 20% de los pacientes con cáncer localizado y 30% de los pacientes con riesgo intermedio presentan recurrencia bioquímica (BCR, por sus siglas en inglés, y definida posterior a la PR como la presencia de PSA de 0,2ng/ml o mayor en los exámenes de seguimiento) posterior al tratamiento estándar con intención curativa6–8. Lo anterior exhibe una gran limitación en el manejo de la enfermedad puesto que una clasificación imprecisa y un tratamiento inadecuado pueden afectar la supervivencia global de los pacientes y aumentar innecesariamente los costos relacionados con el tratamiento.
Clasificación del riesgo de BCR. Parámetros clínicos/patológicos establecidos para la clasificación del riesgo de BCR al momento del diagnóstico6,13,14
| Riesgo de recurrencia bioquímica | Parámetros clínicos/patológicos |
|---|---|
| Riesgo bajo | Estadio T1c-T2a, y |
| PSA ≤ 10ng/ml, y | |
| Puntaje de Gleason ≤ 6 | |
| Riesgo intermedio | Estadio T2b, o |
| PSA>10ng/ml ≤ 20ng/ml, o | |
| Puntaje de Gleason 7 | |
| Riesgo alto | Estadio T2c, o |
| PSA>20ng/ml, o | |
| Puntaje de Gleason ≥ 8 |
Con el propósito de abordar estas limitaciones, se han estudiado una serie de marcadores genéticos con el fin de precisar la estratificación del riesgo mediante mediciones estandarizadas y delimitaciones establecidas que no dependan del observador; esto con el fin de ofrecer un tratamiento oportuno y así evitar el sobretratamiento en casos innecesarios. Este manuscrito hace una revisión de nuevos marcadores propuestos para predecir con mayor precisión el pronóstico de los pacientes con PC localizado al momento del diagnóstico.
MetodologíaSe realizó una búsqueda en la base de datos Pubmed con los términos “prostate cancer prognosis biomarkers”, con los siguientes filtros: fechas de publicación en los últimos cinco años y artículos en idioma inglés. Se seleccionaron únicamente artículos originales cuyas investigaciones involucraran específicamente el análisis de muestras de PR de pacientes con PC, con el fin de seleccionar y describir los biomarcadores de pronóstico más relevantes relacionados con el pronóstico en estos pacientes.
Actuales herramientas en el diagnóstico y seguimiento del cáncer de próstata y sus limitacionesActualmente, existen dos pruebas de tamización que permiten una detección temprana del PC: el PSA y el examen del tacto rectal. Resultados anormales en alguno de estos exámenes conllevan a la realización de la biopsia para establecer el diagnóstico que puede ir desde una enfermedad confinada al órgano o localizada hasta una enfermedad avanzada o metastásica. En la biopsia se establece el puntaje de Gleason que define el grado de diferenciación histológica del tumor y en el que se reportan los dos patrones de diferenciación más prevalentes en la muestra9. Esta clasificación se correlaciona con el pronóstico del paciente, tanto en la predicción de BCR como en la aparición de metástasis, donde el patrón más diferenciado tiene un mejor pronóstico, y el patrón más indiferenciado tiene un peor pronóstico6,9–12.
El estadio clínico tumoral (cT) evaluado en el examen del tacto rectal, junto con el puntaje de Gleason y el nivel de PSA inicial, permiten clasificar al momento del diagnóstico el riesgo de BCR del paciente en tres grupos: bajo, intermedio y alto 6,13,14 (tabla 1). Esta clasificación contribuye a la toma de decisión del tratamiento al que debe ser sometido el paciente una vez que es diagnosticado, ya que la BCR siempre precede a la recurrencia clínica con un tiempo promedio de 8 a 10 años15. Además, el uso del PSA y del puntaje de Gleason se han extendido en diversos métodos de estimación del riesgo pronóstico, no solo en la clasificación desarrollada por D’Amico6,14, sino también en el puntaje CAPRA (Determinación del riesgo de PC) 16,17 y en los diferentes nomogramas que involucran modelos matemáticos y variables de riesgo 18,19. Con respecto al seguimiento, las mediciones de PSA posteriores al tratamiento, trimestralmente durante el primer año y semestralmente en el segundo año20, son una de las principales herramientas de seguimiento de la enfermedad en la práctica clínica21,22.
Sin embargo, todas estas herramientas exhiben limitaciones. Por ejemplo, los niveles de PSA no siempre conservan una relación directa con el diagnóstico de la enfermedad o su recurrencia, ya que estos también aumentan en respuesta a otras afectaciones de la próstata como prostatitis, y también porque algunos casos pueden recurrir en ausencia del incremento de PSA23,24. De otra parte, el examen del tacto rectal permite la palpación únicamente de la zona periférica de la próstata y es examinador dependiente, por lo que puede no detectar algunos tumores. Finalmente, el uso del Gleason se ve limitado por la variabilidad de la lectura histopatológica (inter-observador e intra-observador)25. Estas imprecisiones conllevan a que la estratificación del riesgo varíe en rangos muy amplios que se superponen entre ellos, con la consecuencia de ser poco útil para diferenciar especialmente los casos de riesgo bajo e intermedio al momento del diagnóstico.
Estas dificultades han conllevado a la búsqueda de marcadores moleculares que sean más sensibles y específicos y que permitan mejorar la clasificación del riesgo de recurrencia, incluso en pacientes con tumores latentes. En la actualidad, se han adelantado esfuerzos de la comunidad científica con el fin de entender mejor la biología del tumor y su pronóstico, mediante la utilización de metodologías de punta para la identificación de nuevos marcadores que permitan determinar de manera más acertada el pronóstico de los pacientes; además, se han asociado específicamente con el puntaje de Gleason dado que se ha constituido como el marcador de pronóstico más ampliamente usado en la clínica6,10,11. Se espera que el hallazgo de una firma o perfil de expresión génica permita clasificar los pacientes como potenciales respondedores o no respondedores al tratamiento inicial, con gran uso clínico en biopsias de diagnóstico iniciales o muestras líquidas, como orina y suero.
Biomarcadores de BCR en cáncer de próstata localizado y asociados con el puntaje de GleasonNumerosos estudios han sido enfocados en la identificación de marcadores moleculares que permitan definir un mejor pronóstico relacionado con BCR. Algunos de estos son descritos en la tabla 2. Dentro de los biomarcadores identificados, se ha observado que una fuerte expresión de la integrina α3, determinada por inmunohistoquímica triplica el riesgo de BCR de la enfermedad26; asimismo, la ausencia de expresión de la integrina β1 se asoció con una mayor probabilidad de BCR (2,8 veces más alta) con supervivencia media de 77 meses frente a 112 meses en pacientes con expresión positiva27. Otro biomarcador estudiado ha sido la proteína interactuante DAB2 (DAB2IP), también conocido como proteína activante de Ras GTPasa28–30, cuya pérdida de expresión citoplasmática junto con una fuerte tinción nuclear del receptor de andrógenos es más frecuente en muestras de pacientes recurrentes y hormonorrefractarios31.
Biomarcadores de pronóstico descritos en cáncer de próstata (PC) localizado, identificados en tejido de PR, CTCs, orina, plasma y suero
| Biomarcador (gen) | Función normal | Función aberrante en cáncer | Cambio en la expresión | Significancia en pronóstico | Referencias | |
|---|---|---|---|---|---|---|
| Identificados en Prostatectomía Radical | Integrina α3 | Interacción entre la célula con la matriz extracelular, adhesión celular | Metástasis | ↑ | MENOR SUPERVIVENCIA DE BCR | 26,80 |
| Integrina b1 | Interacción entre la célula con la matriz extracelular, adhesión celular | Metástasis | ↓ | 26,80 | ||
| DAB2IP | Inhibe transición epitelial-mesenquimal, induce apoptosis | Crecimiento celular y supervivencia | ↑ | 81 | ||
| TMPRSS2-ERG | Fusión génica ausente en células normales | Desarrollo y progresión de cáncer de próstata | ↑ | 33 | ||
| NUCB2 | Mantenimiento de niveles de calcio, homeóstasis energética y control del apetito en el hipotálamo | Secreción gástrica en ca. gástrico. Metástasis ganglionar en ca. seno ER+ | ↑ | 82–84 | ||
| SPOP | Regula expresión génica por ubiquitinación | ↓ | 36,37 | |||
| HMGB1 | Regulación de transcripción génica, inflamación, diferenciación celular | Proliferación, angiogénesis, metástasis | ↑ | 85,86 | ||
| ARSB | Regula expresión de proteoglicanos de la matriz extracelular | Proliferación celular | ↓ | 42,87 | ||
| EZH2 | Remodelamiento de cromatina y silenciamiento de genes | Proliferación, invasión, formación tumoral y metástasis | ↑ | 88,89 | ||
| CNR1 | Modula liberación de neurotransmisores | Proliferación celular | ↑ | 90,91 | ||
| APN | Procesamiento postsecretorio de neuropéptidos y su acceso a los receptores en la célula | Invasión, metástasis, neoangiogénesis | ↓ | 46–48 | ||
| MAP3K7 | Controla crecimiento celular, diferenciación y apoptosis | Desregulación de crecimiento celular y apoptosis | ↓ | 51 | ||
| PTEN | Supresor tumoral, regula proliferación, supervivencia celular, estabilidad genómica, migración celular, auto-renovación de células madre | Regula el microambiente tumoral | ↓ | 92 | ||
| Cbx3 | Reparación del DNA, empaquetamiento de cromatina, regulación génica | Regulación génica alterada | ↑ | 55,93 | ||
| Cyr61 | Adhesión, migración, proliferación, diferenciación, supervivencia celular | Migración, invasión y proliferación | ↓ | 94,95 | ||
| SOX9 | Proliferación, diferenciación celular en embriogénesis | Proliferación | ↑ | 58 | ||
| SOX7 | Regulación de la transcripción, degradación de B-catenina | Proliferación | ↓ | 96,97 | ||
| Identificado en CTCs y orina | TMPRSS2-ERG | Fusión génica ausente en células normales | Desarrollo y progresión de cáncer de próstata | ↑ | 33 | |
| Identificado en plasma | hTERT | En células germinales mantiene la longitud telomérica y actividad basal en células somáticas normales | Síntesis de DNA telomérico para el mantenimiento de la longitud telomérica y capacidad proliferativa de células tumorales | ↑ | 62,98 | |
| Identificado en orina | SPINK1 | Inhibición de proteasas serina, como la tripsina, para prevención prematura de zimógenos | Estimula crecimiento celular | ↑ | 68,99 | |
| Identificado en suero | mir-26a | Regulación de expresión génica | Expresión reducida en carcinoma hepatocelular, de tiroides, rabdomiosarcoma y carcinoma renal de células claras. Expresión aumentada en gliomas | ↓ | 100–103 |
En un estudio realizado por Zhang et al. 32, se observó que aumento en la expresión génica de nucleobindina 2 (NUCB2) en tejido tumoral prostático comparado con tejido adyacente no canceroso se correlaciona con un mayor puntaje de Gleason, así como un mayor nivel de PSA prequirúrgico, metástasis a los ganglios linfáticos e invasión linfovascular. Igualmente, pacientes con niveles incrementados presentan una menor supervivencia libre de BCR, lo cual permite identificar pacientes con un mayor riesgo y que probablemente necesitarían un tratamiento más agresivo.
La fusión génica entre TMPRSS2 (21q22.2) y ERG (21q22.3), TMPRSS2-ERG (proteasa transmembrana serina 2 - gen relacionado con ETS), presente en el 30% a 70% de los casos de PC, favorece la expresión oncogénica de ERG33. Asimismo, se ha reportado que la fusión TMPRSS2-ERG está altamente asociada con una mayor BCR en el 58% de los pacientes positivos para la fusión, frente al 8% de recurrencias observadas en pacientes sin la fusión en un periodo de 5 años posterior al tratamiento34. Sin embargo, un estudio publicado recientemente que fue realizado en muestras de biopsias no encontró asociación entre la expresión de TMPRSS2-ERG con un riesgo incrementado en la mortalidad por PC35.
El gen SPOP (proteína speckle-type POZ) codifica la proteína adaptadora de la ubiquitin ligasa E3 “speckle-type” POZ (siglas en inglés de: proteína de dedos de zinc y virus de pox)36,37, y presenta mutaciones somáticas en el 6% al 15% de los casos de PC38. Su baja expresión fue asociada con menor supervivencia libre de BCR y supervivencia libre de progresión, así como con patrones de Gleason más avanzados. Esta expresión no se asoció con las mutaciones del gen que fueron una variable independiente de mal pronóstico en casos negativos para la fusión génica TMPRSS2-ERG39. En otro estudio no se encontró asociación entre las mutaciones del gen con el puntaje de Gleason ni con la BCR de la enfermedad40.
La proteína de alta movilidad del grupo de caja 1 (HMGB1) o anfoterina es una proteína de unión al DNA nuclear de la cromatina. Una correlación positiva de la proteína fue observada con una menor supervivencia de BCR, con una tasa de supervivencia a 5 años del 6%, frente a un 54% en los casos con ausencia de expresión. Sin embargo, este mismo estudio encontró que la expresión positiva estuvo más representada por casos con puntaje de Gleason favorable ≤ 641.
Otra proteína estudiada como marcador es la enzima arisulfatasa B (ARSB), que fue analizada en casos pareados con y sin BCR. Una expresión disminuida fue observada en el 82% de las muestras de PR de casos recurrentes42. Al comparar los niveles de PSA iniciales, estos estuvieron disminuidos en el 65% de los casos, por lo que al combinar la expresión aumentada de ARSB junto con los bajos niveles de PSA, se encontró una predicción del 95% de las recurrencias. Adicionalmente, la expresión de ARSB estuvo inversamente asociada con el puntaje de Gleason, encontrándose mayor expresión en los puntajes 6 y 7 y menor expresión en Gleason 8 y 9.
Por su parte, el gen enhancer de zeste 2 asociado al complejo represor polycomb 2 (EZH2) codifica una enzima lisina metiltransferasa de histonas43. Su expresión en pacientes con riesgo intermedio y tratados con PR constituye un predictor de BCR en PC localizado44. Sin embargo, una mayor precisión en la predicción de la BCR posterior a la PR se observó al identificar la expresión en conjunto del protooncogen c-Myc y EZH2 con ausencia de expresión de la enzima inhibidora de quinasa dependiente de ciclina (CDKN1B), codificada por el gen p27Kip144.
La alta expresión del gen receptor canabinoide tipo 1 (CNR1), encargado de inhibir la actividad de la adenilato ciclasa en PC fue asociada con mal pronóstico y se determinó, mediante inmunohistoquímica, que en pacientes con riesgo intermedio los niveles de expresión de la proteína discriminan el pronóstico45.
La expresión de la proteína de membrana celular aminopeptidasa N (APN), codificada por el gen ANPEP46–48, está disminuida en tejido tumoral respecto a tejido normal. En cáncer avanzado o metastásico se ha observado que la expresión se pierde por completo 49 y se asocia con menores tiempos de supervivencia libre de recurrencia tumoral. Por lo tanto, la expresión de APN fue confirmada como un factor de pronóstico independiente en pacientes con PC clínicamente localizado49. Sin embargo, la predicción de supervivencia mejora considerablemente al analizar la expresión conjunta de APN con la de densidad de microvasos (MVD), o con la del factor de crecimiento endotelial vascular (VEGF)49.
Una de las alteraciones genéticas encontrada en tumores de PC es la deleción de la región 6q14-21 que implica la pérdida de expresión de la proteína Tak1 en el 35% de los casos50. Esta es una proteína supresora tumoral, miembro de la familia de proteínas MAPKKK y codificada por el gen MAP3K7 (proteína 3 quinasa activada por mitógeno 7)51. Esta proteína ha sido asociada con: altos valores de PSA prequirúrgicos (≥10ng/μl); estadios avanzados del tumor (desde estadios pT3 establecidos por patología); presencia de ganglios linfáticos metastásicos, y PC de alto grado (Gleason ≥8)50,52. Asimismo, las deleciones de este gen se asocian con una BCR más rápida y mayor agresividad tumoral50.
Otro de los genes que se asocia con una menor supervivencia libre de progresión en pacientes tratados con PR, específicamente en pacientes positivos para ERG, es el gen supresor tumoral que codifica para la proteína fosfatasa y homólogo de tensina (PTEN) y que presenta deleción o mutaciones puntuales en PC53. Constituye un marcador de pronóstico independiente en los subgrupos tumorales con estadio pT2, puntaje de Gleason 7 y ERG positivos, así como en tumores pT2, Gleason <7 y niveles de PSA iniciales entre 10 y 30ng/ml. Dentro de estos subgrupos, los casos con expresión negativa de PTEN tienen una menor supervivencia libre de progresión54.
En PC también se reporta expresión incrementada de la proteína gamma 1 de la heterocromatina (HP1γ), codificada por el gen Cromobox homólogo 3 (Cbx3)55, la cual se asocia con patrones de Gleason superiores a 4+3, y cuya expresión citoplasmática predice BCR en pacientes con PR y permite separar pacientes con Gleason 7 con alta y baja probabilidad de BCR55.
Otra proteína secretada a la matriz extracelular que se encuentra aumentada en tumores con Gleason ≥8 es la proteína inductor angiogénico rico en cisteína 61 (Cyr61)56. Tumores con al menos una región con una fuerte tinción de la proteína, medida por inmunohistoquímica, fueron 56% menos propensos a presentar BCR. Sin embargo, la intensidad de la tinción no se asoció linealmente con el riesgo de BCR. Al analizar la asociación entre el nivel de expresión de Cyr61 con las características clínicopatológicas de los pacientes, se encontró una mayor proporción de estadios pT3b o superiores con una mayor expresión proteica57.
Los genes SOX (caja HMG - grupo conservado de alta movilidad relacionada con SRY - región Y determinante del sexo) son una familia de factores de transcripción involucrados en el desarrollo embrionario de la próstata que posteriormente en células diferenciadas restringe su expresión a células basales58. La alta expresión de Sox9 (caja 9 de SRY) y baja expresión de Sox7 (caja 7 de SRY) en PC localizado predicen una supervivencia más corta libre de BCR59, en la que expresión de Sox7 se asoció con altos niveles séricos de PSA iniciales y presencia de metástasis. La expresión incrementada de la proteína Sox9 se asoció con mayores puntajes de Gleason y estadios clínicos, y la baja expresión de Sox10 se asoció con mayores niveles séricos de PSA y estadios patológicos avanzados.
Biomarcadores de pronóstico identificados en células tumorales circulantes (CTRCs), suero y orinaEl gen fusión TMPRSS2-ERG también ha sido reportado en células tumorales circulantes (CTCs) y tumor de pacientes con PC hormonorrefractario, independientemente si los pacientes recibieron tratamiento con un inhibidor selectivo de la enzima 17α-hidroxilasa/C17, 20-liasa (CYP17), la cual interviene en la biosíntesis de los andrógenos. Estos resultados sugieren que la fusión no cambia durante la progresión del PC ni durante el tratamiento con el inhibidor60. También se halló en el 50% de muestras de orina, lo que permite aumentar el valor predictivo del Gleason y del estadio clínico en biopsias, y en la predicción de extensión extraprostática en la pieza quirúrgica; esta metodología podría usarse para seleccionar casos con PC clínicamente significativos61.
Por su parte, hTERT, subunidad catalítica con actividad transcriptasa inversa de la enzima telomerasa, se ha encontrado elevada en el 85-100% de los pacientes con cáncer62. También, la expresión génica de hTERT plasmático y el PSA están asociados con las características tumorales del PC, como puntaje de Gleason, el estadio tumoral, y la invasión vascular y perineural63. Por ejemplo, niveles aumentados de hTERT se asocian con una menor supervivencia de BCR, por lo que se sugirió como marcador de seguimiento y en la detección de enfermedad mínima residual; no obstante, debe validarse en estudios a gran escala que incluya más eventos de recurrencia64,65.
La expresión incrementada del inhibidor de proteasa serina Kazal tipo 1 (SPINK1) en muestras de orina de pacientes con tumores negativos para rearreglos ETS, es un predictor de BCR66; sin embargo, no todos los estudios son concordantes con estos hallazgos54,67,68. Por otro lado, llama la atención que a nivel de proteína, solo se identificó su expresión en los casos de PC resistentes positivos para rearreglos ETS.
Uno de los microRNAs que regula la expresión del gen EZH2 en próstata es miR-26a43. Este microRNA es silenciado por metilación en lesiones de HGPIN y PC, lo que permite una sobrerregulación del gen EZH2 involucrado en carcinogénesis de próstata. Su asociación con el pronóstico ha sido evaluada en suero de pacientes con PC localizado e identifica pacientes con PC de alto riesgo69,70.
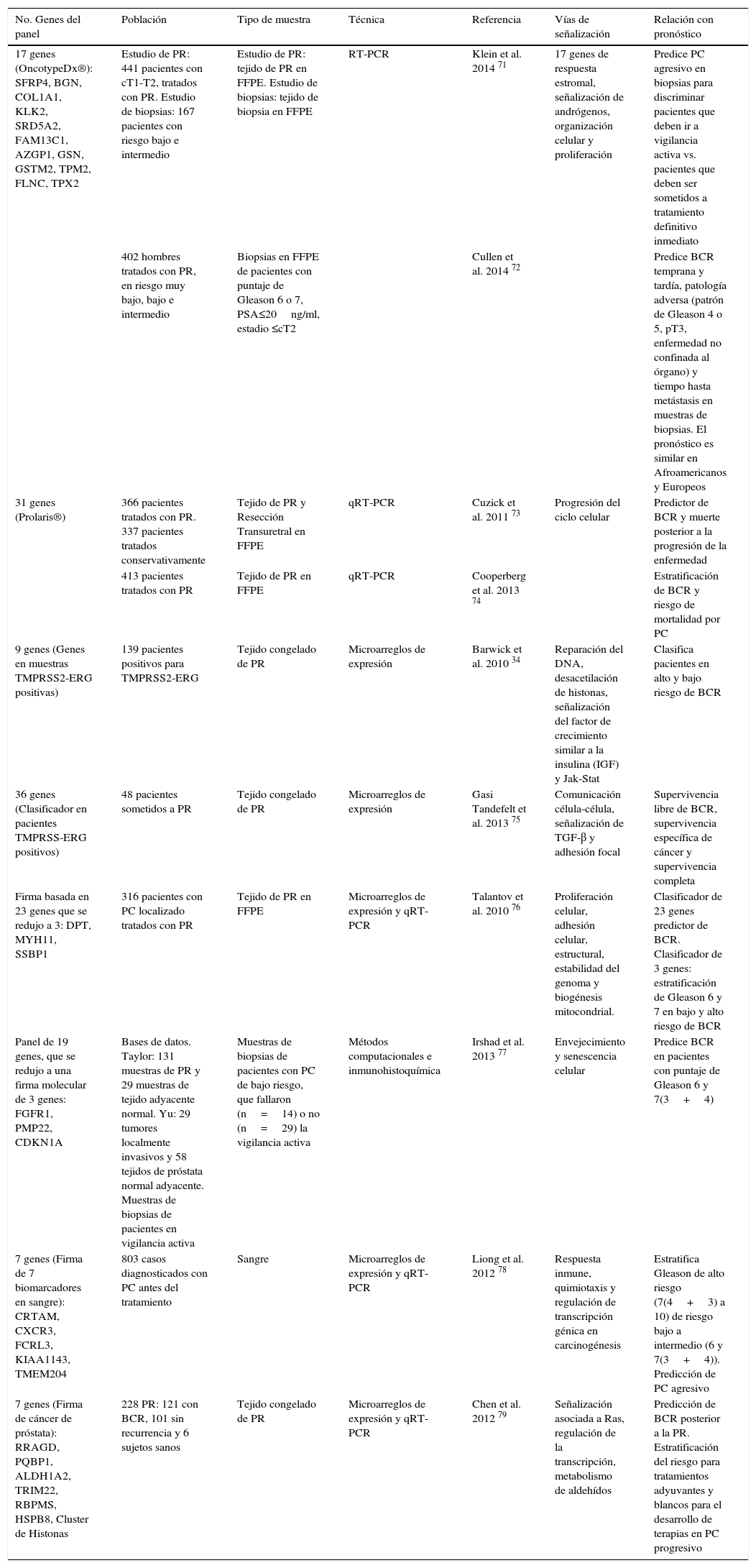
Firmas génicas o panel de biomarcadores de pronóstico de BCR en cáncer de próstata localizadoLos avances recientes en técnicas a gran escala y en bioinformática han permitido identificar paneles de biomarcadores o firmas génicas más acertadas para la estratificación del riesgo pronóstico de PC, que incluso han llevado a la comercialización de pruebas actualmente disponibles como lo son OncotypeDx® y Prolaris® (tabla 3).
Principales firmas génicas o biomarcadores en panel para el pronóstico en cáncer de próstata (PC)
| No. Genes del panel | Población | Tipo de muestra | Técnica | Referencia | Vías de señalización | Relación con pronóstico |
|---|---|---|---|---|---|---|
| 17 genes (OncotypeDx®): SFRP4, BGN, COL1A1, KLK2, SRD5A2, FAM13C1, AZGP1, GSN, GSTM2, TPM2, FLNC, TPX2 | Estudio de PR: 441 pacientes con cT1-T2, tratados con PR. Estudio de biopsias: 167 pacientes con riesgo bajo e intermedio | Estudio de PR: tejido de PR en FFPE. Estudio de biopsias: tejido de biopsia en FFPE | RT-PCR | Klein et al. 2014 71 | 17 genes de respuesta estromal, señalización de andrógenos, organización celular y proliferación | Predice PC agresivo en biopsias para discriminar pacientes que deben ir a vigilancia activa vs. pacientes que deben ser sometidos a tratamiento definitivo inmediato |
| 402 hombres tratados con PR, en riesgo muy bajo, bajo e intermedio | Biopsias en FFPE de pacientes con puntaje de Gleason 6 o 7, PSA≤20ng/ml, estadio ≤cT2 | Cullen et al. 2014 72 | Predice BCR temprana y tardía, patología adversa (patrón de Gleason 4 o 5, pT3, enfermedad no confinada al órgano) y tiempo hasta metástasis en muestras de biopsias. El pronóstico es similar en Afroamericanos y Europeos | |||
| 31 genes (Prolaris®) | 366 pacientes tratados con PR. 337 pacientes tratados conservativamente | Tejido de PR y Resección Transuretral en FFPE | qRT-PCR | Cuzick et al. 2011 73 | Progresión del ciclo celular | Predictor de BCR y muerte posterior a la progresión de la enfermedad |
| 413 pacientes tratados con PR | Tejido de PR en FFPE | qRT-PCR | Cooperberg et al. 2013 74 | Estratificación de BCR y riesgo de mortalidad por PC | ||
| 9 genes (Genes en muestras TMPRSS2-ERG positivas) | 139 pacientes positivos para TMPRSS2-ERG | Tejido congelado de PR | Microarreglos de expresión | Barwick et al. 2010 34 | Reparación del DNA, desacetilación de histonas, señalización del factor de crecimiento similar a la insulina (IGF) y Jak-Stat | Clasifica pacientes en alto y bajo riesgo de BCR |
| 36 genes (Clasificador en pacientes TMPRSS-ERG positivos) | 48 pacientes sometidos a PR | Tejido congelado de PR | Microarreglos de expresión | Gasi Tandefelt et al. 2013 75 | Comunicación célula-célula, señalización de TGF-β y adhesión focal | Supervivencia libre de BCR, supervivencia específica de cáncer y supervivencia completa |
| Firma basada en 23 genes que se redujo a 3: DPT, MYH11, SSBP1 | 316 pacientes con PC localizado tratados con PR | Tejido de PR en FFPE | Microarreglos de expresión y qRT-PCR | Talantov et al. 2010 76 | Proliferación celular, adhesión celular, estructural, estabilidad del genoma y biogénesis mitocondrial. | Clasificador de 23 genes predictor de BCR. Clasificador de 3 genes: estratificación de Gleason 6 y 7 en bajo y alto riesgo de BCR |
| Panel de 19 genes, que se redujo a una firma molecular de 3 genes: FGFR1, PMP22, CDKN1A | Bases de datos. Taylor: 131 muestras de PR y 29 muestras de tejido adyacente normal. Yu: 29 tumores localmente invasivos y 58 tejidos de próstata normal adyacente. Muestras de biopsias de pacientes en vigilancia activa | Muestras de biopsias de pacientes con PC de bajo riesgo, que fallaron (n=14) o no (n=29) la vigilancia activa | Métodos computacionales e inmunohistoquímica | Irshad et al. 2013 77 | Envejecimiento y senescencia celular | Predice BCR en pacientes con puntaje de Gleason 6 y 7(3+4) |
| 7 genes (Firma de 7 biomarcadores en sangre): CRTAM, CXCR3, FCRL3, KIAA1143, TMEM204 | 803 casos diagnosticados con PC antes del tratamiento | Sangre | Microarreglos de expresión y qRT-PCR | Liong et al. 2012 78 | Respuesta inmune, quimiotaxis y regulación de transcripción génica en carcinogénesis | Estratifica Gleason de alto riesgo (7(4+3) a 10) de riesgo bajo a intermedio (6 y 7(3+4)). Predicción de PC agresivo |
| 7 genes (Firma de cáncer de próstata): RRAGD, PQBP1, ALDH1A2, TRIM22, RBPMS, HSPB8, Cluster de Histonas | 228 PR: 121 con BCR, 101 sin recurrencia y 6 sujetos sanos | Tejido congelado de PR | Microarreglos de expresión y qRT-PCR | Chen et al. 2012 79 | Señalización asociada a Ras, regulación de la transcripción, metabolismo de aldehídos | Predicción de BCR posterior a la PR. Estratificación del riesgo para tratamientos adyuvantes y blancos para el desarrollo de terapias en PC progresivo |
Abreviaciones. Genes: SFRP4: Proteína 4 relacionada con frizzled secretada. BGN: biglicano. COL1A1: Colágeno tipo 1 alfa 1. KLK2: Peptidasa 2 relacionada con Kalikreína. SRD5A2: Esteroide-5-alfa-reductada, Polipéptido alfa 2. FAM13C1: Familia con semejanza en secuencia 13, Miembro C. AZGP1: zinc alfa-2-glicoproteína 1. GSN: gelsolina. GSTM2: Glutation S Transferasa Mu 2. TPM2: Tropomiosina 2. FLNC: Filamina C gama. TPX2: TPX2 asociada a microtúbulos. DPT: Dermatopontina. MYH11: Miosina de músculo liso, cadena pesada 11. SSBP1: Proteína 1 de unión al DNA de una cadena, mitocondrial. FGFR1: Receptor del factor de crecimiento de fibroblastos 1. PMP22: Proteína 22 de la mielina periférica. CDKN1A: Inhibidor de quinasa dependiente de ciclina 1A (también nombrado P21, Cip1). CRTAM: Molécula de células T reguladoras y citotóxicas. CXCR3: Receptor 3 de quimiocina (motivo C-X-C). FCRL3: Proteína 3 similar al receptor Fc. KIAA1143: Gen codificante de proteína no caracterizada. TMEM204: Proteína transmembranal 204. RRAGD: Proteína D de unión a GTP relacionada con Ras. PQBP1: Proteína 1 de unión a poliglutamina. ALDH1A2: Miembro A2 de la familia aldehído deshidrogenasas 1. TRIM22: Motivo tripartito 22. RBPMS: Proteína de unión a RNA con múltiples variantes. HSPB8: Proteína de choque térmico 8 de 22kDa.
PR: prostatectomía radical. FFPE: siglas en inglés de tejidos fijados en formalina y embebidos en parafina. RT-PCR: Reacción en cadena de la polimerasa acoplada a transcripción inversa. qRT-PCR: Reacción en cadena de la polimerasa cuantitativa, acoplada a transcripción inversa.
OncotypeDX® Prostate Cancer Assay (Genomic Health®, Redwood, California, Estados Unidos) es un clasificador genómico que incluye 17 genes, 12 relacionados con cáncer y 5 genes de referencia, involucrados en las vías de señalización de andrógenos, de respuesta estromal, de organización celular y de proliferación. La medición de la actividad de estos genes arroja un resultado personalizado, denominado Genomic Prostate Score (GPS, Puntaje Genómico de Próstata), relacionado directamente con el grado de agresividad de la enfermedad. El uso de esta prueba en Estados Unidos contempla la determinación del riesgo del paciente de acuerdo con lo establecido en las guías de práctica clínica, como la NCCN (National Comprehensive Cancer Center–Red Nacional Integral del Cáncer, USA), e indica que pacientes con riesgo alto no son candidatos mientras que para los demás grupos de riesgo la prueba confirma la clasificación o establece el verdadero grupo de riesgo de BCR al que pertenecen los pacientes, con un área bajo la curva (AUC, por sus siglas en inglés) de 0,68, el cual supera al del riesgo definido por las guías de NCCN de 0,59, y permite personalizar las decisiones del tratamiento en el momento del diagnóstico71,72.
Por su parte, Prolaris® incluye 31 genes relacionados con ciclo celular y tiene la capacidad de predecir tanto BCR a 10 años en pacientes tratados con PR como muerte en pacientes en vigilancia activa y diagnosticados mediante resección transuretral. Al combinar el puntaje de la prueba con el puntaje de los factores clínicos el valor AUC es de 0,842 para BCR a 10 años en pacientes con PR, y de 0,878 para muerte en pacientes en vigilancia activa, frente a 0,825 y 0,806 en los factores clínicos, respectivamente73. Este marcador demostró además ser un factor pronóstico del puntaje de Gleason y de los niveles de PSA73, y fue validado en dos cohortes de pacientes sometidos a PR y resección transuretral, respectivamente, donde permitió subestratificar a los pacientes de riesgo bajo y alto clasificados mediante el puntaje CAPRA-S (posquirúrgico), que es otro método de determinación del riesgo pronóstico en PC74. Actualmente está disponible comercialmente para uso sobre biopsias.
Otros trabajos en investigación han conducido a la identificación de posibles clasificadores del pronóstico. A partir del análisis comparativo en diferentes cohortes de pacientes con y sin la fusión TMPRSS2-ERG se identificó un panel de expresión de 9 genes que permite clasificar los pacientes TMPRSS2-ERG positivos en alto y bajo riesgo de BCR. La segregación de los pacientes fue mejorada por la combinación con el puntaje de Gleason y el estado de fusión de TMPRSS2-ERG34. En el 2013, se reportó un clasificador de 36 genes en muestras TMPRSS2-ERG positivas y con altos niveles de expresión de ERG que segregó a los pacientes en grupos de alto y bajo riesgo de BCR. Los resultados fueron validados en una cohorte externa con un seguimiento a 5 años, y se sugiere corroborar el clasificador mediante qPCR o coloración de inmunohistoquímica en biopsias, para aquellos pacientes positivos para la fusión y con altos niveles de expresión de ERG75.
Un clasificador basado en genes conformado por los genes DPT, MYH11 y SSBP1, logró también estratificar el riesgo de BCR que fue validado en un grupo independiente de pacientes, y clasificó pacientes con Gleason 6 y 7. El DPT y MYH11 fueron nuevos marcadores de pronóstico de PC mientras que SSBP1 ya había sido previamente asociado con PC agresivo76.
Recientemente, se estableció un predictor molecular de tres genes (FGFR1, PMP22 y CDKN1A) a partir del análisis de expresión de genes relacionados con envejecimiento y senescencia, que segregó en grupos de alto y bajo riesgo la BCR de pacientes con tumores con Gleason bajo (puntaje 6 y 7 3+4), y superó al nomograma de clasificación de D’Amico. Sin embargo, el mejor nomograma a nivel de expresión de RNA fue el predictor molecular de tres genes junto con el puntaje de Gleason, y a nivel de expresión proteica solo el predictor. Estos resultados fueron validados en biopsias y en grupos de datos de cohortes independientes provenientes de pacientes monitoreados mediante vigilancia activa y que habían sido sometidos a resección transuretral77.
En muestras de sangre también se ha realizado la búsqueda de un panel de biomarcadores de pronóstico que discrimine el riesgo de BCR, de tal forma que pueda ser útil desde el momento del diagnóstico sin realizar procedimientos invasivos en el paciente. En estas muestras un clasificador de siete biomarcadores logró discriminar a los pacientes en dos grupos: un grupo de pacientes con Gleason de alto riesgo de 7 (4 + 3) a 10 y otro grupo con riesgo bajo a intermedio con Gleason 6 (3 + 3) y 7 (3 + 4)78. Otro grupo reportó un pronosticador constituido por siete genes diferentes, con una mayor sensibilidad y precisión en la predicción de BCR posterior a la PR en comparación a otros clasificadores; estos indicadores mejoraron al combinarse con el puntaje de Gleason y fueron validados en dos sets de datos independientes79.
Finalmente, en esta revisión se concluye que el uso de variables clínicas y patológicas en la predicción del pronóstico en pacientes con PC sigue siendo la herramienta más empleada actualmente en la clínica para la clasificación del riesgo de BCR. No obstante, existe la necesidad de nuevos biomarcadores de pronóstico que permitan una estratificación más precisa, no solo del riesgo de BCR sino también del riesgo de recaída clínica y de enfermedad hormonorrefractaria. Algunos de los biomarcadores estudiados ya tienen pruebas disponibles comercialmente, otros están validándose y otros requieren de una validación en sets de datos o pacientes con tamaños de muestra que sean grandes e independientes antes de usarse en la clínica. Las nuevas aproximaciones metodológicas permitirán dirigir los esfuerzos de una manera más acertada para identificar el verdadero riesgo pronóstico y así guiar el manejo de forma personalizada para contribuir al control de la enfermedad con una mayor supervivencia y mejor calidad de vida, y eventualmente, a la optimización en los costos en el tratamiento.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.