El virus de la enfermedad de Aujeszky infecta a los cerdos y genera estados de latencia, en ausencia de signos clínicos, dificultando su diagnóstico. Debido a las grandes pérdidas económicas ocasionadas por esta enfermedad, Argentina ha implementado un plan para su control y erradicación. Como parte del monitoreo de la evolución del plan de saneamiento en una granja con estatus positivo, usando cuerdas se obtuvieron muestras de fluido oral de animales de 40, 70, 90, 120 y 130-160días de vida transcurrido un año de aplicación de una vacuna deleteada e inactivada. Las muestras se analizaron mediante PCR. Se obtuvieron resultados positivos en porcinos de 130-160días de vida. La detección de ADN del virus de la enfermedad de Aujeszky a partir de muestras de fluidos orales obtenidas a campo podría servir para la vigilancia epidemiológica en programas de control y erradicación.
Aujeszky's disease virus infects pigs and causes latency in the absence of clinical signs, making its diagnosis very difficult. Due to the huge economic losses related to this disease, the Argentinian government has created and implemented a control and eradication plan to manage it. As part of the monitoring plan to assess the progress of the control and eradication plan for a positive status farm, oral fluid samples were obtained from animals at 40, 70, 90,120 and 130-160days of age, after one year of vaccination with a deleted and inactivated vaccine. These samples were processed using an antigen-PCR technique and positive results were obtained from 130-160day-old animals. Recovering DNA from Aujeszky's disease virus in oral fluids from field samples could enable the use of this technique for epidemiological surveillance in Aujeszkýs control and eradication programs.
La enfermedad de Aujeszky (EA) es producida por un virus ADN perteneciente al género Varicellovirus, familia Orthoherpesviridae. Este virus, también conocido como virus de la pseudorrabia porcina o herpesvirus porcino tipo1 (HVS-1), puede infectar diferentes especies de mamíferos, produciendo una encefalitis mortal. Por su parte, los cerdos domésticos y salvajes son los hospedadores naturales y reservorios del virus, el cual logra pasar a estados de latencia al establecerse en los ganglios de Gässer, bulbo olfatorio y amígdalas de porcinos que sobreviven a la primoinfección. En los estados de latencia el genoma viral puede persistir indefinidamente, incluso después de la desaparición de los signos clínicos, lo que dificulta el diagnóstico. A su vez, en el proceso de reactivación de la replicación viral en los cerdos aumenta la probabilidad de contagio a través del contacto directo entre los hospedadores portadores y los sensibles1,8,13,14.
El virus de la EA (VEA) puede detectarse empleando diferentes técnicas diagnósticas, como, por ejemplo, la detección del ADN viral mediante la reacción en cadena de la polimerasa (PCR). Esta técnica presenta como ventajas su rapidez, sensibilidad y capacidad de diagnosticar la infección en etapas tempranas de la enfermedad utilizando muestras de tejidos, secreciones, exudados, líquido cefalorraquídeo o sangre de animales infectados, incluso antes de la formación de los anticuerpos (Ac) específicos o luego de haberse establecido el estado de latencia5,7,13.
Si bien la PCR puede realizarse a partir de diferentes tipos de muestras, en las últimas décadas ha cobrado relevancia el desarrollo de protocolos para el diagnóstico de enfermedades infecciosas y no infecciosas a partir de muestras de fluidos orales (FO) tanto en personas como en animales10. Para la especie porcina, existen reportes de detección de Ac y antígenos (Ag) de diferentes virus y bacterias empleando FO3,6,9,11. La obtención de muestras de FO a través de la introducción de una cuerda es un método de muestreo sencillo, eficiente y de bajo costo que se ve facilitado por el interés natural de morder que presentan los cerdos cuando se ingresan objetos nuevos a los corrales5,6,10,12.
En 2010, Kittawornrat et al.6 realizaron un estudio del cual concluyeron que el método de la cuerda es una forma de muestreo de fácil implementación, que permite obtener muestras diarias de forma no invasiva (la mayoría de los animales se adaptaron rápidamente) para detectar el virus del síndrome respiratorio reproductivo porcino (VPRRS) y realizar así el diagnóstico temprano de la enfermedad, favoreciendo la vigilancia epidemiológica. La técnica de la cuerda abre la posibilidad de reemplazar el monitoreo serológico establecido por el programa de control y erradicación del VPRRS en Estados Unidos.
En relación con el HVS-1, Cheng et al.2-4 reportaron la detección de Ac y ADN viral en muestras de FO a través de la técnica de ELISA y PCR, respectivamente, bajo condiciones experimentales. Por ello, el objetivo del presente trabajo fue evaluar la capacidad para detectar el HVS-1 mediante una técnica de PCR dirigida a una secuencia de ADN que codifica glicoproteínas virales, aplicada en muestras de FO obtenidas con cuerdas de algodón en corrales de cerdos ubicados en establecimientos infectados naturalmente.
El estudio se desarrolló en un establecimiento de producción porcina comercial de la provincia de Santa Fe, Argentina, que presenta un sistema de producción confinado de ciclo completo, en un solo sitio, con aproximadamente 360 hembras en producción. El establecimiento cuenta con escasas medidas de bioseguridad.
En 2011, los cerdos de la granja presentaron signología clínica compatible con la infección por el HVS-1 y se confirmó el diagnóstico a través de un estudio serológico. En diciembre de 2016 se comenzó a utilizar una vacuna deleteada en gE e inactivada establecida por Senasa (AUSKIPRA®BK, Laboratorio HIPRA), que se aplicó según el protocolo presentado y aprobado previamente por la coordinación de enfermedades de los porcinos de Senasa.
Como parte del monitoreo de la evolución del plan de control en la granja, se obtuvieron muestras de FO de corrales de porcinos de 40, 70, 90, 120 y 130-160días de vida (ddv) a través de la técnica de la cuerda según lo descrito por Prickett et al.10, para su posterior procesamiento por la técnica de PCR. Para ello, se suspendieron cuerdas trenzadas de algodón (al 100%) en una de las rejas que dividían dos corrales contiguos. De esta forma, se muestrearon en total 75 lechones de 40ddv, 50 porcinos de 70, 90 y 120ddv y 75 cerdos de 130 a 160ddv. Las cuerdas se ataron a la altura del hombro de los cerdos, alejadas de comederos y bebederos. Se dejaron al acceso de los animales durante 20 a 30minutos. Si se observaba que algún cerdo permanecía por mucho tiempo mordiendo la cuerda, se realizaban movimientos de animales para facilitar el acceso a la cuerda de otros cerdos y así obtener un muestreo representativo del corral. Transcurrido dicho lapso, cada una de las cuerdas se introdujo en una bolsa de polietileno y se escurrió hasta obtener el FO, que luego se colocó en tubos de centrífuga plásticos con tapa a rosca de 10ml. Las muestras se remitieron refrigeradas y en posición vertical al Servicio de Biología Molecular de la Facultad de Ciencias Veterinarias, Universidad Nacional de Rosario. De cada muestra se obtuvo 1ml de sobrenadante, que fue colocado en un tubo tipo eppendorf y almacenado a −80°C hasta su procesamiento. Para la extracción del ADN se utilizó el kit comercial QIAamp DNA Mini kit (QIAgen GMBH, Ref. 51304) y para la PCR el kit Top Taq Master Mix kit (Qiagen GMBH, Alemania).
Los cebadores que se utilizaron fueron diseñados para detectar secuencias específicas de genes que codifican la gD del HVS-1. Estos fueron seleccionados con un programa informático (Primer Express 1.0 Software, Perkin-Elmer) a partir de una secuencia genómica publicada en la base de datos GenBank (AY217094) y fueron sintetizados por Genbiotech SRL (Buenos Aires, Argentina). Las secuencias utilizadas fueron las siguientes: gD sentido (5’- GTG CAC GGA GGA CGA GCT GGG GCT -3’) y gD antisentido (5’- GAC GTC CAC GCC CCG CTT GAA GCT -3’).
Para la PCR propiamente dicha, se utilizó un termociclador (Multigene Mini Personal Thermal Cycler) con el siguiente protocolo: un ciclo inicial a 94°C durante 3min, 35 ciclos de 30segundos a 94°C, 30segundos a 60°C, seguido de 72°C por 1min, y un último ciclo a 72°C por 10min. Se realizó la corrida electroforética de las muestras sometidas a amplificación sobre un gel de agarosa al 1% teñido con SYBR Safe. Los parámetros utilizados fueron los siguientes: 150V; 320A; 30min. El volumen sembrado fue de 5μl/muestra. Se utilizó un transiluminador (DyNA LIGHT UV) para visualizar las bandas. Como control positivo (C+) de extracción de ADN, se utilizó tejido de encéfalo de lechones infectados de forma natural por HVS-1 que resultaron positivos a las técnicas de PCR, inmunohistoquímica y aislamiento en cultivo celular. El control negativo (C−) de extracción se realizó con agua estéril. Como control positivo de PCR, se utilizó un extracto de vacuna deleteada [gE(−)] e inactivada (AUSKIPRA®BK, Laboratorio HIPRA) y como control negativo se sustituyó el ADN por agua estéril.
De los mismos corrales donde se obtuvieron las muestras de FO se tomaron muestras de sangre de 10 animales de 20, 40, 70 y 90ddv y de 60 animales de 120-160ddv por punción de la vena cava craneal. Las muestras se colocaron en tubos tipo Kahn descartables y se remitieron al Servicio de Asesoramiento Sanitario en Porcinos de la FCV-UNR. Se obtuvo el suero mediante centrifugación a 1.500rpm durante 5min, y este se almacenó en tubos tipo eppendorf a una temperatura de entre 3 y 8°C. Las muestras de suero se estudiaron mediante la técnica de ELISA de bloqueo para la detección de Ac anti-gE del HVS-1. Se utilizó un kit comercial (Idexx Pseudorabies Virus gpl Antibody test kit, Idexx Laboratories, Inc. Hoofddorp, Países Bajos), de acuerdo con las especificaciones del laboratorio elaborador del kit.
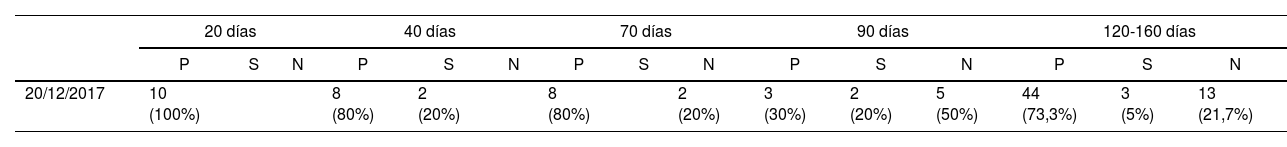
La tabla 1 y la figura 1 muestran los resultados de la PCR para la detección del HVS-1 a partir de muestras de FO. La tabla 2 detalla los resultados obtenidos a partir de las muestras de suero procesadas por la técnica de ELISA de bloqueo a 1año de introducida la vacuna. Se detectaron Ac anti-gE del HVS-1 a lo largo de toda la línea de producción.
Resultados de serología
| 20 días | 40 días | 70 días | 90 días | 120-160 días | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| P | S | N | P | S | N | P | S | N | P | S | N | P | S | N | |
| 20/12/2017 | 10 (100%) | 8 (80%) | 2 (20%) | 8 (80%) | 2 (20%) | 3 (30%) | 2 (20%) | 5 (50%) | 44 (73,3%) | 3 (5%) | 13 (21,7%) | ||||
P: positivo; S: sospechoso; N: negativo.
Las muestras fueron procesadas con el kit Idexx Pseudorabies Virus gpl Antibody test kit.
En los últimos años, diferentes grupos de investigadores han evaluado el uso de FO como método de monitoreo de enfermedades en la producción porcina5,6,10,11. El uso de una cuerda para obtener muestras de FO en la especie porcina y su posterior procesamiento por técnicas de PCR para detectar agentes como el virus de la influenzaA y el VPRRS, entre otros, es muy común en el monitoreo sanitario de granjas. En el establecimiento evaluado se realizó un monitoreo de la circulación del VEA mediante el procesamiento de FO y se obtuvieron resultados positivos a los 130-160ddv.
Si bien no hemos encontrado información a nivel mundial sobre la detección del VEA mediante PCR de muestras FO, los resultados obtenidos a campo se consideran un aporte que resultaría de gran utilidad en situaciones en las que se necesita conocer la dinámica de transmisión y de infección de animales expuestos en una granja.
Aun cuando los resultados por serología dan cuenta de que el establecimiento evaluado presentaba circulación viral, el ADN viral solo se detectó en la etapa de terminación de los animales. Esto sugiere que, en las condiciones del establecimiento, podría haber una mayor excreción viral en animales de 130-160ddv, entre los cuales probablemente exista la mayor presión de infección del agente. Esta información resulta útil para la granja a efectos de evitar el contacto entre animales eliminadores y animales expuestos.
De esta forma, concluimos que la técnica de PCR en FO resultó ser eficaz para la detección de material genético del HVS-1 en muestreos obtenidos a campo. Esta información resulta relevante debido a que no hay reportes anteriores del uso de dicha técnica en el diagnóstico de EA. Si bien se deberán realizar estudios adicionales para determinar la sensibilidad de la PCR en la detección viral del VEA a partir de muestras de FO y su utilidad en la vigilancia epidemiológica en el marco de programas de control y erradicación de la EA, esta metodología podría ser útil en forma complementaria a la serología para comprender mejor la dinámica de transmisión e identificar los puntos críticos de control en las granjas.
FinanciaciónLa presente investigación no ha recibido ninguna beca específica de agencias de los sectores público, comercial o sin ánimo de lucro.
Conflictos de interesesNinguno.
AgradecimientosAgradecemos a las personas propietarias y encargadas de la granja por permitirnos el acceso para recolectar las muestras utilizadas en este estudio.