La actividad cervecera en la Patagonia andina argentina tiene un rol muy importante en la economía de la región; una de las problemáticas que enfrenta, en términos de calidad, son las contaminaciones microbianas. La presencia de bacterias y levaduras contaminantes en la cerveza produce cambios microbiológicos, físicos y químicos, que impactan en sus atributos sensoriales. No obstante, pocas cervecerías establecen criterios y políticas que garanticen la calidad microbiológica de sus productos. El propósito de este trabajo fue estudiar por primera vez la incidencia de contaminantes microbianos en cervezas artesanales embotelladas producidas en la Patagonia andina argentina, además de identificar los principales microorganismos involucrados y determinar posibles relaciones entre los eventos de contaminación y variables fisicoquímicas de la cerveza. Para ello se analizaron 75 cervezas provenientes de 37 cervecerías de 12 localidades andinas. El 69,3% de las muestras analizadas evidenció crecimiento de microorganismos en los medios de cultivo empleados para la detección de contaminantes cerveceros. La bacteria Levilactobacillus brevis y levaduras del género Saccharomyces fueron los principales contaminantes identificados. Se comprobó que las contaminaciones microbianas impactaron sobre el perfil sensorial de la cerveza y que el cambio de pH fue un indicador de contaminación por bacterias lácticas. De cada 10 fábricas estudiadas, 8 presentaron problemas de contaminación, lo que pone en evidencia la necesidad de diseñar estrategias de prevención y control de contaminaciones en microcervecerías.
The brewing activity in Andean Patagonia plays a very important role in the region's economy, being microbial contamination one of the main problems in terms of quality. The presence of contaminant bacteria and wild yeasts in beer generate microbiological, physical and chemical changes that impact on its sensory attributes. However, few breweries establish criteria and policies to guarantee the quality of their products in a microbiological sense. The purpose of this work was to study for the first time the incidence of microbial contaminants in bottled craft beers from Andean Patagonia, identify the main microorganisms involved and establish relationships between contamination and the physicochemical variables of beer. We analyzed 75 beers from 37 breweries from 12 different Patagonian cities. Our results showed that 69.3% of the analyzed beer exhibited contaminant microorganism growth. Bacteria Levilactobacillus brevis and wild yeasts of Saccharomyces were the main microorganisms responsible for these contaminations. In addition, we found that microbial contamination had an impact on beer sensory profile and also that pH was correlated with the presence of lactic acid bacteria in beer, being an indicator of contamination for these bacteria. In conclusion, we observed that 8 out of 10 breweries studied showed contamination problems, highlighting the need to design prevention and control strategies in microbreweries.
La contaminación microbiana ha demostrado ser uno de los factores más importantes que afectan la calidad de la cerveza, en general, y de las cervezas artesanales, en particular7,14,26. La producción de cerveza artesanal en Argentina es parte de un sector emergente y ha experimentado un continuo crecimiento durante los últimos 20 años53,54. Este crecimiento significa un gran impacto en las bioeconomías regionales, ya que, además, genera múltiples puestos de trabajo, ayuda a diversificar las matrices productivas e impacta positivamente en otros sectores económicos, como el de servicios, metalúrgica, transporte, gastronomía, entre otros12,20.
La región andina de la Patagonia se destaca en materia de antecedentes cerveceros y Bariloche es una de las localidades con mayor impacto y reconocimiento en cuanto a producción de cerveza artesanal en el país. San Carlos de Bariloche presenta la mayor densidad de microcervecerías por habitante de Argentina (40 en el año 2018) y la mayor cantidad de litros de cerveza artesanal elaborada por habitante de todo el país (17 litros por persona)49.
El sector productivo de Bariloche y su zona de influencia está integrado por actores que presentan fuertes asimetrías en relación con el tamaño de producción, tecnologías incorporadas y mercados en los que operan25. El rápido crecimiento en el volumen y la cantidad de cervecerías artesanales, así como la mayor demanda de cervezas de calidad por parte del consumidor, obligan a buscar constantemente estrategias de control de calidad eficientes y asequibles, que les garantice a los productores competitividad y permanencia en el mercado.
Aunque esta bebida presenta características que restringen el crecimiento microbiano (contiene etanol, iso-α-ácidos, alto contenido de dióxido de carbono, bajo pH, bajo contenido de oxígeno y pocos nutrientes), existen bacterias y levaduras contaminantes capaces de tolerar dichas condiciones y producir cambios sensoriales, microbiológicos, químicos y físicos que alteran la calidad de la cerveza18,26,38,46. La presencia de bacterias en la cerveza puede causar turbidez, sedimentos, exopolisacáridos, compuestos sulfurados, diacetilo y diversos ácidos orgánicos, entre otros compuestos. El 90% de las contaminaciones reportadas son causadas por bacterias ácido lácticas (BAL) de los géneros Levilactobacillus, Lactobacillus y Pediococcus30,40,41,48; Levilactobacillus brevis —antes conocida como Lactobacillus brevis— es la especie de mayor incidencia40,41,47. La contaminación con bacterias gram negativas, como Acetobacter y Gluconobacter (acetobacterias), también ha sido reportada, aunque en los últimos años, la incidencia de estas bacterias fue desplazada por bacterias anaerobias estrictas, como Megasphaera y Pectinatus15,19. A su vez, se ha observado que algunos géneros de la familia Enterobacteriaceae, como Obesumbacterium, Hafnia, Klebsiella y Citrobacter, son capaces de alterar el mosto y la cerveza en estadios tempranos de la fermentación33,35.
En la elaboración de cerveza, la cepa de levadura utilizada para la fermentación es uno de los factores claves que influyen en el perfil de aromas y sabores. Las levaduras empleadas tradicionalmente en la elaboración de cerveza son cepas domesticadas de Saccharomyces cerevisiae y Saccharomyces pastorianus. Las primeras se emplean para fabricar cervezas de tipo Ale, ya que fermentan entre los 18 y 24°C, y se suelen caracterizar por una mayor complejidad de aromas y sabores (flavor) frutales, florales o especiados. Por su parte, la especie híbrida Saccharomyces pastorianus (S. cerevisiae x S. eubayanus) se utiliza para producir cervezas de tipo Lager, que fermentan a bajas temperaturas (entre 5 y 15°C) y resultan sensorialmente más neutras45. Dado el rol fundamental que tienen las levaduras en el perfil sensorial de la cerveza, es importante asegurar que la fermentación sea conducida solo por la levadura cervecera elegida43.
La presencia de levaduras que no han sido inoculadas por el productor y que, por tanto, no están bajo su control, puede derivar en alteraciones de la cerveza; a estas se las considera «levaduras contaminantes». Las levaduras contaminantes se pueden dividir en dos grandes grupos: las del género Saccharomyces y las que pertenecen a otros géneros, como Brettanomyces, Pichia, Candida, Debaryomyces, Filobasidium, Zygosaccharomyces y Wickerhamomyces, entre otros. Las contaminaciones causadas por levaduras pueden producir en la cerveza turbidez, fenoles, ésteres, alcoholes superiores, superatenuación y un exceso de carbonatación, entre otros efectos36. En general, las contaminaciones causadas por levaduras pueden evidenciarse sensorialmente porque muchas de ellas (Saccharomyces y no Saccharomyces) producen compuestos fenólicos (POF+), como el 4-vinilguaiacol, 4-vinilfenol, 4-etilguaiacol y 4-etilfenol, a partir de la transformación de ácidos orgánicos como el cinámico, el ferúlico y el cumárico, presentes en la malta. Por el contrario, las levaduras cerveceras, salvo algunas excepciones, no son capaces de producir este tipo de compuestos (son POF−). Las levaduras contaminantes que pertenecen al género Saccharomyces representan el mayor riesgo debido a su similitud fisiológica y morfológica con la levadura inoculada, y su detección y control representan un gran desafío para las cervecerías48. A diferencia de las levaduras cerveceras, muchas S. cerevisiae contaminantes (por ejemplo, las cepas diastáticas de S. cerevisiae previamente conocidas como variedad diastaticus) son capaces de metabolizar carbohidratos residuales de la cerveza, como dextrinas complejas y almidones, gracias a la enzima extracelular glucoamilasa, codificada por el gen STA11,22,27. Sin embargo, la presencia del gen STA1 no siempre es indicativo de poder diastásico, ya que se ha observado variabilidad en el metabolismo de estos azúcares e incluso incapacidad de consumir dextrinas, aun en presencia del citado gen1,22.
Actualmente, el límite de tolerancia para la presencia de microorganismos contaminantes en cerveza es un tema en discusión. No existen especificaciones oficiales en el Código Alimentario Argentino respecto de criterios microbiológicos en cerveza, dado que se considera que estos influyen exclusivamente en la calidad del producto y no en su inocuidad, siempre que se cumplan los parámetros fisicoquímicos de alcohol y pH11. Sin embargo, en la práctica, se considera que todo microorganismo contaminante presente en la cerveza representa un potencial riesgo para el producto18. Algunos autores proponen como límites de tolerancia para contaminantes microbianos en cerveza 0 a 50UFC (unidades formadoras de colonias) por cada 100-200ml de cerveza7,18. Otros autores sugieren un límite de hasta 10UFC/ml para bacterias ácido lácticas y acéticas y menor de 1UFC/ml para levaduras contaminantes en la cerveza terminada16,34,50.
En Argentina, pocas cervecerías establecen criterios microbiológicos y políticas que garanticen la calidad y estabilidad de sus productos. Además, la investigación en materia de ciencia y tecnología cervecera es muy reciente y la calidad microbiológica de la cerveza artesanal argentina está muy poco estudiada. En dicho contexto, y teniendo en cuenta que la Patagonia andina es uno de los principales polos cerveceros del país, este trabajo tuvo como objetivo estudiar por primera vez la incidencia de contaminantes microbianos en las cervezas artesanales embotelladas producidas en la Patagonia andina. Para ello se planteó aislar e identificar los principales microorganismos involucrados y determinar posibles relaciones entre los eventos de contaminación y variables fisicoquímicas de la cerveza.
Materiales y métodosCervezas estudiadasSe estudiaron 75 muestras de cervezas artesanales de múltiples estilos, producidas y embotelladas por 37 microcervecerías comerciales de 12 localidades andinas de la Patagonia. De norte a sur, las localidades y el número de muestras (entre paréntesis) fueron Caviahue (1), San Martín de los Andes (8), Villa La Angostura (4), Dina Huapi (3), San Carlos de Bariloche (27), El Bolsón (9), El Hoyo (3), Lago Puelo (3), Epuyén (6), Esquel (7), Trevelín (1) y Ushuaia (3), abarcando una distancia de 2.800km entre las localidades más lejanas. Las cervezas estudiadas (no pasteurizadas) fueron elaboradas y analizadas durante la temporada primavera-verano de 2014-2015. Las muestras fueron obtenidas directamente de los productores o en locales de venta al público y conservadas a 4°C hasta su análisis, por no más de un mes y antes de su fecha de caducidad.
Detección y cuantificación de microorganismos contaminantesSe analizó la presencia de bacterias aeróbicas totales, bacterias ácido lácticas (BAL) y levaduras contaminantes en todas las muestras. La detección de bacterias totales se realizó en medio diferencial de Wallerstein (WLD) Sigma®. Se sembraron 100μl de cerveza en superficie utilizando una espátula de Drigalski, y luego de 48-72h a 30°C, se realizó el recuento de UFC por ml de cerveza. Para la detección de BAL, se utilizó el medio de cultivo de Hsu semisólido para Levilactobacillus, Lactobacillus y Pediococcus (Siebel Institute's HLP)2. Para esta determinación, se colocaron 14ml del medio de cultivo y 1ml de la muestra en tubos Falcon de 15ml, la incubación fue a 30°C durante 48-72h. Transcurrido ese lapso, se determinó la presencia o ausencia de colonias dentro del tubo. Cabe mencionar que la detección de BAL utilizando HLP no permite el recuento de UFC, por lo que el recuento de bacterias se realizó únicamente sobre el medio de cultivo WLD.
Para la detección de levaduras contaminantes se inocularon 100μl de la muestra mediante siembra en superficie en el medio de sulfato de cobre de Lin (LCSM)24. Las placas se incubaron durante 48-72h a 25°C y luego se realizó el recuento de UFC/ml. Para descartar el posible crecimiento en LCSM de las principales levaduras cerveceras utilizadas en la región, se incluyeron como controles las cepas Safale s 04 (S-04) y Safale us 05 (US-05) de la marca Fermentis-Lesaffre y la cepa Nottingham de la marca Lallemand Brewing.
Se consideró que la muestra de cerveza estaba contaminada cuando el recuento de UFC fue mayor o igual a 10UFC/ml en los medios sólidos o cuando se registró crecimiento de colonias en el medio semisólido HLP. Este límite se eligió según lo sugerido por Hill16 para la detección de bacterias y se extrapoló para la detección de levaduras.
Análisis sensorialSe realizó el análisis sensorial de las muestras de cerveza con el fin de detectar descriptores asociados a contaminación (presencia de fenoles, percibida como aroma a clavo de olor o establo, y de ácidos orgánicos, percibida como acidez), así como otros descriptores indeseados que reducen la tomabilidad de la cerveza, entre ellos diacetilo (aroma y sabor a manteca), compuestos sulfurados, como el dimetil sulfuro (DMS) (aroma y sabor a choclo, otras verduras hervidas y huevo) y acetaldehído (aroma y sabor a manzana verde), entre otros. Para esto se contó con personal entrenado en la cata de cerveza y se siguieron los lineamientos propuestos por la American Society of Brewing Chemists3.
Aislamiento y conservación de microorganismos contaminantesLas bacterias y levaduras obtenidas a partir de los cultivos selectivos fueron conservadas a −80°C utilizando glicerol (20% v/v) como crioprotector. Estos microorganismos contaminantes fueron ingresados al cepario del Centro de Referencia en Levaduras y Tecnología Cervecera (CRELTEC), perteneciente al Instituto Andino Patagónico de Tecnologías Biológicas y Geoambientales (IPATEC), CONICET–UNComahue.
Identificación molecularLa identificación molecular de los microorganismos aislados se realizó mediante la secuenciación de genes ribosomales (ADNr). La extracción del ADN de bacterias y levaduras no sacaromicéticas se realizó a partir de un cultivo puro de 24-48h, del cual se tomó una ansada de biomasa, que se resuspendió en 500μl de EDTA 5mM. Luego se centrifugó por 5min a 8.000rpm y se descartó el sobrenadante. El pellet obtenido se congeló a −80°C por 30min y luego se colocó en baño termostatizado a 65°C. Se agregaron perlas de vidrio y 500μl de buffer de lisis (100mM NaCl, 10mM Tris-HCl pH 8.0, 1mM EDTA, 2% Tritón X-100, 1% SDS) y 250μl de una mezcla de cloroformo:alcohol isoamílico (24:1 v/v). Se incubó en agitación durante 20min y se separaron las fases mediante centrifugación (10min a 13.000rpm). Se recuperó la fase acuosa, se agregaron 2 vol de etanol al 70% (frío) y se incubó 30min a −80°C para precipitar el ADN. Por último, se centrifugó durante 5min a 13.000rpm y se descartó el sobrenadante. El pellet de ADN se lavó con 500μl de etanol absoluto frío y luego se resuspendió con 50μl de agua libre de ADNasa/ARNasa.
Las bacterias fueron identificadas mediante la amplificación de la subunidad ribosomal 16S por PCR con los cebadores 27F (5’-GAGAGTTTGATCCTGGCTCAG-3’) y 1495r (5’-CTACGGCTACCTTGTTACGA-3’)21. La amplificación de 3 aislamientos bacterianos presentó resultados no concluyentes, por lo cual estos fueron identificados en el Centro de Referencia para Lactobacilos (CERELA, CONICET), según Nediani et al.31.
La identidad de las levaduras no sacaromicéticas se estudió mediante la amplificación de la región ITS del ARNr utilizando el cebador ITS1 (5́-TCCGTAGGTGAACCTGCGG-3’) e ITS4 (5́-TCCTCCGCTTATTGATATGC -3’), según White et al.52.
Los productos de amplificación de levaduras y bacterias fueron secuenciados por Macrogen Inc. (Corea). Las secuencias obtenidas se editaron manualmente y se compararon contra las secuencias tipo equivalentes en bases de datos públicas accesibles (GenBank) usando la herramienta computacional BLAST, disponible en http://www.ncbi.nlm.nih.gov/BLAST/Blast.cgi.
Las levaduras sacaromicéticas fueron identificadas genéticamente en un estudio previo, en el que se utilizó la técnica de real time PCR para la identificación a nivel de género y especie, seguida de la tipificación de la cepa por electroforesis capilar de amplicones del fragmento espaciador intergénico 2 (IGS2) del ARN ribosomal, según Latorre et al.23.
Caracterización fisicoquímicaLa caracterización fisicoquímica se realizó utilizando los métodos propuestos por la American Society of Brewing Chemists (ASBC). Para cada muestra, se determinó el pH4, las unidades internacionales de amargor (IBUs, por sus siglas en inglés)5 y el color6.
Se midió la concentración de los azúcares residuales en cada cerveza como medida indirecta del grado de atenuación (consumo de azúcares del mosto); este parámetro puede dar indicios de la eficiencia de la fermentación. La presencia de azúcares residuales (superior a 7 grados Brix) sugiere una fermentación deficiente, mientras que una superatenuación (por debajo de 4 grados Brix) puede sugerir la presencia de contaminantes, ya que las levaduras cerveceras no son capaces de fermentar, por ejemplo, las dextrinas (en general, 4% del extracto fermentable del mosto de cerveza). La concentración de azúcares residuales disueltos en la cerveza se midió utilizando un refractómetro de mano (Alla France-ATC), cuya unidad de medida son los grados Brix (°Bx, g azúcar/100g cerveza). En los casos en que se contaba con el dato de la densidad inicial, aportado por el productor, se realizó la corrección por presencia de alcohol del valor de lectura del refractómetro.
Se evaluó si existían correlaciones entre la presencia/ausencia de contaminación y las variables fisicoquímicas analizadas utilizando un modelo de regresión logística binomial y el paquete estadístico R-project37. Se estableció el nivel de significación en valores de p inferiores a 0,01.
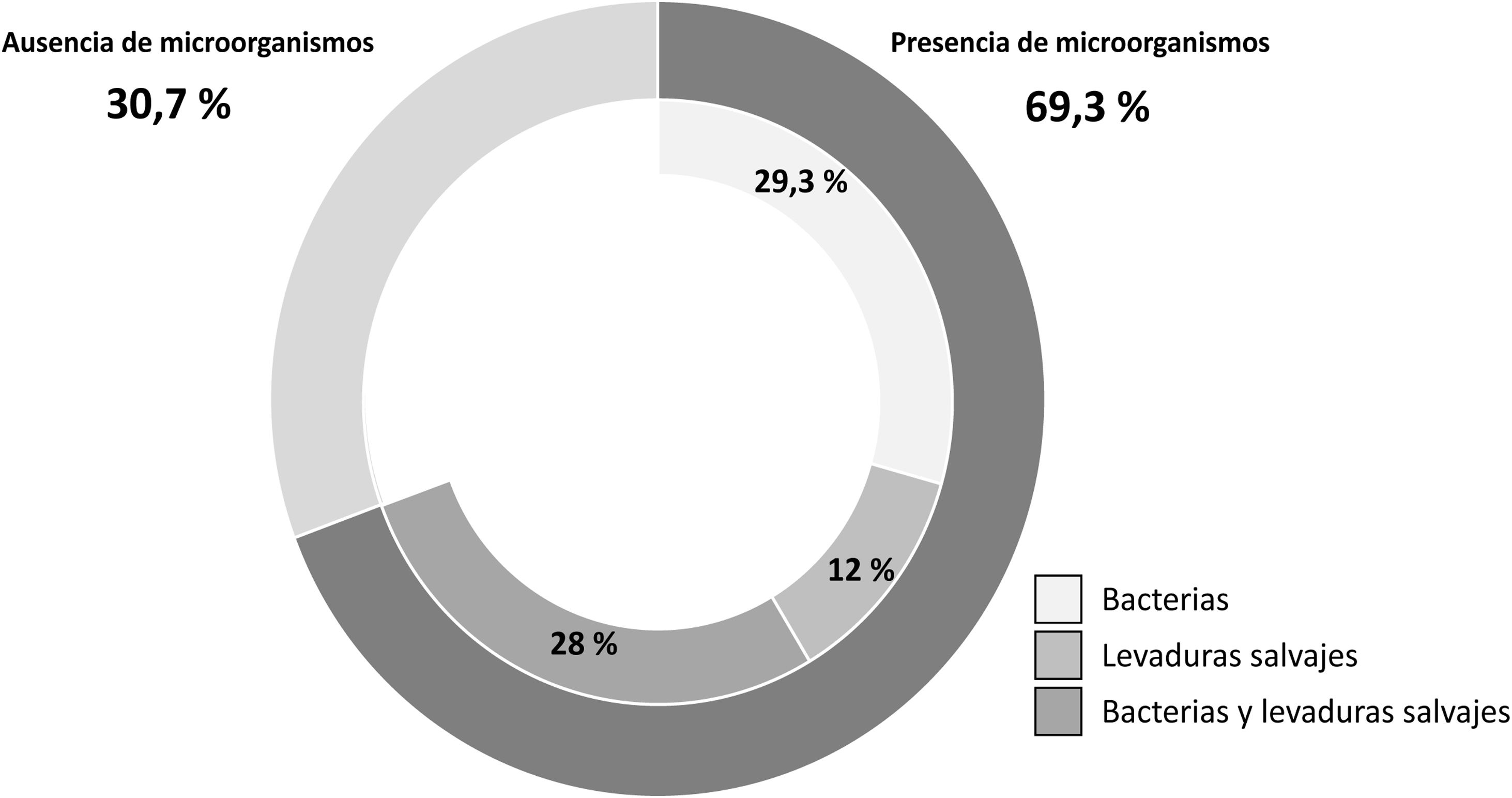
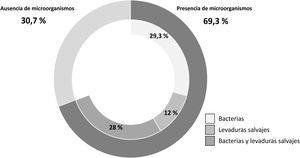
ResultadosDe las 75 muestras analizadas, 52 (69,3%) evidenciaron crecimiento de microorganismos (>10UFC/ml de cerveza) en por lo menos uno de los medios de cultivo empleados para la detección de contaminantes cerveceros. Asimismo, 34 de las 75 cervezas estudiadas (45,3%) mostraron resultados positivos en el medio HLP, selectivo para BAL de los géneros Levilactobacillus, Lactobacillus y Pediococcus. Con la excepción de 2 muestras, todos los casos positivos en HLP también lo fueron en WLD, medio que permite la detección y el recuento de bacterias acéticas, de especies de Flavobacterium, Proteus y otras bacterias termofílicas, y de BAL. Se registraron 9 muestras positivas en el medio WLD que fueron negativas en el medio HLP, totalizando así 43 muestras con presencia de bacterias contaminantes (57,3%). En cuanto a la detección de levaduras contaminantes, se observó crecimiento de colonias en 31 cervezas (39,7%). En el 29,3% de las muestras se detectaron exclusivamente bacterias, mientras que el 12% presentó únicamente levaduras contaminantes y el 28% ambos tipos de microorganismos (fig. 1).
Incidencia de microorganismos contaminantes en 75 muestras de cerveza embotellada producidas por microcervecerías de la Patagonia andina, durante la primavera-verano de 2014-2015. Una cerveza se consideró contaminada cuando presentó >10UFC/mL (en medio WLD para bacterias totales y LCSM para levaduras contaminantes) y/o cuando se detectó presencia de bacterias en el medio HLP.
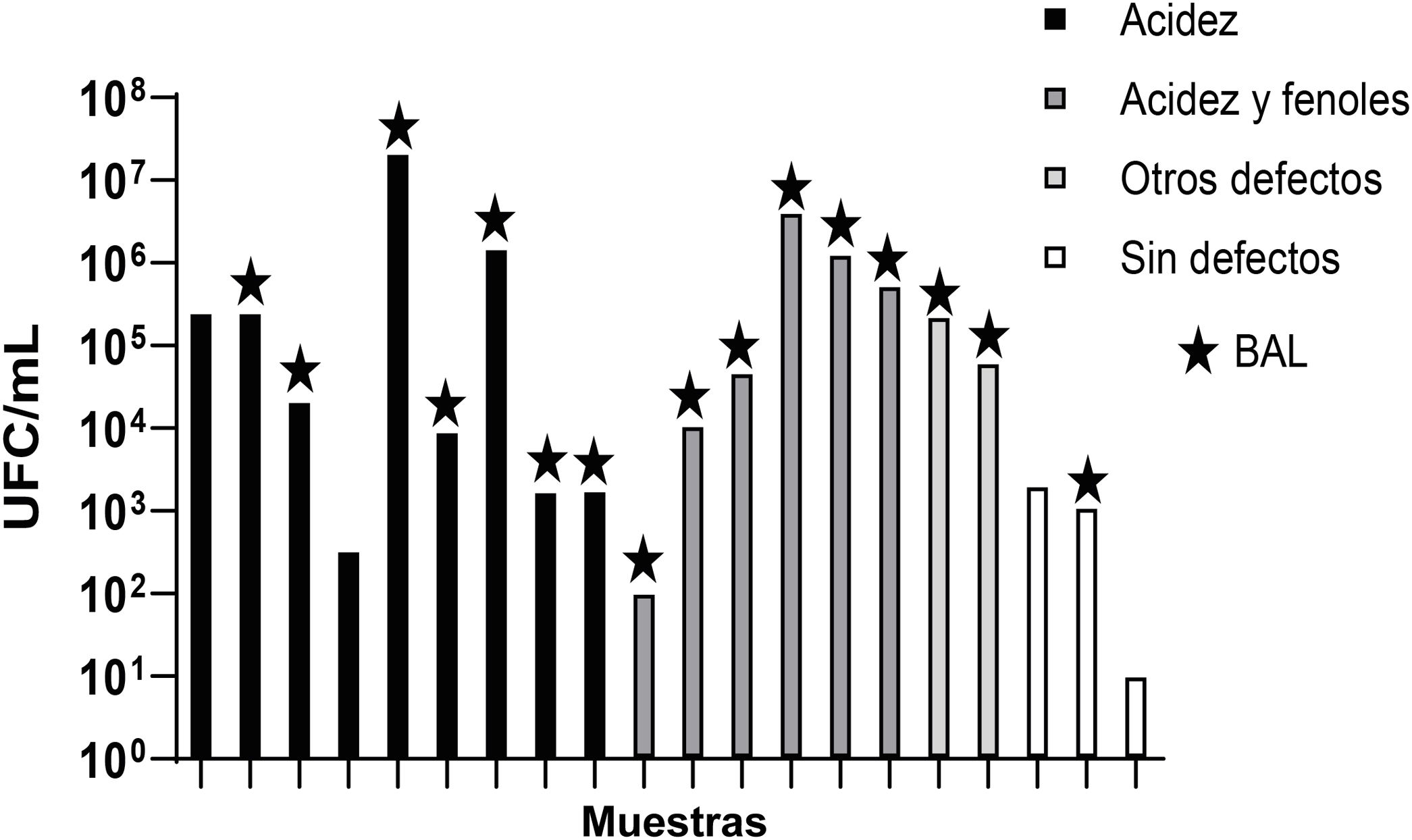
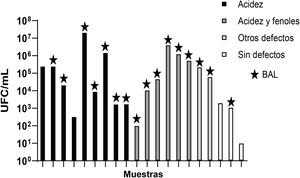
El número de colonias en las 41 muestras con crecimiento de bacterias en WLD varió desde 10UFC/ml hasta 2,1×107UFC/ml. Por otro lado, las 31 muestras positivas para levaduras contaminantes presentaron valores de recuento que oscilaron desde 10UFC/ml hasta 1×104UFC/ml (con un valor promedio de 1,2×102UFC/ml).
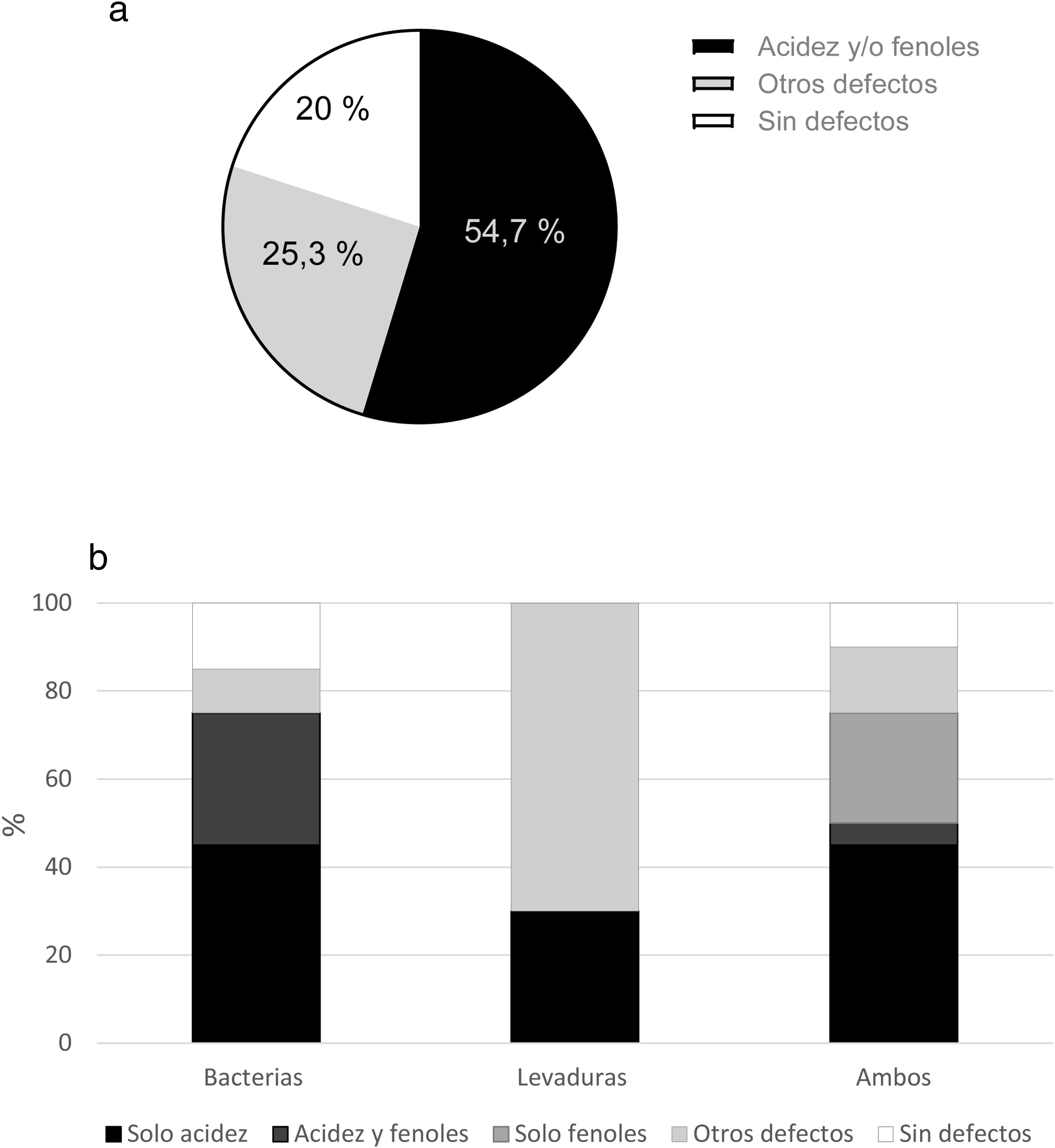
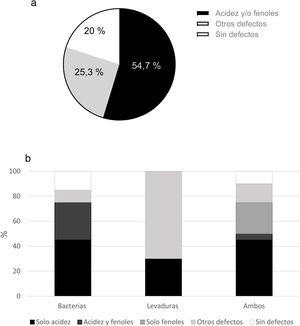
Como análisis complementario a los estudios microbiológicos, las cervezas fueron evaluadas sensorialmente para detectar posibles defectos asociados a contaminaciones microbianas y luego agrupadas en 3 grupos, según los descriptores percibidos. Se detectó que el 54,7% de las cervezas catadas (41 de las 75) presentaban los descriptores básicos de contaminación que se consideran inaceptables para el estilo de cerveza definido por el productor y conformaron el grupo «acidez y/o fenoles» (fig. 2a), es decir, exhibían una acidez pronunciada (comúnmente asociada a la contaminación bacteriana) y/o aroma y sabor (flavor) a compuestos fenólicos (aromas especiados o a clavo de olor, normalmente atribuidos a contaminación por levaduras). El 25,3% de las cervezas (19 muestras) presentaron descriptores que podrían o no estar vinculados a eventos de contaminación («otros defectos»), pero que influyen de forma negativa en la tomabilidad; estos fueron aroma y sabor a manteca (diacetilo), aroma y sabor a choclo (dimetil sulfuro) y huevo (compuestos sulfurosos). El 20% (15/75) de las cervezas analizadas conformaron el grupo «sin defectos», ya que no se percibieron deméritos a nivel sensorial; sin embargo, se registró la presencia de contaminantes en 6 de las 15 muestras.
a) Resultados del análisis sensorial de 75 cervezas artesanales de la Patagonia andina. La categoría «acidez y/o fenoles» incluye las muestras que presentaron los descriptores comúnmente asociados a contaminación. La categoría «otros defectos» agrupa las muestras que presentaron defectos como DMS, diacetilo y sulfuros, y «sin defectos» hace referencia a las muestras que no presentaron deméritos sensoriales. b) Descriptores sensoriales hallados según el tipo de microorganismo contaminante encontrado en la cerveza.
La figura 2b relaciona los resultados del análisis sensorial con el tipo de microorganismo encontrado. La presencia de acidez fue el principal descriptor que pudo asociarse con los casos de contaminación, ya que se percibió en el 75% de las muestras contaminadas solo con bacterias, en el 30% de las muestras contaminadas solo con levaduras y en el 50% de las muestras contaminadas con ambos tipos de microorganismos (fig. 2b). Por otro lado, la presencia del descriptor acidez no parece depender del número de UFC/ml registrado, ya que el valor promedio de UFC/ml en las muestras contaminadas exclusivamente con bacterias y que presentaron acidez no mostró diferencias significativas respecto del de las muestras contaminadas exclusivamente con bacterias que no presentaron dicho descriptor (5,1×105UFC/ml vs. 4,3×106UFC/ml, respectivamente, p=0,4145). Tampoco se observaron diferencias entre las muestras con y sin acidez contaminadas únicamente con levaduras (p=0,6112). Sin embargo, la presencia de acidez parece estar vinculada con la presencia de BAL, ya que 13 de las 15 muestras con acidez registraron crecimiento de BAL (86,6%) en HLP, como se muestra en la figura 3. En el caso de las levaduras, la asociación con un descriptor particular no fue tan clara, ya que los deméritos asociados a cervezas contaminadas con levaduras exclusivamente pertenecen al grupo «otros defectos», entre los cuales los más destacados fueron DMS, diacetilo y sulfuros.
De las 43 cervezas que registraron crecimiento de bacterias fue posible obtener e identificar genéticamente 37 aislamientos bacterianos. Los resultados de identificación (tabla 1) mostraron que 18 aislamientos (48,6%) pertenecían a Levilactobacillus brevis, presente en el 24% de las muestras de cerveza analizadas. Un aislamiento adicional de bacteria ácido láctica (BC17) fue identificado como Lactobacillus backii.
Identificación por secuenciación de la región 16S del ADN ribosomal de aislamientos bacterianos obtenidos de cervezas artesanales contaminadas
| Grupo taxonómico | Especie | Cervecería de origen(n.o de aislamientos) | % identidad | Secuencia de comparación (GeneBank) | Códigointerno |
|---|---|---|---|---|---|
| Bacterias ácido lácticas(gram+) | Levilactobacillus brevis | 2 (1), 4 (2), 6 (1), 9 (1), 14 (2),21 (2), 22 (1),24 (2), 25 (1),26 (3), 29 (1), 30 (1) | >99,28 | LC062897.1(JCM 1059T) | BC15, BC16, BC60, BC19, BC28, BC32, BC36, BC48, BC64, BC49, BC51, BC53, BC55, BC58, BC59, BC62, BC65, BC67 |
| Lactobacillus backii | 4 (1) | 99,91 | NR_114385.1(JCM 18665T) | BC17 | |
| Bacterias ácido acéticas(gram−) | Acetobacter malorumAcetobacter cerevisiae | 12 (1), 14 (1),15 (1), 17 (1),20 (1), 31 (1) | >99,61 | NR_113553.1(JCM 17274T)NR_113552.1(JCM 17273T) | BC29, BC35, BC45, BC69, BC39, BC42 |
| Acetobacter fabarumAcetobacter lovaniensis | 18 (1), 24 (1) | >99,71 | NR_042678.1(LMG 24244T)NR_113551.1(JCM 17121T) | BC43, BC50 | |
| Acetobacter acetiAcetobacter sicerae | 14 (2) | >99,25 | NR_113673.1(NBRC 14818T)NR_133949.1(LMG 1531T) | BC31, BC38 | |
| Asaia bogorensisAsaia lannensis | 14 (1) | 85,24 | AP014690.1(NBRC 16594T)NR_114144.1(NBRC 102526T) | BC41 | |
| Otras bacterias | Raoultella ornithinolytica | 6 (3) | 99,80 | MH071137.1(JCM 6096T) | BC23, BC25, BC26 |
| Cronobacter dublinensis | 37 (1) | 98,89 | MF118668.1 (DSM 18707T) | BC81 | |
| Klebsiella oxytoca | 33 (1) | 99,89 | NR_112010(JCM 1665T) | BC82 | |
| Staphylococcus epidermidis | 28 (1) | 99,89 | KT989845.1(ATCC 14990T) | BC63 | |
| Pseudomonas hunanensis | 4 (1) | 99,51 | JX545210.1(LV 1247546 T) | BC18 |
El segundo grupo más representado correspondió al género de bacterias ácido acéticas Acetobacter sp., con 10 aislamientos (27%) presentes en el 13,3% de las muestras. En este género, no fue posible la identificación a nivel de especie, ya que la comparación de las secuencias arrojó resultados ambiguos en 8 de los 10 aislamientos. Sin embargo, fue posible discriminar entre pares de especies. Así, los aislamientos BC29, BC35, BC45 y BC69 presentaron similitud con Acetobacter malorum y A. cerevisiae, los aislamientos BC31 y BC38 con A. aceti y A. sicerae, y los aislamientos BC43 y BC50 con A. fabarum y A. lovaniensis. El aislamiento BC41 presentó similitud con las especies Asaia bogorensis y Asaia lannensis, sin embargo, el porcentaje de identidad fue menor del 90%.
Un tercer grupo estuvo integrado por 5 enterobacterias, 3 de estos aislamientos fueron identificados como Raoultella ornithinolytica (BC23, BC25 y BC26) y provenían de la misma cervecería. Los otros dos correspondieron a dos cervecerías distintas y se identificaron como Cronobacter dublinensis (BC81) y Klebsiella oxytoca (BC82). Las muestras de origen de estas enterobacterias se caracterizaron por poseer un pH elevado (4,42-4,89) en comparación con el promedio de cervezas analizadas (4,42±0,27). Por último, se obtuvieron aislamientos individuales pertenecientes a las especies Pseudomonas hunanensis y Staphylococcus epidermidis.
En la tabla 1 se puede observar que algunas cervecerías presentaron diversidad de contaminantes en sus cervezas. Por ejemplo, de las 3 cervezas provenientes de la cervecería 14, se aislaron 3 especies bacterianas. Asimismo, se aisló el mismo microorganismo en diferentes muestras de la misma cervecería; por ejemplo, la cervecería 26 presentó contaminación con la misma bacteria en los 3 estilos de cerveza analizados.
Aislamiento e identificación molecular de levaduras contaminantesDe las 75 muestras analizadas, 31 registraron crecimiento de levaduras contaminantes, a partir de las cuales se aislaron e identificaron genéticamente 24. Dieciséis aislamientos (66,7%) provenientes de 10 cervecerías diferentes fueron identificados como Saccharomyces spp. sobre la base del análisis de las secuencias de la región ITS (LC16 cervecería 4, LC21 cervecería 8, LC23 cervecería 10, LC24 cervecería 10, LC25A cervecería 10, LC25B cervecería 10, LC26 cervecería 12, LC34 cervecería 12, LC35 cervecería 1, LC38 cervecería 15, LC42 cervecería 20, LC43 cervecería 20, LC45 cervecería 21, LC47 cervecería 23, LC50 cervecería 4, LC54 cervecería 33).
A su vez, se obtuvieron 8 levaduras no sacaromicéticas (33,3%) (tabla 2), 6 de ellas pertenecientes al filum Ascomycota, identificadas como Wickerhamomyces anomalus. Además, se identificaron aislamientos pertenecientes a las especies Clavispora lusitaniae y Wickerhamiella pararugosa, con un aislamiento cada una. Dos aislamientos pertenecieron al filum Basidiomycota y fueron identificados como Trichosporon ovoides y Naganishia adeliensis.
Identificación por secuenciación de la región ITS de levaduras no sacaromicéticas obtenidas de cervezas artesanales contaminadas
| Grupo taxonómico | Especie | Cervecería de origen(n.o de aislamientos) | % identidad | Secuencia de comparación (GeneBank) | Código interno |
|---|---|---|---|---|---|
| Ascomycota | Wickerhamomyces anomalus | 6 (1), 18 (1), 14 (1),20 (1) | >99,82 | MH545921.1(CBS 5759T) | LC 19, LC32, LC40, LC44 |
| Clavispora lusitaniae | 6 (1) | 100 | MH545926.1(CBS 6936T) | LC 18 | |
| Wickerhamiellapararugosa | 14 (1) | 99,89 | MH545932.1(CBS 1010T) | LC 33 | |
| Basidiomycota | Trichosporon ovoides | 14 (1) | 99 | NR_073254.1(CBS 7556T) | LC 31 |
| Naganishia adeliensis | 33 (1) | 100 | NR_111050.1(CBS 8351T) | LC52 |
Como en el caso de las bacterias, se puede observar diversidad de levaduras contaminantes en una misma fábrica, como es el caso de la cervecería 14, que presentó 3 levaduras contaminantes de diferente especie, además de las 3 especies de bacterias mencionadas anteriormente.
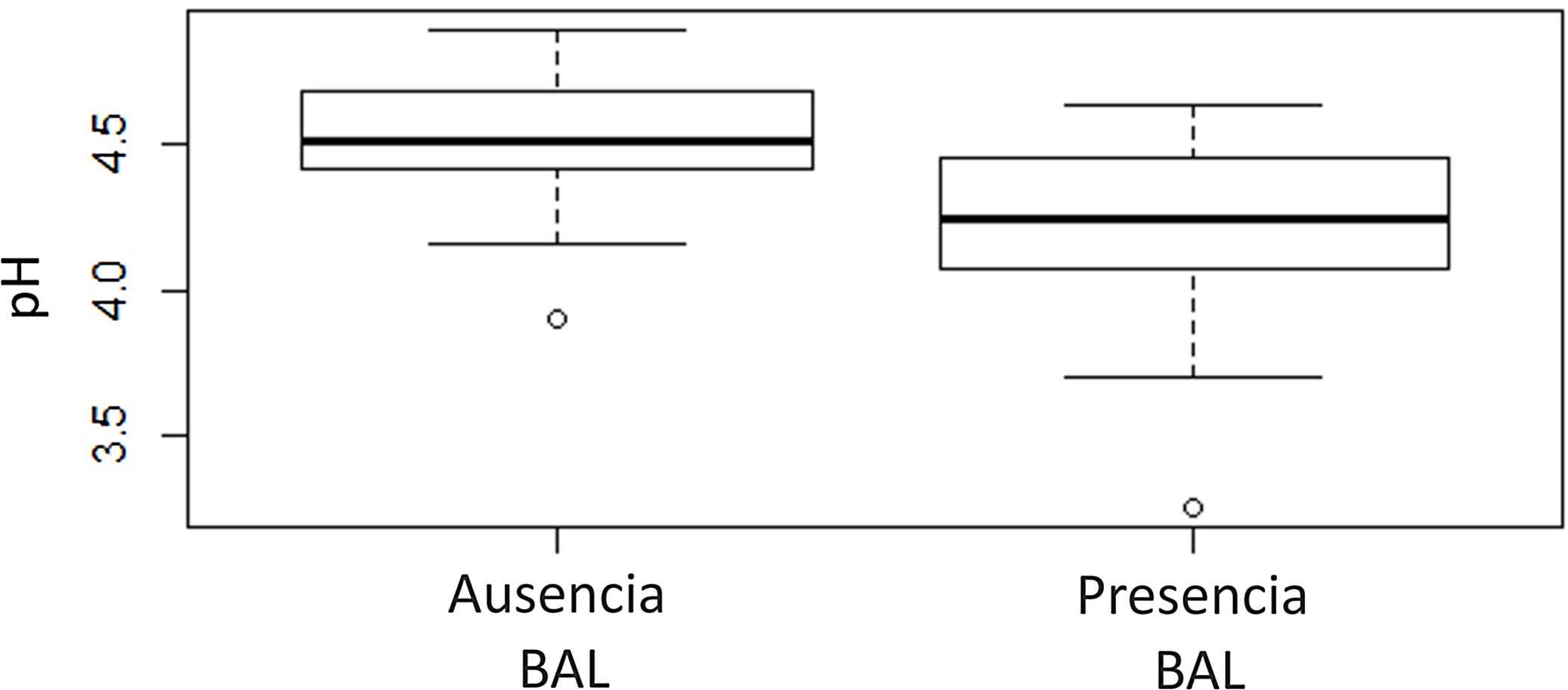
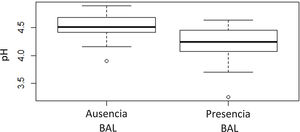
Caracterización fisicoquímica de las cervezas y su relación con la presencia de contaminantesLas cervezas estudiadas mostraron valores de pH entre 3,30 y 4,90, con un promedio de 4,42±0,27. La presencia de contaminantes, en general (bacterias, levaduras o ambas), no mostró correlación con el pH (p=0,0509). Sin embargo, al analizar la relación entre el pH y la presencia de bacterias ácido lácticas (BAL), sí se observó correlación entre estos parámetros, con un nivel de significación de p=0,003 (fig. 4).
En cuanto a las unidades de amargor, las cervezas variaron entre 15 y 65IBUs (en promedio, 23,41±10,55IBUs), y no se observó correlación entre la presencia de microorganismos contaminantes y el amargor de la cerveza (p=0,5278). En relación con el color, las muestras variaron entre 2 y 70SRM (en promedio, 12,88±13,94SRM) y no se encontró correlación entre los eventos de contaminación y el color de la cerveza (p=0,1570). Tampoco se observó una correlación entre los solutos residuales medidos en grados Brix y la contaminación microbiana (p=0,1224).
DiscusiónEste trabajo constituye el primer estudio formal que abarca la evaluación de la calidad microbiológica, fisicoquímica y sensorial de cervezas artesanales patagónicas, por lo que determina un punto de partida para entender y dimensionar la incidencia de esta problemática en las microcervecerías de la región.
La evaluación de la calidad microbiológica utilizando métodos tradicionales de cultivo evidenció la presencia de contaminantes en un gran número (ca. 70%) de las cervezas artesanales de la Patagonia andina producidas y envasadas durante la temporada 2014-2015. Esta incidencia de contaminaciones es comparable con el valor del 73% informado recientemente para cervezas artesanales en la Ciudad Autónoma de Buenos Aires y Gran Buenos Aires13. La mayoría de las cervecerías implicadas en nuestro estudio presentaron al menos una cerveza con evidencias de contaminación, causada principalmente por bacterias (57,3%), aunque también se registró una elevada tasa de contaminaciones mixtas (bacterias y levaduras). Las cervezas contaminadas solo con levaduras fueron las minoritarias (12%). Los valores obtenidos, nuevamente, son comparables con los informados con anterioridad en Buenos Aires y denotan la importancia del control microbiológico y la falta de procedimientos adecuados para la limpieza y desinfección de las cervecerías artesanales argentinas.
Algunas cervecerías andino-patagónicas presentaron diversidad de contaminantes en sus cervezas, como es el caso de la cervecería 14. Ese resultado sugiere diversas fuentes o eventos de contaminación. Asimismo, se aisló el mismo microorganismo en diferentes muestras de una misma cervecería, por ejemplo, la cervecería 26 presentó contaminación con la misma bacteria en los 3 estilos de cerveza analizados. Esto indica que esas cepas se encuentran bien establecidas en la fábrica y se extienden a los diferentes lotes, posiblemente debido a la reutilización de levaduras cerveceras contaminadas o la mala sanitización de superficies en las líneas de circulación de mosto, cerveza o equipos de producción. Por otro lado, se constató que las cervezas contaminadas presentaron, en general, valores de carga microbiana muy superiores a los valores menos exigentes recomendados en cerveza (de hasta 10UFC/ml), incluso excedieron ampliamente los límites más exigentes (<1UFC/ml)16,34.
El análisis sensorial puso en evidencia que las contaminaciones microbianas tienen un impacto considerable a nivel organoléptico, ya que en más de la mitad de las cervezas estudiadas se detectaron descriptores negativos, como acidez y aromas y sabores fenólicos. Estos defectos estuvieron correlacionados con la presencia de bacterias y levaduras contaminantes. Asimismo, Moretti30 confirmó que la presencia de BAL, aun en bajas densidades celulares (<1UFC/ml), causa defectos sensorialmente notables.
El resto de los defectos sensoriales registrados en este trabajo correspondieron a descriptores que podrían o no estar vinculados a una contaminación, pero igualmente influyeron de forma negativa en la tomabilidad de la cerveza. Las cervezas que no presentaron defectos sensoriales, pero sí contaminantes (6 cervezas), pertenecían a estilos de cerveza con caracteres y sabores fuertes, que pudieron haber enmascarado los efectos causados por la contaminación. Por ejemplo, de las 6 muestras en esta situación, 3 cervezas pertenecían al estilo Indian Pale Ale (IPA), con alto grado de amargor (45-65IBUs) y con elevada presencia de lúpulo en aroma; una al estilo Dubbel, de origen belga, con alta graduación alcohólica y fuertes notas frutales y especiadas; y las dos restantes al estilo Strong Ale. La aparición de indicadores sensoriales asociados a contaminantes en el producto terminado y su correlación con los resultados de los análisis microbiológicos demuestran la utilidad del análisis sensorial como herramienta complementaria para la detección de contaminaciones, aunque en el caso de los estilos con caracteres fuertes o con alta graduación alcohólica, los defectos pueden verse enmascarados.
La utilización de medios selectivos para la detección de bacterias contaminantes permitió el aislamiento de BAL en el 25,3% de las muestras, valor similar al encontrado por Menz et al.29 en cervezas artesanales de Australia (27,5%). En Italia, se ha reportado la presencia de BAL en el 41% de las cervezas artesanales30 y en el 15% en EE. UU.51. Estos datos, en su conjunto, confirman que las BAL dominan los eventos de contaminación a nivel mundial y que las cervezas de la Patagonia andina responden a ese mismo patrón.
La identificación molecular confirmó que L. brevis es la especie que predomina en las cervezas contaminadas con bacterias, en coincidencia con lo descripto por Suzuki48. Dicho autor mostró que L. brevis fue y sigue siendo la principal bacteria que contamina la cerveza en Europa; esta se halló en el 26-51% de los incidentes de contaminación, según múltiples estudios realizados entre 1980 y 2016. Lactobacillus backii también es una bacteria descripta frecuentemente en los incidentes de contaminación8,40, aunque en este trabajo estuvo representada por un solo aislamiento.
El segundo grupo de bacterias halladas con alta frecuencia en este trabajo estuvo conformado por miembros del género Acetobacter. Esto indica que las microcervecerías de la Patagonia andina no cuentan con el equipamiento adecuado para impedir el ingreso de oxígeno en el proceso de producción o en la etapa de envasado, a fin de reprimir el crecimiento de estos microorganismos, aerobios obligados. Este tipo de bacterias poseen un rol importante en la formación de biopelículas32,44, los cuales, debido a su mayor resistencia a la limpieza mecánica y química19, actúan como reservorio de otros microorganismos contaminantes42. Con la información proporcionada por la secuenciación de la región 16S fue posible determinar al menos 3 tipos de Acetobacter diferentes que afectaron a las cervezas patagónicas. Su identificación a nivel de especie requiere del análisis de secuencias de otras regiones del ADN10.
En tercer lugar, se aislaron bacterias pertenecientes a la familia Enterobacteriaceae, las cuales funcionan como indicadores del nivel de higiene y sanitización en las plantas de producción, debido a que normalmente son sensibles al etanol y a pH bajos28. Por esta razón, algunas enterobacterias como Salmonella, Serratia, Shigella, Escherichia y Klebsiella normalmente no son capaces de desarrollarse en la cerveza terminada28,29,33, pero en algunos casos pueden sobrevivir por tiempos prolongados en el producto si hubo reproducción en las etapas iniciales de la fermentación28. Por ejemplo, enterobacterias patógenas como Escherichia coli y Salmonella typhimurium son capaces de sobrevivir hasta 30 días cuando se las conserva a 4°C, incluso en presencia de un 5% de alcohol, o hasta 20 días a un pH inferior a 428. Esto explicaría el aislamiento de enterobacterias en las cervezas analizadas en este trabajo, que presentaron densidades finales elevadas (y, por lo tanto, valores de alcohol menores de lo esperado) o pH relativamente altos (4,4-4,9), o ambos atributos, que son indicativos de fermentaciones ineficientes o inconclusas. Estas situaciones atentan contra la calidad y, sobre todo, contra la inocuidad de la cerveza artesanal, al no generarse las barreras fisicoquímicas normales de la cerveza, principalmente los niveles de alcohol y pH adecuados.
En relación con la identificación de levaduras, más de la mitad de los aislamientos obtenidos en medio LCSM correspondieron al género Saccharomyces, y todos ellos fueron confirmados como pertenecientes a la especie S. cerevisiae mediante métodos moleculares en un trabajo complementario, que incluyó su clasificación a nivel de cepa, lo que nos permitió verificar que no se trataba de cepas cerveceras comerciales23. En consonancia con nuestros hallazgos, Suzuki48 informó que en el período 1992-2002, el género Saccharomyces fue el dominante en los incidentes de contaminación en Europa. Por otro lado, se pudo determinar que muchas de esas cepas (68%) corresponden a cepas diastáticas de S. cerevisiae, ya que se comprobó la presencia del gen STA1, involucrado en el metabolismo de dextrinas23.
La levadura Wickerhamomyces anomalus fue la levadura salvaje no sacaromicética más frecuente en las muestras contaminadas. Esta levadura ascomicética es comúnmente aislada de cerveza y otras bebidas17,39. También se la ha observado como levadura dominante en las biopelículas de superficies de acero de los equipos en las líneas de llenado de cerveza42. La importancia de las contaminaciones con W. anomalus radica en que se trata de una levadura que produce significativas cantidades de acetato de isoamilo (asociado con descriptores como banana), acetato de etilo (manzana roja) y ácido acético, lo que redunda en notorios defectos en las cervezas que contamina39. Sin embargo, no fue posible correlacionar directamente estos descriptores con W. anomalus, ya que las muestras de origen contenían otros microorganismos que podrían haber enmascarado el flavor esperado para cervezas contaminadas con esta levadura. El resto de las levaduras no sacaromicéticas identificadas estuvieron representadas por un solo aislamiento, correspondiente a levaduras no frecuentes en eventos de contaminación cervecera.
Aunque los tipos de cerveza tradicionales todavía dominan el mercado, actualmente existe una tendencia que empuja a las cervecerías al desarrollo de productos novedosos9. En este sentido, la venta de cervezas no convencionales, cervezas ácidas, cervezas bajas en calorías y cervezas sin alcohol aumentó significativamente. Los aislamientos obtenidos en este trabajo fueron incorporados a la colección microbiana del IPATEC y representan un gran recurso, tanto para el diseño de estrategias de detección y control de microorganismos contaminantes como para profundizar en el estudio de su fisiología, características genéticas y potencial para la innovación cervecera.
De los análisis fisicoquímicos realizados, solo el pH demostró ser un parámetro que se correlaciona con la presencia de BAL, lo que señala su relevancia como herramienta complementaria y accesible para el control de calidad frente a las principales bacterias contaminantes. El resto de los parámetros fisicoquímicos evaluados en este trabajo no se correlacionaron con los eventos de contaminación.
ConclusionesNuestros resultados muestran que 8 de cada 10 fábricas de cerveza artesanal de la Patagonia andina argentina presentaron problemas de contaminación, lo que sugiere que la presencia de bacterias y levaduras es una problemática común en estas microcervecerías y pone en evidencia la necesidad de implementar o ajustar sus protocolos de calidad. Los aislamientos de bacterias y levaduras contaminantes obtenidos en este trabajo conforman la primera colección de microorganismos contaminantes de cerveza de la Patagonia andina y servirán como punto de partida para el diseño de estrategias de detección y control de contaminantes cerveceros. Asimismo, contribuirán a la optimización de los procesos de pasteurizado. Dado que actualmente existe una tendencia a elegir bacterias y levaduras «no convencionales» que aumenten el bioflavoring9, algunos de los géneros de levaduras y bacterias encontrados en este trabajo podrían ser de interés para producir nuevos estilos de cerveza, con el objetivo de fomentar la innovación y la diferenciación productiva.
FinanciaciónEste estudio fue financiado con fondos provenientes del CONICET (PIP 424), FONCyT (PICT 3677), UNComahue (B199), SPU (proyecto Amilcar Herrera «Contaminaciones microbianas en la producción de cerveza artesanal: desarrollo de estrategias de control microbiológico y transferencia a micro-cervecerías», Ministerio de Educación, UNComahue 2014 y Diseño productivo «Contaminantes en Cerveza», Ministerio de Educación, UNComahue 2014.
Conflicto de interesesLos autores declaran no tener conflictos de interés.
Agradecemos a los productores de cerveza artesanal de la Patagonia andina, quienes aportaron muchas de las cervezas para realizar el estudio y a los colegas del CRELTEC, quienes siempre mostraron su predisposición para ayudar en la realización del trabajo.