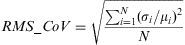Estudiar la viabilidad del tiempo de relajación longitudinal (T1) en RM del cartílago patelar como biomarcador del grado de degeneración.
Material y métodosSe incluyeron 15 sujetos clasificados mediante criterios clínicos (dolor, limitación funcional y duración de la sintomatología) y de imagen como normales (3 hombres, 2 mujeres, 30±14 años), con degeneración inicial del cartílago patelar (3 hombres, 2 mujeres, 30±6 años) y con degeneración avanzada (3 hombres, 2 mujeres, 57±10 años). A todos se les realizó un estudio de RM con secuencias especiales eco de gradiente para segmentar el cartílago y calcular los mapas de T1. Se seleccionó el cartílago completo y las regiones de interés clasificadas en base a criterios clínico-radiológicos como normalidad, degeneración inicial y degeneración avanzada. Los valores de T1 del cartílago se obtuvieron píxel a píxel y se calcularon como la media para todo el cartílago o por subregiones (normal, inicial, avanzada). Las diferencias entre grupos para el cartílago completo y las regiones se analizaron mediante ANOVA Student-Newman-Keuls. La reproducibilidad se estudió mediante el coeficiente de varianza.
ResultadosEl análisis global del cartílago no presentó diferencias estadísticamente significativas entre los 3 grupos (normal: 1.003±172ms; inicial: 1.064±124ms; avanzada: 1.041±308ms, p=0,665). En cambio, en el análisis por regiones se obtuvieron diferencias significativas (normal: 908±53ms; degeneración inicial: 1.057±157 ms; degeneración avanzada: 1.133±116 ms; p=0,029). El estudio de reproducibilidad ofreció variaciones del 1,3% para el cálculo global, del 3,7% para el regional, y del 8,2% para la adquisición.
ConclusiónEn este estudio preliminar, el cálculo del T1 del cartílago permite diferenciar regiones con diferente grado de degeneración.
To study the viability of longitudinal relaxation time (T1) of patellar cartilage as a biomarker of the degree of degeneration.
Material and methodsWe included 15 subjects classified into three groups according to clinical criteria (pain, functional limitation, and duration of symptoms) and imaging criteria as follows: (a) normal (3 men, 2 women; age 30±14 years), (b) with initial degeneration of the patellar cartilage (3 men, 2 women; age 30±6 years), or (c) with advanced degeneration (3 men, 2 women; age 57±10 years). All underwent MRI examination using special echo-gradient sequences to segment the cartilage and calculate the T1 maps. We selected the entire cartilage and the regions of interest classified according to clinical and imaging criteria as normal, initial degeneration, and advanced degeneration. The T1 values of the cartilage were obtained pixel by pixel and were calculated as the mean for the entire cartilage or by subregions (normal, initial, advanced). Differences between groups for the entire cartilage and the regions were analyzed using Student-Newman-Keuls post-hoc ANOVA. Reproducibility was evaluated using the coefficient of variance.
ResultsNo significant differences in the overall analysis of the entire cartilage were found between the three groups (normal: 1003±172ms, initial: 1064±124ms, advanced: 1041±308ms, p=0.665). However, the analysis by regions revealed significant differences (normal: 908±53ms, initial degeneration: 1057±157ms, advanced degeneration: 1133±116ms, p=0.029). The reproducibility analysis found variations of 1.3% for the overall calculation, 3.7% for the regional calculation, and 8.2% for the acquisition.
ConclusionIn this preliminary study, calculating the T1 of the cartilage enabled regions with different degrees of degeneration to be differentiated.
En los estudios de RM, los tiempos de relajación de los protones varían según los componentes tisulares y el campo magnético aplicado. Su conocimiento es importante de cara a planificar secuencias que potencien un determinado contraste en imágenes de RM1. En los últimos años ha habido un creciente interés por el uso clínico de los tiempos de relajación, ya que su cálculo puede ayudar a caracterizar y cuantificar eficazmente diferentes enfermedades, sirva de ejemplo su uso en la valoración de parámetros de permeabilidad capilar y perfusión mediante estudios de farmacocinética (conversión de intensidad a concentración)2. En este contexto, se han desarrollado diferentes técnicas y secuencias para medir los tiempos de relajación, basadas en pulsos de inversión-recuperación3, saturación progresiva4, single-shot Look-Locker5, lecturas directas del espaciok6 y las más usuales secuencias espín eco multieco y eco de gradiente multieco o multiángulo de inclinación. En general, estas secuencias presentan notables diferencias por la diferente resolución espacial y, principalmente, los largos tiempos necesarios para su cuantificación. Entre las secuencias más precisas se encuentran las técnicas basadas en pulsos de inversión-recuperación, aunque requieren de un tiempo de estudio muy elevado. Las técnicas que más han reducido el tiempo de adquisición para la obtención del valor de T1 manteniendo la resolución espacial7,8 se basan en la repetición de secuencias eco de gradiente con distinto ángulo de inclinación9.
El estudio del cartílago articular por RM se ha basado tradicionalmente en la modificación del sistema propuesto por Outerbridge10 aplicado a RM, que permite estratificar el estado del cartílago en cuatro categorías, desde la normalidad hasta la degeneración avanzada (tabla 1). Sin embargo, los avances en las técnicas de cuantificación y la utilización de secuencias de adquisición más rápidas, han permitido incorporar al diagnóstico diversos biomarcadores cuantitativos extraídos de imágenes específicas. Entre estos parámetros están el espesor, el volumen y el cálculo de los tiempos de relajación del cartílago. Con su variación causada por los efectos directos de la enfermedad, se pretende obtener una información menos subjetiva y, por tanto, más reproducible del estado global y regional del cartílago.
Clasificación modificada de Outerbridge aplicada a RM
| Grado | Imagen por RM |
| 0 | Cartílago normal |
| 1 | Señal intracondral alterada, superficie condral normal |
| 2 | Menos de un 50% de pérdida de espesor del cartílago (2A: focal/2B: difusa) |
| 3 | Más de un 50% de pérdida de espesor de cartílago, pero sin exposición del hueso subcondral (3A: focal/3B: difusa) |
| 4 | Pérdida completa del cartílago con exposición del hueso subcondral |
Algunos de estos biomarcadores se centran en las propiedades bioquímicas del cartílago, como la calidad de la matriz de colágeno y sus proteoglicanos, relacionados directamente con los estados iniciales de degeneración del cartílago11,12. En la actualidad se pueden calcular los valores T2, que se correlacionan con la presencia y gravedad de la enfermedad degenerativa del cartílago13; y los valores de T1rho (relajación longitudinal en el marco rotativo, obtenido mediante técnicas spin-lock) que están relacionados con la destrucción de la matriz de colágeno y el aumento de la cantidad y movilidad del agua14. En este estudio se ha propuesto la utilidad del tiempo de relajación longitudinal T1 en la valoración del cartílago, no del tiempo T1rho. Aunque el mecanismo de relajación es longitudinal en ambos casos, ambas magnitudes difieren. El valor de T1 se cálculo teniendo en cuenta el efecto del campo magnético principal (B0) y empleando múltiples ángulos de inclinación, mientras que para el cálculo de T1rho se desecha la contribución de B0 y se emplean pulsos de radiofrecuencia que crean un entorno magnético en el tejido independiente de B0. De esta forma, la relajación de los protones se produce también por interacciones espín-entorno modificado por la utilización de campos magnéticos pequeños, de tal manera que el tejido no está sujeto a los efectos de B0. Es cierto que con técnicas spin-lock se consigue una mayor sensibilidad para evaluar las diferencias entre los distintos entornos macromoleculares, que pasan a caracterizarse mediante el tiempo T1rho. Sin embargo, las técnicas spin-lock requieren secuencias de adquisición más largas y complejas en comparación a las eco de gradiente utilizadas en este artículo para calcular el T1.
En la actualidad se pueden calcular los valores T2, que se correlacionan con la presencia y gravedad de la enfermedad degenerativa del cartílago13; y los valores de T1rho (relajación longitudinal en el marco rotativo, obtenido mediante técnicas spin-lock) que están relacionados con la destrucción de la matriz de colágeno y el aumento de la cantidad y movilidad del agua14. Además, la utilización de medios de contraste con análisis de la captación tardía (dGEMRIC) permite detectar diferencias en la concentración de proteoglicanos mediante el cálculo del T1 del cartílago. Estas diferencias se producen por el acoplamiento por polarización que el medio de contraste sufre al disminuir la concentración de proteoglicanos15.
Hasta la fecha los trabajos existentes se han centrado principalmente en el estudio de los parámetros T2, T1rho y T1 calculado de manera tardía tras la inyección de contraste intravenoso (dGEMRIC). El propósito de este trabajo es calcular el parámetro T1 nativo (sin inyección previa de contraste intravenoso) y validar su utilidad como biomarcador del grado de degeneración del cartílago patelar.
Material y métodosPacientesSe incluyeron 15 sujetos evaluados por traumatólogos con experiencia en patología degenerativa articular, que presentaban dolor o limitaciones funcionales de la rodilla de probable causa degenerativa y con una prolongada duración de su sintomatología.
El cartílago articular se valoró en RM con criterios de intensidad de señal, integridad de la superficie, alteración del hueso subcondral y variaciones en su perfusión analizada con modelos farmacocinéticos tras la administración de un medio de contraste16. La base de la estratificación fue la clasificación modificada de Outerbridge para RM10 (tabla 1), combinada con los criterios de perfusión de permeabilidad capilar16. El estado del cartílago patelar se clasificó con los criterios mencionados como normal (grado I, 3 hombres, 2 mujeres, 30±14 años), con degeneración inicial (grado II, 3 hombres, 2 mujeres, 30±6 años) o con degeneración avanzada (grados III y IV, 3 hombres, 2 mujeres, 57±10 años).
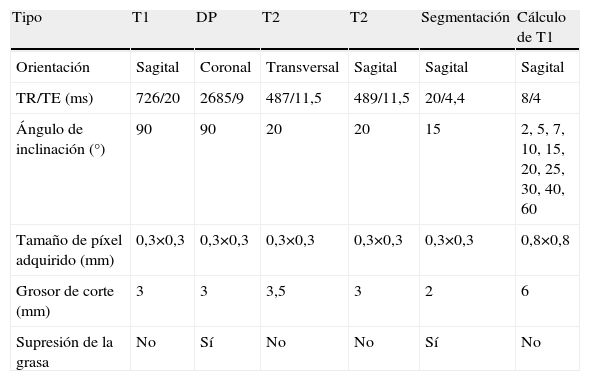
Adquisición de las imágenesLas imágenes se adquirieron en un equipo de RM de 3,0T (Philips Cuidados de la Salud, Best, Países Bajos). Junto con las secuencias habituales del estudio estándar de rodilla (T1 sagital, DP coronal, T2 transversal y T2 sagital) se adquirieron dos secuencias 3D eco de gradiente adicionales (tabla 2). La primera, de alta resolución espacial, se empleó para segmentar el cartílago mediante supresión de señal de la grasa y una muy alta resolución espacial, basándose en la maximización de su contraste con el hueso, el líquido sinovial y los ligamentos. La segunda secuencia eco de gradiente se empleó para el cálculo de los valores de T1, para lo cual se adquirió repetidamente el mismo volumen con destrucción de la magnetización residual, manteniendo constantes los valores de TR y TE, pero variando progresivamente el ángulo de inclinación. El tiempo total de adquisición fue de aproximadamente 17min para el estudio completo.
Parámetros de adquisición de las secuencias de RM utilizadas
| Tipo | T1 | DP | T2 | T2 | Segmentación | Cálculo de T1 |
| Orientación | Sagital | Coronal | Transversal | Sagital | Sagital | Sagital |
| TR/TE (ms) | 726/20 | 2685/9 | 487/11,5 | 489/11,5 | 20/4,4 | 8/4 |
| Ángulo de inclinación (°) | 90 | 90 | 20 | 20 | 15 | 2, 5, 7, 10, 15, 20, 25, 30, 40, 60 |
| Tamaño de píxel adquirido (mm) | 0,3×0,3 | 0,3×0,3 | 0,3×0,3 | 0,3×0,3 | 0,3×0,3 | 0,8×0,8 |
| Grosor de corte (mm) | 3 | 3 | 3,5 | 3 | 2 | 6 |
| Supresión de la grasa | No | Sí | No | No | Sí | No |
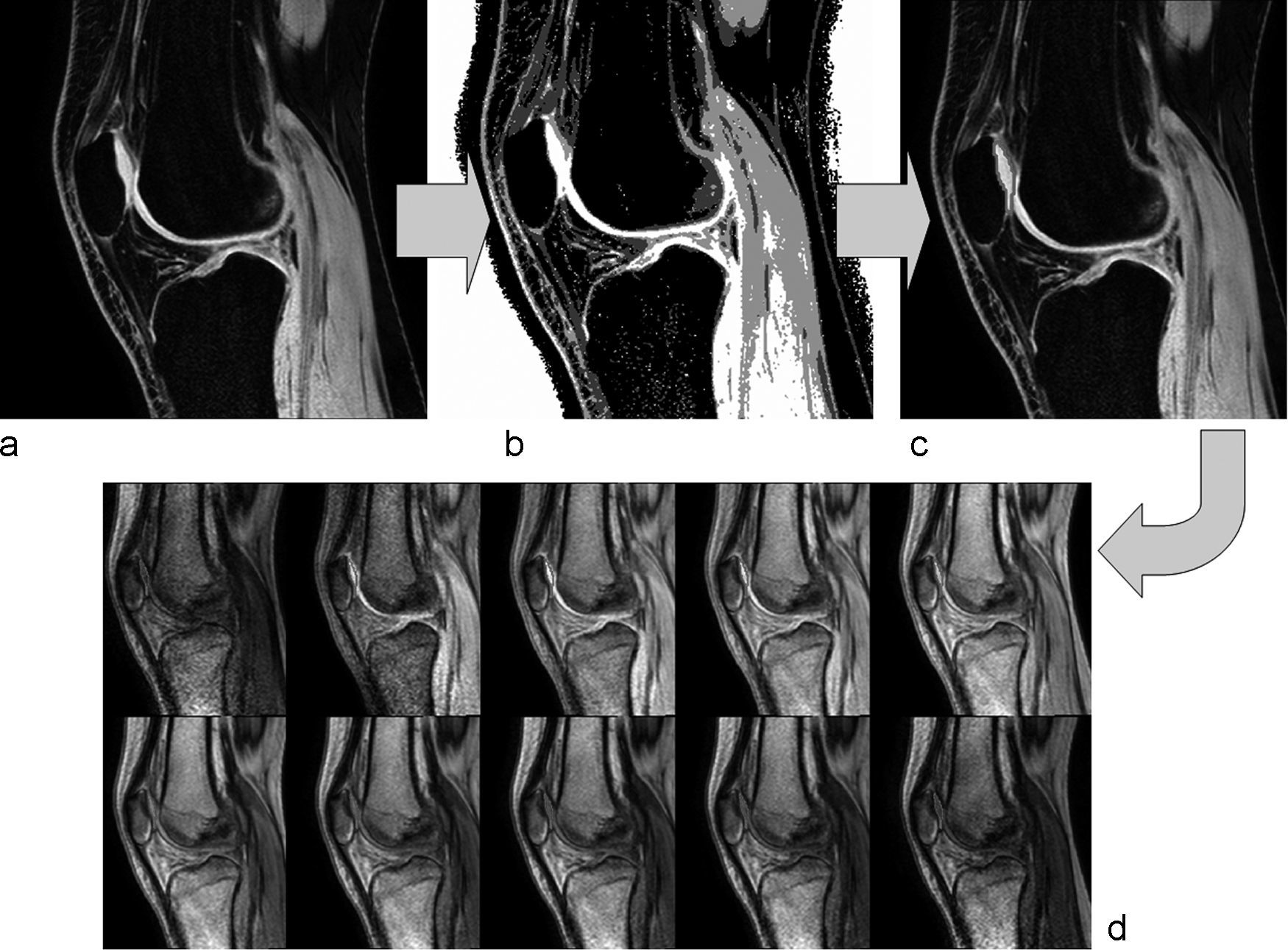
En las imágenes de la primera secuencia eco de gradiente se seleccionó el cartílago mediante una segmentación parcialmente automática basada en la umbralización por intensidades para minimizar la variabilidad debida al usuario17. Tras separar el cartílago se realizaron ajustes por correcciones manuales en aquellos casos en que el método no separó automáticamente el cartílago patelar del femoral. Una vez realizada la segmentación completa del cartílago patelar, esta se utilizó como máscara para el cálculo del T1. Dado que las imágenes para el cálculo de T1 presentaban una menor resolución espacial, fue necesario realizar una interpolación bicúbica para asegurar la coherencia espacial. En la figura 1 se puede ver un ejemplo de segmentación del cartílago patelar mediante esta metodología.
El cálculo del valor de T1 para cada píxel se realizó en base a la ecuación que relaciona la intensidad con el ángulo de inclinación y los tiempos TR y T1 para una secuencia eco de gradiente con supresión de la magnetización y un TE⪡T2* para el cartílago9:
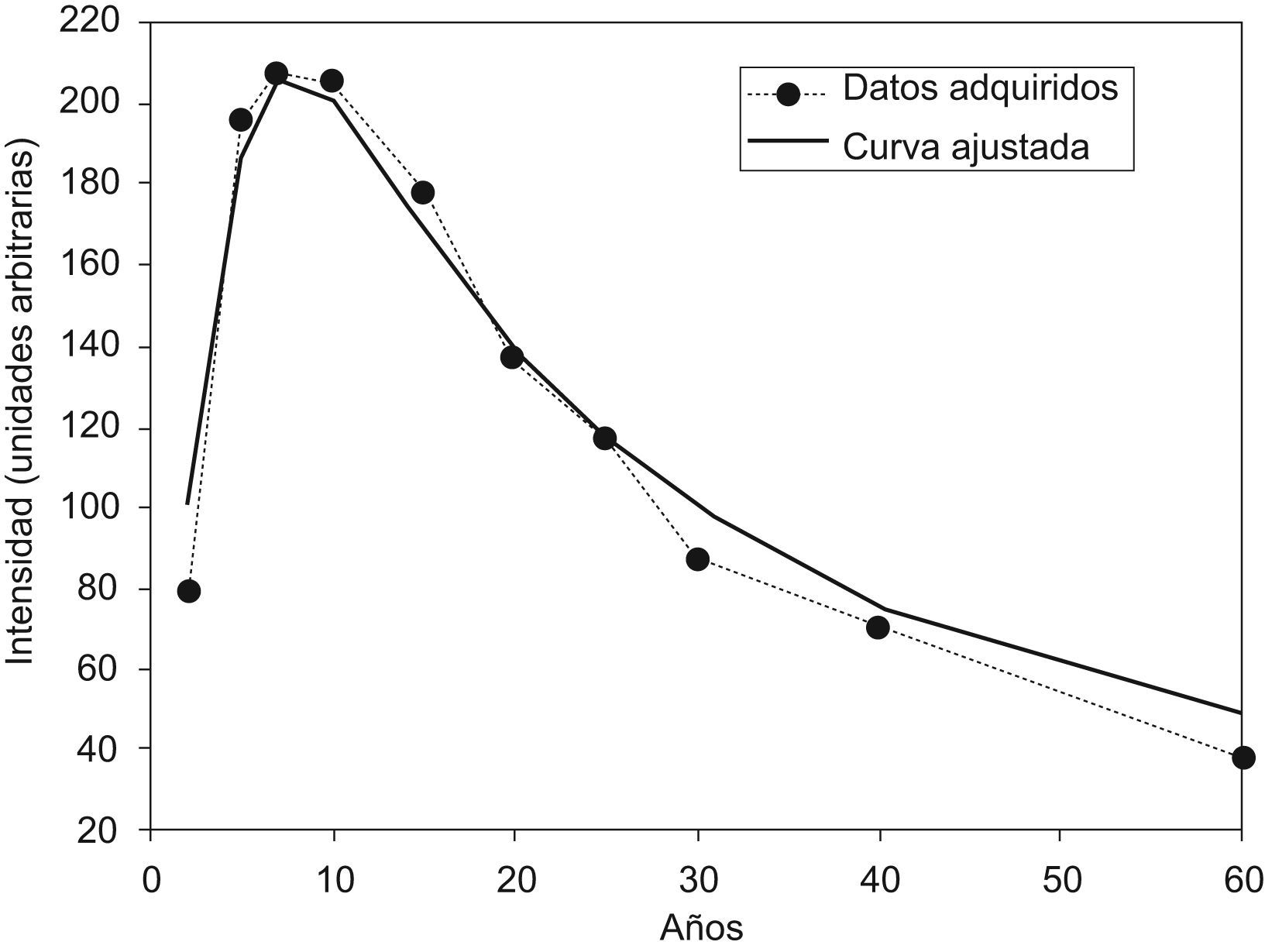
donde S(α, x, y) es la señal para un píxel situado en las coordenadas (x, y), M0(x, y) corresponde a la magnetización en equilibrio térmico y T1(x, y) es el valor de T1 nativo que se desea calcular. De esta expresión se conocen los valores de S para los distintos píxeles y valores del ángulo de inclinación y TR, que no varía a lo largo de la secuencia. Por tanto es posible calcular para cada píxel del cartílago los valores de M0 y T1. Para ello se utilizó un método de ajuste de curvas por mínimos cuadrados18. De esta forma, al generar una curva ajustada a la variación de la señal con el ángulo de inclinación, se obtuvo directamente el valor de T1 de cada píxel (fig. 2). Tras realizar el ajuste para cada píxel se genera de forma automática un mapa paramétrico en color que muestra las variaciones regionales de los valores de T1 para el cartílago patelar.Las medidas de T1 se realizaron para todo el cartílago (T1 promediado global) y también para aquellas áreas etiquetadas como normales, degeneración inicial o avanzada según los criterios modificados de Outerbridge para RM (T1 específico por áreas). Estas áreas específicas se seleccionaron manualmente por un radiólogo experimentado sobre el cartílago previamente segmentado. Todo el análisis de las imágenes se realizó mediante una herramienta informática de cuantificación propia.
Análisis estadísticoPara el estudio de las diferencias se utilizó la prueba ANOVA con análisis post hoc de Student-Newman-Keuls para evaluar las diferencias grupo a grupo (normal-inicial-avanzado). Se consideró estadísticamente significativo un valor de p<0,05.
Para el estudio de la reproducibilidad del cálculo de T1 se evaluaron las imágenes de 5 casos, seleccionados de forma aleatoria (2 normales, 2 iniciales y 1 avanzada), con dos medidas separadas una semana. Para obtener la variabilidad se empleó el coeficiente de variación, calculado como:
donde σi es la desviación típica, μi la media para cada par de medidas y N el número de casos. Valores bajos de este parámetro se corresponden con una reproducibilidad elevada.También se analizó la variabilidad asociada a la adquisición RM, de manera que para 5 sujetos (2 normales, 1 inicial y 2 avanzada) se adquirieron dentro de cada estudio dos veces las imágenes con la secuencia para el cálculo de T1, calculándose los valores medios de T1 para cada adquisición y obteniéndose el coeficiente de variación asociado.
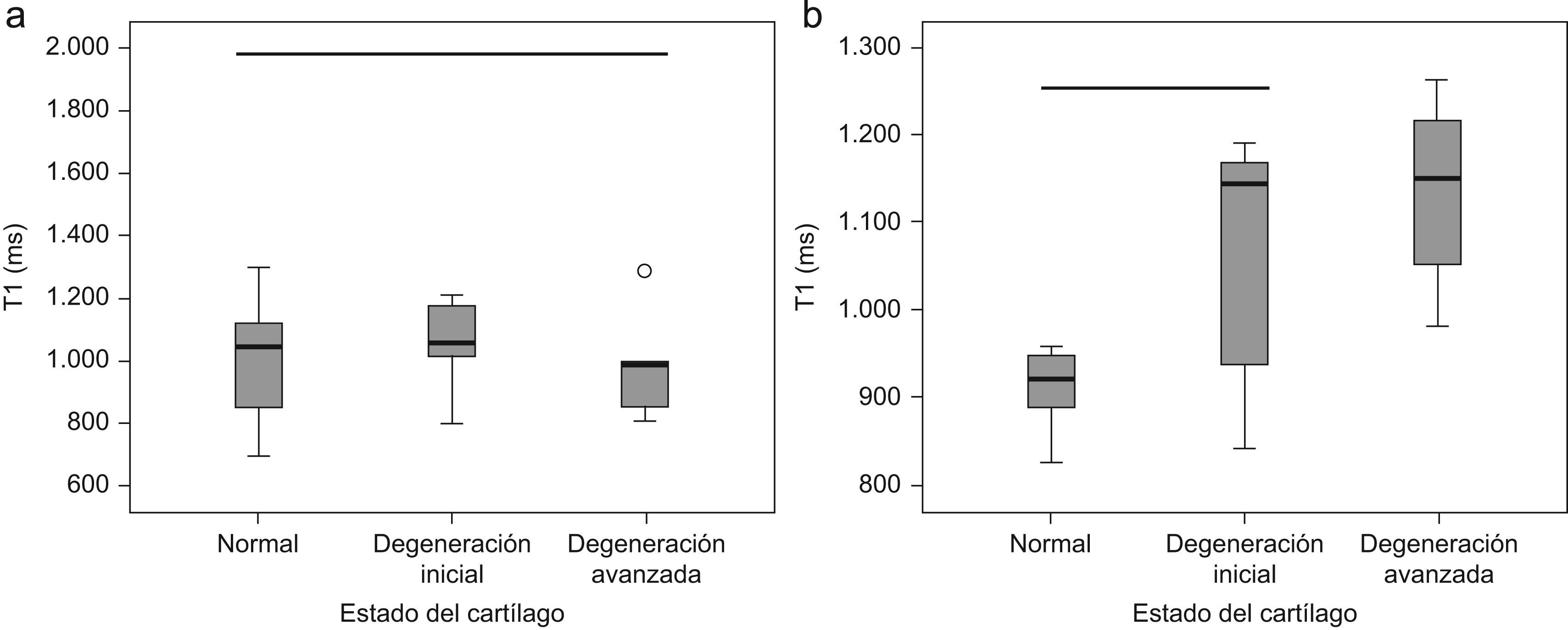
ResultadosEn la tabla 3 se muestran los resultados obtenidos para las distribuciones estudiadas: análisis global y análisis regional. Solo hay diferencias significativas entre los grupos (ANOVA, p=0,029) para el análisis regional, pero no para el global.
Valores de T1 en milisegundos obtenidos mediante el análisis global y el análisis regional para cada grupo de sujetos
| Normal | Degeneración inicial | Degeneración avanzada | p | |
| Análisis global | 1.003±172 | 1.064±124 | 1.041±308 | 0,665 |
| Análisis regional | 908±53 | 1.057±157 | 1.133±116 | 0,029 |
Los valores se expresan como media±desviación típica.
Las pruebas post-hoc de Student-Newman-Keuls demostraron que las diferencias estadísticamente significativas se producían entre los grupos normal y degeneración inicial con el grupo de degeneración avanzada (fig. 3).
En el análisis de la reproducibilidad se obtuvieron unos valores de coeficiente de variación de T1 del 1,3% para la metodología de análisis global, del 3,7% para la metodología de análisis por regiones y del 8,2% para la adquisición, pudiéndose considerar estos resultados como indicadores de una alta reproducibilidad19.
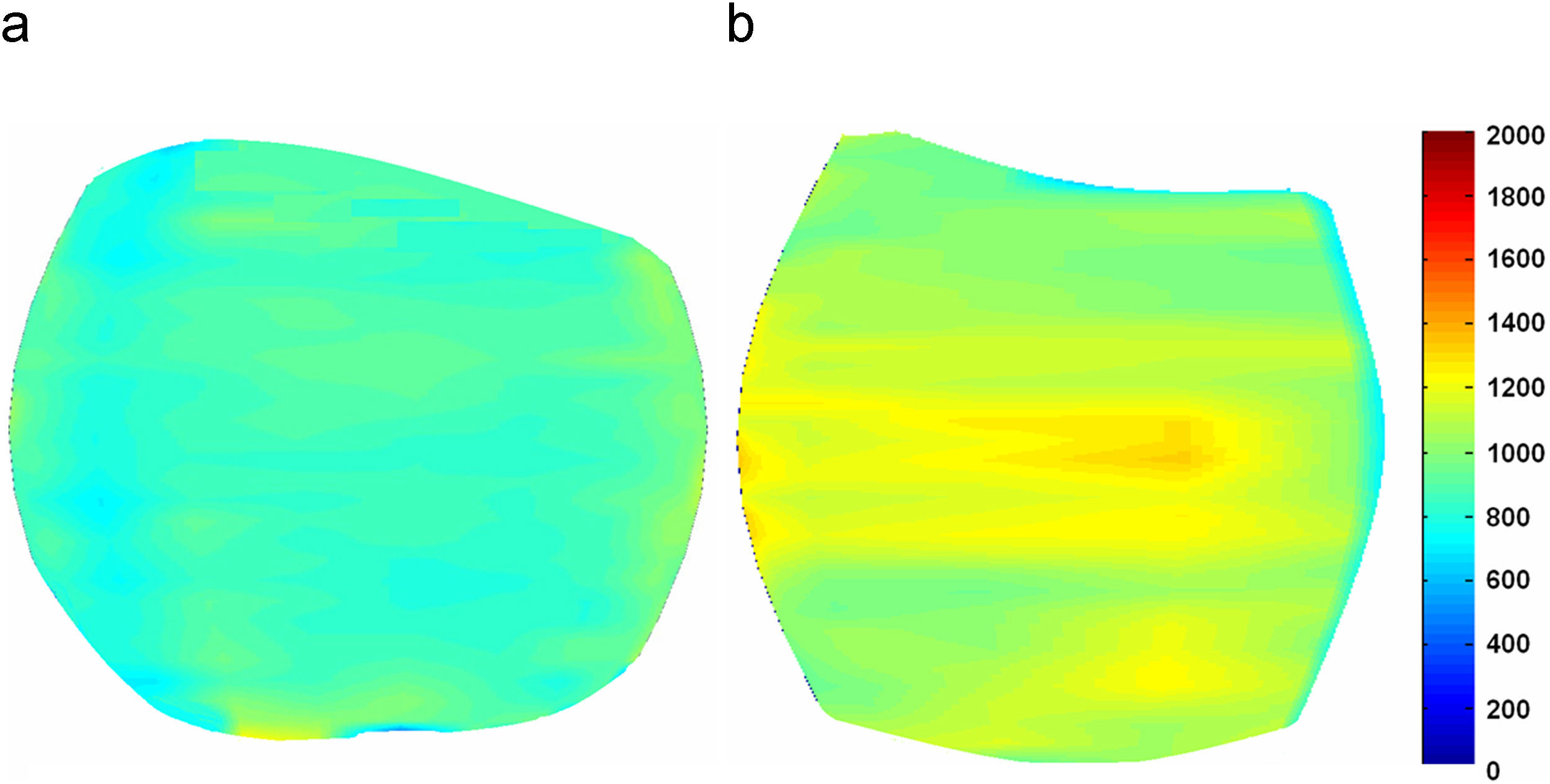
DiscusiónEn este estudio se ha demostrado la utilidad del cálculo del tiempo de relajación longitudinal T1 como biomarcador de imagen por RM para evaluar diferencias entre los distintos grados de degeneración del cartílago patelar. Los mapas paramétricos de T1 permiten visualizar la distribución regional de las anomalías (fig. 4) y realizar medidas objetivas mediante regiones de interés sobre las áreas más afectas. La utilidad es mayor cuando se analizan regionalmente los datos de T1 del cartílago.
Los estudios que han ido apareciendo en los últimos años sobre parámetros cuantitativos del cartílago derivados de la imagen por RM se han centrado principalmente en el cálculo del valor de T2 y de T1rho15. Esta información, junto con la cuantificación volumétrica del cartílago, ha venido a complementar la valoración radiológica convencional, basada en el sistema de gradación propuesto por Outerbridge10. Sin embargo, hasta la fecha no se han presentado estudios sobre la viabilidad del T1 nativo como biomarcador de la degeneración del cartílago. Esta deficiencia probablemente se deba a la excesiva duración de las secuencias RM tradicionales necesarias para su cálculo, del tipo inversión-recuperación con varios tiempos de inversión del orden de segundos, lo cual impedía su utilización en estudios con una población amplia de sujetos. Afortunadamente, las mejoras en los equipos y las secuencias de RM han permitido aplicar técnicas de adquisición mucho más rápidas basadas en eco de gradiente con destrucción de la magnetización residual. Esta ventaja, unida al conocimiento de las bases físicas asociadas y al diseño de la metodología adecuada9, ha permitido el cálculo de mapas de T1 en tiempos del orden de minutos.
La posibilidad de calcular los valores regionales de T1 del cartílago brinda la oportunidad de obtener un mayor conocimiento sobre la composición molecular de los tejidos, así como de aplicar nuevas metodologías para obtener biomarcadores adicionales (como poder convertir valores de intensidad de señal a valores de concentración de contraste para el cálculo de parámetros farmacocinéticos que caracterizan la microvascularización de los tejidos16). Otro valor añadido de conocer los valores del T1 de los tejidos es que permiten optimizar la adquisición de secuencias RM en base a dichos tiempos.
En este trabajo se han estudiado solamente 15 pacientes con diferentes condiciones de degeneración del cartílago patelar. Esta muestra es relativamente baja para una enfermedad de tan alta prevalencia, por lo que trabajos futuros deberían incluir grupos de estudio mayores. Además, la clasificación de los pacientes en cada uno de los grupos se ha establecido solamente a partir de criterios convencionales, tanto clínicos como de imagen, ya que a ninguno se le había realizado una exploración por artroscopia, considerada como la técnica estándar para la valoración clínica de la degeneración del cartílago articular.
La representación de los mapas de T1 como imágenes paramétricas coloreadas (fig. 4) permite estudiar el cartílago de manera regional y realizar una caracterización mucho más exacta y localizada de la degeneración. En este estudio ha quedado demostrado que la utilización de los estadísticos tradicionales (media y desviación típica) no es adecuada para caracterizar el cartílago a nivel global. En estos casos el efecto de la media enmascara cualquier variación en los valores que indique algún grado mayor de degeneración del cartílago. Se sugiere por tanto la utilización de estos mapas paramétricos, que permiten evaluar el cartílago a nivel global y localizar posibles áreas de afectación severa de una forma rápida.
En conclusión, en este estudio preliminar se ha demostrado que el tiempo de relajación longitudinal T1 del cartílago permite discriminar entre cartílagos normales o con degeneración inicial y cartílagos con degeneración avanzada. Los resultados obtenidos deben ser validados en estudios futuros incluyendo un mayor número de pacientes.
FinanciaciónEste trabajo ha sido financiado parcialmente por una beca de la Sociedad Española de Radiología (ref. 02-MVM-INVESTIGACION-SERAM-2007).
AutoríaLos autores Roberto Sanz Requena y Luis Martí Bonmatí han diseñado el estudio y redactado el presente trabajo; Vicente Hervás y María Vega han contribuido en el análisis clínico y en el diagnóstico mediante imágenes; José Miguel Carot ha contribuido en la realización del apartado de estadística; Ángel Alberich Bayarri y Gracián García Martí han contribuido con sus revisiones críticas. Todos los autores han leído y aprueban la versión final del artículo.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores agradecen al Dr. Javier Sánchez González (Philips Cuidados de la Salud) su valiosa ayuda para el diseño de las secuencias de RM.