La interpretación de la imagen médica es una de las principales tareas que realiza el radiólogo. Conseguir que los ordenadores sean capaces de realizar este tipo de tareas cognitivas ha sido, durante años, un reto y a la vez un objetivo en el campo de la visión artificial. Gracias a los avances tecnológicos estamos ahora más cerca que nunca de conseguirlo y los radiólogos debemos involucrarnos en ello para garantizar que el paciente siga siendo el centro de la práctica médica.
Este artículo explica de forma clara los conceptos teóricos más importantes de esta área y los principales problemas o retos actuales; además, aporta información práctica en relación con el desarrollo de un proyecto de inteligencia artificial en un servicio de Radiología.
The interpretation of medical imaging tests is one of the main tasks that radiologists do. For years, it has been a challenge to teach computers to do this kind of cognitive task; the main objective of the field of computer vision is to overcome this challenge. Thanks to technological advances, we are now closer than ever to achieving this goal, and radiologists need to become involved in this effort to guarantee that the patient remains at the center of medical practice.
This article clearly explains the most important theoretical concepts in this area and the main problems or challenges at the present time; moreover, it provides practical information about the development of an artificial intelligence project in a radiology department.
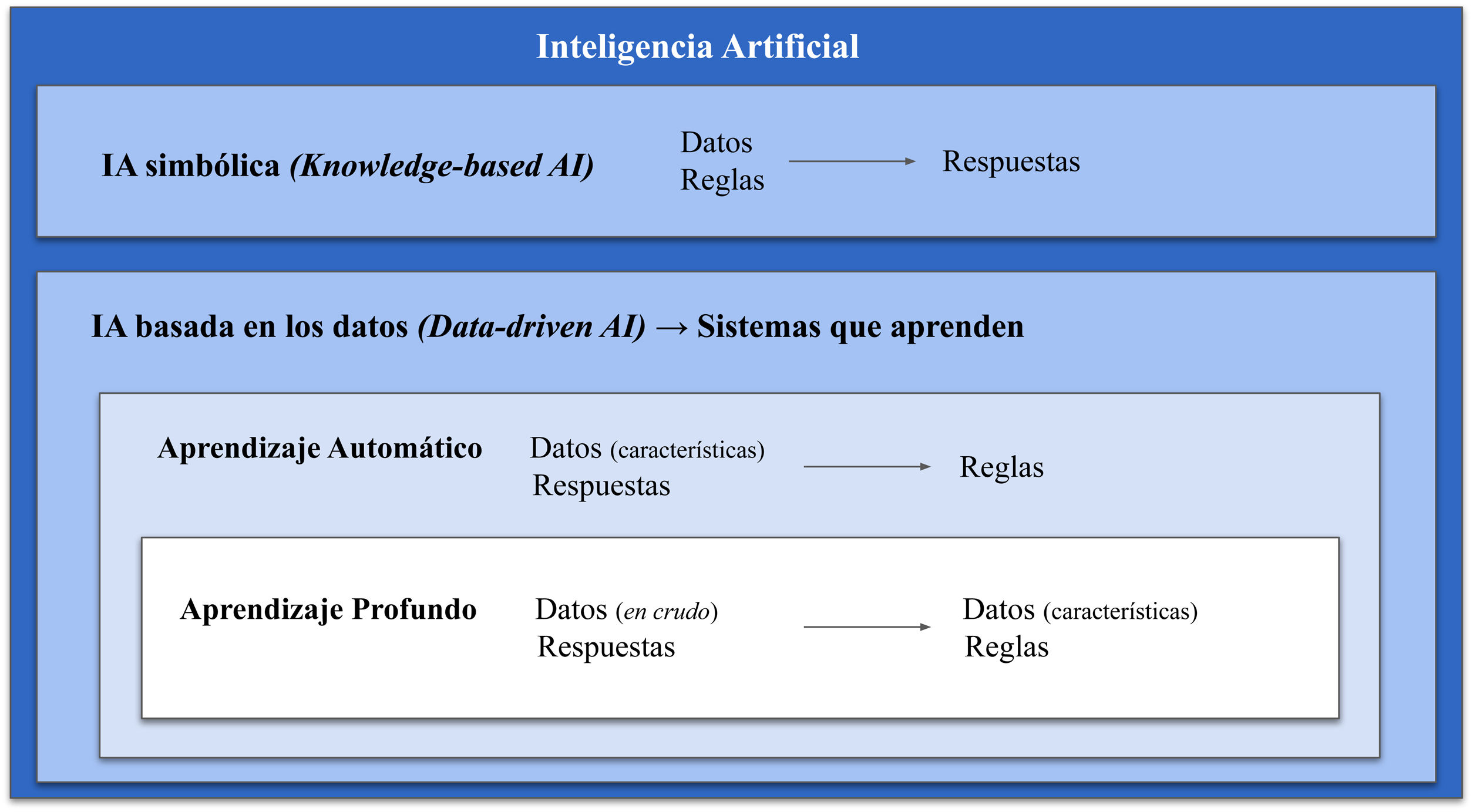
La inteligencia artificial (IA) se define como la capacidad de las máquinas de realizar tareasintelectuales habitualmente realizadas por humanos1. Este término se utiliza como concepto general que engloba tanto el aprendizaje automático (AA) como el aprendizaje profundo (AP). Ambos conceptos pertenecen a un subcampo de la IA que se caracteriza por crear sistemas que son capaces de aprender, es decir, capaces de generar sus propias reglas sirviéndose únicamente de los datos (Data-driven Artificial Intelligence)2.
Algunos autores diferencian entre el AA y el AP basándose en que, en el primero, existe intervención humana en el entrenamiento del algoritmo a través de la manipulación de los datos (extracción y selección de las características más importantes), mientras que en el AP la intervención humana es mínima dado que no existe este paso previo2. Sin embargo, un enfoque más correcto es considerar el AP como una evolución del AA: los sistemas de AP son sistemas de AA, pero más profundos (y de ahí viene su nombre), es decir, constan de muchas más capas y, precisamente, son estas capas extra las que les confieren la capacidad de extraer las características más relevantes de los datos por sí solos (fig. 1)2. En este campo, el concepto de característica se refiere a aquellas variables o propiedades de los datos que son mensurables como, por ejemplo, el valor del píxel o la edad del paciente. Las características más relevantes serán aquellas que ayuden en la resolución del problema que se plantea. Otro concepto importante es el modelo de aprendizaje del sistema de AA o AP, ya que se distinguen 3 tipos (fig. 2)3.
Esquema que engloba los diferentes tipos de inteligencia artificial (IA). Se distinguen 2 campos principales: la IA simbólica, formada por sistemas que necesitan la programación previa de unas reglas, y la IA basada en datos, formada por sistemas que aprenden, dentro de la cual se encuentra el aprendizaje automático, con algoritmos a los que hay que ofrecerles los datos ya depurados y, dentro de este, el aprendizaje profundo con sistemas que, gracias a la mayor cantidad de capas, son capaces de hacer esto ellos mismos.
AI: artificial intelligence.
La IA y, especialmente el AP, han protagonizado innumerables artículos en los últimos años, muchos de ellos relacionados con la Radiología. Sin embargo, estos conceptos no son tan novedosos como se cree. En realidad, la IA surgió a mediados de los años 504 y, a lo largo de su historia, ha presentado momentos de estancamiento (los llamados inviernos de la IA) y momentos de repunte. Actualmente, estamos viviendo uno de esos momentos de repunte sin precedentes, principalmente gracias al desarrollo de la tecnología necesaria para su funcionamiento óptimo como, por ejemplo, las unidades de procesamiento gráfico. En el ámbito médico, la Radiología es una de las especialidades que más se está viendo revolucionada por estos nuevos sistemas5.
Principales hitos en el desarrollo de la inteligencia artificialDesde finales de los años 50, una serie de sucesos relacionados con la IA tuvieron gran impacto mediático.
En 1970-1976, el teorema de los 4 colores, problema matemático no resuelto, consigue probarse gracias a la ayuda de un ordenador, convirtiéndose así en el primer ejemplo de la inclusión de los ordenadores en la resolución de problemas humanos6. Es en esta misma década cuando nace el término inteligencia aumentada para expresar el uso de los ordenadores dedicados a ensalzar la cognición humana7.
Posteriormente, en 1997, el ordenador Deep Blue de IBM consigue vencer en un torneo de ajedrez al campeón del mundo, Garry Kasparov. Deep Blue poseía información de miles de partidas previas y era capaz de analizar todas las posibles situaciones de los siguientes6–8 movimientos: se trataba de IA simbólica(knowledge-based artificial intelligence) (fig. 1), puesto que estos sistemas no aprendían nada, sino que simplemente aplicaban las reglas del juego programadas por humanos, con la ventaja que tienen los ordenadores de poder procesar muchos datos en poco tiempo8.
En 2015, el sistema AlphaGo, desarrollado por Google DeepMind, se convierte en el primer sistema en vencer a uno de los mejores jugadores de go, un juego con reglas sencillas, pero más complejo que el ajedrez en el aspecto estratégico9. Este sistema se basaba ya en técnicas de AA implementadas a través de redes neuronales de AP, puesto que el algoritmo era capaz de inferir o aprender él solo las reglas del juego con base en los datos de partidas previas.
Acercándonos al momento actual, en 2017, se presenta AlphaZero que, a diferencia de AlphaGo, es capaz de aprender enfrentándose a sí mismo, es decir, se basa en aprendizaje por refuerzo y no se le proporcionan previamente datos de partidas anteriores, evitando, por tanto, cualquier intervención humana. Con tan solo unas pocas horas de entrenamiento autónomo, este algoritmo fue capaz de ganar al go a otros programas y versiones previas10.
En la actualidad, son las redes neuronales profundas, generalmente entrenadas mediante aprendizaje supervisado, los sistemas con mayor éxito en el ámbito médico y científico11. Estas redes se engloban dentro del AP dado que aprenden directamente de los datos y sin necesidad de estar estos previamente seleccionados por los humanos. El desarrollo de estas técnicas ha supuesto un cambio de paradigma en este campo y, sobre todo, en el análisis de imágenes y es por ello que son el foco de este artículo.
Las redes neuronalesLas redes neuronales son modelos de predicción, es decir, dados unos datos previos, son capaces de producir una predicción al enfrentarse a datos nuevos. Otros modelos de predicción más conocidos son la regresión lineal simple, la múltiple o la regresión logística. Las redes neuronales obtienen mejores resultados que los anteriores ante problemas más complejos. En general, podemos dividir los modelos de predicción en modelos de clasificación y modelos de regresión. Los modelos de clasificación se basan en encontrar una predicción discreta para la variable de entrada como, por ejemplo, predecir la presencia o no de una enfermedad concreta a partir de una imagen. Mientras que los modelos de regresión se utilizan para encontrar predicciones continuas para la variable de entrada como, por ejemplo, predecir el valor del dímero D a partir de unas variables de entrada (a saber, la edad, la presencia de enfermedad oncológica, etc.). A continuación, se explican las bases de las redes neuronales, comenzando con la neurona artificial y terminando con las redes neuronales convolucionales, el tipo de red neuronal que más éxito ha demostrado en visión artificial.
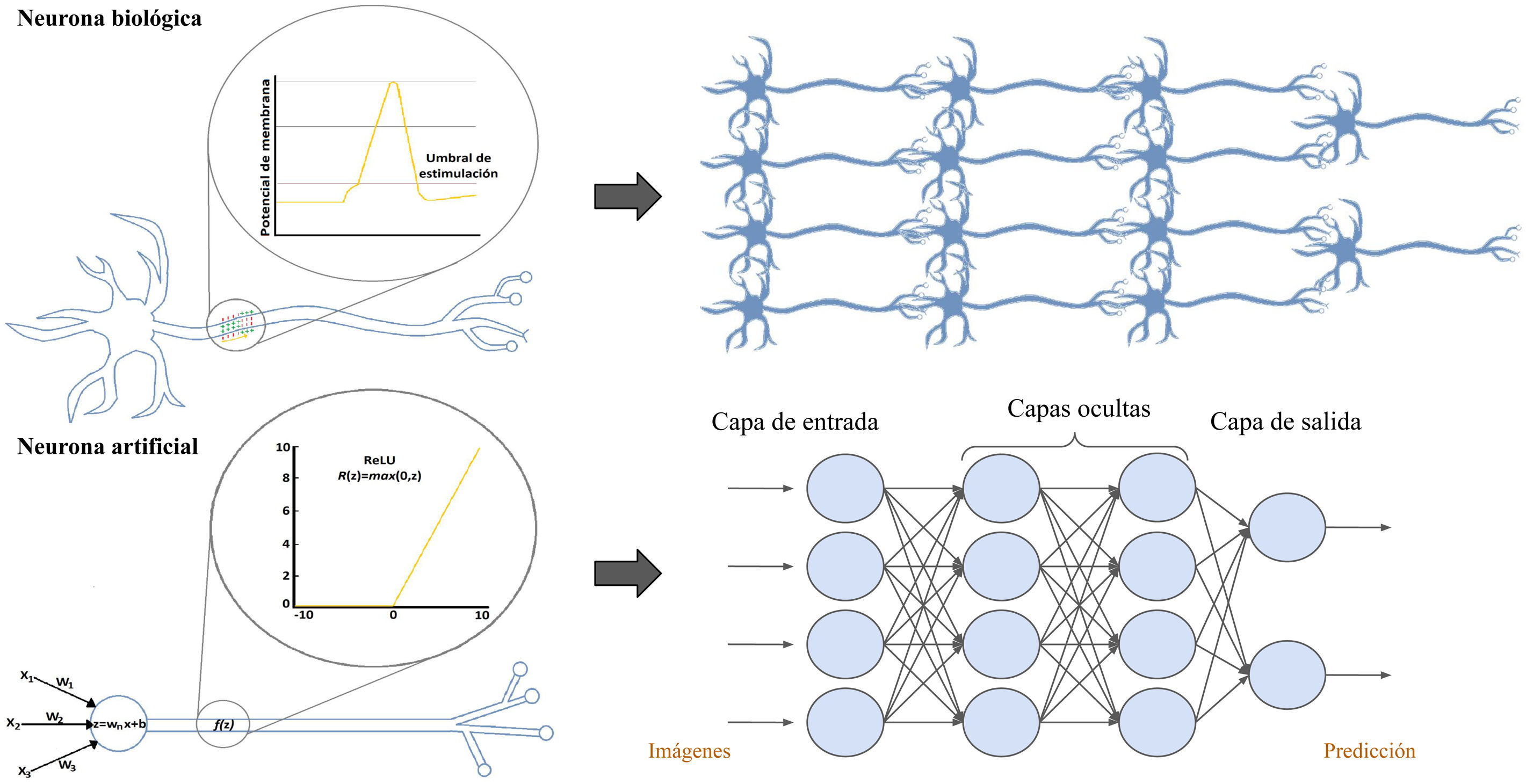
La neurona artificialLas redes neuronales artificiales están compuestas por múltiples neuronas artificiales interconectadas, también llamadas perceptrones simples, que se pueden comparar con las neuronas biológicas (fig. 3). La neurona artificial o perceptrón simple consta de varias vías de entrada, que se asemejan a las dendritas de las neuronas biológicas y que transmiten la información hacia el soma. El soma de la neurona artificial es una función que integra toda la información de las entradas y que, tras aplicar una función de activación, genera una salida3.
Comparativa entre la neurona biológica y la artificial y la red neuronal biológica y la artificial. La red neuronal artificial se divide en 3 partes principales: la capa de entrada es una capa de perceptrones especializados en recibir la información; las capas ocultas son aquellas capas capaces de extraer las características de los datos e ir transformándolos en busca de la mejor representación del problema a resolver, y la capa de salida es una capa preparada para ofrecer la información de salida, como, por ejemplo, en los problemas de clasificación, la clase a la que corresponde la imagen de entrada según la predicción de la red.
La función de activación se podría asemejar al proceso biológico de despolarización de las membranas, que no sigue una función lineal, sino que responde a la ley del todo o nada. Las neuronas biológicas reciben muchos impulsos que no consiguen activarlas o despolarizarlas, hasta que llega uno con suficiente potencia para despolarizarla consiguiendo generar una salida o potencial de acción que viaja a través del axón, el cual transmitirá el impulso a las neuronas contiguas12. A las neuronas artificiales también les llegan una serie de estímulos y, si alguno de ellos logra activar la función de activación, esta dará lugar a una salida. La razón matemática que explica que estas funciones sean indispensables es que son las encargadas de introducir la no linealidad en la neurona, lo que permite poder aproximarse a funciones mucho más complejas y así resolver, por ejemplo, problemas de clasificación que no sean separables por una recta3.
La red neuronal artificial clásicaAl igual que las neuronas biológicas se organizan en capas para formar redes neuronales biológicas, las neuronas artificiales hacen lo mismo formando redes artificiales, por lo tanto, la asociación de perceptrones en capas y la concatenación de sucesivas capas es lo que da lugar a una red neuronal (fig. 3)13.
La arquitectura de las redes neuronales profundas se puede asemejar al modelo biológico de la corteza visual primaria propuesto por D. H. Hubel y T. Wiesel, ambos premios Nobel, en 1959. Según este modelo biológico, la corteza visual primaria está compuesta por 2 tipos de células: las células simples y las células complejas. Las células simples, también llamadas detectoras de bordes, responden positivamente al detectar el borde de un objeto en una determinada orientación, mientras que las células complejas utilizan la contribución de las anteriores para encontrar todos los bordes del objeto. La organización de estas células en capas, de modo jerárquico, hace que los objetos se vayan reconociendo de forma secuencial, empezando por las características más simples para acabar por las más complejas. Las redes neuronales mantienen una arquitectura similar: la primera capa de la red se encarga de extraer características groseras de la imagen, como los bordes, el contraste de color, etc. Para, posteriormente, pasar la información por sucesivas capas que van extrayendo detalles más finos14.
El proceso de aprendizaje o entrenamientoAntes de comenzar el entrenamiento de la red, se deben seleccionar unas variables llamadas hiperparámetros. Los hiperparámetros son variables que determinan la estructura de la red y de cómo se entrena, por lo tanto, se definen antes de comenzar el entrenamiento y se van ajustando en función de los resultados del mismo. El tipo de función de activación utilizada y el número de capas ocultas del algoritmo son ejemplos de hiperparámetros2.
El proceso de aprendizaje o entrenamiento de una red neuronal consiste en ajustar unos parámetros llamados pesos. Los pesos se entienden como la intensidad de las conexiones existentes entre las neuronas artificiales. En la neurona biológica, los pesos se podrían asemejar a la intensidad de las sinapsis entre neuronas. Así, ajustando las sinapsis conseguiremos un resultado final óptimo15.
Cada vez que una imagen entra en la red, se activan secuencialmente todas las neuronas de todas las capas de la red, es decir, se van generando unos pesos para cada conexión neuronal, a lo que se le llama propagación hacia delante. Finalmente, al llegar a la última capa, gracias a este proceso, se genera una predicción para esa imagen. Pero, dado que todavía estamos entrenando la red, ¿cómo podemos saber si esa predicción es correcta o errónea? ¿Cómo podemos hacer que la red vaya mejorando con cada imagen de entrenamiento?
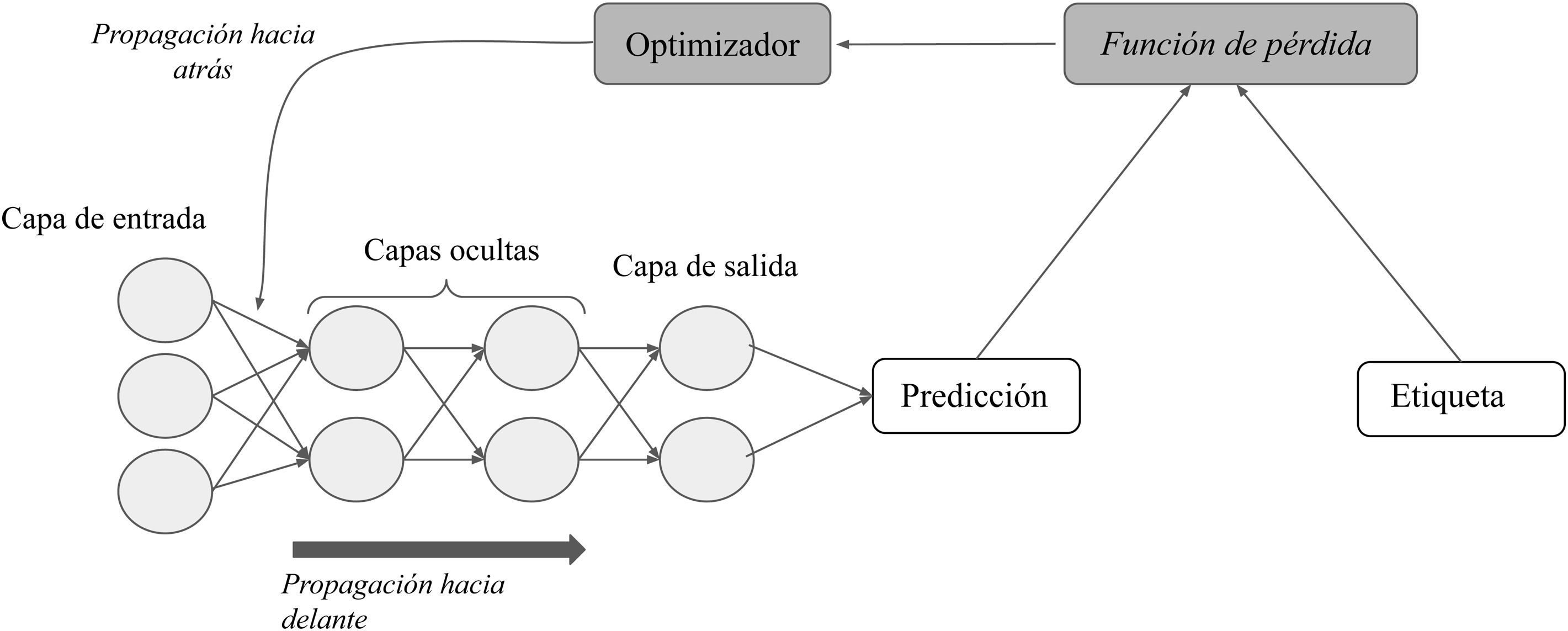
En un modelo de aprendizaje supervisado, la red neuronal comienza utilizando unos pesos aleatorios y aprende cuando esos pesos se van ajustando al comparar los resultados de la red para un ejemplo con el resultado de referencia o etiqueta11. Para ello necesita, por un lado, las referencias o etiquetas; por otro, una función que mida el error generado (función de pérdida); un algoritmo de optimización que calcule la magnitud y la dirección en la que deben modificarse los pesos con el objetivo de minimizar ese error (descenso de gradiente), y, por último, otra función capaz de trasladar este ajuste de forma retrógrada a través de la red y que modifique los pesos de cada neurona en función de cuánto de responsable sea esa neurona del resultado final (propagación hacia atrás) (fig. 4)15.
El proceso de aprendizaje supervisado (el entrenamiento). Mediante la función depérdida se cuantifica la diferencia entre la predicción de la red y la etiqueta de cada entrada; a continuación, a través de la propagación hacia atrás de este error y del algoritmo de optimización, se ajustan los pesos de las distintas neuronas hasta que correspondan con un mínimo en la función de pérdida.
Este proceso de ajuste de pesos es lo que se llama aprendizaje de la red y se da principalmente en las capas ocultas. A lo largo de la red, y mediante el ajuste de los pesos, las capas ocultas van formando representaciones de los datos cada vez más complejas, pero que se ajustan cada vez más al problema (fig. 5)2. Así, la base tanto del AA como del AP es transformar los datos sucesivamente hasta encontrar la mejor representación que permita resolver el problema. El término profundo no hace referencia a un entendimiento más profundo de los datos, sino a aprender capas sucesivas de representaciones cada vez más significativas y el número de estas capas es lo que se conoce como la profundidad del modelo3.
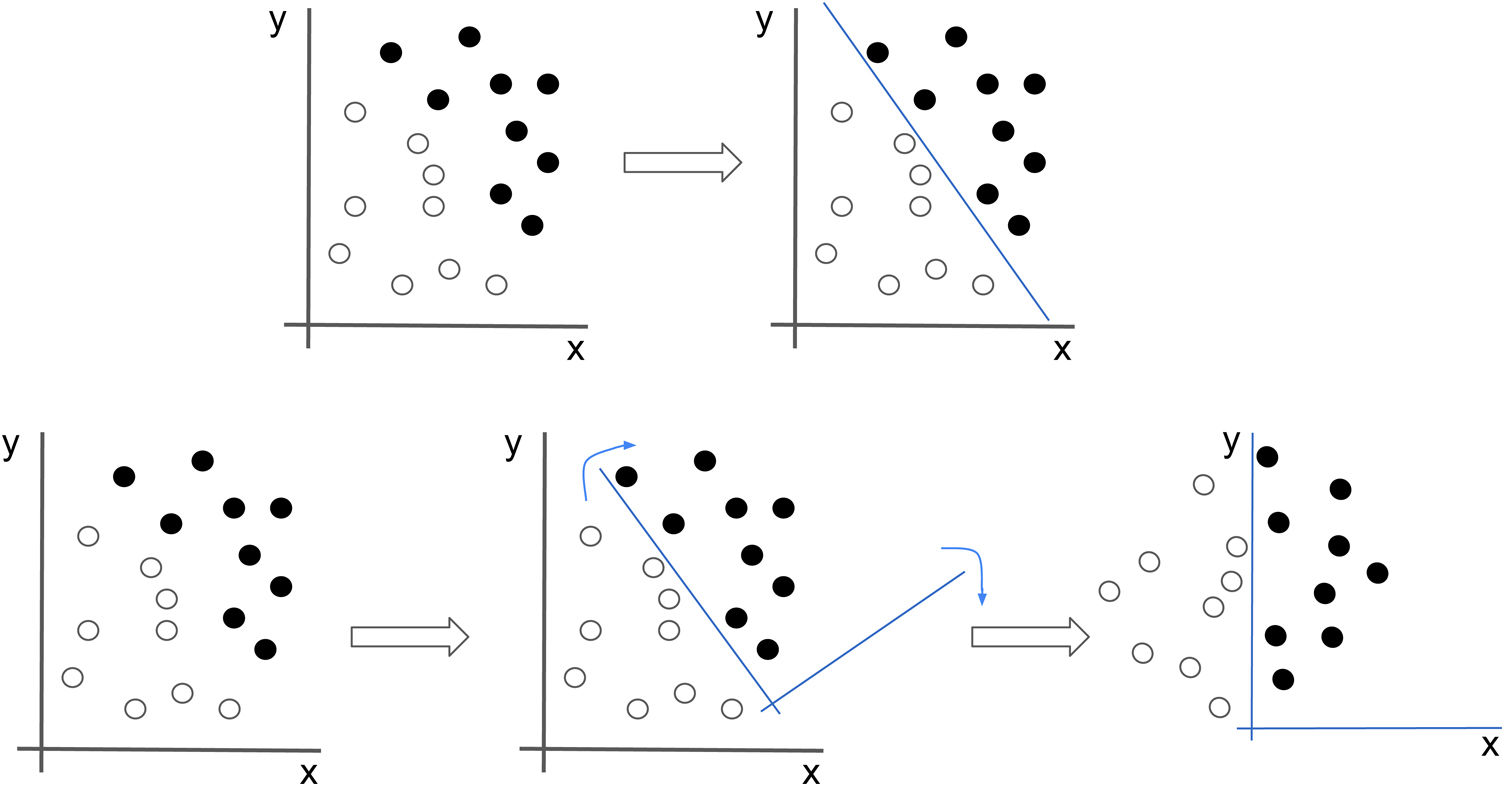
Las redes neuronales buscan la mejor representación de los datos que les permita resolver el problema. En este ejemplo, si tratamos de clasificar los puntos en negros y blancos tendríamos que trazar una recta que correspondería con una ecuación no intuitiva. Sin embargo, si aplicamos una transformación en los datos que hagan que la imagen rote, el problema, de repente, se vuelve mucho más sencillo (x=0).
Para entrenar una red neuronal, es necesario disponer de al menos 2 subconjuntos de datos: un grupo para el entrenamiento propiamente dicho, con el que el modelo ajustará sus pesos de acuerdo con un mínimo en la función de pérdida; y otro grupo de datos con el que evaluar el rendimiento de este, denominado el conjunto de validación. Así, se van realizando iteraciones (llamadas épocas) sobre estos grupos de datos y el modelo irá obteniendo cada vez mejores resultados que se irán observando en la evaluación del rendimiento del modelo con el conjunto de validación en cada iteración. Si el rendimiento del modelo no es bueno, el experto puede realizar cambios en los hiperparámetros.
Finalmente, una vez concluido el entrenamiento, es decir, una vez ajustados tantos los pesos como los hiperparámetros, se prueba el modelo con datos nuevos (conjunto de datos denominado test) para evaluar su rendimiento real. Es decir, se expone al modelo a datos nuevos, por ejemplo, imágenes nuevas y no etiquetadas, y se obtiene una predicción; por ejemplo, la clase a la que pertenece esa imagen en un problema de clasificación. Los datos del test no deben nunca utilizarse para modificar pesos o hiperparámetros del modelo.
Las redes neuronales convolucionalesCon la extensión del uso de las redes neuronales clásicas, empezaron a surgir problemas que impulsaron el desarrollo de formas más complejas de redes neuronales. En el caso de la imagen y el reconocimiento de objetos, el principal problema era que, generalmente, el mismo objeto podía tener formas y posiciones diferentes, lo que reducía el rendimiento de las redes. Así, surgieron las redes neuronales convolucionales (RNC), las más usadas para imagen médica15.
Las redes neuronales clásicas mencionadas anteriormente están compuestas por capas totalmente conectadas. Esto significa que todas las neuronas de una capa están conectadas con las de la siguiente capa y, por lo tanto, la imagen se interpreta en su totalidad, tomando como entrada el valor de todos los píxeles y realizando operaciones que incluyen toda la información de la imagen. Así, si por ejemplo la red tiene como objetivo aprender a identificar coches y en una de las imágenes aparece un coche en la esquina superior izquierda y en otra en la esquina inferior derecha, la red tendrá que aprender unos pesos diferentes para cada una de esas imágenes, dado que la diferente localización del mismo objeto hace que sean interpretados como objetos diferentes, cada uno con sus pesos y representaciones específicas. Esto hace que estas redes no funcionen bien ni sean eficientes en tareas como la interpretación de la imagen o la identificación de objetos. Por el contrario, las RNC disponen de unas matrices denominadas filtros capaces de analizar la composición de la imagen y que conceden a la red la capacidad de identificar el coche independientemente de su localización, lo que las hace mucho más eficientes que las redes neuronales clásicas para la interpretación de la imagen15.
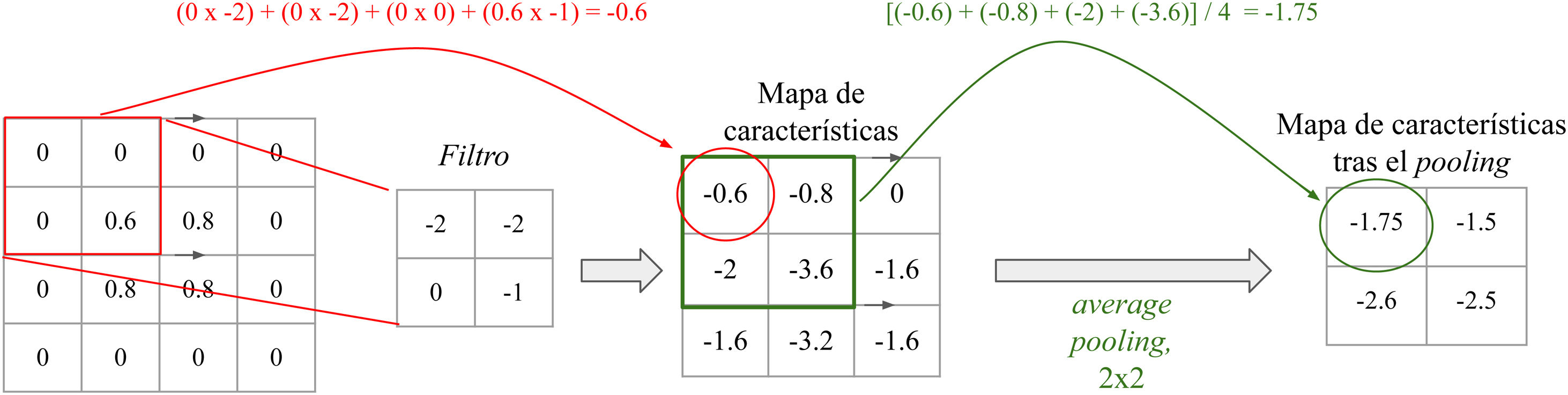
Cada capa convolucional de una RNC puede constar de varios filtros. Estos filtros son matrices numéricas que van recorriendo la imagen realizando operaciones de convolución sobre grupos de píxeles, dando lugar a mapas de características. Cada filtro representa una característica (fig. 6). Así, capa tras capa, se van extrayendo características cada vez más complejas y se van formando representaciones cada vez más groseras de los datos de entrada. Es habitual que las capas convolucionales se sigan de capas de pooling, capas que reducen la dimensionalidad de los mapas y así el coste computacional (fig. 7). Las representaciones de la última capa de la parte convolucional son transformadas a un vector final a través de una o más capas completamente conectadas y, finalmente, a una predicción. A esta segunda parte de la red se le denomina comúnmente el clasificador15.
Filtros y pooling: El filtro es una matriz que va recorriendo la imagen y va realizando una operación de multiplicación elemento a elemento y suma para obtener un valor (convolución). Obtenemos valores altos cuando aplicamos el filtro sobre una característica similar al mismo y valores bajos cuando lo aplicamos sobre una distinta. En un filtro que detecte bordes verticales, si obtenemos valores altos significa que se ha detectado un borde vertical. ¿Recuerda esto a las células de la corteza visual primaria? Por otro lado, la capa de pooling, de tipo average y de tamaño 2 × 2 en este ejemplo, realiza la media de los valores del rango, reduciendo la dimensionalidad del mapa de características.
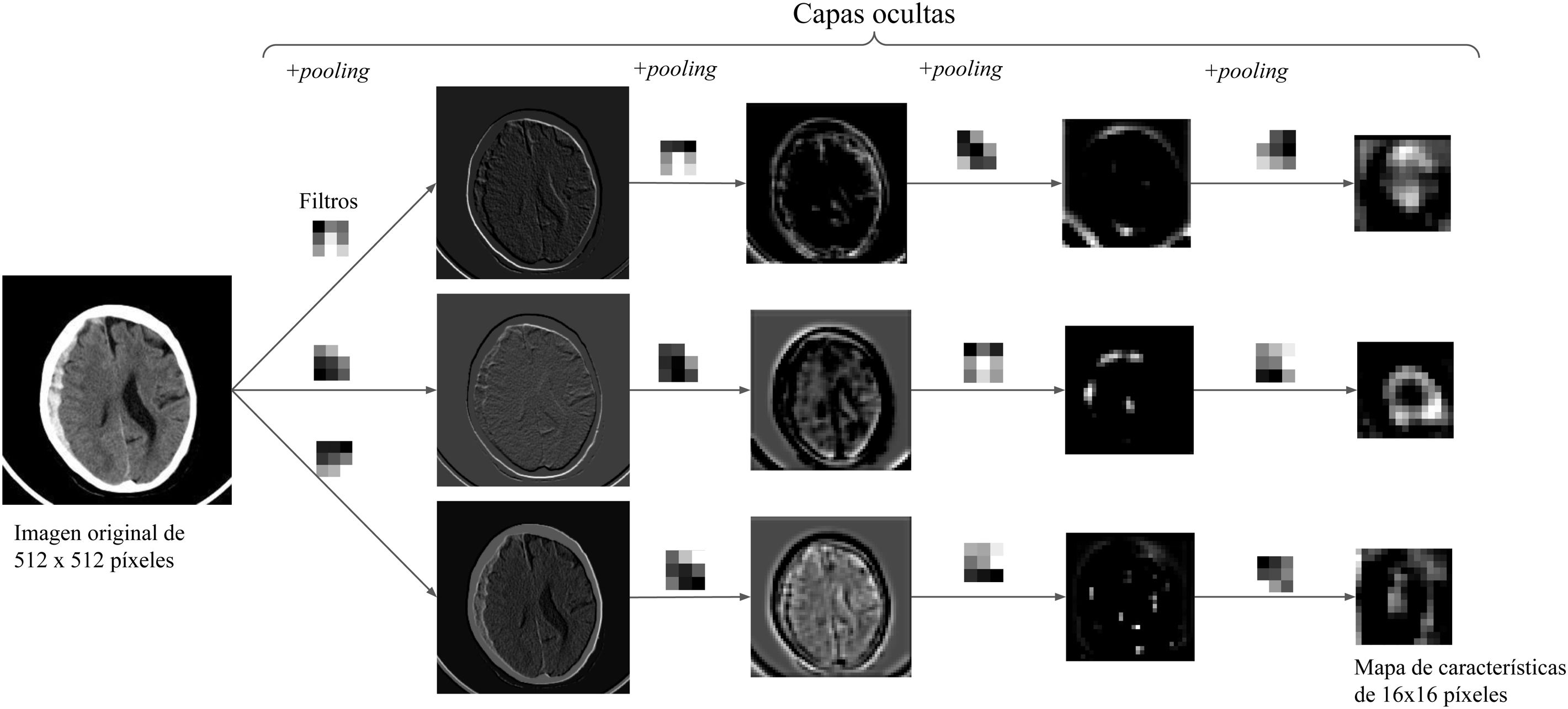
Ejemplo con una imagen: se muestra un ejemplo en el que la imagen de una TC de cráneo va pasando por sucesivas capas que contienen 3 filtros de 3 × 3 y la función de pooling, lo que da lugar a diferentes mapas de características cada vez de menor resolución espacial. A mayor profundidad en la red, los mapas son cada vez más groseros y de menor tamaño, transmitiéndose hacia delante solo la información más relevante.
En conclusión, las RNC, gracias a los filtros, aprenden patrones locales y, por lo tanto, son capaces de reconocer dicho patrón independientemente de que se realice una traslación, mientras que las redes neuronales clásicas aprenden patrones globales y no son capaces de abstraerse de la localización, orientación o forma del objeto en la imagen. Así pues, las RNC han demostrado ser las más adecuadas para trabajar con imagen médica, siendo capaces de realizar tareas complejas como la clasificación de imágenes2.
Principales problemas de las redes neuronales y algunas solucionesEntre los obstáculos a los que nos podemos enfrentar durante el entrenamiento de una red neuronal y que somos capaces de detectar gracias al conjunto de validación destacan el sobreajuste y el subajuste. El sobreajuste ocurre cuando el modelo se especializa tanto en los datos de entrenamiento que no es capaz de generalizar y, por lo tanto, al enfrentarse a datos nuevos no obtiene buenos resultados. El subajuste, por su parte, se refiere a un modelo que, debido a su insuficiente especialización o excesiva simplicidad, no es capaz de obtener buenos resultados ni siquiera con los datos de entrenamiento. En ninguno de los casos el modelo habrá encontrado aquellas características importantes que le permitirían resolver el problema de forma general o con datos nuevos, es decir, en ninguno de los casos el modelo habrá aprendido a generalizar. En conclusión, las redes neuronales deben aprender transformaciones y no ejemplos concretos3.
El sobreajuste está directamente relacionado con uno de los obstáculos más importantes con los que nos encontramos a la hora de desarrollar sistemas relacionados con la imagen médica: la escasez de datos etiquetados. Una de las razones de este problema es que la creación de amplias bases de datos de imágenes debidamente etiquetadas requiere mucho tiempo y esfuerzo por parte del experto, en nuestro caso, del radiólogo. Además, dado que no siempre concuerdan el diagnóstico por imagen con el histológico, se debe tener muy en cuenta qué prueba es la que se debe considerar diagnóstica de la enfermedad a estudio para así crear la etiqueta. La imagen se considera diagnóstica en algunas entidades como las fracturas. Sin embargo, la mayoría de las enfermedades necesitan de otras pruebas para realizar el diagnóstico definitivo, ya sea la histología o los hallazgos clínico-analíticos, como, por ejemplo, el cáncer, en el que en la mayoría de los casos es necesario el resultado histológico para realizar su diagnóstico16. A su vez, esta falta de imágenes etiquetadas muchas veces se ve acentuada debido al complicado marco ético y legal en la transferencia de datos de carácter médico17.
No obstante, existen varios proyectos en marcha con el objetivo de crear amplias bases de datos con imágenes médicas etiquetadas, como el Cancer Imaging Archive18, o empresas como Savana19, que ofrecen soluciones de IA para la explotación de los datos médicos en formato de texto libre. También se plantean estrategias como el informe interactivo, en el que el radiólogo puede crear vínculos (hipertexto) a otros textos o etiquetas en el propio informe16; el informe estructurado; o incluso proyectos de colaboración internacionales que involucran a muchos radiólogos, como la preparación del conjunto de datos para el RSNA 2019 Brain CTHemorrahge Challenge20; plataformas como OpenNeuro21, que facilita el acceso a bases de datos tanto de imágenes cerebrales como de electroencefalogramas; la red europea de imagen de tumores cerebrales ENBIT22, o consorcios como ENIGMA23, que une a investigadores en genómica e imagen cerebral.
Otra de las soluciones que más éxito está demostrando, principalmente en el ámbito médico, es la transferencia de aprendizaje (transfer learning). Esta técnica consiste en poder trasladar a nuestro modelo, desde una red ya entrenada, tanto la arquitectura como los pesos de las primeras capas. Se elige trasladar los pesos de las primeras capas debido al aprendizaje jerárquico de las redes neuronales, ya comentado anteriormente, según el cual son las primeras capas las que se encargan de extraer características más simples, es decir, menos específicas del problema a resolver y que se asume que son comunes para ambos conjuntos de imágenes. En este sentido, se puede o bien entrenar únicamente la última parte de la red, el clasificador, y mantener la parte convolucional congelada, o bien entrenar también un número variable de capas de la parte convolucional, a lo que se denomina descongelar capas. En cualquier caso, este nuevo modelo inicia su proceso de aprendizaje con ventaja, al tener que ajustar los pesos desde una posición favorable en lugar de partir de valores aleatorios. Es por ello que estos modelos pueden obtener buenos resultados con menos datos que aquellos modelos completamente nuevos24.
Y, por último, uno de los problemas más importante de estos sistemas y, al mismo tiempo, más difícil de solucionar es su escasa transparencia. Ya que, aunque se pueda explicar el proceso matemático mediante el cual se construyen los algoritmos, no se conoce claramente cómo llegan a sus conclusiones. Es por ello que, todavía a día de hoy, las RNC son consideradas cajas negras25 y mejorar su explicabilidad es motivo de estudio. Una de las soluciones que está siendo muy utilizada son las Grad-CAM26, sistemas de localización mediante gradiente de las áreas de la imagen en las que el algoritmo se fija para tomar la decisión final. Al mismo tiempo, esta escasa explicabilidad y transparencia dificultan el desarrollo de un marco ético-legal para la regulación de la implementación de estos sistemas en la práctica médica habitual27.
Desarrollo de sistemas de inteligencia artificial en los servicios de RadiologíaDentro de un servicio de Radiodiagnóstico, los sistemas de IA pueden aplicarse en múltiples áreas, como en tareas relacionadas con la citación de los pacientes28, la selección del mejor protocolo de imagen y dosis de radiación29, la colocación del paciente en el equipo30, el posprocesado de la imagen (reconstrucciones, mejora de la calidad de la imagen, etc.)31 y, por supuesto, y como ya hemos explicado, en la interpretación de la imagen32. En este último campo, no solo se están desarrollando sistemas de IA que realicen un diagnóstico, sino también sistemas capaces de segmentar órganos y detectar lesiones, así como monitorizarlas33. Y, yendo un poco más allá, se están estudiando sistemas que predigan, por ejemplo, la supervivencia estimada o la gravedad de la enfermedad en función del tipo de lesión u otros datos clínico-analíticos del paciente32–34. La suma de datos clínicos del paciente a los datos propios de la imagen puede aportar mejoras sustanciales en los resultados de estos modelos de IA, lo que ha llevado a crear redes que combinan métodos de AP y de AA, las redes híbridas34.
Cuando se plantea el desarrollo de un sistema de IA relacionado con la interpretación de imagen médica, lo primero es obtener la aprobación del comité de ética del hospital. Generalmente, para estudios retrospectivos en los que la obtención del consentimiento informado no es factible y los riesgos de fuga de datos médicos son mínimos, el consentimiento informado del paciente suele ser prescindible35.
Posteriormente, se procede a la selección de aquellos pacientes a incluir en el estudio y a la recolección de sus imágenes. Este es uno de los pasos más importantes en el desarrollo de estos sistemas que, como ya hemos mencionado, dependen, en gran medida, de la cantidad y la calidad de los datos (data-driven systems). A día de hoy, debido a que el etiquetado de las imágenes radiológicas no está extendido y que los sistemas de información radiológicos no están preparados para este tipo de búsquedas, la obtención de imágenes de una enfermedad concreta no es una tarea fácil35. Aquí, el procesamiento del lenguaje natural proporciona técnicas capaces de obtener datos estructurados de los informes radiológicos, con resultados muy prometedores36. Asimismo, es de gran relevancia la desidentificación de las imágenes que, en el caso del formato DICOM, puede ser compleja. Una vez obtenidos los datos, estos deben ser preprocesados en función del tipo de red neuronal que se vaya a entrenar16.
Cuando la base de datos ya está preparada, esta se divide en los 3 subconjuntos mencionados anteriormente: el de entrenamiento, el de validación y el de test, con un porcentaje aproximado del 80, el 10 y el 10%, respectivamente16.
Ofrecemos un repositorio en GitHub donde ponemos a su disposición un ejemplo de una neurona que resuelve un problema de regresión y otro ejemplo de una red neuronal que resuelve un problema de clasificación, en el siguiente enlace: https://github.com/deepMedicalImaging/RedNeuronalArtificial.git.
Autoría- 1.
Responsable de la integridad del estudio: AP, PM, PS, LL y DR.
- 2.
Concepción del estudio: AP, PM, PS, LL y DR.
- 3.
Diseño del estudio: AP.
- 4.
Obtención de los datos: no procede.
- 5.
Análisis e interpretación de los datos: no procede.
- 6.
Tratamiento estadístico: no procede.
- 7.
Búsqueda bibliográfica: AP.
- 8.
Redacción del trabajo: AP.
- 9.
Revisión crítica del manuscrito con aportaciones intelectualmente relevantes: AP, PM, PS, LL y DR.
- 10.
Aprobación de la versión final: AP, PM, PS, LL y DR.
Los autores declaran no tener ningún conflicto de interés.