Esencialmente, todos los niños se infectan con el herpesvirus 6 humano (HHV6) en los tres primeros años de vida, presentando la mayoría muestras de infección a los 12 meses de edad1. Se cree que el HHV6 hallado en la saliva de los contactos asintomáticos es la principal vía de transmisión a los niños pequeños2,3. Más recientemente, la infección HHV6 congénita, detectada por ADN de HHV6 en sangre de cordón, se ha identificado también como medio de transmisión, y ocurre en el 1% de los niños, una tasa similar a la de la infección por citomegalovirus (CMV) congénita4-11. Sin embargo, al contrario que con el CMV, se ha demostrado que el genoma de HHV6 se integra en los cromosomas de algunos individuos, un fenómeno único del HHV6 entre los herpesvirus humanos12-20. No está claro cuán a menudo el HHV6 cromosómicamente integrado (CI-HHV6) está presente en individuos normales, pero se ha estimado que ocurre en el 0,2-0,8% de las poblaciones de Japón y Reino Unido17,18,20. Estos individuos se han identificado principalmente por cargas virales elevadas (µ 1 copia de ADN de HHV6 genómico por leucocito) en sus células mononucleares de sangre periférica (PBMC), que eran persistentes y marcadamente más altas que los niveles presentes en la infección latente habitual no asociada con virus integrado cromosómicamente17-22 (1 copia genómica por 104-105 leucocitos).
En algunos de estos individuos, el CI-HHV6 ha sido confirmado por FISH o detección de HHV6 en sus folículos capilares14,16-19,23.
Las infecciones por HHV6 congénitas se han considerado generalmente resultado de una infección por HHV6 materna reactivada que ha cruzado la placenta. Sin embargo, el HHV6 integrado cromosómicamente podría ser un modo importante y único de transmisión de infección congénita. El propósito de este estudio fue en primer lugar determinar en una cohorte de niños seguida prospectivamente la aparición de CI-HHV6 como modo de infección congénita identificado por la presencia de cargas virales elevadas en células mononucleares de sangre de cordón (CBMC) y confirmado por detección de HHV6 en sus folículos capilares.
Nosotros intentamos entonces delinear los rasgos virales e inmunológicos asociados con la infección por HHV6 entre los niños infectados congénitamente con CI-HHV6 en comparación con los niños con infección congénita transplacentaria (TPI) y con los niños con infección por HHV6 adquirida posnatalmente (PNI).
En tercer lugar, intentamos determinar la presencia de CI-HHV6 entre los padres de niños con infección por HHV6 congénita.
MÉTODOSDiseño del estudioDe nuestros estudios en marcha con niños con infección por HHV61,4, incluimos desde julio de 2003 hasta el 15 de abril de 2007 a niños (µ 36 semanas de gestación) con y sin infección por HHV6 congénita, identificados por cribado de sangre de cordón en el momento de nacer para detectar la presencia de ADN de HHV6. Por cada niño incluido con infección congénita, se incluyeron un promedio de 5 niños sin infección congénita, que se igualaron por género, raza y nacimiento en 8 semanas, y fueron seguidos hasta la adquisición de infección por HHV6 primaria. Sólo se pidió permiso a las familias para la inclusión de los niños después de la aprobación de sus médicos particulares. Las visitas del estudio al Golisano Children’s Hospital en la Universidad del Rochester Medical Center se pautaron a los 4-6, 12-15, 24-30 y 30-36 meses de edad. Desde que se puso en marcha la inclusión, variaron las edades y número de visitas entre los niños. Los niños fueron examinados y se obtuvieron muestras de todos ellos para ser analizadas según se describe más abajo.
Se utilizó la determinación cuantitativa de ADN de HHV6 en CBMC para identificar entre los niños infectados congénitamente aquellos con cargas virales elevadas características del CI-HHV6 y aquellos con cargas virales más bajas características de TPI17-22. Los grupos se confirmaron por la detección o ausencia de ADN de HHV6 en muestras de folículos capilares. Además, se utilizó la hibridación fluorescente in situ (FISH) para demostrar la integración del genoma de HHV6 en los cromosomas de una muestra representativa de niños infectados congénitamente. Se analizaron entonces los rasgos virales e inmunológicos entre estos dos grupos de niños con infección congénita CI-HHV6 y TPI en comparación con niños controles con infección posnatal (PNI).
Grupos de individuosLos niños con infección por HHV6 congénita eran aquellos con ADN de HHV6 presente en su sangre de cordón. Los niños con infección congénita por CI-HHV6 eran los que presentaban cargas virales elevadas de ADN de HHV6, µ 1 copia genómica/leucocito (equivalente a 1-2 µ 105 copias genómicas de HHV6/µg ADN celular), que han sido utilizadas para identificar a los individuos con CI-HHV6 como se ha publicado previamente17-20,22 y/o aquellos en los que se había detectado ADN de HHV6 en sus muestras de folículos capilares. Los niños con TPI eran aquellos con cargas virales más bajas encontradas en una infección latente no asociada con CI-HHV6, 1 copia genó-mica/104-105 leucocitos21 (equivalente a < 1 copia genómica/µg ADN celular) y sin ADN de HHV6 en sus muestras de folículos capilares. Los niños controles eran aquellos con sangre de cordón negativa para ADN de HHV6 y que adquirieron el HHV6 posnatalmente.
Análisis de laboratorioPreparación de las muestrasA partir de 0,5-3 ml de sangre total recogida en EDTA, se separaron las células mononucleares y se utilizaron para 1) PCR va-riante-específica de ADN; 2) PCR cuantitativa en tiempo real (q PCR); 3) PCR de la transcriptasa inversa (RT-PCR), y 4) análisis FISH. Se purificó ácido nucleico a partir de 0,05 ml de saliva y 0,2 ml de orina utilizando kits de aislamiento de ARN de Promega Corp. (Madison, WI) o Qiagen Inc. (Valencia, Ca) sin tratamiento DNase. A partir de 2-3 folículos de cabello con raíz se aisló el ADN utilizando el microkit de ADN Qiagen QIAmp®.
PCR variante-específica de ADN
Se realizó PCR anidada con tipaje virus-específico con pruebas de oligonucleótidos específicas para HVV6A y HHV6B, tal como se había publicado previamente1,4. La fiabilidad de la prueba detecta µ 10 copias genómicas. Todos los experimentos se llevaron a cabo con controles positivos y negativos, así como con cebo de µ-globulina para confirmar la presencia de material celular y excluir la presencia de inhibidores.
RT-PCR anidada para HVV6
El ensayo de RT-PCR amplifica el gp82-105 del ARNm del HHV6 y se realizó como se había publicado previamente, excepto en que la secuencia de la prueba se modificó ligeramente basándose en la información de la secuencia de la variante B del HHV624,25. La secuencia de la prueba en estos experimentos fue 5’-GCTCCCGAAAGCGCCATA-3’. El ensayo para HHV6 detecta < 10 copias de ARNm.
PCR cuantitativa a tiempo real
La qPCR para el gen HHV6 U38 se llevó a cabo tal como se había publicado previamente26, excepto para nuestro cebo, 5’-TGCTTCTGTAACGTGTCTTGGA-3’, que contenía una molécula de adenina menos al final del 3’. Sólo las muestras positivas de ADN de HHV6 por PCR cualitativa se analizaron por PCR cuantitativa. Las muestras se purificaron utilizando un sistema de purificación de ADN genómico Wizard SV (Promega, Madison, WI) y se hicieron por triplicado. Los resultados medios de µ 2 huecos se analizaron y comunicaron como copias genómicas de HHV6/µg de ADN celular21,27,28 (1-2 µ 105 copias genómicas de HHV6/µg de ADN celular = 1 copia genómica de HHV6/leucocito).
Hibridación fluorescente in situ (FISH)
Se prepararon diseminaciones cromosómicas en la metafase de PBMC a partir de líneas celulares linfoblastoides transformadas de EBV (LCLs) utilizando protocolos establecidos29. Las pruebas de ADN fueron biotiniladas con dATP y se llevó a cabo FISH según métodos publicados30. Se observaron las señales de FISH bajo microscopia de fluorescencia y se fotografiaron.
Serología
Los niveles de anticuerpos HVV6 se determinaron por inmunofluorescencia indirecta utilizando un aislado clínico de HHV6 crecido en células HSB-2 que contenían genomas de HHV6A y HHV6B1. Se consideraron positivos los títulos > 3,32 log2 (dilución > 1:10).
Consentimiento informadoEl University of Rochester’s Research Subjects Review Board aprobó este estudio. Todas las familias proporcionaron el consentimiento informado y fueron compensadas por los costes de transporte y otros costes asociados con cada visita.
Análisis estadísticoLas características demográficas se compararon entre los tres grupos de niños (aquellos con CI-HHV6, TPI y PNI) utilizando pruebas de chi cuadrado, pruebas exactas de Fisher o t-tests (por edad), según fueran apropiados. Las pruebas exactas de Fisher se usaron para comparar 1) grupos CI-HHV6 y con infección transplacentaria con respecto a la proporción de individuos con la variante A en contraposición con la variante B detectada en sangre de cordón; 2) grupos CI-HHV6 y con infección posnatal con respecto a la proporción de individuos con la variante A detectada en una muestra de al menos un sitio en cualquier momento postinfección, y 3) madres y padres con CI-HHV6 detectada por ADN de HHV6 en folículos capilares con respecto a la proporción de muestras que contenían la variante A de HHV6. La prueba de McNemar se usó para comparar muestras emparejadas de madres y padres de niños con HHV6 con respecto a la frecuencia relativa de detección de ADN de HHV6.
Se comparó el título medio de anticuerpos HHV6 (log2 transformado) en sangre de cordón entre los grupos CI-HHV6 y con infección posnatal utilizando un t-test. Se utilizó un análisis de un modelo de varianza para llevar a cabo comparaciones emparejadas del título medio de anticuerpos HHV6 en sangre de cordón entre niños con CI-HHV6 derivada de la madre, CI-HHV6 derivada del padre e infección posnatal. Se realizaron comparaciones similares utilizando muestras de PBMC, pero se utilizó un análisis de mediciones repetidas de un modelo de varianza, con una estructura de correlación de simetría de compuestos, para permitir las mediciones repetidas de títulos de anticuerpos en el mismo individuo. Se utilizó una estrategia de análisis similar para comparar la carga viral media (log10 transformado) en muestras de PBMC y saliva entre los grupos de infección por HHV6 transplacentaria y con infección posnatal. Todos los valores comunicados son de dos colas.
RESULTADOSGrupos de estudioDe los 254 niños incluidos, 43 tenían infección por HHV6 congénita. De los 211 niños sin infección congénita, 42 fueron niños controles que adquirieron la infección por HHV6 durante el período de estudio. Ninguno mostró evidencias de infección por HHV6 adquirida hasta 1,5 meses de edad o más.
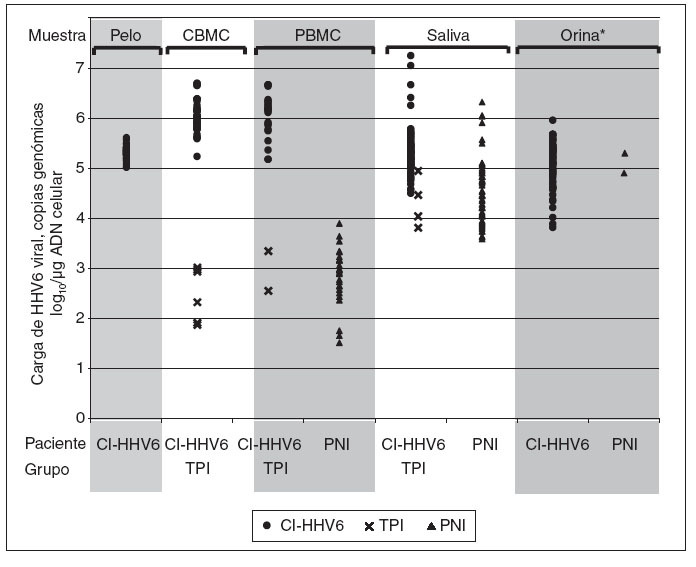
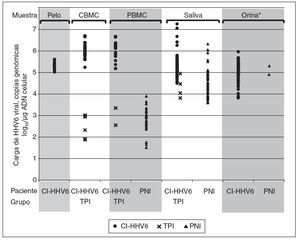
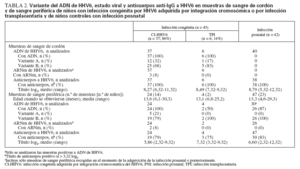
Entre los 43 niños infectados congénitamente, 37 (86%) se identificaron como CI-HHV6 y 6 (14%) como TPI según sus niveles de ADN de HHV6 cuantitativos en CBMC. El promedio de carga viral en CBMC entre los 43 niños infectados congénitamente fue de 5,57 log10 (rango 1,90-6,72) copias genó-micas/µg ADN celular. La distribución de las cargas virales mostró dos grupos separados: 37 (86%) tenían una carga viral media notablemente más alta, 6,06 log10 (rango 5,25-6,72) copias genómicas/µg ADN celular, y 6 (14%) tenían una carga viral media más baja de 2,53 log10 (rango 1,90-3,04) copias genómicas/µg de ADN celular (p < 0,0001) (fig. 1). El primero comprendía el grupo con infección congénita por CIHHV6 y el último aquellos con infección congénita adquirida por vía placentaria. Las características y hallazgos de laboratorio de los niños se presentan a continuación según los tres grupos de niños, niños congénitamente infectados con CI-HHV6, niños infectados congénitamente con TPI y los niños controles.
Fig. 1. Carga viral de HHV6 en células mononucleares de sangre de cordón (CBMC), células mononucleares de sangre periférica(PBMC), folículos capilares, orinas y saliva de niños con infección congénita por HHV6 adquirida por integración cromosómica (CIHHV6) o por infección transplacentaria (TPI) y de niños controles con infección posnatal (PNI). *Todas las orinas TPI fueron negativas en ADN HHVY y todas excepto dos de cien orinas PNI analizadas fueron negativas.
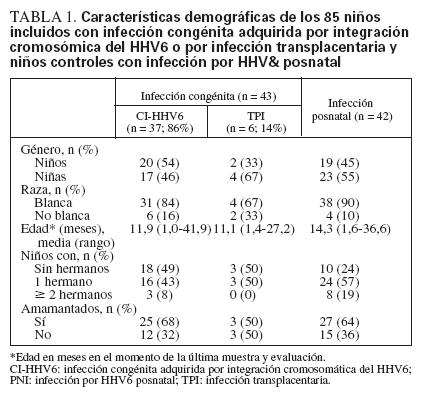
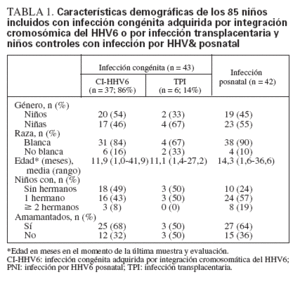
Características demográficasLos niños infectados congénitamente y posnatalmente eran en general similares, aunque los niños controles en comparación con los congénitamente infectados eran más mayores, aunque no significativamente, y tenían más posibilidades de tener uno o más hermanos que los niños infectados congénitamente (76% vs. 51%, p = 0,02) (tabla 1).
TABLA 1. Características demográficas de los 85 niños incluidos con infección congénita adquirida por integración cromosómica del HHV6 o por infección transplacentaria y niños controles con infección por HHV& posnatal
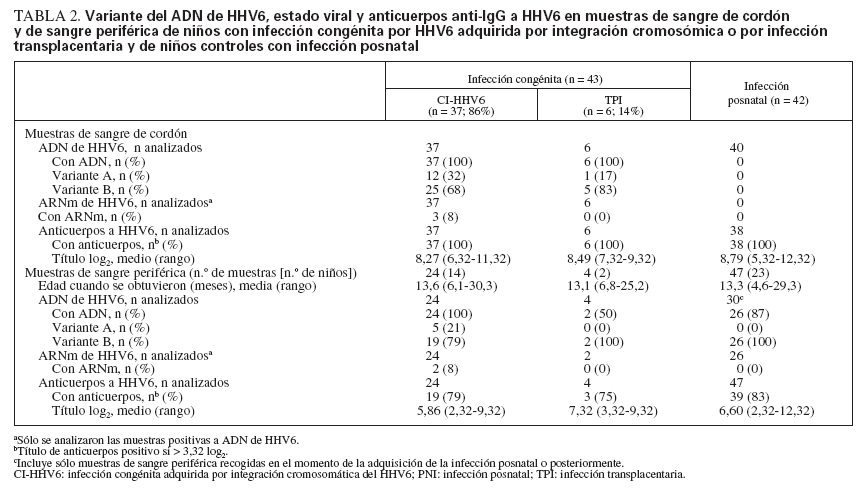
Detección de ADN de HHV6, variante y estado replicativoEntre las muestras de sangre de cordón, el ADN de HHV6 detectado fue variante A en 12 (32%) de los niños CI-HHV6 y en 1 (17%) de los niños infectados transplacentariamente (p = 0,44) (tabla 2).
TABLA 2. Variante del ADN de HHV6, estado viral y anticuerpos anti-IgG a HHV6 en muestras de sangre de cordón y de sangre periférica de niños con infección congénita por HHV6 adquirida por integración cromosómica o por infección transplacentaria y de niños controles con infección posnatal
Entre las muestras seguidas de PBMC de niños CIHHV6, todas contenían ADN de HHV6, que era la misma variante detectada en su CBMC. En comparación, el 50% de las muestras de PBMC de TPI y el 87% de las de niños PNI contenían ADN de HHV6. El HHV6 B comparación con el 2% de los niños infectados posnatalmente (p = 0,0004). Los ensayos RT-PCR para una transcripción tardía de genes que indicara replicación viral fueron positivos en el 8% de las muestras de CBMC y PBMC de los niños con CI-HHV6 y en ninguna de las muestras de los otros grupos de niños.
Anticuerpos a HHV6Se determinaron los títulos de anticuerpos en muestras de sangre de cordón, como un indicador de anticuerpos maternos pasivos, así como en las subsiguientes muestras de sangre periférica obtenidas, con el fin de comparar el declive y adquisición de anticuerpos HHV6 entre los tres grupos de niños (tabla 2). Se detectaron anticuerpos HHV6 en todas las muestras de sangre de cordón obtenidas de cada grupo. El título log 2 medio (± desviación estándar) de anticuerpos a HHV6 en las muestras de sangre de cordón no difirió significativamente entre los niños CI-HHV6 y los niños PNI controles sin infección congénita (8,27 ± 1,15 vs. 8,79 ± 1,39, p = 0,27).
Puesto que la respuesta humoral a la infección por HHV6 podría empeorar entre los individuos con CI-HHV6, el nivel de anticuerpos a HHV6 pasivos en los niños CI-HHV6 cuyas madres tenían CI-HHV6 podría diferir del observado entre los niños con CI-HHV6 cuyos padres tenían CI-HHV6 y de aquellos del grupo de infección posnatal cuyos padres no tenían CI-HHV6. De los niños con CI-HHV6, 18 tenían madres con CIHHV6, y 11 tenían padres con CI-HHV6, basándose en la detección de ADN de HHV6 en las muestras de cabello de los progenitores. Los niveles medios de anticuerpos presentes en la sangre de cordón no difirieron significativamente entre los niños con CI-HHV6 derivada de la madre (8,43 ± 1,37 log2), y los niños con CI-HHV6 derivada del padre (8,32 ± 1,10 log2), y los niños con infección posnatal (8,79 ± 1,39 log2) (p > 0,30 para cada comparación). El nivel medio de anticuerpos en las 15 muestras de sangre periférica de niños con CI-HHV6 derivada de la madre fue más bajo que el nivel en las 47 muestras de sangre periférica de los niños con infección posnatal, aunque esta diferencia no fue estadística-mente significativa (5,85 vs. 6,60 log2, p > 0,35). Las muestras se obtuvieron a una edad media de 13 meses en los dos grupos.
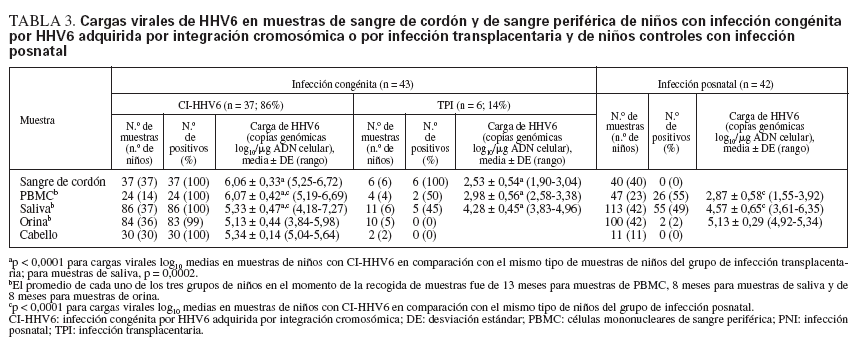
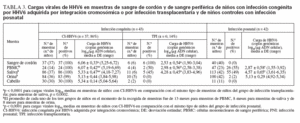
Cargas virales de ADN de HHV6 en muestras de niños infectados congénitamente y posnatalmenteSe detectaron altas cargas virales de ADN de HHV6 en todas las muestras de PBMC, saliva, orina y folículos capilares de niños con CI-HHV6 (tabla 3; fig. 1). Las cargas virales medias de cada sitio oscilaron de 5,13 a 6,07 log10 copias genómicas/µg de ADN celular. La media y rango de las cargas virales en PBMC de niños con CI-HHV6 fueron similares a aquellas detectadas en CBMC. Las cargas medias de HHV6 en PBMC y saliva de niños con CI-HHV6 fueron significativamente más elevadas (p µ 0,0002) que los niveles detectados en las muestras correspondientes de niños con infección HHV6 transplacentaria y posnatal.
TABLA 3. Cargas virales de HHV6 en muestras de sangre de cordón y de sangre periférica de niños con infección congénita por HHV6 adquirida por integración cromosómica o por infección transplacentaria y de niños controles con infección posnatal
Además, las distribuciones de las cargas virales detectadas en CBMC y PBMC de niños con CI-HHV6 no se superponían con las detectadas en las muestras correspondientes de niños con infección transplacentaria o posnatal. Se detectó HHV6 en las 86 muestras de saliva de niños con CI-HHV6 en comparación con el 45% y 49% de las muestras de saliva de niños infectados por vía transplacentaria y posnatalmente, respectivamente.
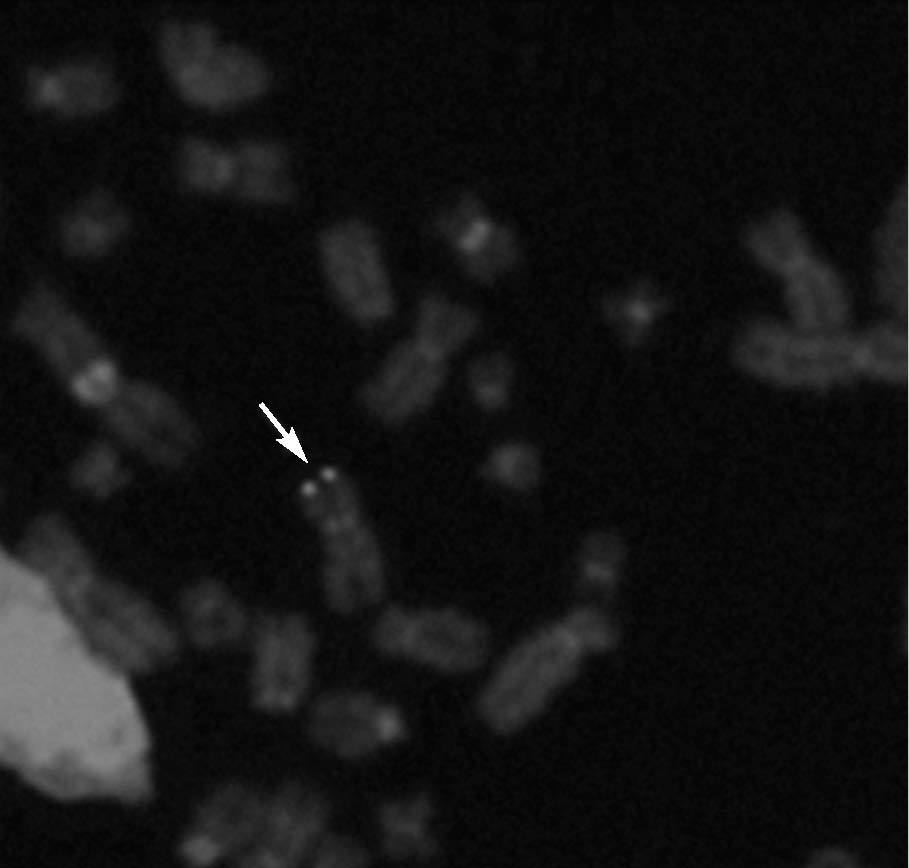
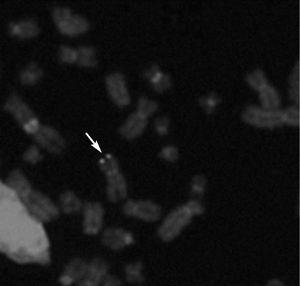
Hibridación fluorescente in situ (FISH)De 5 niños infectados congénitamente se examinaron muestras LCL de PBMC por FISH. Se detectaron señales positivas de FISH para HHV6 en 4 líneas celulares (fig. 2). Todas fueron del grupo de CI-HHV6, definido por la presencia de HHV6 en cargas virales elevadas en muestras de CBMC y folículos capilares. El único espécimen negativo era de un niño infectado congénitamente clasificado como TPI por bajas cargas virales en CBMC y ausencia de ADN de HHV6 en muestras de folículos capilares.
Fig. 2. Hibridación fluorescente in situ (FISH) representativa en cromosomas en metafase de un niño con elevados niveles de ADN de HHV6 en sangre de cordón (> 5 copias genómicas log10µg de ADN celular) y detección de ADN de HHV6 en sus folículos capilares. La flecha muestra señales simétricas de ADN de HHV6 en ambas cromátides de un único cromosoma.
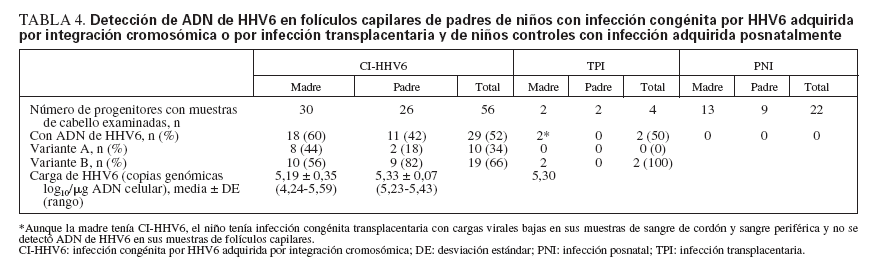
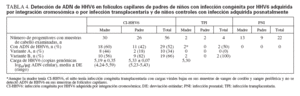
ADN de HHV6 en muestras de folículos capilares de los padresSe detectó ADN de HHV6 en muestras de folículos capilares en un padre de todos los niños CI-HHV6 con muestras disponibles de los progenitores (tabla 4). En una familia con dos niños incluidos en el estudio, se detectó ADN de HHV6 en las muestras de folículos capilares de la madre y de ambos niños.
TABLA 4. Detección de ADN de HHV6 en folículos capilares de padres de niños con infección congénita por HHV6 adquirida por integración cromosómica o por infección transplacentaria y de niños controles con infección adquirida posnatalmente
Entre las 56 muestras de folículos capilares examinadas, una proporción similar de las muestras de cabello maternas y paternas contenían ADN de HHV6, 60% y 42%, respectivamente (p = 0,56). La carga viral media en las muestras de folículos capilares de las madres fue más baja que en las muestras de los padres, aunque no significativamente (p = 0,12). La variante A se detectó en el 44% de las muestras de cabello maternas y en el 18% de las muestras de cabello paternas (p = 0,23). La variante identificada entre las muestras de cabello de los miembros de la familia fue el mismo que el identificado en sus hijos en todos los casos.
No se detectó ADN de HHV6 en las muestras de cabello obtenidas de padres de niños PNI o TPI, con una excepción. Aunque la madre tenía CI-HHV6, la infección del niño pareció ser transplacentaria, puesto que sus muestras de folículos capilares no contenían ADN de HHV6, y las cargas virales en sus muestras de CBMC y PBMC fueron bajas y no presentes de forma constante. Así mismo, se obtuvieron muestras de cabello de 21 padres de un grupo adicional de niños controles que fueron seguidos por adquisición posnatal de la infección, pero que no se infectaron durante el período de estudio, y que por tanto no podían ser incluidos en el grupo de infección posnatal. Ninguna de estas muestras de padres y de niños contenía ADN de HHV6.
DISCUSIÓNEsta evaluación prospectiva de niños desde su nacimiento ha proporcionado observaciones novedosas en relación con la infección congénita por HHV6. La infección congénita por HHV6 puede ocurrir tanto por transmisión transplacentaria como por un mecanismo único de integración cromosómica del virus. Además, el CIHHV6 causó la gran mayoría, 86%, de las infecciones congénitas por HHV6. En tercer lugar, entre los niños infectados congénitamente, el CI-HHV6 podría ser identificado como el mecanismo de transmisión por las elevadas cargas virales en las muestras de sangre de cordón y en las otras muestras, así como por detección de AND de HHV6 en las muestras de folículos capilares. Esto fue confirmado entre 5 (12%) niños infectados congénitamente por análisis FISH. Además, es más probable que la infección congénita por CI-HHV6 ocurra vía transmisión por línea germinal, tal como se demuestra por la detección de ADN de HHV6 en folículos capilares de uno de los progenitores en familias con niños con CI-HHV6, pero en ninguno de las muestras de folículos capilares de progenitores de niños con infección adquirida posnatalmente. Aunque el HHV6 puede integrarse cromosómicamente por otros mecanismos, estos hallazgos indican que era poco probable que la integración espontánea durante la gestación o después del nacimiento fuera el mecanismo de infección por CI-HHV6 en estos niños12,31.
Puesto que todas las madres de niños con infección por HHV6 congénita han tenido infección por HHV6 previa, se ha asumido que la infección por HHV6 congénita, similar a la infección por CMV congénita, es también resultado principalmente del paso transplacentario de la infección materna reactivada o de la reinfección por HHV6. Ambos virus ocasionan infección congénita en tasas notablemente similares de alrededor del 1%8,9, siendo también el CMV una infección común aunque no universal. Sin embargo, existen importantes diferencias entre las infecciones por HHV6 y CMV, como señaló Pass32. La infección por CMV congénita es resultado de una infección materna, que podría ser una infección primaria, reinfección o reactivación del virus latente32,33. Para que se produzca la infección congénita después de la infección materna, sería fundamental la transmisión transplacentaria del virus replicante activo. Sin embargo, pocas veces hemos sido capaces de documentar replicación activa del HHV6 en muestras maternas de PBMC y otros sitios o en muestras de sangre de cordón o neonatal de niños congénitamente infectados4,34. Esto podría apoyar nuestros actuales hallazgos, que apuntan que sólo una pequeña proporción de las infecciones congénitas por HHV6 serían resultado de la reactivación o reinfección en la madre.
La importancia de la infección congénita por CIHHV6 dependerá en parte de las ramificaciones biológicas del CI-HHV6. Se sabe poco, sin embargo, sobre el estado viral del CI-HHV6, y sobre si los individuos con CI-HHV6 producen una respuesta inmune normal o demuestran tolerancia inmunológica a la infección subsiguiente con HHV6.
La respuesta humoral inmune ha sido variable en los pocos individuos con CI-HHV6 en los que se ha comunicado13,17,22,23. Entre cuatro artículos que examinaron 37 pacientes con CI-HHV6, la mayoría, aunque no todos, mostraban anticuerpos HHV6 detectables, aunque el anticuerpo no era neutralizante en algunos pacientes13,17,22,23,35. Todos nuestros niños con infección congénita tenían anticuerpos a HHV6 en su sangre de cordón, lo que sugiere que todas las madres producían una respuesta humoral a HHV6. Los niños con CI-HHV6 tenían niveles medios más bajos de títulos de HHV6 en sus muestras de sangre de cordón y de sangre periférica en comparación con los niveles medios detectados en los niños con infección transplacentaria o en los niños controles con infección posnatal, pero las diferencias no fueron estadísticamente significativas. Así mismo, la cinética de los títulos de anticuerpos a HHV6 pareció ser similar entre los niños con y sin CI-HHV6. Ambos mostraron el esperado declive de anticuerpos pasivos seguido por un aumento de los títulos en muestras obtenidas después de unos meses de edad. La mayoría de los niños con CI-HHV6, por tanto, parecían ser capaces de mostrar una respuesta humoral inmune a HHV6 bien de una infección adquirida después del nacimiento o posiblemente a la producción del genoma integrado de partículas virales. El hallazgo de que los títulos medios de anticuerpos a HHV6 en la sangre de cordón de niños cuyas madres tenían CI-HHV6 era similar a los de los niños con CI-HHV6 adquirido por vía paterna y a las de los niños sin infección congénita sugiere de nuevo que los individuos con CI-HHV6 pueden producir una respuesta humoral específica a HHV6.
Sin embargo, los anticuerpos a HHV6 en individuos con CI-HHV6 pueden no ser protectores. Una de nuestras madres tenía CI-HHV6, pero la infección congénita del niño ocurrió por transmisión transplacentaria del virus, probablemente como resultado de una reinfección o reactivación del HHV6 en la madre. Por eso, las madres con CI-HHV6 podrían producir niños congénitamente infectados por paso de la línea germinal o virus integrado cromosómicamente o por infección transplacentaria, o posiblemente por ambos mecanismos.
El estado viral del CI-HHV6 es potencialmente integral a la respuesta inmune y a las consecuencias de la infección congénita por CI-HHV6. Se ha demostrado que el CI-HHV6 no es replicante in vitro, y no se ha detectado replicación en un número limitado de pacientes con enfermedades linfoproliferativas, o en miembros de familias con CI-HHV612,13,15,17,36. Entre nuestros niños con CI-HHV6, se detectó ARNm de HHV6 en el 8% de las muestras de sangre de cordón y de PBMC, pero en ninguna de las muestras de niños con infección transplacentaria o posnatal. La detección de ARNm de HHV6 en sangre de cordón podría indicar replicación de virus integrado cromosómicamente o transcripción de todo o parte del genoma viral durante la transcripción génica humana. Sin embargo, la detección de ARNm en PBMC podría ser resultado también de reinfección con una nueva cepa de HHV6. Nosotros hemos demostrado previamente la presencia de ARNm en muestras de PBMC de una pequeña proporción (3,3%) de niños normales años después de la infección inicial, lo que indica que la reinfección o reactivación del virus puede producirse en niños inmunocompetentes34.
La tasa de transmisión de CI-HHV6 no pareció estar afectada por la vía parental, igual que la proporción de niños con CI-HHV6 con herencia materna versus paterna no fue significativamente diferente, siendo sus cargas virales similares. No obstante, la variante A se detectó significativamente más a menudo en muestras sanguíneas de niños con CI-HHV6 que en muestras de niños infectados posnatalmente. Esto podría indicar que el HHV6A tendría una mayor propensión que el HHV6B a convertirse en cromosómicamente integrado en sitios cromosómicos específicos. Cada uno de nuestros pacientes con CI-HHV6 con muestras analizadas por FISH demostró integración simétrica de HHV6 al final de ambas cromátides de un único cromosoma. Muy pocos individuos o familias clasificados como CI-HHV6 habían sido analizados por FISH y sólo dos niños, ambos con problemas neonatales, fueron examinados durante la infancia14,17-19. La señal de integración de HHV6 en estos niños vino también de un solo homólogo de un cromosoma19. De los pocos cromosomas identificados con CIHHV6, los finales de los telómeros habían sido los sitios identificados predominantemente12,13,16-19,23. Teóricamente esto podría tener relevancia clínica puesto que los telómeros parecen ser integrales al mantenimiento de la función inmune normal37.
Se desconocen las potenciales manifestaciones inmunológicas, biológicas y a largo plazo asociadas con el CI-HHV6. Nuestros hallazgos demuestran que la transcripción de genes virales, tanto a partir del genoma integrado como de la falta de protección frente a la reinfección, puede ocurrir en niños con CI-HHV6. La actividad génica viral persistente en múltiples sitios, incluyendo el sistema nervioso central, podría posiblemente intensificar el riesgo de déficits de desarrollo subsiguientes, como ocurre con la infección congénita por CMV8,10,11,32. Esto debe ser más estudiado. Si el 86% del 1% de los niños nacidos con infección por HHV6 congénita tienen CI-HHV6, como sugiere este estudio, 1 de cada 116 niños nacidos vivos podría tener CI-HHV6, lo que se podría traducir en una carga de salud apreciable, aunque actualmente no reconocida.
AGRADECIMIENTOSEste trabajo fue financiado por becas del National Institute of Child Health and Development (RO1 HD 44430-01), y en parte por becas del General Clinical Research Center (GCRC), 5 MO1 RR00044 del National Center for Research Resources, NIH, y de la HHV6 Foundation.
Correspondencia: Caroline Breese Hall, MD, Department of Pediatrics, University of Rochester School of Medicine and Dentistry, 601 Elmwood Ave, Box 689, Rochester, NY 14642, Estados Unidos.
Correo electrónico: caroline_hall@urmc.rochester.edu