Determinar los grupos filogenéticos y la diversidad clonal de cepas de Escherichia coli aisladas de productos lácteos artesanales elaborados y comercializados en Mérida, Venezuela.
Materiales y métodosSe analizaron 45 cepas de E.coli provenientes de productos lácteos artesanales (crema de leche, cuajada y requesón). Estas cepas se aislaron según la norma de la Comisión Venezolana de Normas Industriales (COVENIN) y la identificación se llevó a cabo por metodologías convencionales. La susceptibilidad antimicrobiana se determinó mediante la técnica de difusión del disco, y los fenotipos con susceptibilidad intermedia o resistente fueron confirmados por concentración inhibitoria mínima (CIM). El grupo filogenético se determinó mediante amplificación por PCR de los genes chuA, yjaA y del fragmento TspE4.C2 y la tipificación de las cepas por Rep-PCR.
ResultadosEl 73,3% (33/45) de las cepas fueron susceptibles a todos los antibióticos probados y el 24,4% (11/45) presentaron resistencia a la ampicilina. La mayoría de las cepas resistentes fueron aisladas de requesón. El 82,2% de las cepas fueron ubicadas en el filogrupoA y el 8,9% en los grupos B1 y D, respectivamente. El filogrupoB2 no fue detectado. La tipificación por Rep-PCR de las cepas demostró una estructura poblacional heterogénea.
ConclusiónLa caracterización molecular de estas cepas demostró que no están relacionadas clonalmente y en su mayoría se distribuyeron entre los filogrupos correspondientes a cepas comensales de baja patogenicidad. La detección de E.coli en alimentos lácteos indica una contaminación de origen fecal, que eventualmente podría estar asociada con la presencia de otros enteropatógenos.
To determine phylogenetic groups and clonal diversity of Escherichia coli strains isolated from several homemade dairy foods produced and marketed in Mérida, Venezuela.
Materials and methodsForty-five E.coli strains isolated from homemade dairy products (cream cheese, curd and cottage cheese) were analyzed. These strains were isolated according to procedures established by the Venezuelan Commission of Industrial Norms (COVENIN) and identification was carried out using conventional methods. Antimicrobial susceptibility was determined by the disk diffusion method and phenotypes with intermediate or resistant susceptibility were confirmed by minimum inhibitory concentration (MIC). Phylogenetic groups were identified by PCR amplification of chuA, yjaA genes and the TspE4.c2 DNA fragment, while molecular typing was carried out by Rep-PCR.
ResultsOf the 45 isolates, 33 (73.3%) were susceptible to all antibiotics tested while 11 (24.4%) were ampicillin-resistant. The phylogenetic groupA was the most common (82.2%), followed by B1 and D (8.9%, respectively). The phylogroupB2 was not detected and Rep-PCR typing of E.coli strains showed a heterogeneous population structure.
ConclusionThe molecular characterization of E.coli strains showed that they were clonally nonrelated and mostly distributed among phylogenetic groups that belong to low pathogenicity commensal strains. Detection of E.coli strains in dairy foods indicates a contamination of fecal origin, which could be associated with the presence of other enteric pathogens.
Escherichia coli es un microorganismo que se encuentra en el intestino de humanos y animales formando parte de la microbiota normal1. La presencia de esta bacteria en alimentos indica generalmente una contaminación directa o indirecta de origen fecal, que en algunas ocasiones pudiera estar acompañada de otros patógenos intestinales. La detección y cuantificación de E.coli constituye uno de los parámetros importantes para evaluar la calidad sanitaria de un alimento1,2. Algunos derivados lácteos que no son sometidos a procesos de higienización pueden tener una cantidad permisible de E.coli, sin que ello signifique necesariamente un peligro para el consumidor o una disminución en la calidad de estos productos3. Sin embargo, valores por encima de los límites recomendados para E.coli como indicador pueden advertir sobre una inadecuada manipulación y condiciones sanitarias deficientes durante la producción, almacenamiento, transporte y comercialización de estos alimentos2,3.
E.coli tiene la habilidad de sobrevivir y desarrollarse en el medio ambiente gracias a su versatilidad para aprovechar distintas fuentes de energía, además de ser un microorganismo poco exigente en sus requerimientos nutricionales. Esta capacidad de adaptación a diversas condiciones favorece su integración a comunidades microbianas en una variedad de ambientes4. Esta propiedad de E.coli constituye uno de los factores que facilita la contaminación de alimentos de origen animal y vegetal, frutas y sus derivados, fuentes de agua potable, así como su intervención y/o persistencia en cualquiera de las etapas de la cadena alimentaria4,5.
Aunque E.coli es el principal componente aerobio de la flora intestinal del hospedero humano, sus interrelaciones con este permiten separarla en 3 grandes grupos: cepas comensales, patotipos intestinales y patógenos extraintestinales6. A su vez, con base en el análisis de isoenzimas (MLEE, del inglés Multilocus Enzyme Electrophoresis) las cepas de E.coli son designadas en 4 grupos filogenéticos principales: A, B1, B2 y D6,7. Las cepas comensales se ubican principalmente en los filogruposA y B1, mientras que las patógenas extraintestinales se agrupan en el B2 y el D, y los patotipos intestinales, en cualquiera de los filogrupos6-8. Algunos estudios señalan que las cepas de E.coli aisladas del ambiente pertenecen al grupo filogenéticoB14,6, mientras que cepas contaminantes de alimentos pueden encontrarse en cualquiera de los 4 filogrupos8.
Por otra parte, E.coli es considerado como un reservorio y transmisor de genes a otros miembros de la microbiota humana o animal. Por consiguiente, el tracto gastrointestinal se convierte en el nicho ideal para la transferencia a gran escala de genes de resistencia antimicrobiana y de factores de patogenicidad8,9. De hecho, en los últimos años, se ha registrado un aumento importante de cepas de E.coli resistentes a los antibióticos provenientes de animales sanos y sus productos destinados al consumo humano10-13. Esta resistencia, al estar codificada principalmente en elementos genéticos móviles, facilita su diseminación en diferentes ecosistemas, incluyendo el humano9,13. En este sentido, la Organización Mundial de la Salud recomienda monitorizar la resistencia antimicrobiana en una «bacteria indicadora» como E.coli para analizar los fenotipos y mecanismos de resistencia que pudieran diseminarse a través de alimentos de origen animal y sus derivados, en diferentes áreas geográficas14.
Actualmente, las técnicas moleculares que analizan las características o polimorfismos genéticos de microorganismos han ampliado notablemente el campo de la tipificación de bacterias de interés epidemiológico; dentro de estas técnicas destacan por su alto poder de discriminación el análisis del polimorfismo de la longitud de los fragmentos de restricción (RFLP, del inglés Restriction Fragment Length Polymorphism) y la electroforesis en gel de campos pulsantes (PFGE, del inglés Pulsed-Field Gel Electrophoresis)1,15. Sin embargo, las técnicas de tipificación basadas en la amplificación de ácidos nucleicos mediante PCR se utilizan mucho más, por ser menos laboriosas, ya que no dependen de enzimas ni de equipos de electroforesis especiales y tienen un aceptable nivel discriminatorio y buena reproducibilidad, y además son de relativo bajo costo. Entre las técnicas alternativas se encuentra la amplificación de elementos repetitivos (Rep-PCR, del inglés repetitive element PCR fingerprinting), para la rápida obtención de huellas genómicas15. En el caso de E.coli, esta técnica tiene la finalidad de determinar la relación y origen clonal de cepas de la misma especie, determinar el número de clones circulantes, reconocer brotes infecciosos de casos no relacionados, así como evaluar la eficacia de las medidas de control dirigidas a evitar la diseminación de clonas con características particulares de patogenicidad o resistencia15-17.
Existen numerosas investigaciones que reportan las características moleculares de cepas de E.coli aisladas de muestras clínicas de humanos y de animales enfermos, y una gran variedad de trabajos analizan el grado de relaciones genéticas entre cepas patógenas en animales con las de origen humano13,18,19. Por el contrario, los estudios que describen las propiedades genéticas de E.coli aisladas de alimentos de origen animal destinados al consumo humano, especialmente de los que no tienen un procesamiento industrial, son escasos11,20,21. Particularmente, en Venezuela existen pocos reportes que involucren técnicas moleculares para estudiar la diversidad genética y la importancia epidemiológica de bacterias presentes en alimentos procesados artesanalmente16. Por tal motivo, el objetivo de este estudio fue determinar los grupos filogenéticos y la diversidad clonal de cepas de E.coli aisladas de productos lácteos procesados artesanalmente y comercializados en Mérida, Venezuela.
Materiales y métodosProductos lácteosSe recolectaron 15 muestras de productos lácteos artesanales distribuidas de la siguiente manera: 5 de crema de leche, 5 de cuajada y 5 de requesón. Estas muestras fueron adquiridas de manera aleatoria a partir de lotes no relacionados y en establecimientos comerciales diferentes ubicados en el área urbana del Municipio Libertador del estado Mérida, Venezuela, durante el período enero a julio 2013. Los productos lácteos seleccionados oscilaron entre 250 y 500g por presentación.
Aislamiento e identificación de las cepas de Escherichia coliSe tomaron porciones de diferentes sitios del producto lácteo para tener una muestra representativa. Se pesaron 10g de cada muestra y se homogeneizaron en 90ml de agua peptonada al 0,1% (dilución 10−1), siguiendo las recomendaciones de la Comisión Venezolana de Normas Industriales (COVENIN)22. Luego, con la finalidad de mejorar la recuperación de las cepas de E.coli se realizó una incubación por 2h a 37°C, y a partir de esta solución se realizaron 3 diluciones (10−2 a 10−4) bajo las mismas condiciones. Posteriormente, de cada dilución se tomó 1ml para inocular las placas rehidratables tipo Petrifilm E.coli/coliformes (3M™, EE.UU.)23 y se incubaron a 35°C por 18 a 24h, siguiendo las recomendaciones del proveedor.
Se escogieron todas las placas que mostraron un crecimiento entre 4 a 10 unidades formadoras de colonias (UFC) de E.coli; estas colonias fueron reconocidas por el color azul y su asociación a la producción de gas. A partir de estas placas, se seleccionaron al azar 4 colonias, las cuales fueron extraídas con el asa en punta, colocadas en caldo BHI (BBL, Cockeysville, Md, EE.UU.) e incubadas a 36°C por 18-24h. Luego del crecimiento en dicho caldo y subcultivando en agar Levine o MacConkey (Himedia, Mumbai, India) a 36°C por 18-24h, se seleccionaron las colonias fermentadoras de la lactosa que tuviesen morfología sugestiva de E.coli, y a estas se les confirmó su identificación mediante el sistema de galerías API 20E (BioMerièux, Marcy l’Etoile, Francia), de acuerdo a lo descrito por el proveedor.
Adicionalmente, a todas las cepas de E.coli seleccionadas se les determinó bioquímicamente la fermentación del sorbitol. Las variantes fenotípicas sorbitol negativas fueron sometidas a ensayos de aglutinación con antisueros específicos (AntiColi O157:K-, Sifin Berlin, Alemania) con el objeto de descartar la presencia de una posible E.coli O157:H7. En estos ensayos se utilizaron como cepas control: E.coli ATCC 25922 y E.coli O157:H7 (CVCM1931), siguiendo el protocolo descrito por el proveedor.
Pruebas de susceptibilidad antimicrobianaLa susceptibilidad antimicrobiana de las cepas de E.coli se realizó utilizando el método de difusión por disco, de acuerdo con los criterios del Clinical and Laboratory Standards Institute (CLSI)24. Los discos de antibióticos utilizados fueron (BBL): ampicilina (10μg), amoxicilina/ácido clavulánico (30/15μg), ceftazidima (30μg), cefotaxima (30μg), gentamicina (10μg), tobramicina (10μg), amikacina (30μg), ciprofloxacino (5μg), ácido nalidíxico (30μg), imipenem (10μg), meropenem (10μg) y ertapenem (10μg). Las cepas de E.coli que resultaron con susceptibilidad intermedia y/o resistente a los antibióticos probados se les confirmó el fenotipo mediante la determinación de la concentración inhibitoria mínima (CIM) en agar, siguiendo las recomendaciones del CLSI20. En estos ensayos se utilizó como cepa control E.coli ATCC 25922.
Extracción del ADNLa extracción y preparación del ADN genómico se realizó mezclado varias colonias provenientes de cultivos frescos en 200μl de agua destilada estéril. Estas suspensiones se colocaron en congelación (–20°C) durante 30min y luego se sometieron a ebullición durante 15min. Los residuos celulares se separaron por centrifugación (13.000rpm durante 5min a temperatura ambiente) y el ADN disuelto en el sobrenadante se recuperó en un tubo eppendorf estéril, el cual se almacenó a –20°C hasta el momento de su uso17.
Determinación de los grupos filogenéticos de Escherichia coliLas cepas fueron clasificadas dentro de los grupos filogenéticos (A, B1, B2 y D) de acuerdo con la presencia de los genes chuA, yjaA y el fragmento génico TspE4.C2, mediante una PCR múltiple utilizando los iniciadores y condiciones descritas previamente25. La mezcla de amplificación se realizó en un volumen final de 25μl y estuvo compuesta por: 2,5μl del buffer de reacción (10X), 1,5μl de MgCl2 (50mM), 1,5μl de dNTPs (10mM; Bioneer, Daejeon, Corea), 2,5μl de cada iniciador (10pmol/μl), 0,3μl de la Taq polimerasa (5U/μl; Bioneer), 2,2μl de agua bidestilada ultrapura y 2μl del ADN extraído. Los resultados fueron interpretados con base en el esquema de Clermont et al.25 mediante la ausencia (–) o presencia (+) de los elementos antes descritos: grupo A: chuA– y TspE4.C2–; B1: chuA– y TspE4.C2+; B2: chuA+ y yjaA+ y el filogrupo D: chuA+ y yjaA–. Para estos ensayos se utilizaron como cepas control E.coli LMM36-ULA (chuA+ y yjaA+) y E.coli LMM32-ULA (TspE4.C2+).
Tipificación de Escherichia coli por Rep-PCRLa relación clonal entre las cepas fue determinada por la amplificación de secuencias repetitivas por PCR (Rep-PCR) a partir del ADN total, utilizando los iniciadores: REP-1 F: 5’- IIIGCGCCGICATCAGGC-3’; REP-2 R: 5’-ACGTCTTATCAGGCCTAC-3’ y las condiciones descritas por Lozano et al.26. La mezcla de reacción para la Rep-PCR se realizó en un volumen final de 25μl y estuvo constituida por 2,5μl de buffer (10X), 2,5μl de MgCl2 (50Mm), 3μl de dNTPs (10Mm), 3μl de cada uno de los iniciadores (10pmol/μl), 0,5μl de Taq polimerasa (5U/μl; Bioneer), 5,5μl de agua bidestilada estéril y 5μl de ADN. Los productos de amplificación se separaron en un gel de agarosa al 1,5%, teñido con bromuro de etidio y fotografiados con el UVP Biodoc-It System. Como marcador de peso molecular se utilizó una escalera de 50 pb (Bioneer). Los patrones obtenidos del Rep-PCR fueron analizados utilizando el Software Treecon 1.3b, el cual generó el dendrograma o árbol de similitud para establecer las relaciones entre las cepas estudiadas. Los patrones con coeficientes de similitud superior al 90% fueron considerados relacionados clonalmente.
ResultadosUn total de 90 cepas de E.coli fueron aisladas a partir de derivados lácteos artesanales. De estas, se seleccionaron aleatoriamente 45 que correspondieron a 15 cepas por cada grupo de producto lácteo: crema de leche, cuajada y requesón. Ninguna de las cepas seleccionadas resultó positiva para la identificación del serotipo patógeno E.coli O157:H7 (datos no mostrados).
Los resultados de las pruebas de susceptibilidad realizadas en las 45 cepas de E.coli se muestran en la tabla 1. El 73,3% (33/45) de las cepas fueron susceptibles a los 12 antibióticos probados. Las cefalosporinas de tercera generación (cefotaxima y ceftazidima), los carbapenemos y la amikacina fueron los antibióticos con mayor actividad sobre las cepas estudiadas (100%), seguido en orden de frecuencia por ácido nalidíxico (97,8%; 44/45), amoxicilina/ácido clavulánico (95,6%; 43/45), gentamicina y tobramicina (ambos 91,1%; 41/45). La resistencia a ampicilina se observó en el 24,4% de las cepas (11/45), mientras que el 8,9% (4/45) fue resistente a gentamicina y tobramicina. La resistencia a amoxicilina/ácido clavulánico y al ácido nalidíxico estuvo representada por un bajo número de cepas (4,4% [2/45] y 2,2% [1/45], respectivamente). La susceptibilidad intermedia solo se evidenció con ciprofloxacino en 17,8% de las cepas (8/45). Los fenotipos de multirresistencia se observaron en 5 cepas (11,1%) y estuvieron conformados por la asociación de 4 o 5 antibióticos, de los cuales la susceptibilidad intermedia a ciprofloxacino y la resistencia a ampicilina fueron los marcadores más frecuentes. Todas las cepas de E.coli con fenotipo resistente fueron aisladas de requesón, representando el 73,3% (11/15) del total de cepas recuperadas en ese producto. Solo una cepa (A25-4) proveniente de crema de leche presentó susceptibilidad intermedia a ciprofloxacino (tabla 2).
Susceptibilidad de 45 cepas de Escherichia coli aisladas de productos lácteos artesanales frente a 12 agentes antimicrobianos
| Susceptibilidad antimicrobiana, n (%) | |||
| Antibiótico | Sensible | Intermedioa | Resistentea |
| Ampicilina | 34 (75,5) | 0 | 11 (24,4) |
| Amoxicilina/ácido clavulánico | 43 (95,6) | 0 | 2 (4,4) |
| Cefotaxima | 45 (100) | 0 | 0 |
| Ceftazidima | 45 (100) | 0 | 0 |
| Imipenem | 45 (100) | 0 | 0 |
| Meropenem | 45 (100) | 0 | 0 |
| Ertapenem | 45 (100) | 0 | 0 |
| Amikacina | 45 (100) | 0 | 0 |
| Gentamicina | 41 (91,1) | 0 | 4 (8,9) |
| Tobramicina | 41 (91,1) | 0 | 4 (8,9) |
| Ácido nalidíxico | 44 (97,8) | 0 | 1 (2,2) |
| Ciprofloxacino | 37 (82,2) | 8 (17,8) | 0 |
Distribución de los patrones de resistencia y procedencia de las cepas de Escherichia coli
| Cepa número | Marcadores de resistencia (n) | Patrón de resistencia | Procedencia |
| A19-3 | AMP | Requesón | |
| A25-4 | CIPI | Crema de leche | |
| A29-5 | 1 | AMP | Requesón |
| A30-5 | AMP | Requesón | |
| A31-5 | AMP | Requesón | |
| A33-7 | CIPI, AMP | Requesón | |
| A36-7 | 2 | CIPI, AMP | Requesón |
| A34-7 | CIPI, AMP, AMC, NA | Requesón | |
| A60-14 | CIPI, AMP, CN, NN | Requesón | |
| A61-14 | 4 | CIPI, AMP, CN, NN | Requesón |
| A63-14 | CIPI, AMP, CN, NN | Requesón | |
| A62-14 | 5 | CIPI, AMP, AMC, CN, NN | Requesón |
AMC: amoxicilina/ácido clavulánico; AMP: ampicilina; CIPI: ciprofloxacino susceptibilidad intermedia; CN: gentamicina; NA: ácido nalidíxico; NN: tobramicina.
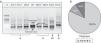
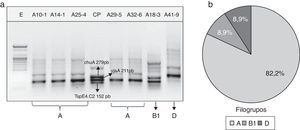
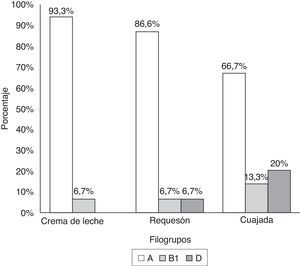
En la figura 1a se muestra un gel electroforético representativo de los productos amplificados por PCR correspondientes a los genes chuA (279pb) y yjaA (211pb) y al fragmento de ADN TspE4.C2 (152pb), cuya presencia o ausencia en las cepas de E.coli determinó la clasificación de los grupos filogenéticos A, B1, B2 y D. La distribución porcentual de los filogrupos en las 45 cepas de E.coli se presenta en la figura 1b. El 82,2% (37/45) de las cepas fueron ubicadas en el grupo filogenéticoA, mientras que los grupos B1 y D se distribuyeron en igual proporción (8,9% [4/45]). Sin embargo, cuando se estudió la frecuencia de los filogrupos de acuerdo con el origen o procedencia de la cepa, se encontró que el grupoA predominó en los 3 tipos de producto lácteo (crema de leche: 93,3%; requesón: 86,6%; cuajada: 66,7%). El filogrupoB1 tuvo una frecuencia del 13,3% en cepas aisladas de cuajada y del 6,7% en crema de leche y requesón. Por el contrario, el grupoD estuvo ausente en las cepas obtenidas a partir de crema de leche, pero su distribución se evidenció con mayor frecuencia en E.coli aisladas de cuajada (20% [3/15]). Ninguna de las cepas clasificó para el grupo B2 (fig. 2).
a) Gel electroforético representativo de la amplificación por PCR de los genes chuA, yjaA y del fragmento de ADN TspE4.C2 en cepas de E.coli aisladas de productos lácteos artesanales. CP: control positivo; E: marcador de peso molecular (100pb). b) Distribución general de los grupos filogenéticos de E.coli aisladas de productos lácteos artesanales.
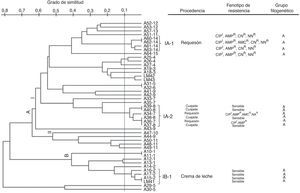
Las relaciones genéticas de las cepas de E.coli y sus respectivas características fenotípicas se muestran en la figura 3. El 95,6% de las cepas estudiadas (43/45) se distribuyeron en 2 grupos principales, A y B, los cuales mostraron niveles de similitud entre un 35 y un 58%, respectivamente. En el grupoA, específicamente en la ramaI, se observaron 2 subgrupos (IA-1 y IA-2) con aproximadamente un 95% de similitud. El IA-1 estaba conformado por 4 cepas aisladas de requesón y con al menos 4 marcadores de resistencia antimicrobiana comunes, y el IA-2 estaba integrado por 6 cepas (4 provenientes de cuajada y 2 de requesón), de las cuales 2 mostraron entre 2 y 4 marcadores de resistencia y 4 fueron susceptibles. En el grupoB se observó un subgrupo (IB-1) con relaciones de similitud cercana al 95%, estructurado por 4 cepas provenientes de crema de leche y sensibles a los antibióticos. Todas las cepas de E.coli que conformaron estos 3 subgrupos (IA-1, IA-2 y IB-1) fueron clasificadas dentro del filogrupoA.
DiscusiónE.coli es uno de los agentes biológicos que con mayor frecuencia se encuentra involucrado en la contaminación de alimentos. Particularmente la leche y sus productos derivados no pasteurizados son potencialmente peligrosos por su alto riesgo de contaminación en todas las etapas de la producción27. En este estudio se analizaron un conjunto de muestras provenientes de 3 grupos de productos lácteos artesanales —crema de leche, cuajada y requesón—, los cuales fueron adquiridos en diferentes establecimientos comerciales de la ciudad de Mérida. En ningún caso estos productos fueron reportados como vehículos en la transmisión o brote de alguna enfermedad de origen alimentario durante el lapso de estudio.
De un total de 90 cepas de E.coli aisladas de los derivados lácteos analizados, 45 fueron seleccionadas para su caracterización posterior. El análisis fenotípico de estas cepas demostró que más del 70% fueron susceptibles a los antibióticos ensayados, destacando la excelente actividad exhibida por las cefalosporinas de amplio espectro, carbapenemos y amikacina. No obstante, un porcentaje menor al 25% de las cepas fueron resistentes a ampicilina, y menos del 10% mostraron baja susceptibilidad a gentamicina, tobramicina, amoxicilina/ácido clavulánico y ácido nalidíxico. Estos resultados son similares a los descrito por Tamang et al.13, quienes reportaron la presencia de E.coli resistente a β-lactámicos en un 16,7% en muestras de leche cruda provenientes de 5 provincias de Corea. Sin embargo, porcentajes superiores a los obtenidos en este estudio fueron registrados en la región de Jammu (India)20, donde la prevalencia de cepas de E.coli multirresistentes en leche y derivados lácteos no pasteurizados fue del 48%, y en Irán se reportó una frecuencia de E.coli resistente a ampicilina, a cefalotina y a los aminoglucósidos mayor del 30% en productos lácteos fermentados con procesamiento artesanal8. Al respecto, se ha descrito que el uso de los antibióticos como terapéuticos, profilácticos y promotores del crecimiento en animales destinados al consumo humano ha contribuido con la aparición de cepas bacterianas resistentes9,12,28.
En este trabajo, las cepas de E.coli con fenotipos resistentes fueron aisladas casi exclusivamente de muestras de requesón, y es posible que este producto haya sido elaborado con lácteos contaminados desde su origen en el animal. Sin embargo, no se descarta que este hallazgo también se encuentre asociado a la naturaleza misma de su elaboración, ya que en las etapas de escurrido y amasado existe una mayor exposición de la materia prima a fuentes de contaminación provenientes del ambiente y/o del manipulador. En este sentido, Saleh et al.21 señalaron que las fuentes de contaminación con patotipos de E.coli multirresistentes de quesos artesanales provenientes del Valle de Beqaa (Líbano) fueron la manipulación y el secado al aire libre de la materia prima. Por el contrario, la evaluación de la calidad microbiológica de 126 quesos naturales de elaboración doméstica en Hokkaido (Japón) reportó un 19,8% de muestras positivas para coliformes con valores menores a 25UFC/g, y solo una muestra (0,8%) fue positiva para E.coli enteroinvasiva. Estos resultados, según los estándares internacionales, fueron considerados satisfactorios, debido al uso de leches pasteurizadas y a la implementación de buenas prácticas higiénicas en todas las etapas de la producción3.
Varios reportes señalan que dentro de la diversidad genética de E.coli, los gruposA y B1 reúnen las cepas comensales con pocos o ningún factor de virulencia, pero que son reservorios de genes de resistencia con una frecuencia mayor que en el resto de los filogrupos (B2 y D)6,7,27,28. Los resultados obtenidos avalan esta afirmación, ya que la mayoría de las cepas de E.coli analizadas pertenecieron al filogrupoA, y las pocas cepas que presentaron algún marcador de resistencia se concentraron en este mismo filogrupo. Aunque Son et al.1 no determinaron el perfil de resistencia de 570 cepas de E.coli aisladas en una finca productora de alimentos lácteos, el 86% de estas fueron consideradas como comensales, distribuyéndose entre los filogruposA y B1; el 11% se agrupó en el filogrupoD y el 4% en el B2. No obstante, Asai et al.11 señalaron que las cepas de E.coli productoras de β-lactamasas aisladas de alimentos provenientes de animales con colibacilosis se clasificaron indistintamente en los grupos filogenéticosA, B1 y D. Al respecto, varios autores coinciden en señalar que la cadena alimentaria es el vehículo de transferencia de genes de resistencia, y se ha sugerido un origen alimentario a la presencia de E.coli portador de diversos mecanismos de resistencia en la microbiota intestinal de humanos3,9,12,13,27,28.
Por otra parte, los resultados obtenidos se ajustan a la hipótesis propuesta por Carlos et al.29, quienes señalaron que la distribución de los grupos filogenéticos de E.coli puede ayudar a la identificación de las principales fuentes de contaminación fecal en productos de origen animal. En este sentido, cepas de E.coli de los gruposA y B1 se encuentran en un amplio espectro de mamíferos herbívoros y carnívoros, respectivamente, mientras que el B2 y D tienen un estrecho y especializado rango de hospederos; particularmente el filogrupoB2 es un buen indicador de contaminación fecal humana6,7. En este estudio, el filogrupo dominante fue el A, correspondiéndose con cepas comensales de E.coli probablemente presentes en un hospedero bovino, mientras que la ausencia de cepas del grupo B2 podría indicar que la fuente de contaminación fecal de los productos lácteos no era de origen humano.
La tipificación por Rep-PCR de las 45 cepas de E.coli aisladas de los 3 productos lácteos artesanales permitió evidenciar una estructura poblacional heterogénea, donde más del 95% de las cepas se distribuyeron en 2 grandes grupos, cuyos índices de similitud no superaron el 60%. Sin embargo, 3 subgrupos (IA-1, IA-2 y IB-1) con una relación genéticamente distante destacaron por rangos internos de similitud de aproximadamente el 95%, sin asociación alguna con las características fenotípicas. Es probable que la distribución policlonal observada en este estudio haya estado influenciada por la participación de diferentes factores intervinientes no controlados, tales como origen de la materia prima para la elaboración del producto, área geográfica donde se realizó el estudio, manufactura, condiciones de preservación del producto y aleatoriedad en la selección de las cepas, entre otros. Sin embargo, desde el punto de vista técnico es probable que utilizando otras herramientas moleculares con mayor poder discriminatorio, como la electroforesis de campo pulsante, y aumentado el número de cepas a estudiar, se pudiera dilucidar mejor la estructura y la distribución clonal de las cepas de E.coli presentes en alimentos lácteos artesanales que se expenden en Mérida, Venezuela. Estos aspectos serán considerados en estudios futuros.
Finalmente, los hallazgos obtenidos en este estudio permitieron concluir que los productos lácteos como crema de leche, cuajada y requesón de procesamiento artesanal disponibles comercialmente para el consumo humano están contaminados con cepas de E.coli clonalmente heterogéneas. Las características fenotípicas y genéticas de estas cepas indican que aunque siendo en su mayoría distribuidas entre los grupos filogenéticos correspondientes a cepas comensales de baja patogenicidad, no se descarta la posibilidad de que estas puedan adquirir nueva información genética y convertirse en reservorios de genes que codifiquen para factores de virulencia y/o diversos mecanismos de resistencia antimicrobiana.
Desde el punto de vista epidemiológico, la detección de cepas de E.coli en alimentos lácteos indica una contaminación de origen fecal, que eventualmente podría estar asociada con la presencia de otros enteropatógenos. Por ende, la información presentada en este estudio revela la necesidad de intensificar los controles higiénico-sanitarios en todas las etapas que conlleva la manufactura artesanal del producto, así como la implementación de medidas que permitan la vigilancia epidemiológica de cepas bacterianas potencialmente patógenas presentes en los productos y derivados lácteos no pasteurizados de fabricación artesanal que se expenden en la ciudad de Mérida, Venezuela.
Si bien el estudio descrito se circunscribe a una localidad geográfica específica, los resultados obtenidos, muy probablemente, son representativos de una problemática nacional que incluso puede ser válida para otras regiones del continente que comparten características socioeconómicas y culturales similares.
FinanciaciónEste trabajo fue parcialmente financiado por el Consejo de Desarrollo Científico Humanístico, Tecnológico y de las Artes, Universidad de Los Andes (CDCHTA-ULA) Proyecto N.° FA-541-13-03-EM y por el Fondo Nacional de Ciencia y Tecnología (FONACIT). Fortalecimiento del Laboratorio de Microbiología Molecular de la Facultad de Farmacia y Bioanálisis, Universidad de Los Andes, Mérida, Venezuela (Proyecto No. 2012002321).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores agradecemos a las profesoras Yolima Rosales y Cándida Díaz, del Laboratorio de Microbiología de Alimentos, y Judith Velasco, del Laboratorio de Síndromes Gastrointestinales de la Facultad de Farmacia y Bioanálisis, Universidad de Los Andes, Mérida, por sus valiosas recomendaciones en la realización de este estudio.