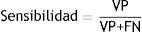En este trabajo se presenta un sistema para la detección de ictus hemorrágico e isquémico en imágenes de tomografía computarizada. En primer lugar, la imagen original se convierte en escala de grises y el ruido se elimina con el filtro bilateral. Después, las regiones del cráneo se eliminan mediante una función morfológica. La imagen se clasifica en hemorrágica o isquémica según la desviación estándar y la media. Un método para la segmentación manual también se implementó. Este método se basa en el uso de modelos deformables, aplicando específicamente el algoritmo de gradiente de flujo vectorial Snake. El resultado son imágenes segmentadas para las regiones anormales. Los experimentos realizados en las imágenes de tomografía computarizada muestran la precisión del sistema propuesto.
This paper presents a system for detection of ischemic and haemorrhagic stroke in computed tomography images. Firstly, the original image is converted into grey scale and the noise is removed using the bilateral filter. Then skull regions are removed by a morphological function. The image is classified into infarct or haemorrhagic stroke based on the mean and standard deviation. A method for manual segmentation was also implemented. This method is based on the use of deformable models, specifically applying the gradient vector flow Snake algorithm. The results are segmented images for abnormal region. Experiments carried out on computed tomography images show the accuracy of the proposed system.
Hoy en día, los accidentes cerebrovasculares (ACV) son la tercera causa de muerte en el mundo después del cáncer y las enfermedades cardiovasculares1, y es una de las principales causas de discapacidad a largo plazo2.
La resonancia magnética y la tomografía computarizada (TC) son las 2 formas principales por las que los médicos pueden diagnosticar esta enfermedad3.
Es conocido que las imágenes de TC tienen ciertas ventajas sobre las de la resonancia magnética, tales como: mayor disponibilidad, menor costo y velocidad más alta. Otras ventajas pueden ser debidas a la claustrofobia, la presencia de implantes metálicos o eléctricos, o a la incapacidad de permanecer inmóvil durante la duración del examen debido a la edad, el dolor o las condiciones médicas1. No obstante, la calidad de las imágenes de TC es lo suficientemente alta como para diagnosticar con precisión los ACV.
Para apoyar el diagnóstico realizado por los especialistas se han venido desarrollando los sistemas de diagnóstico asistido por ordenador, conocidos como CAD por sus siglas en inglés, los cuales usan los ordenadores para ayudar a los médicos a llegar a un diagnóstico rápido y preciso1. Estos sistemas suelen ser de dominio específico, ya que están optimizados para ciertos tipos de enfermedades, partes del cuerpo o métodos de diagnóstico. Ellos analizan diferentes tipos de información, tales como síntomas, resultados de las pruebas de laboratorio y las imágenes médicas. Uno de los tipos más comunes de diagnóstico de las enfermedades cerebrovasculares es la que depende de las imágenes médicas.
Dentro de estos sistemas se encuentran los de detección asistida por ordenador (CADe por sus siglas en inglés)1,4, término que se refiere al reconocimiento de regiones con características sospechosas en la imagen que aportan un llamado de atención al radiólogo sobre esta zona, con el fin de disminuir los falsos negativos.
Las motivaciones para la construcción de tales herramientas son: (i) la reducción de errores observacionales humanos, y (ii) la reducción del tiempo y el esfuerzo asociado con el diagnóstico1,5. En este trabajo presentamos un prototipo de software para detectar ACV en imágenes de TC.
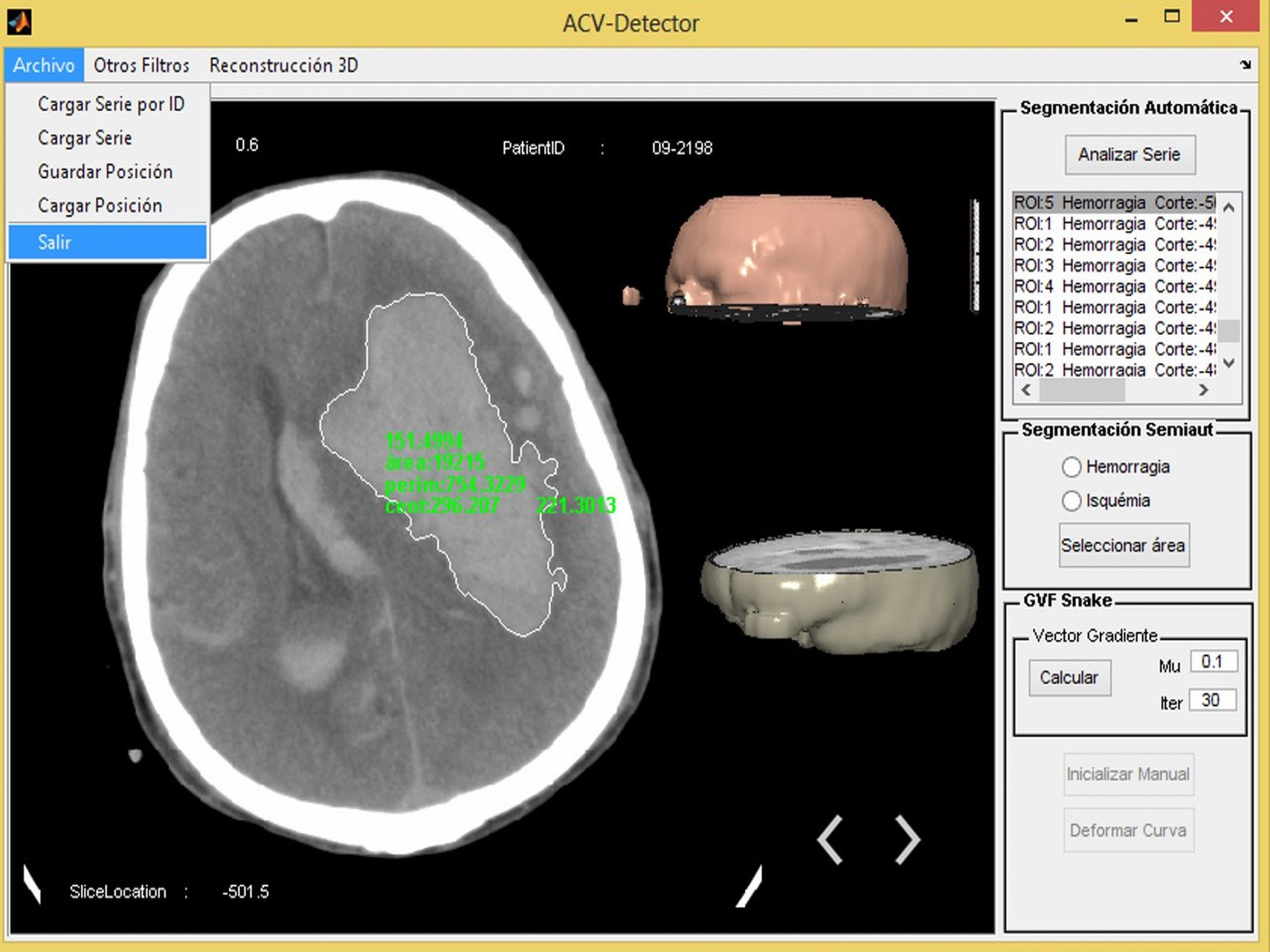
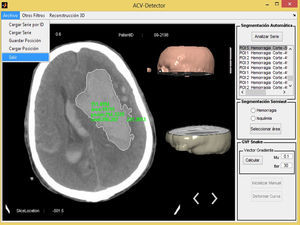
Sistema propuestoLas imágenes médicas son deterioradas por el ruido, lo que puede afectar la detección de regiones patológicas, por lo que el ruido se debe eliminar antes de que las imágenes sean utilizadas con fines de diagnóstico. En primer lugar, la imagen original fue convertida a escala de grises ajustando los valores de interés (VOI), y el ruido fue eliminado utilizando el filtro bilateral. El filtro bilateral es muy eficaz para reducir el ruido y, al mismo tiempo, preservar los bordes de las estructuras. Entonces, regiones del cráneo que pueden afectar la detección durante la etapa de segmentación se extraen mediante una función morfológica. Todos los algoritmos utilizados en nuestra herramienta se implementaron usando Matlab R2012a. La figura 1 muestra el prototipo del software propuesto.
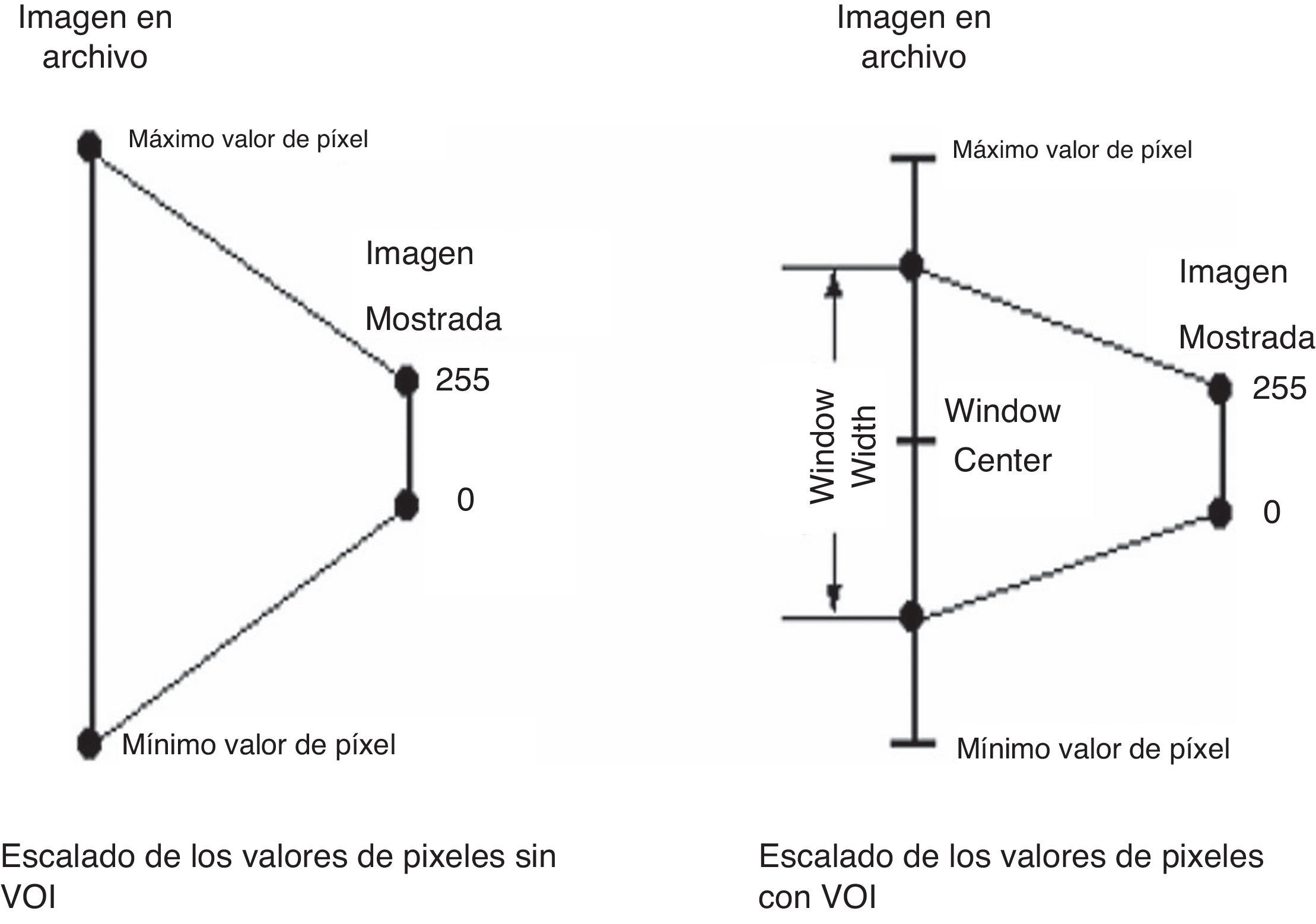
Valores de interésLas imágenes en formato DICOM usualmente contienen datos codificados en la máxima resolución alcanzable por la modalidad que las creó6.
La representación visual de esta gran cantidad de niveles de gris es posible solamente en monitores especializados, mientras que los comunes de PC solo pueden mostrar hasta 256 niveles de gris. Como resultado, los valores originales de los píxeles de una imagen deben ser «encogidos» en un rango de 0 a 2556.
Los tejidos que les interesan a los médicos usualmente ocupan una pequeña parte del rango disponible de niveles de gris, así que es razonable tratar de asignar estos rangos interesantes al rango de salida disponible. Esto es logrado exactamente aplicando las transformaciones VOI.
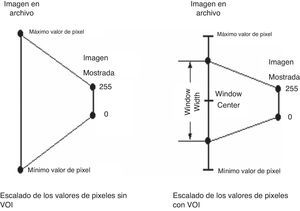
Las transformaciones VOI especifican que solo los valores de píxeles dentro del parámetro WindowWidth en la imagen original centrada alrededor del parámetro WindowCenter ocuparán completamente el rango de salida disponible, que en la mayoría de los casos es de 0 a 2556. La figura 2 muestra el escalado de los valores de píxeles con transformación VOI y sin esta.
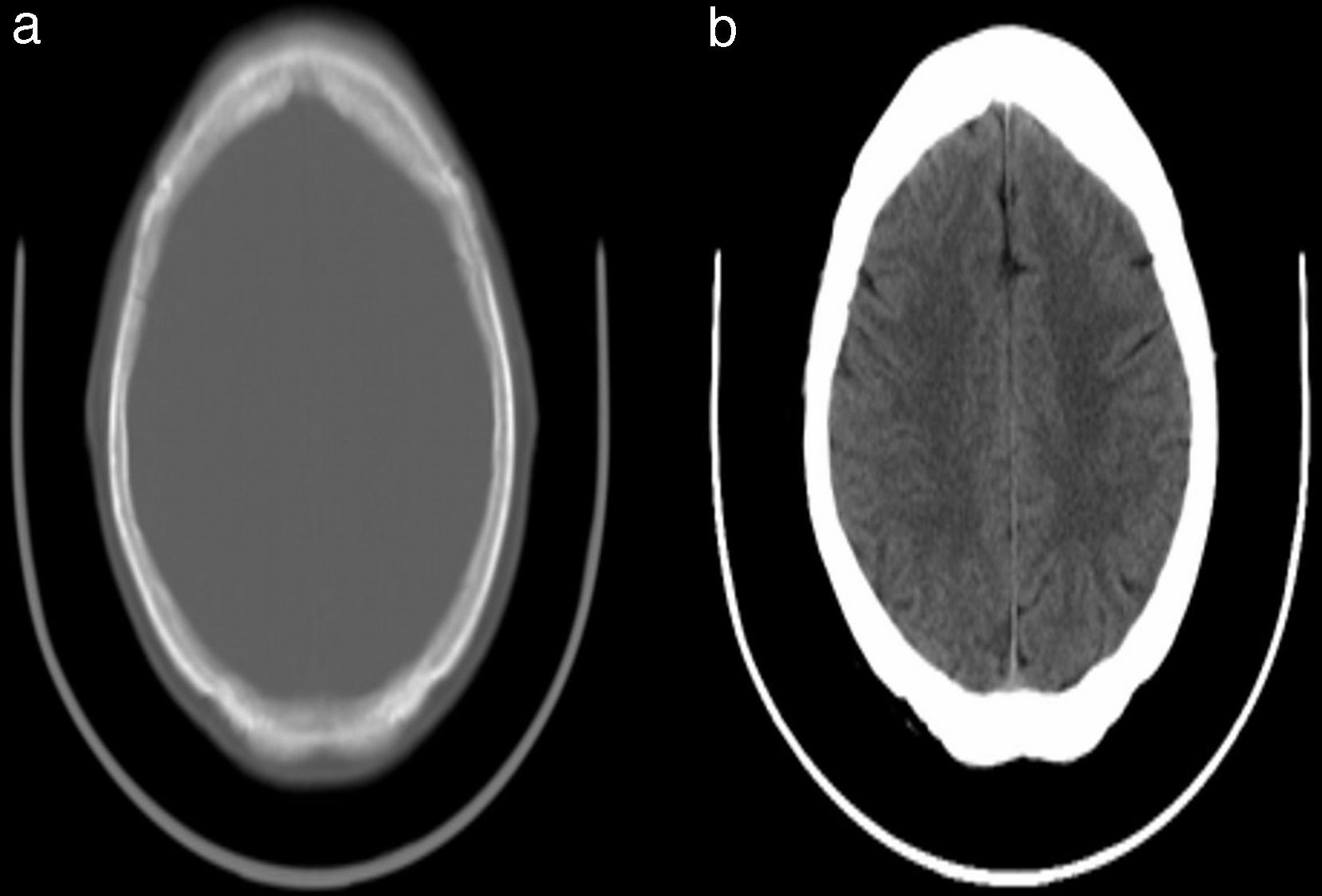
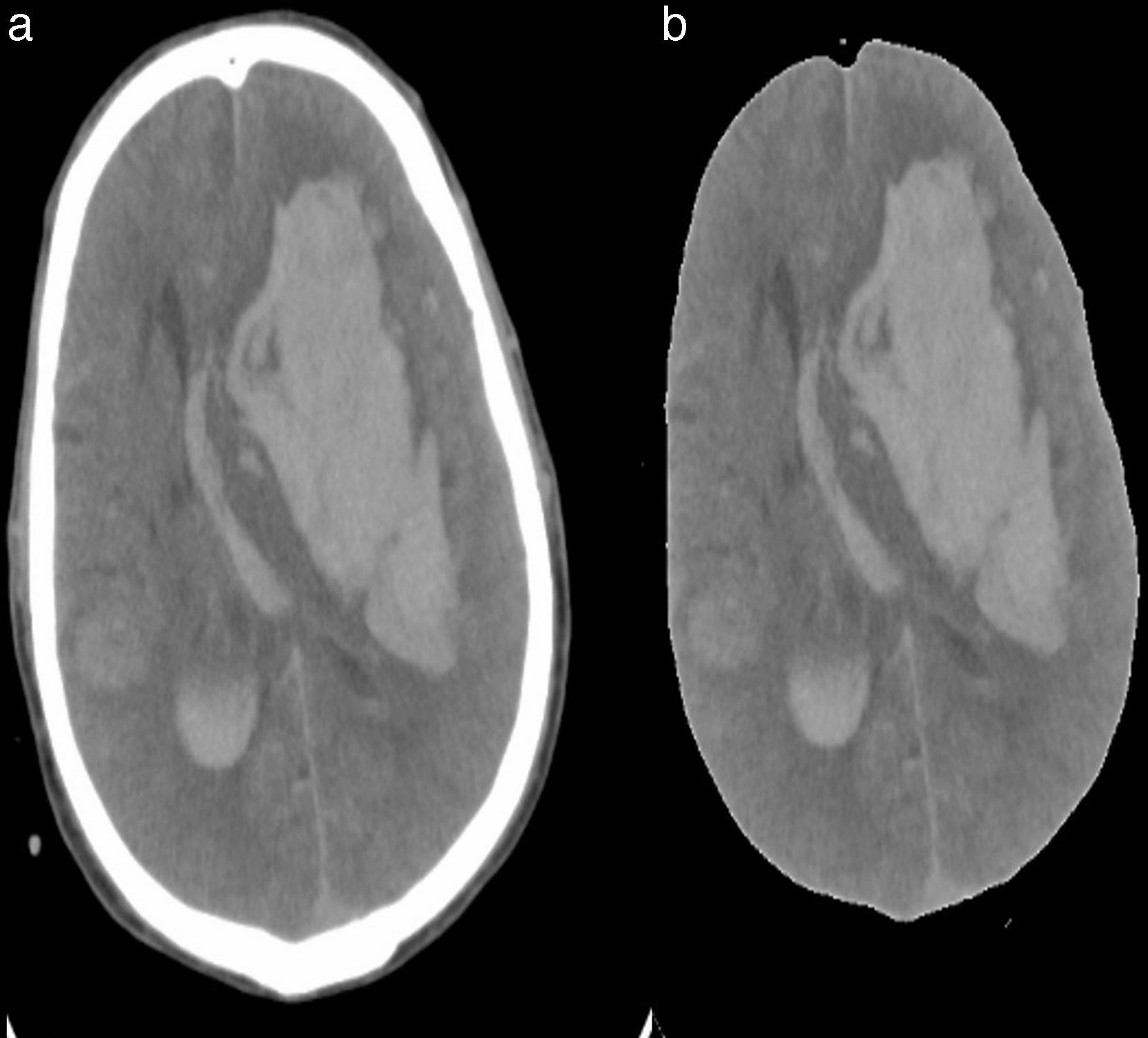
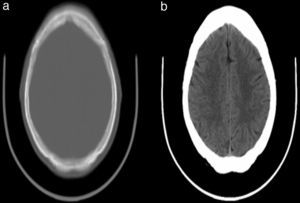
Mediante la manipulación apropiada de los parámetros VOI se puede afectar dramáticamente la manera en que los datos de la imagen son mostrados; varias características de la imagen pueden hacerse visibles o desaparecer a medida que WindowCenter y WindowWidth son modificados. Por ejemplo, consideremos las 2 imágenes que aparecen en la figura 3, ambas derivadas de la misma imagen. Nótese que en la imagen de la izquierda, a la cual no se le ha aplicado una transformación VOI, el área del cerebro es casi uniformemente gris y ninguna estructura es aparente. Sin embargo, en la imagen de la derecha, a la cual se le aplicó una transformación VOI, la estructura interna del tejido del cerebro se hace sorprendentemente evidente.
Segmentación de imágenesEl sistema incluye 3 formas de realizar la segmentación y, por tanto, la detección de las enfermedades en las imágenes. Esto se realizó para cubrir todos los casos posibles, o sea, si se necesita analizar todas las imágenes de la serie a la vez, si se desea analizar una imagen en específico de manera rápida o si se desea analizar una imagen centrándose solamente en una detección más exacta de los bordes de la afección. A estas 3 formas de trabajar se les llamó segmentación automática, segmentación semiautomática y segmentación manual, respectivamente.
Segmentación automáticaComo su nombre indica, se refiere a una segmentación donde todo el procesamiento se realiza con un solo clic; a través de esta se puede analizar toda una serie de imágenes que hayan sido cargadas previamente en la aplicación, dando como resultado todas y cada una de las afecciones encontradas en las imágenes, mostrando finalmente solo aquellas en las que se encuentra el ictus. El resto de las imágenes no se muestra, aunque el usuario puede analizarlas manualmente si lo desea.
Las imágenes fueron procesadas a través de un ciclo para cada una de las de la serie. Primero el complemento de la imagen es calculado, con el objetivo de resaltar el área patológica. Después de esto el filtro bilateral es aplicado para eliminar el ruido sin perder mucha información en la imagen.
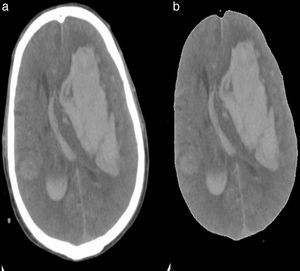
Un algoritmo fue creado para eliminar la región ósea de la imagen y dejar solo la parte que corresponde al cerebro; esto fue creado para eliminar cualquier falso positivo que pueda aparecer en otra parte de la imagen, como los cortes donde aparecen la región nasal y los globos oculares (fig. 4). Esta función produce una nueva imagen que contiene solo la región del cerebro que procedemos a binarizar con los umbrales de niveles de intensidad relacionados con el ictus.
Después del paso anterior se guardan las posiciones de las regiones y se crea una máscara de cada región. Esta máscara es multiplicada por la imagen original, obteniéndose una nueva imagen que contiene solo las regiones con sus niveles de intensidad. Con los valores de los niveles de intensidad se pasa a calcular la media y la desviación estándar, valores que nos permiten caracterizar el área y especificar el tipo de ictus.
Una vez son obtenidos estos valores el criterio para determinar si la región es un ictus o no es el siguiente:
Abs (mean_ROI−mean_ictus)≤std_average_ictusdonde mean_ROI es la media de la región de interés (ROI) segmentada, mean_ictus es la media del nivel de intensidad que representa una hemorragia o una isquemia. Esto fue determinado por el análisis de 100 imágenes con presencia de ictus hemorrágico e isquémico (50 hemorrágicas y 50 isquémicas). Finalmente, la std_average_ictus es la media de las desviaciones estándar de los niveles de intensidad de las regiones que representan un ictus de las 100 imágenes analizadas.
Si es determinado que la zona es un ictus, la información específica de esta ROI es calculada.
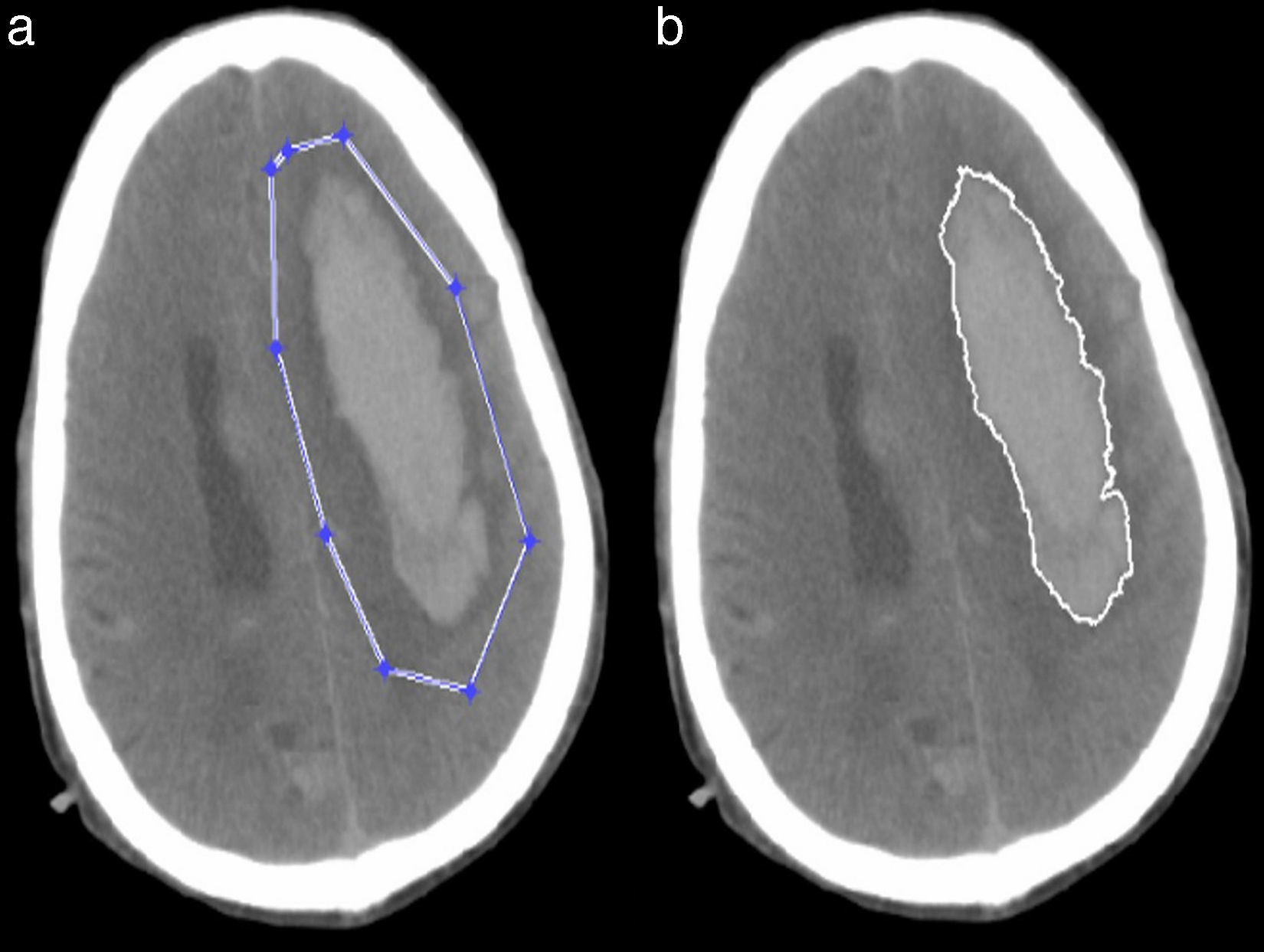
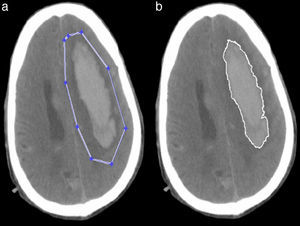
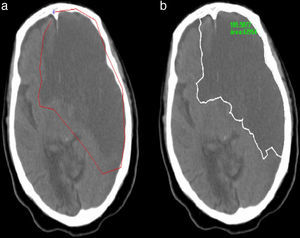
Segmentación semiautomáticaEste tipo de segmentación es muy simple y más rápida porque no analiza la imagen completa, ya que se enfoca en una región específica de la misma. En este método se usó la función del Matlab roipoly.m que permite seleccionar un polígono dentro de la imagen y hace una máscara de la región. Con esta máscara la segmentación se realizó como en la segmentación automática (fig. 5).
Segmentación manualEn este caso la segmentación se ha realizado de forma similar a la anterior; el análisis se apunta solo a una región específica de la imagen en que el usuario enfoca su atención.
Se basa en el uso de modelos deformables, aplicando específicamente el algoritmo gradient vector flow Snake7,8; de esta manera el resultado es una aproximación más exacta de los bordes de la afección buscada.
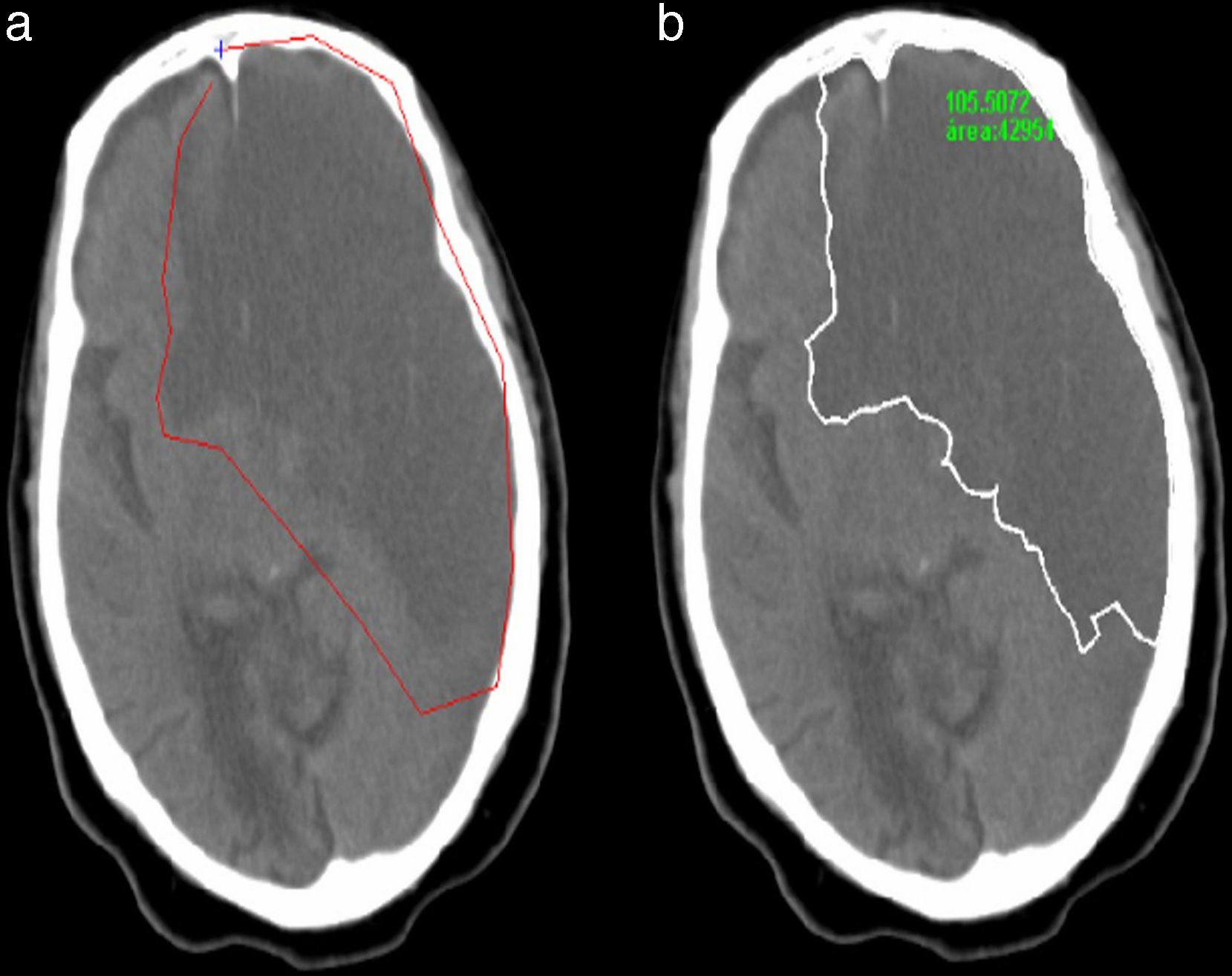
Primero calculamos el vector gradiente de la imagen para definir las fuerzas internas y externas que deformarán la curva; después, el usuario traza una curva alrededor de la región que quiere segmentar.
Finalmente, la curva trazada por el usuario se deformará, atrayéndola cada vez más cerca de los bordes de la región patológica. El resultado final no solo depende de las fuerzas calculadas, sino también de cuán cerca esté la curva inicial de la región a segmentar (fig. 6).
Una parte importante del algoritmo está en el establecimiento de un ciclo que depende del número de iteraciones definidas por el usuario en la aplicación.
Reconstrucción 3D de tejidos óseos y blandosA menudo, un ACV hemorrágico puede ser producido por lesiones en la cabeza causadas por accidentes de diversa naturaleza; en esos casos es de vital importancia mostrar el cráneo del paciente para poder analizar y tener una mejor visión de los daños.
Debido a que una serie de un paciente contiene una cantidad bastante grande de imágenes, la representación 3D de estas no es una tarea difícil, ya que toda la información se detalla en ellas, bajo el concepto de colocar una encima de la otra y conectar los puntos de cada una de las estructuras representadas; con esto se consigue una reconstrucción similar a la estructura original.
El primer paso de la reconstrucción es cargar la serie de imágenes en el sistema para crear la matriz 3D con la que se va a trabajar. Como el código permite la operación de matrices 3D, el conjunto de imágenes se concatena una encima de la otra en una matriz de tamaño 512×512×n, donde n es el número de imágenes de la serie.
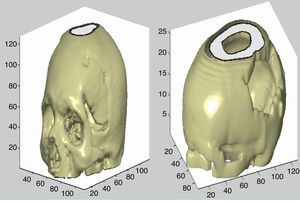
La matriz de cráneo fue creada a partir de la segmentación de este tejido en las imágenes, donde se muestra solo el hueso; para ello la imagen se binariza con un umbral alto. Después de esto se dibujan los contornos de las estructuras segmentadas, se define el grosor de las líneas representadas, se suavizan los datos, se procede a la creación de una isosuperficie y se calcula la geometría final.
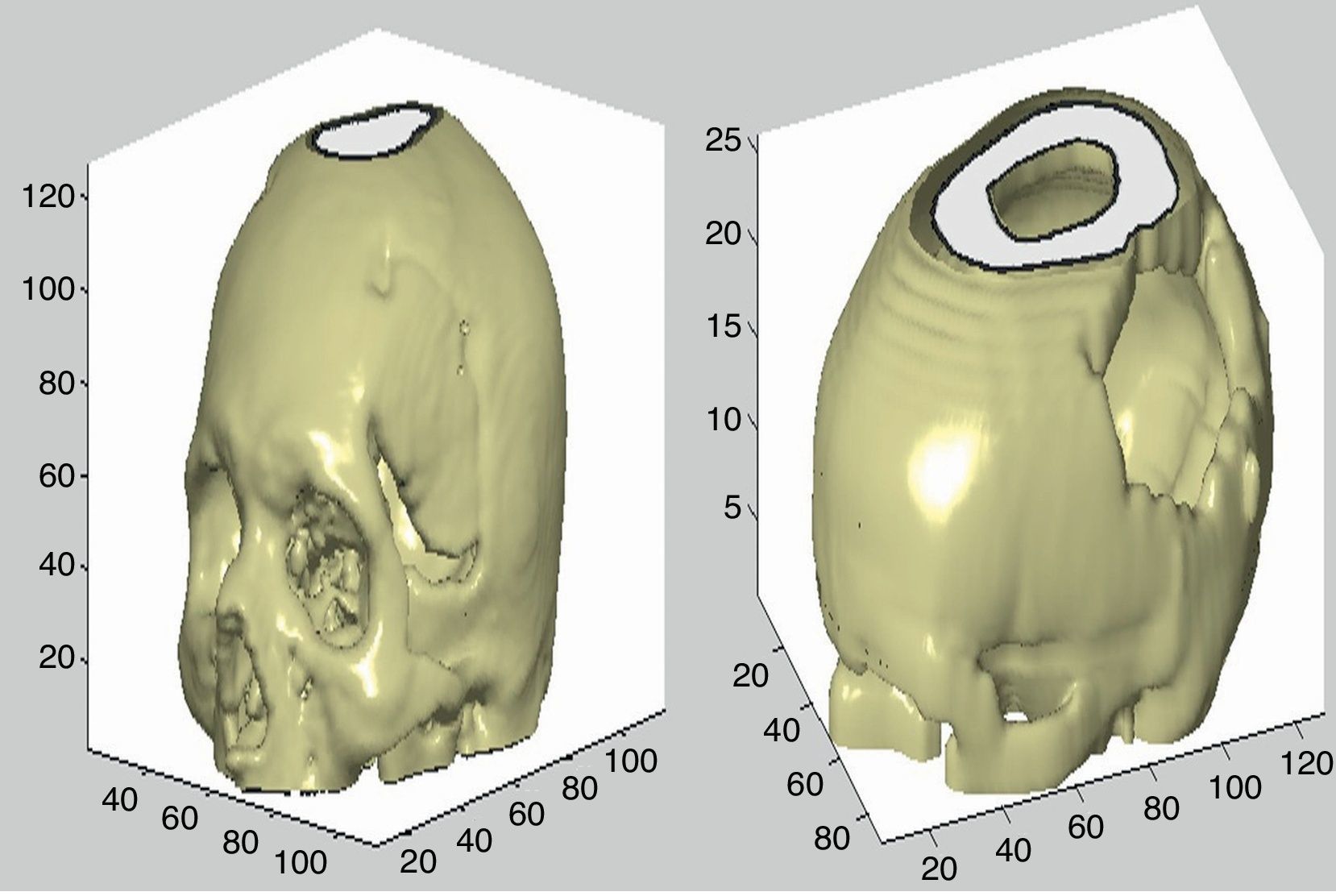
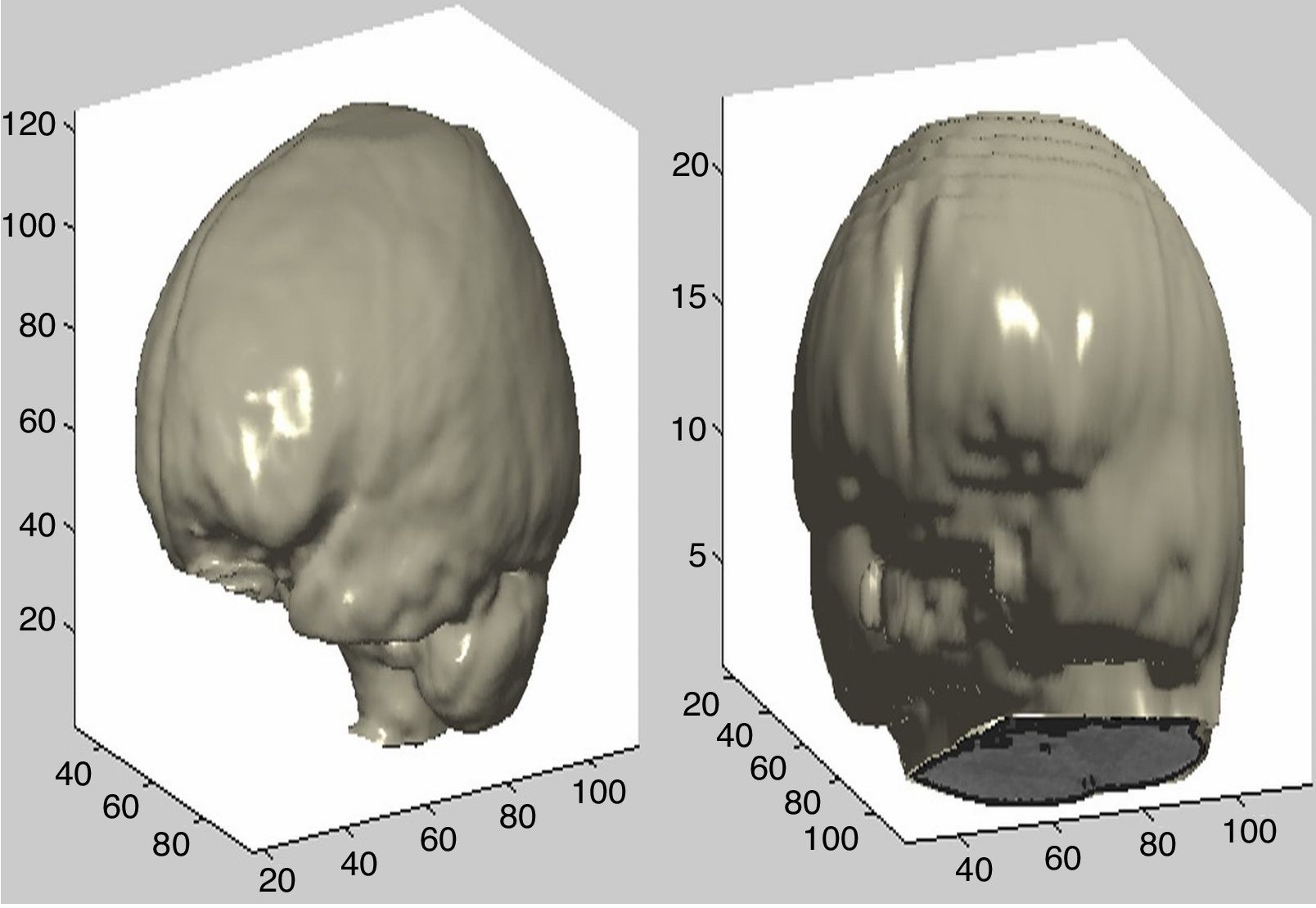
Los pasos posteriores se utilizan para añadir una fuente de luz para la visualización con brillo del objeto y la configuración de los ejes en la figura, así como los ángulos de visión, el tamaño, etc. En la figura 7 se muestran 2 ejemplos de reconstrucción ósea.
Con el uso del algoritmo descrito anteriormente para remover la parte que corresponde al tejido óseo del resto de la imagen se consigue la representación del cerebro. En la figura 8 se muestran 2 ejemplos de esta reconstrucción.
Cuanto mayor es el número de imágenes, o si el espesor de corte es más pequeño, mayor es el nivel de detalle con el que se realiza la reconstrucción 3D de las estructuras.
Resultados experimentalesEl rendimiento del sistema se ha probado en un conjunto de imágenes recogidas en el Hospital Provincial de Santiago de Cuba, junto con los resultados del diagnóstico verificados por 2 radiólogos expertos. Los datos de la TC fueron obtenidos mediante el escáner Siemens Sensation Cardiac 64.
En total, hay 345 imágenes patológicas (122 hemorrágicas, 109 isquémicas, 114 con tumores cerebrales) y 229 de pacientes sin enfermedad. Se incluyen imágenes de tumores cerebrales sin la presencia de ACV para evaluar la capacidad del sistema para detectarlos en presencia de otras enfermedades. Las medidas utilizadas para evaluar el desempeño del sistema propuesto son la precisión, la sensibilidad, la especificidad y el área bajo la curva receiver-operating characteristic. Las siguientes ecuaciones definen la precisión, la sensibilidad y la especificidad, respectivamente9:
donde: VP, verdadero positivo=casos anormales correctamente clasificados; VN, verdadero negativo=casos normales correctamente clasificados; FP, falso positivo=casos normales clasificados como anormales; FN, falso negativo=casos anormales clasificados como normales.El área bajo la curva fue obtenida por la regla trapezoidal, que es simplemente la integral de la curva.
La tabla 1 muestra el desempeño en la detección del sistema propuesto. Los falsos positivos y falsos negativos se deben principalmente a errores de detección causados en algunos casos por una pequeña diferencia entre las regiones normales y anormales, y a la presencia de imágenes en las que aparecen las regiones de la cavidad nasal con valores similares a los ACV.
Las imágenes utilizadas en nuestro experimento fueron de las últimas etapas de los procesos isquémicos. La detección de isquemias en estado prematuro es difícil, y la región se puede perder debido al nivel de contraste, especialmente cuando las imágenes de TC se examinan rápidamente y la región del infarto es demasiado pequeña.
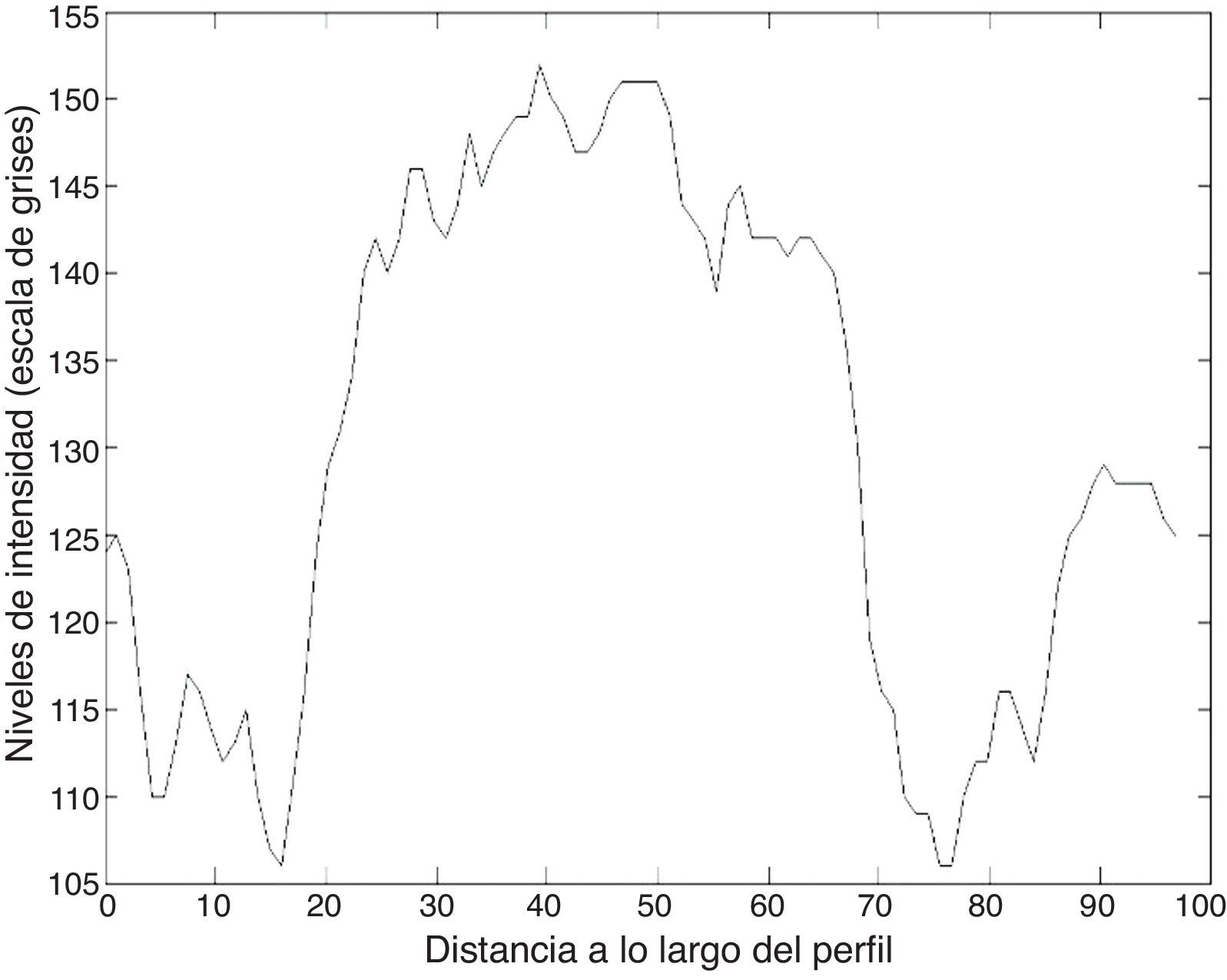
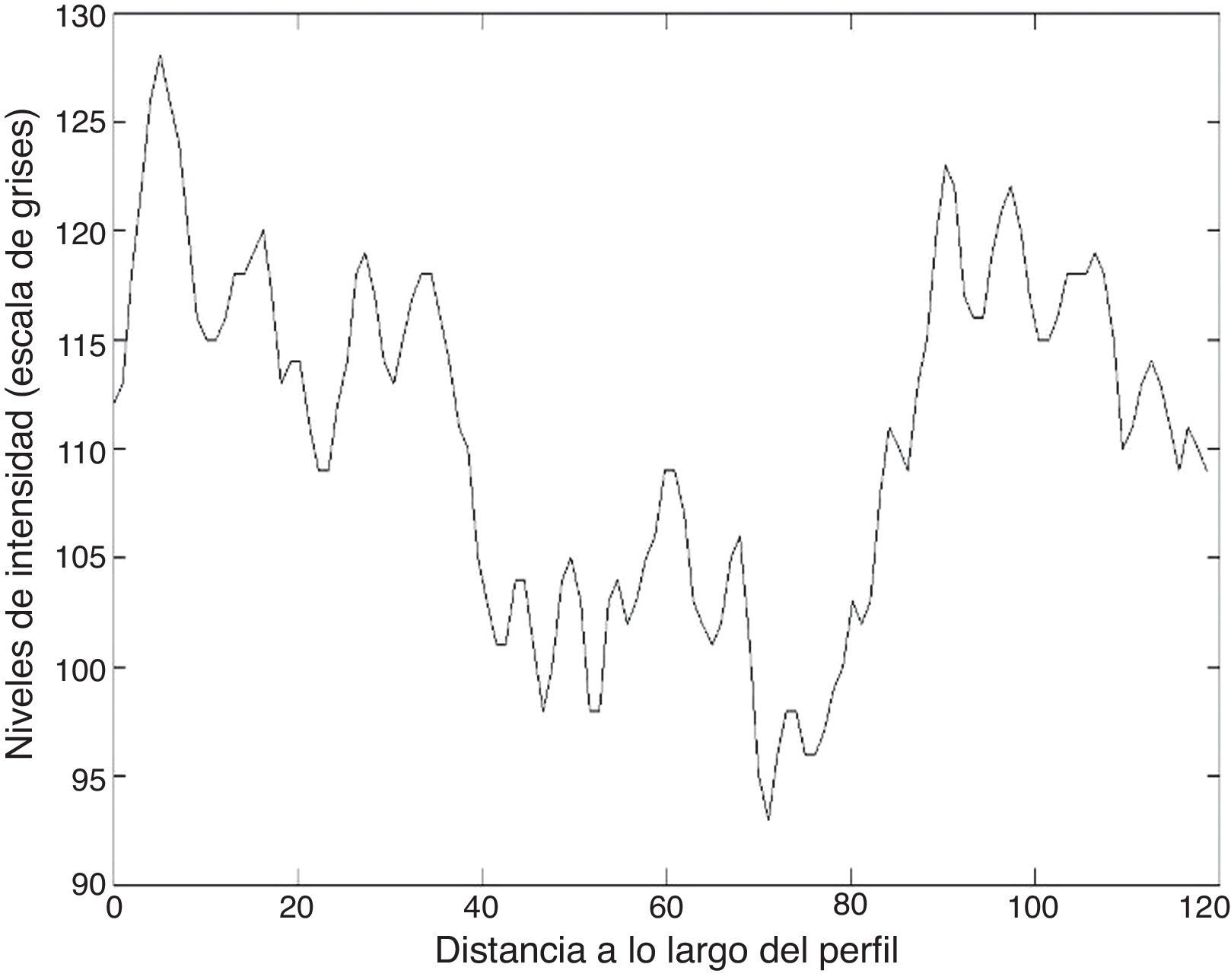
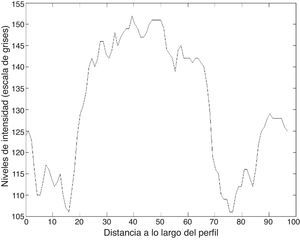
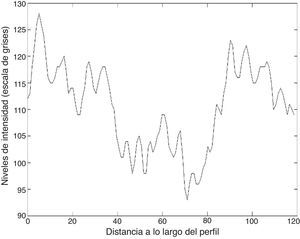
Las figuras 8 y 9 muestran los perfiles de intensidad de las regiones con presencia de ictus isquémico y hemorrágico; en ellas se pueden observar las diferencias y similitudes con otros tejidos cercanos. En el perfil hemorrágico se observa una marcada diferencia entre el tejido sano y el patológico; en cambio, en el perfil isquémico no es así, ya que se pueden observar regiones con valores similares a los de la región patológica (fig. 10).
Los valores del área bajo la curva receiver-operating characteristic para la hemorragia e isquemia fueron 0,976±0,01 y 0,01±0,864, lo que indica que la capacidad de detectar correctamente una lesión fue del 97,6 y 86,4%, respectivamente.
La tasa de errores de detección se puede reducir con una mejor caracterización de las regiones. A pesar de esto, el sistema presentado mejora la visualización de estas afecciones en las imágenes de TC y, por lo tanto, la precisión del diagnóstico.
ConclusionesEl sistema que se presenta en este trabajo permite segmentar con precisión la región afectada, así como mejorar la visualización de las distintas dolencias en imágenes del cerebro.
El ictus se pudo clasificar correctamente para el caso de las isquemias y hemorragias con una precisión del 88,6 y 98,5%, respectivamente. El sistema propuesto puede ser útil como una herramienta de aprendizaje para los estudiantes de Medicina y radiólogos en formación. Además, el sistema puede servir de ayuda a los radiólogos en la identificación del ictus, y permitir llegar a una decisión más rápida y precisa.
Los resultados preliminares muestran que los algoritmos utilizados son aplicables a otras imágenes de la TC cerebral similares. Se necesitan otros trabajos en nuestro sistema para desarrollar nuevas técnicas de segmentación con un mayor nivel de precisión que permita ampliar la detección a otras lesiones.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.