El cáncer es una de las principales causas de muerte en todo el mundo, y en muchos casos esto se debe a la detección tardía de la enfermedad, complicando su tratamiento. Los microARN son secuencias de ARN no codificantes cortos de ∼19-23 nucleótidos que juegan un papel crítico en múltiples procesos celulares a través de la regulación postranscripcional y traduccional de genes específicos, y una alteración en este mecanismo de regulación conduce al cáncer. En esta revisión se discute la desregulación de los microARN en el cáncer y, además, nosotros proponemos las aplicaciones de estas pequeñas moléculas en el tratamiento y diagnóstico del cáncer.
Cancer is one of the leading causes of death worldwide, and in many cases this is due to late detection of the disease, which complicates its treatment. MicroRNAs are short non-coding RNA sequences of ∼19-23 nucleotides that play a critical role in multiple cellular processes through post-transcriptional and translational regulation of specific genes, and an alteration in this regulatory mechanism leads to cancer. In this review, we discuss microRNA dysregulation in cancer, and propose applications of these small molecules in the treatment and diagnosis of cancer.
El Proyecto Genoma Humano reveló la existencia de aproximadamente 25,000 genes codificantes de proteínas. En tanto que dichas proteínas mayoritariamente median funciones biológicas, existen diversas barreras regulatorias a la transcripción y a la traducción génica requeridas para una función celular adecuada. Entre estas se encuentran los factores de transcripción, los cuales desempeñan un papel importante en la activación génica a través de interacciones con la región 5’ no traducida (5’UTR) del ARN mensajero (ARNm). De manera interesante, y más recientemente, también se ha encontrado que la región 3’ no traducida (3’UTR) del ARNm es importante en la regulación de la expresión génica. Los microARN (miARN) son reguladores de la eficiencia traduccional que ejercen su efecto mediante la interacción con las 3’UTR de los ARNm1 y en casos especiales con las 5’UTR. Los microARN (miARN o miR) son pequeños ARN no codificantes que regulan los perfiles de expresión de distintos genes. En consecuencia, los miARN controlan y regulan diversos procesos celulares, como la proliferación, la apoptosis y la diferenciación, todos ellos involucrados en el desarrollo y metástasis del cáncer.
La evidencia actual sugiere que los miARN se encuentran presentes en el suero y otros líquidos corporales en pacientes con diversos procesos patológicos, especialmente en el cáncer. Los diferentes perfiles de miARN específicos de pacientes con cáncer podrían conducir a una nueva manera de detectar y predecir el cáncer3-5. En la presente revisión se analizan los mecanismos de regulación de miARN involucrados en la carcinogénesis y metástasis de algunos tipos de cáncer. Finalmente, también se examinan las posibles aplicaciones terapéuticas y diagnósticas de algunos miARN.
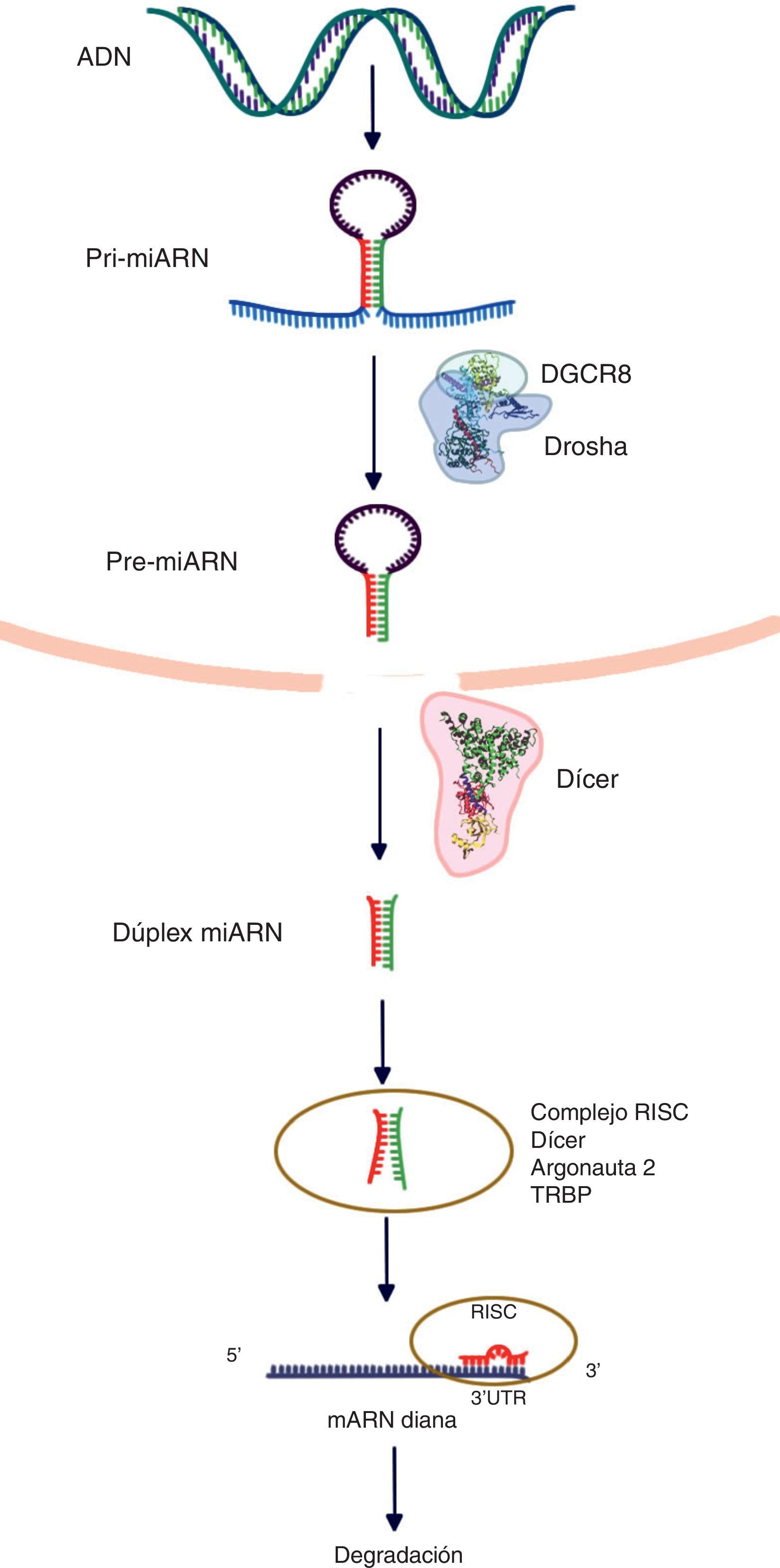
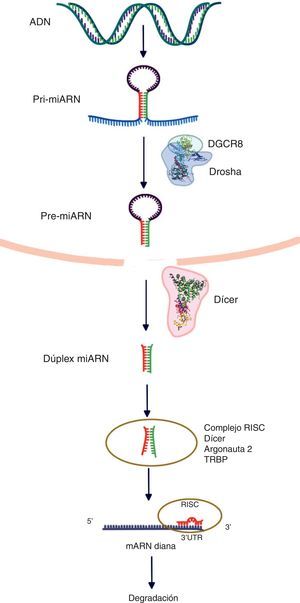
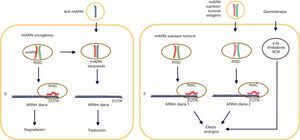
Biogénesis de los microARNLos genes de los miARN son transcritos por la ARN polimerasa II (polimerasa III en el caso de algunos miARN) y a continuación forman los nacientes, «encasquetados» y poliadenilatados transcritos primarios (3’), llamados «pri-miARN», los cuales presentan una longitud variable que oscila entre cientos y miles de ribonucleótidos. Dichos transcritos pueden ser monocistrónicos (con una sola horquilla) o policistrónicos (varias horquillas). Los miARN presentan 3 características definitorias: 1) estructura de asa; 2) protuberancias internas, y 3) estructura bicatenaria. Los pri-miARN son procesados posteriormente para formar precursores más cortos (de aproximadamente 70 nucleótidos, ahora llamados pre-miARN) por una enzima RNasa tipo III llamada «Drosha» junto con al menos otros 20 polipéptidos, como la proteína de la región crítica del gen 8 del síndrome DiGeorge (Pasha en las plantas). Subsecuentemente a la formación de pre-miARN, estos son trasportados del núcleo al citoplasma, lo cual es facilitado por la proteína exportina-5. En el citoplasma, el pre-miARN es escindido por Dicer (RNasa III), generando un dúplex de miARN de ∼22nt. En la vía canónica de procesamiento de los miARN, una de las cadenas es tomada por la proteína Argonauta (Ago) a través de un mecanismo aún desconocido. Diversos modelos hipotéticos explican la incorporación de una sola cadena para dar forma al complejo de silenciamiento inducido por miARN (miRNA-induced silencing complex [miRISC])6.
La supresión postranscripcional se logra predominantemente mediante la unión del complejo miARN-RISC en la 3’UTR del ARNm. La complementariedad de las bases entre el miARN y el ARNm influye en el resultado final de esta represión, en tanto que un emparejamiento completo produce una degradación del ARNm diana, no obstante, un emparejamiento imperfecto deriva en un secuestro de los «cuerpos P» por el ARNm diana en el interior del citoplasma. Es preciso tener presente que existe una secuencia «semilla» que debe ser estrictamente complementaria al ARNm diana (2-8 nucleótidos en el extremo 5’ de un miARN maduro)7. Las secuencias «semilla» se conservan en muchas especies y existe variación dependiendo de los genes que regulan, y generalmente se utilizan para clasificar familias de miARN. Las mutaciones en la región semilla podrían presentar cambios dramáticos en la expresión génica, conduciendo al cáncer3 (fig. 1).
Desregulación de los microARN en el cáncerLa transformación maligna es un proceso de etapas múltiples donde las células normales adquieren diferentes alteraciones genéticas y epigenéticas. En dicho proceso, las células adquieren alteraciones que favorecen el crecimiento y la supervivencia celular y afectan a los genes supresores tumorales. Por ejemplo, p53 es un reconocido supresor tumoral que monitoriza el ciclo celular e induce la apoptosis, pero se encuentra ausente o mutado en el 50% de todos los cánceres humanos1. Como resultado de dichos procesos de transformación, las células transformadas pueden volverse independientes del crecimiento, resistentes a la apoptosis, invasoras de tejidos y/o metastásicas.
El papel del miARN fue examinado por Mohammadi et al.8. Estos investigadores, utilizando un enfoque con un vector lentiviral que contenía miARN, sobreexpresaron el miARN-340 en la línea celular «MDA-MB-231» de cáncer de mama triple negativo, mostrando una declinación en la expresión del factor de transcripción SOX2 y de los inhibidores de cinasa P16 y P27 dependientes de ciclina. Asimismo, se registró un incremento en la expresión de la ciclina A2, de las cinasas dependientes de la ciclina 2 (cyclin-dependent kinases 2 [CDK2]), del inhibidor de cinasa dependiente de ciclina P18, del factor de transcripción SOX17 y de SMAD 4. Por lo tanto, miARN-340 inhibe genes asociados al cáncer de mama y debería ser investigado más ampliamente en cuanto a su uso terapéutico.
MiARN-21 es uno de los miARN oncogénicos más conocidos que regulan la expresión de diversos genes diana relacionados con el cáncer. Reportes previos señalan que miARN-21 se encuentra más elevado en el suero, plasma y tejidos de pacientes con cáncer de mama, pulmón, ovario, colon y gástrico9-11. Junto con miARN-21, miARN-146a también se muestra significativamente más elevado en muestras de plasma de pacientes con cáncer de mama12. Los niveles de expresión de miARN-21 son significativamente más altos en pacientes con cáncer de mama con enfermedad en estadio III y mal pronóstico13.
Los polimorfismos de un solo nucleótido (single nucleotide polymorphisms [SNP]) que residen dentro de los genes del miARN potencialmente podrían alterar diversos procesos biológicos al influir en la biogénesis del miARN y alterar la selección de dianas. Los SNP y las mutaciones en las secuencias de los miARN o en los sitios diana de los miARN pueden afectar el proceso de maduración o la selección de dianas, respectivamente. Varios estudios investigaron las repercusiones de los polimorfismos de los miARN y el riesgo de diversos cánceres14,15. Li et al.16 encontraron que las asociaciones entre los SNP rs3746444 en miARN-499 y rs4919510 en miARN-608 se asocian significativamente a un incremento en el riesgo de cáncer pulmonar. Dichos polimorfismos producen un decremento significativo en la expresión de miARN-499 y miARN-608 en comparación con los tejidos normales adyacentes. Además, el polimorfismo en miARN-499 incrementa el riesgo de cáncer de próstata14.
En el carcinoma colorrectal, Christensen et al.17 encontraron que el incremento en la expresión de miARN-362-3p se relaciona con la interrupción del ciclo celular y la inhibición del crecimiento tumoral, lo cual sugiere que este incremento en la expresión se asocia a un mejor pronóstico en aquellos pacientes que han recibido algún tratamiento previo como, por ejemplo, quimioterapia. Nordentoft et al.18 publicaron un análisis de casi 600 miARN en tumores de pacientes con cáncer de vejiga en progresión donde los pacientes fueron tratados con cisplatino y encontraron que 15 miARN desempeñan un papel importante en dicha respuesta, 5 en el intervalo de supervivencia y 3 en ambos aspectos. En células de cáncer de ovario sensibles a cisplatino, la sobreexpresión de miARN-21 disminuyó los niveles de «muerte celular programada 4» (programmed cell death 4 [PDCD4]) e incrementó la proliferación celular. Sin embargo, el bloqueo de JNK-1, el principal activador de la fosforilación de c-Jun, redujo la expresión de pre-miARN-21 e incrementó la expresión de su gen diana, PDCD4. Por lo tanto, la vía JNK-1/c-Jun/miARN-21 contribuye a la resistencia al cisplatino en el cáncer ovárico11.
Papel de los microARN en la metástasisLa metástasis se refiere a la diseminación de un centro canceroso a un lugar distinto de la parte del organismo en que se inició. Normalmente esto ocurre a través de las vías sanguínea o linfática. Aproximadamente el 98% de las muertes causadas por cáncer no detectado se deben a este proceso de migración de las células cancerígenas19. El papel de los miARN en el desarrollo de metástasis fue observado inicialmente por Ma et al.20. Años después, estos investigadores descubrieron que la sobreexpresión de miARN-10b era el responsable del inicio del cáncer de mama y de su metástasis a pulmón21,22. Asimismo, en el glioma, miARN-10b presenta una expresión elevada, especialmente en los gliomas de grado avanzado23. La inhibición de miARN-10b produce efectos pleiotrópicos en el crecimiento de líneas celulares de glioma, mas no en el crecimiento de neuronas y astrocitos normales24.
La sobreexpresión de miARN-320a inhibe la capacidad de invasión y migración del cáncer de mama in vitro, en tanto que el silenciamiento de miARN-320a podría favorecerla. Este efecto se debe a que miARN-320a suprime el potente oncogén metadherina. Experimentos adicionales con xenoinjertos también mostraron que miARN-320a podía inhibir la metástasis del cáncer de mama in vivo25. Asimismo, en el cáncer pancreático, la sobreexpresión de miARN-320a contribuyó fuertemente a la adquisición de características tales como altos niveles de proliferación, invasión, metástasis y farmacorresistencia, así como a la transición epitelio-mesenquimal al actuar selectivamente sobre PDCD426.
Publicaciones recientes muestran que la sobreexpresión de miARN-335 suprime la metástasis y la migración27 por medio del silenciamiento del factor de transcripción SOX4, la «tenascina C» (proteína de la matriz celular) y el gen de secuencia pareada 6 (paired box 6 [PAX6]) en la línea celular MCF-7 de cáncer de mama. La señalización no canónica del factor de crecimiento transformante β (TGF-β) es regulada por la acción selectiva sobre la proteína cinasa 1 asociada a Rho con motivo estructural de hélice superenrollada (Rho-associated coiled-coil containing protein kinase [ROCK1]) y MAPK1, así como por la inhibición de la expresión de ambas por miARN-335, lo cual deriva en un decremento de la fosforilación de la proteína motora de la cadena ligera de miosina (myosin light chain [MLC]), conduciendo a una inhibición significativa del potencial invasivo y migratorio de las células de neuroblastoma28.
El fármaco hipoglucemiante metformina (clorhidrato de 1,1-dimetilbiguanida) mata las células precursoras del cáncer. El cáncer de mama triple negativo es más sensible a los efectos de metformina, y la ácido graso sintasa (fatty acid synthase [FASN]) es uno de los genes cuya expresión se ve más significativamente reducida tras 24h de tratamiento por un incremento en la expresión de uno de los miembros de la familia miARN-193, miARN-193b29. La expresión de miARN-193b actúa como supresor tumoral en el cáncer pancreático y se encuentra notoriamente disminuida en los tejidos con neoplasia avanzada. Líneas celulares transfectadas con miARN-193b exhibieron un decremento significativo en la proliferación, la migración y la invasividad30.
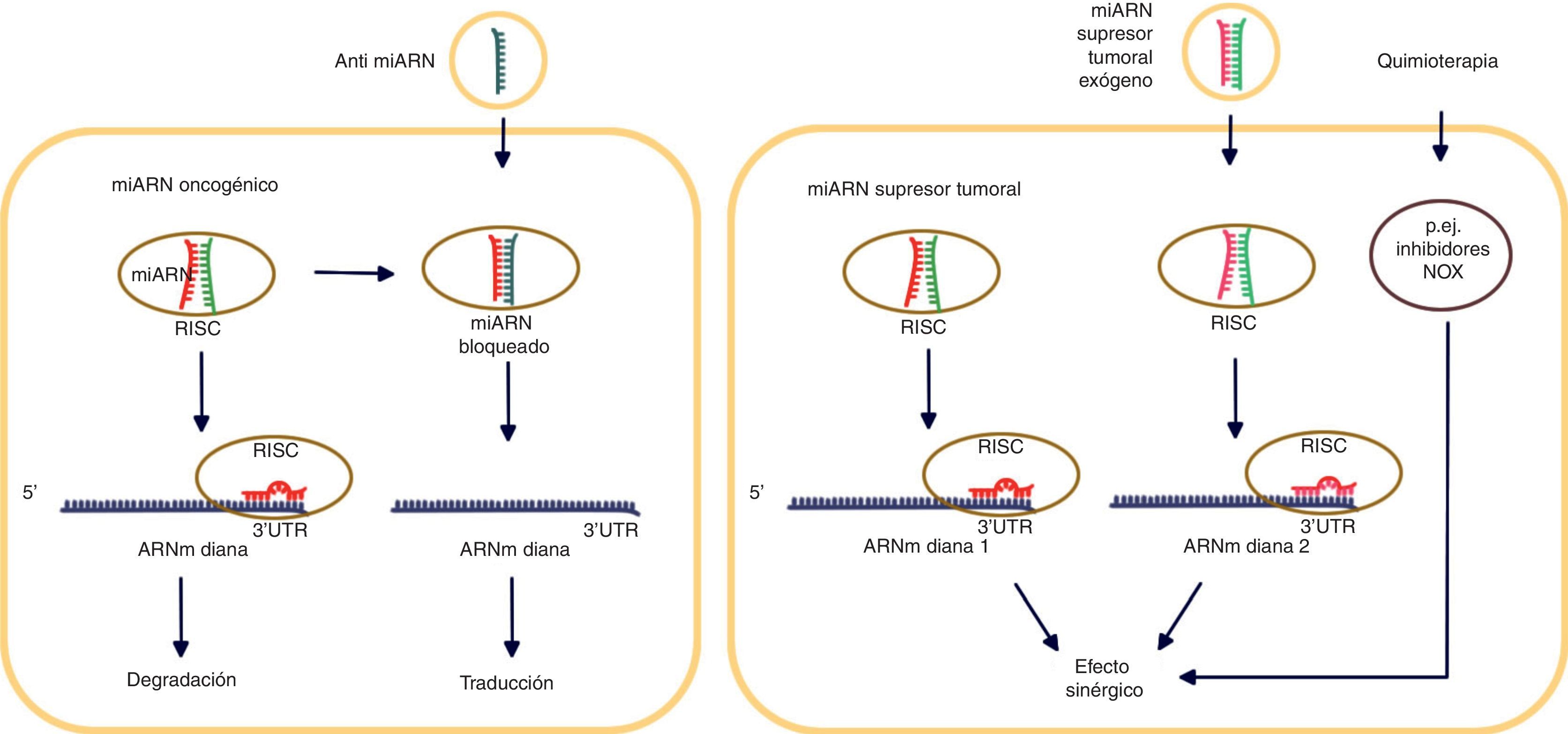
Los microARN en el tratamiento del cáncerDescubrimientos recientes muestran que cada cáncer posee una combinación específica de miARN, ya sea oncomiARN sobreexpresados o miARN supresores tumorales con expresión inhibida31. Este perfil de cada uno de ellos podría establecerse como una «huella digital» para diseñar terapéuticas personalizadas a fin de contar con tratamientos específicos para pacientes con cáncer. Algunas de las estrategias terapéuticas antineoplásicas más efectivas in vitro consisten en el diseño de anti-miARN para el silenciamiento de genes que se sobreexpresan en el cáncer, como el receptor del factor de crecimiento epidérmico (epidermal growth factor receptor [EGFR])32 (fig. 2). Sin embargo, barreras fisiológicas y celulares obstaculizan la eficacia in vivo de las tecnologías anti-miARN.
MicroARN en el tratamiento del cáncer. Se emplean distintas estrategias dependiendo del efecto del miARN. Los anti-miARN bloquean la unión de los miARN oncogénicos con su ARN diana. Los miARN supresores tumorales pueden actuar sinérgicamente cuando se combinan los miARN endógenos con los exógenos. La quimioterapia también muestra propiedades sinérgicas que actúan junto con los miARN.
Como se mencionó previamente, se ha constatado que uno de los primeros miARN detectados en el genoma humano, miARN-21, se sobreexpresa en el glioblastoma33 y podría ser utilizado como objetivo terapéutico en este tipo de cáncer. En las células de glioblastoma, la interacción aditiva de inhibidores tanto de miARN-21 como de miARN-10b (oligonucleótido antisentido) puede constituir una estrategia terapéutica efectiva para controlar el crecimiento del glioblastoma mediante la inhibición de la expresión de oncogenes y la sobreexpresión de genes supresores tumorales. De manera independiente, un inhibidor de miARN-21 puede interrumpir la actividad de las vías del EGFR, incrementando de este modo la expresión de PDCD4 y TPM1 y reduciendo las actividades de las MMP. El valor IC 50 descendió dramáticamente en células tratadas con una combinación de inhibidores de miARN-10b y miARN-21. Además, dichos inhibidores, combinados sinérgicamente, incrementaron la apoptosis de manera significativa y redujeron la capacidad de invasión34.
Existen 2 retos relacionados con la manipulación de la función de los miARN. El primero concierne a la identificación adecuada de miARN (diseño in silico) que pueden inhibir o imitar de manera efectiva la función de miARN específicos, con el objeto de lograr la pérdida o ganancia de funciones de los miARN, respectivamente. El segundo reto consistiría en la eficiencia de la liberación por tiempo prolongado de dichas moléculas en sus sitios diana específicos. Sin embargo, Cheng et al.35 desarrollaron recientemente una nueva forma de liberación de miARN específicamente dirigida al miARN-155 en un microambiente tumoral ácido en linfoma murino, evadiendo la barrera hepática y facilitando una ruta mediante una vía endocítica no específica.
En la actualidad se están desarrollando estrategias para hacer llegar el miARN al interior de la célula recurriendo a diferentes enfoques. Por ejemplo, Song et al.36 evaluaron el péptido R3V6 como transportador de oligodeoxinucleótidos antisentido. El ensayo de estabilidad en suero mostró que R3V6 los protegió de las nucleasas más eficientemente que la polietilenimina (PEI; 25kDa, PEI25k). En un ensayo de transfección in vitro, el péptido R3V6 transportó el oligodeoxinucleótido antisentido anti-miARN-21 al interior de las células más eficientemente que PEI (25kDa, PEI25k) y lipofectamina.
La quimioterapia y la terapia con miARN mostraron actividades antineoplásicas sinérgicamente incrementadas. Por ejemplo, doxorubicina (DOX) previene la replicación del ADN en las células de cáncer mediante la regulación epigenética de la transcripción génica a través de la inhibición de la expresión de la ADN metiltransferasa 1 (DNMT1); posteriormente, DOX reactiva genes supresores tumorales silenciados por la metilación del ADN en el glioblastoma33. Asimismo, taxol incrementa su eficacia en células de glioblastoma tratadas con un inhibidor de miARN-21. La combinación de un inhibidor de miARN-21 y taxol podría ser una estrategia terapéutica efectiva para controlar el crecimiento del glioblastoma multiforme (GBM) mediante la inhibición de la expresión y la fosforilación de STAT337. De manera similar, la inhibición de NADPH oxidasa (NOX) abatió drásticamente el potencial invasivo del cáncer pulmonar in vitro a través del incremento de PTEN y el decremento de MMP9 inducidos por miARN-2138 (fig. 2).
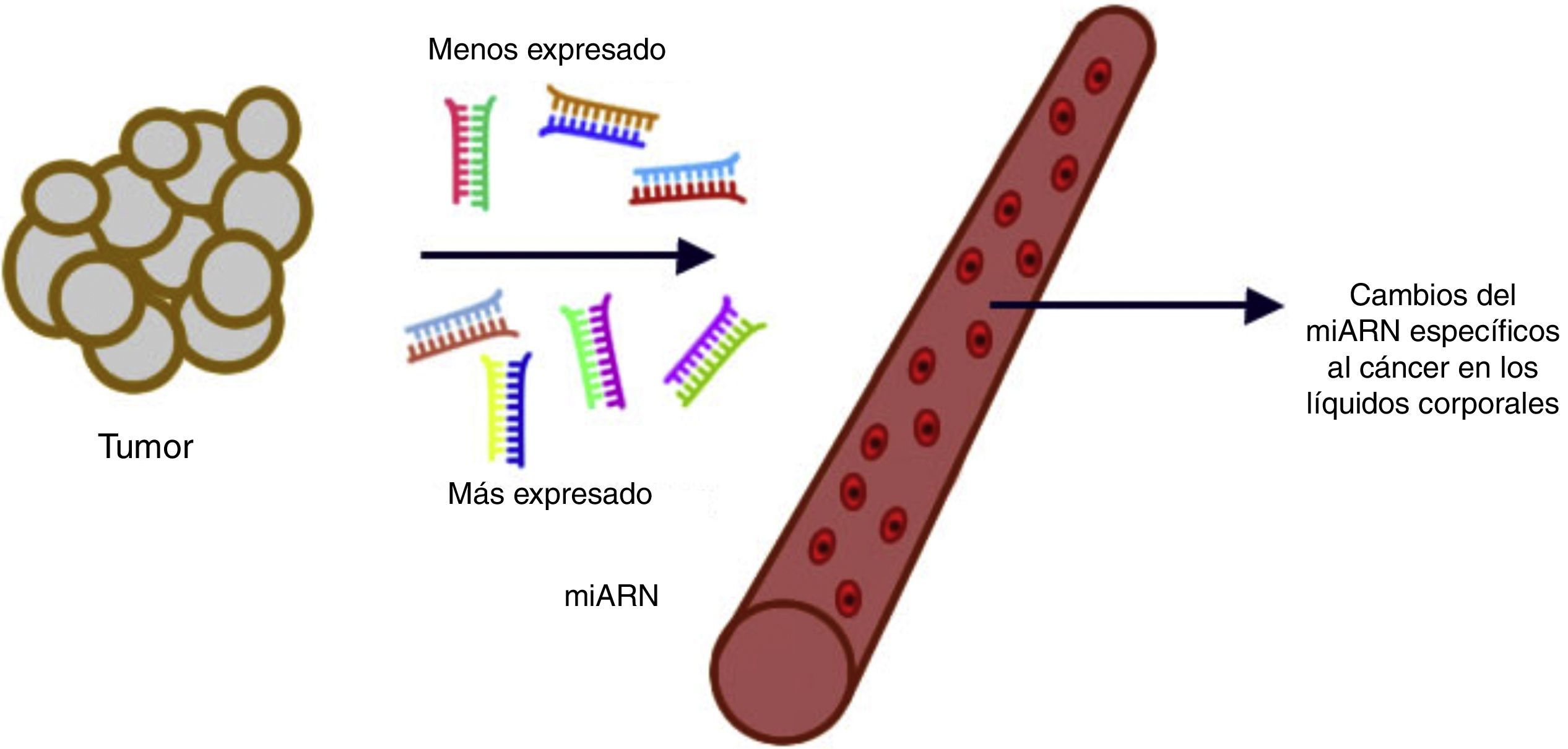
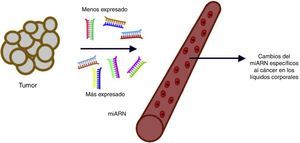
Los microARN en el diagnóstico del cáncerPara el diagnóstico oportuno del cáncer, es crítica la caracterización de biomarcadores sensibles y específicos, preferiblemente de aquellos que circulan en los líquidos del organismo. La detección temprana de la enfermedad con una prueba de escrutinio mínimamente invasiva incrementará de manera significativa la efectividad del tratamiento y disminuirá su costo. En la actualidad, los miARN están cobrando creciente importancia como los principales reguladores de la expresión génica, lo cual los convierte en una poderosa herramienta para la detección de transformaciones malignas incipientes. Los niveles disminuidos de algunos miARN con supresores tumorales se deben a las proteínas disfuncionales involucradas en su biogénesis; este resultado es consecuente con la observación de una disminución general en la expresión de miARN maduro en células cancerosas en comparación con su expresión en tejidos normales39. La desregulación de la expresión del miARN puede ser el resultado de una alteración genética. Se encontró que el 53% de los genes de miARN se localizaban en sitios frágiles, donde es mucho más probable que presenten susceptibilidad a las mutaciones, lo cual respalda firmemente la idea de que los miARN desempeñan un papel crucial en el desarrollo del cáncer. Se ha reportado que en algunos tumores se presenta una pérdida de expresión de Dicer. La inhibición de la expresión de maquinarias importantes de la biogénesis del miARN como Drosha, DCGR8 y Dicer disminuye sustancialmente la producción de miARN y promueve un fenotipo de células más transformado1-5,40.
Se pueden asignar perfiles de miARN significativamente diferentes a diversos tipos de tumores, los cuales podrían servir como firmas fenotípicas en distintos cánceres para su explotación en el diagnóstico, el pronóstico y el tratamiento del cáncer. Si los perfiles de miARN pueden predecir neoplasias malignas con precisión, esta tecnología podría explotarse como una herramienta para superar los retos diagnósticos41. Varios estudios recientes han revelado que los miARN son detectables establemente en plasma/suero. Debido a su liberación a la circulación y a su extraordinaria estabilidad, los niveles de miARN en plasma y en otros líquidos biológicos pueden servir como biomarcadores diagnósticos y pronósticos de enfermedades42 (fig. 3).
ConclusiónLa expresión de los miARN se encuentra alterada en el proceso de carcinogénesis y puede ser inhibida o estimulada dependiendo del papel de cada miARN en particular. Por lo tanto, se pueden diseñar oncomiARN para inhibir a los miARN involucrados en el silenciamiento de genes supresores tumorales y miARN para silenciar oncogenes y de algún modo tratar el cáncer. Asimismo, algunos miARN podrían predecir el desarrollo de algunos tipos de cáncer y, en un futuro cercano, esta tecnología podría explotarse como herramienta para superar los retos diagnósticos.
AutoríaEl presente manuscrito se llevó a cabo parcialmente con la tutoría de Rodríguez W. en la Universidad Nacional de Piura.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
AgradecimientosA CONCYTEC a través de CIENCIACTIVA por su apoyo financiero mediante una beca de maestría en la Universidad Peruana Cayetano Heredia.