Introducción
La sensibilidad a los antibióticos betalactámicos difiere entre las distintas especies del género Proteus.
P. mirabilis es uno de los miembros de la familia Enterobacteriaceae más sensibles, debido entre otras causas a la ausencia de betalactamasas cromosómicas. La resistencia en esta especie se debe fundamentalmente a la adquisición de betalactamasas plasmídicas, siendo TEM-1 la más frecuentemente detectada1.
El aislamiento de cepas resistentes a cefalosporinas de tercera generación se ha ido incrementando, alcanzando un 18% en esta especie en Argentina2.
Las betalactamasas de espectro extendido (BLEE) de mayor prevalencia en este país son CTX-M-2, una cefotaximasa con escasa actividad sobre ceftazidima y aztreonam, y PER-2, que a diferencia de CTX-M-2 tiene también actividad de ceftazidimasa2-4. De hecho, el primer aislamiento productor de PER-2 en nuestro país fue P. mirabilis5.
Las otras dos especies del género presentan naturalmente una mayor resistencia a los betalactámicos, debido a la producción de una cefuroximasa cromosómica inducible, que confiere resistencia a penicilinas, cefalosporinas de primera generación y cefuroxima.
Los objetivos de este trabajo fueron evaluar el perfil de sensibilidad a los antibióticos betalactámicos en el género Proteus, detectar las enzimas responsables de la resistencia a estos antimicrobianos y evaluar los fenotipos y genotipos de resistencia a las cefalosporinas de tercera y cuarta generación en P. mirabilis.
Métodos
Microorganismos
Se estudiaron 99 aislamientos únicos, consecutivos e ininterrumpidos de Proteus (87, P. mirabilis; 10 pertenecientes al complejo P. vulgaris, y 2, P. penneri) aislados de pacientes atendidos en el Hospital de Clínicas José de San Martín de Buenos Aires, Argentina, durante el período comprendido entre septiembre de 1998 y mayo de 2000.
Pruebas de sensibilidad a los antimicrobianos y detección de BLEE
La sensibilidad a diferentes antibióticos betalactámicos (solos y en combinación con inhibidores de betalactamasas) se determinó por el método de dilución en agar, siguiendo las recomendaciones del National Committee for Clinical Laboratory Standards (NCCLS)6. Los antibióticos ensayados fueron: amoxicilina, amoxicilina-ácido clavulánico (2:1), piperacilina, piperacilina-tazobactam (concentración de tazobactam: 4 mg/l), cefalotina, cefoxitina, cefuroxima, imipenem y ácido clavulánico. Cefotaxima, ceftazidima, cefepima y aztreonam se ensayaron solos y asociados a 4 mg/l de ácido clavulánico.
Se detectó la presencia de BLEE utilizando el ensayo de sinergia de doble disco, colocando discos de ceftazidima (30 mg) y ceftriaxona (30 mg) a 25 mm de un disco de amoxicilina-ácido clavulánico (20/10 mg) (Difco, EE.UU.)7. Se incluyó también el ensayo confirmatorio de BLEE propuesto por el NCCLS para Escherichia coli, Klebsiella pneumoniae y K. oxytoca determinando la concentración inhibitoria mínima (CIM) por dilución en agar de cefotaxima, ceftazidima, cefepima y aztreonam solos y asociados a 4 mg/l de ácido clavulánico6.
Se sospechó la presencia de BLEE cuando la CIM de una o más cefalosporinas de tercera o cuarta generación fue mayor o igual a 1 mg/l. Se confirmó fenotípicamente la presencia de BLEE cuando se observó sinergia entre los discos de las cefalosporinas de tercera generación y amoxicilina-ácido clavulánico y/o cuando la relación entre la CIM de cefotaxima, ceftazidima, cefepima y/o aztreonam asociados al inhibidor de betalactamasa disminuyó por lo menos tres diluciones con respecto a la CIM de las cefalosporinas solas.
Determinación de los puntos isoeléctricos de las betalactamasas
Los extractos enzimáticos se obtuvieron a partir de cultivos de los microorganismos en 50 ml de caldo cerebro-corazón con 100 mg/l de ampicilina (Argentia, Argentina) o 50 mg/l de cefotaxima (Roche, Argentina) para las cepas productoras de BLEE. Los cultivos se centrifugaron a 8.000 rpm (Sorvall, Rotor SS 34) a 4 °C durante 20 min y las células se resuspendieron en 2 ml de búffer fosfato 20 mM de pH 7,0. Las suspensiones celulares se sometieron a rotura ultrasónica en baño de hielo para evitar la desnaturalización proteica, utilizando un sonicador Vibra Cell (Sonics & Materials Inc, EE.UU.). Se eliminaron los restos celulares por centrifugación a 15.000 rpm (Sorvall Rotor SS 34) a 4 °C durante 30 min.
Se determinó el punto isoeléctrico (pI) de las enzimas por isoelectroenfoque analítico8 empleando geles de poliacrilamida de amplio rango (pH 3-10). Se incluyeron enzimas de pI conocido como patrón. Se empleó un equipo LKB 2117 Multiphor II (LKB Produkter AB, Suecia). Las betalactamasas fueron reveladas por el método iodométrico en agar utilizando ampicilina (500 mg/l) y cefotaxima (1.000 mg/l)9.
Detección molecular de los genes codificantes de BLEE
Se amplificó el gen codificante de la BLEE utilizando iniciadores deducidos a partir de la secuencia del gen blaCTX-M-24: bla CTX-M-2 I: 5'TTAATGATGACTCAGAGCATTC 3' (GIBCO BRL, EE.UU.), bla CTX-M-2 II: 5' GATACCTCGCTCCATTTATTG 3'(GIBCO BRL, EE.UU.)10.
Se utilizó ADN plasmídico obtenido por lisis alcalina11 de las cepas de P. mirabilis resistentes a cefalosporinas de tercera y cuarta generación.
Los parámetros de amplificación cumplidos fueron: desnaturalización del ADN a 95 °C durante 5 min, annealing a 48 °C durante 15 min y 30 ciclos que incluían 1 min a 95 °C, 1 min a 48 °C y 90 s a 72 °C; y un período de extensión final de 20 min a 72 °C. Se empleó un termociclador Matercycler 5330 (Eppendorf, Alemania).
Los fragmentos amplificados por reacción en cadena de la polimerasa (PCR) se secuenciaron automáticamente en ambas cadenas empleando el ABI PRISM 3700 DNA.
Resultados
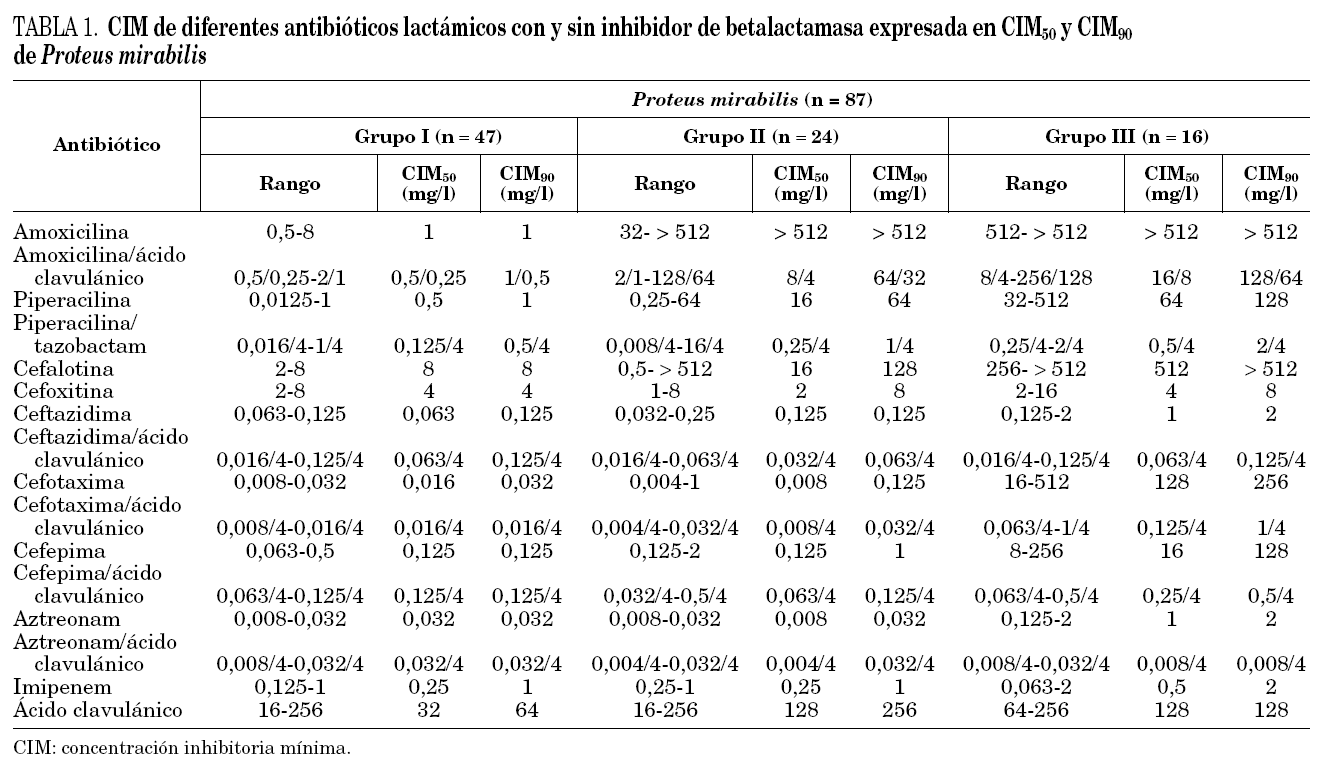
Los aislamientos de P. mirabilis se dividieron en 3 grupos de acuerdo a su sensibilidad a los antibióticos betalactámicos. De las 87 cepas estudiadas, 47 (54%) fueron sensibles a todos los antibióticos betalactámicos ensayados (grupo I); 24 (28%) fueron sólo resistentes a las aminopenicilinas y cefalosporinas de primera generación (grupo II), y 16 (18%) resultaron resistentes a alguna de las cefalosporinas de tercera generación ensayadas (grupo III). Los valores de CIM50, CIM90 y los valores de rango de CIM de cada antibiótico se detallan en la tabla 1.
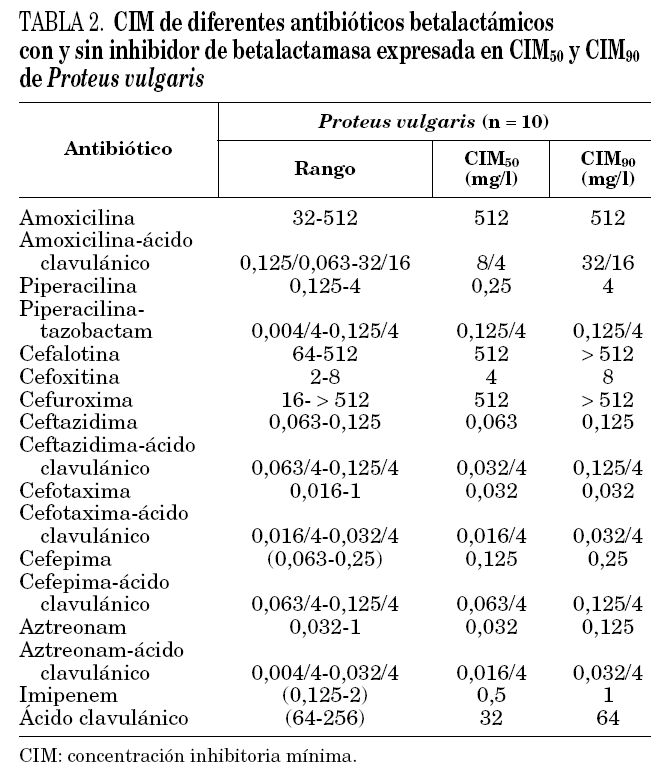
Todas las cepas de P. vulgaris y P. penneri estudiadas presentaron la resistencia natural a amoxicilina, cefalotina y cefuroxima. Un solo aislamiento de P. vulgaris presentó una CIM de cefotaxima de 1 mg/l (tabla 2), y una de las cepas de P. penneri fue resistente a cefotaxima (CIM = 64 mg/l).
En 2 de los 24 aislamientos de P. mirabilis del grupo II se observó una CIM de cefepima mayor o igual a 1 mg/l, siendo en uno de ellos la CIM de cefotaxima de 1 mg/l. Sin embargo, en ninguna de las cepas de este grupo disminuyó la CIM de las cefalosporinas de tercera y cuarta generación cuando se asoció el ácido clavulánico, ni se observó sinergia con este inhibidor en la prueba de doble disco. En todas las cepas del grupo II se detectó una enzima de pI 5,4, la cual hidrolizó ampicilina (500 mg/l) pero no cefotaxima (1.000 mg/ml) cuando se reveló por el método iodométrico. Cuatro de ellas poseían además una segunda enzima con actividad sobre ampicilina (500 mg/l), de pI 5,6.
Todas las cepas del grupo III presentaron sinergia con ácido clavulánico cuando fue utilizado en la prueba de doble disco y también mostraron una disminución en la CIM de por lo menos tres diluciones al asociar este inhibidor con las cefalosporinas de tercera y cuarta generación y aztreonam. Por otra parte, la CIM de cefotaxima y cefepima en todas las cepas fueron mayores a 1 mg/l, mientras que sólo 10 de 16 aislamientos presentaron CIM de ceftazidima y aztreonam mayor o igual a 1 mg/l. Las seis cepas restantes presentaron CIM de ceftazidima y aztreonam entre 0,125 y 0,5 mg/l.
Por isoelectroenfoque, en todas las cepas del grupo III se detectó la presencia de dos betalactamasas diferentes, activas sobre ampicilina (500 mg/l), una de ellas de pI 5,4 y otra de pI 8,2. Esta última hidrolizó también cefotaxima (1.000 mg/l), al ser revelada por el método iodométrico en agar.
El fragmento de amplificación obtenido por PCR tuvo un tamaño aproximado de 0,9 kb en concordancia con el gen blaCTX-M-2. La secuenciación de 12 de los productos amplificados por PCR correspondieron a CTX-M-2 (100% identidad) y ninguno correspondió a otra enzima relacionada10.
Todos los aislamientos de P. vulgaris y P. penneri presentaron una enzima de pI 5,4 que hidrolizó ampicilina (500 mg/l).
El aislamiento de P. vulgaris con CIM de cefotaxima de 1 mg/l, produjo además una enzima de pI 7,4, que correspondería a una de las cuatro betalactamasas cromosómicas descritas en esta especie12, que hidrolizó tanto ampicilina (500 mg/l) como cefotaxima (1.000 mg/l) al ser revelada por el método iodométrico.
En P. penneri la resistencia a cefotaxima (CIM, 64 mg/l), se asoció con la detección de una enzima de pI 6,8, coincidente con la betalactamasa cromosómica, propia de esta especie12, que hidrolizó cefotaxima (1.000 mg/l) en el ensayo iodométrico.
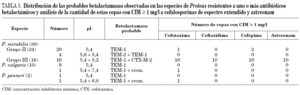
La distribución de las betalactamasas detectadas en las diferentes especies del género Proteus, resistentes a uno o más antibióticos betalactámicos se observa en la tabla 3.
Los 18 aislados de Proteus resistentes a cefalosporinas de tercera generación (P. mirabilis [16], P. penneri [1], P. vulgaris) fueron aislamientos únicos provenientes de 5 pacientes ambulatorios y 13 pacientes internados en diferentes salas del hospital: clínica médica (7), terapia (5) y neurocirugía (1).
Discusión
Las cefalosporinas de tercera generación constituyen una opción terapéutica fundamental en el tratamiento de las infecciones por bacilos gramnegativos. Debido a su amplia utilización se ha incrementado notablemente la resistencia en la mayoría de los miembros de la familia Enterobacteriaceae, incluyendo P. mirabilis13. El principal mecanismo de resistencia a dichos antibióticos en microorganismos que carecen de enzima cromosómica de clase C lo constituye la adquisición de betalactamasas plasmídicas3,14-18.
Las enzimas de espectro extendido más frecuentemente halladas en P. mirabilis, en Estados Unidos y Europa son derivadas de enzimas tipo TEM, como TEM-3, TEM-10 y TEM-2619-21. En Argentina, en cambio, las enzimas del tipo CTX-M son prevalentes en esta especie2,3,16. Recientemente en Israel se ha descrito por primera vez la presencia de CTX-M-2 en cepas de P. mirabilis de origen hospitalario22.
En este estudio la enzima CTX-M-217 resultó la única BLEE hallada en P. mirabilis, no encontrándose derivadas de SHV o de TEM, en concordancia con lo descrito previamente por Quinteros et al3. La BLEE hallada se detectó con el punto de corte recomendado por el NCCLS para K. pneumoniae, K. oxytoca y E. coli (CIM de cefotaxima mayor o igual a 2 mg/l6) y el recomendado por la Subcomisión de Antimicrobianos de SADEBAC (CIM de cefotaxima mayor o igual a 1 mg/l) para las mismas enterobacterias y P. mirabilis23. En este trabajo, utilizando ceftazidima y aztreonam sólo se detectó el 63% de las cepas de P. mirabilis del grupo III productoras de esta enzima, ya que el 37% restante presentaron CIM menor a 1 mg/l, como ya se ha demostrado previamente3. Asimismo, la CIM de las cefalosporinas de tercera y cuarta generación y de aztreonam asociadas a ácido clavulánico descendían en todos los casos por lo menos tres diluciones con respecto a las cefalosporinas y aztreonam solos, lo que confirmó fenotípicamente la presencia de BLEE.
En todos los aislamientos de P. mirabilis de los grupos II y III, se detectó la presencia de una enzima de pI 5,4 compatible con una betalactamasa de amplio espectro, probablemente TEM-1. Además, el 17% de ellos producía una segunda enzima de pI 5,6, compatible con TEM-2.
Por otra parte, en P. vulgaris y P. penneri el mecanismo más frecuente de resistencia a las cefalosporinas de tercera generación se debe a la adquisición de betalactamasas plasmídicas2,14. Sin embargo, en nuestro estudio los aislamientos resistentes presentaron enzimas de pI coincidentes con los de las betalactamasas cromosómicas propias de estas especies. Estas enzimas hidrolizaron cefotaxima, pero no ceftazidima, cefepima ni aztreonam y fueron inhibidas por ácido clavulánico. La hiperproducción de esta betalactamasa cromosómica es fenotípicamente indistinguible de las cefotaximasas halladas en nuestros aislamientos de P. mirabilis ya que ambas elevan la CIM de cefotaxima y no la de ceftazidima y aztreonam y son inhibidas por ácido clavulánico. La imposibilidad de diferenciarlas por el fenotipo de resistencia obliga a realizar su confirmación por isoelectroenfoque o por métodos genéticos.
Se puede concluir que la betalactamasa de amplio espectro más difundida en el género Proteus fue la enzima TEM-1 y la de BLEE correspondió en P. mirabilis a CTX-M-217, la cual es indistinguible fenotípicamente de la betalactamasa cromosómica de P. vulgaris y P. penneri.
Si bien no se ha intentado la caracterización clonal de los aislamientos resistentes analizados, si cabe recordar que corresponden a diversas salas del hospital y que un número importante de ellos proviene de pacientes ambulatorios, parece improbable la diseminación de un único clon.
La metodología recomendada por el NCCLS para detección de BLEE en E. coli, K. pneumoniae y K. oxytoca resulta adecuada para P. mirabilis empleando cefotaxima, así como también el ensayo de doble disco que sería de gran utilidad en los laboratorios que no tienen acceso a métodos cuantitativos.
Agradecimientos
Este trabajo fue financiado en parte por las becas: ANPCyT-PICT 06931 a Gabriel Gutkind, UBACyT BO66 a Pilar SantiniÝ, Beca Carillo Oñativia a Gabriel Gutkind y Fundación Roemmers a Marcela Radice.
Gabriel Gutkind es miembro de la carrera de investigador científico del Consejo Nacional de Investigaciones Científicas y Tecnológicas (CONICET).