En esta revisión se ofrece una información sucinta, precisa y completa de acuerdo con los datos disponibles sobre nuevas especies anaerobias estrictas implicadas en enfermedades infecciosas humanas. Se consideró “nueva” a toda aquella especie no descrita anteriormente, a la especie que no había sido implicada en clínica humana, a la especie que conociendo esta implicación no se había caracterizado y a la especie que había tenido cambios taxonómicos.
This review offers succinct, precise, and complete information based on the available data concerning new anaerobic bacterial species involved in infectious diseases in humans. All hitherto undescribed species, those not previously implicated in clinical conditions, those with confirmed implication in human disease that have not been characterized, and those that have undergone taxonomic changes are considered to be “new”.
Los avances tanto en la genómica y en la quimiotaxonomía como en su aplicación técnica han permitido conocer mejor a las bacterias y sus relaciones. Esto ha llevado a definir y situar adecuadamente tanto a especies conocidas como a nuevas especies y a caracterizar a grupos que no habían sido situados específicamente.
Entre las técnicas genómicas que se emplearon en anaerobios están la hibridación ácido desoxirribonucleico (ADN)-ADN y la secuenciación total o parcial de los genes del ácido ribonucleico ribosómico (ARNr) 16S: rpoB y dnaK. Estos estudios complementan a las propiedades fenotípicas (morfológicas, bioquímicas, enzimáticas y cromatográficas) y a los estudios de quimiotaxonomía (lípidos celulares y menaquidonas). De esta forma, una vez definida genéticamente una especie se deben buscar las características fenotípicas que permitan —si es posible— su caracterización. Para esto, se tiende a utilizar diversos microsistemas (Rapid ID 32A, API 20A, API ZYM; Vitek ANI Card [bioMérieux, Marcy l’Etoile, Francia]; RapID ANA II [Remel, Inc., Lenexa, Kans.]; BBL Cristal Anaerobe Identification System [Becton Dickinson Microbiology System, Cockeysville, Md]; MicroScan rapid anaerobe identification system [Dade MicroScan Inc. Sacramento, CA]) y la sensibilidad a discos diagnósticos de vancomicina (5μg), colistina (10μg) y kanamicina (1.000μg) y en el caso de los cocos grampositivos de metronidazol (5μg) y de polianetol sulfonato sódico (SPS). En ocasiones, es necesario recurrir a la cromatografía gas líquido para determinar los ácidos grasos finales del metabolismo.
La información sobre las “nuevas” especies se encuentra muy diseminada y esta revisión pretende aglutinar la información existente y favorecer el acercamiento a ellas, en concreto a las que no han sido recogidas o sólo son mencionadas en el Manual of Clinical Microbiology1.
Aunque cada año se describen numerosas especies aisladas de muestras clínicas y de la flora normal, aquí sólo se analizan las que se han aislado en muestras clínicas de pacientes sintomáticos. La patogenicidad de las bacterias anaerobias es difícil de demostrar porque son oportunistas, proceden habitualmente de la propia flora y se aíslan frecuentemente asociadas a otras bacterias. Por último, se incluyen los cambios en las denominaciones de algunas especies, derivados, fundamentalmente, del conocimiento de sus relaciones genéticas (tabla 1). Esta revisión pretende ofrecer información adicional y puesta al día del Procedimiento de Microbiología Clínica dedicado a las bacterias anaerobias2.
Cambios taxonómicos recientes en las bacterias anaerobias
| Denominación antigua | Denominación actual |
| Peptostreptococcus micros/Micromonas micros | Parvimonas micra |
| Bacteroides splanchnicus | Odoribacter splanchnicus |
| Bacteroides distasonis | Parabacteroides distasonis |
| Bacteroides goldsteinii | Parabacteroides goldsteinii |
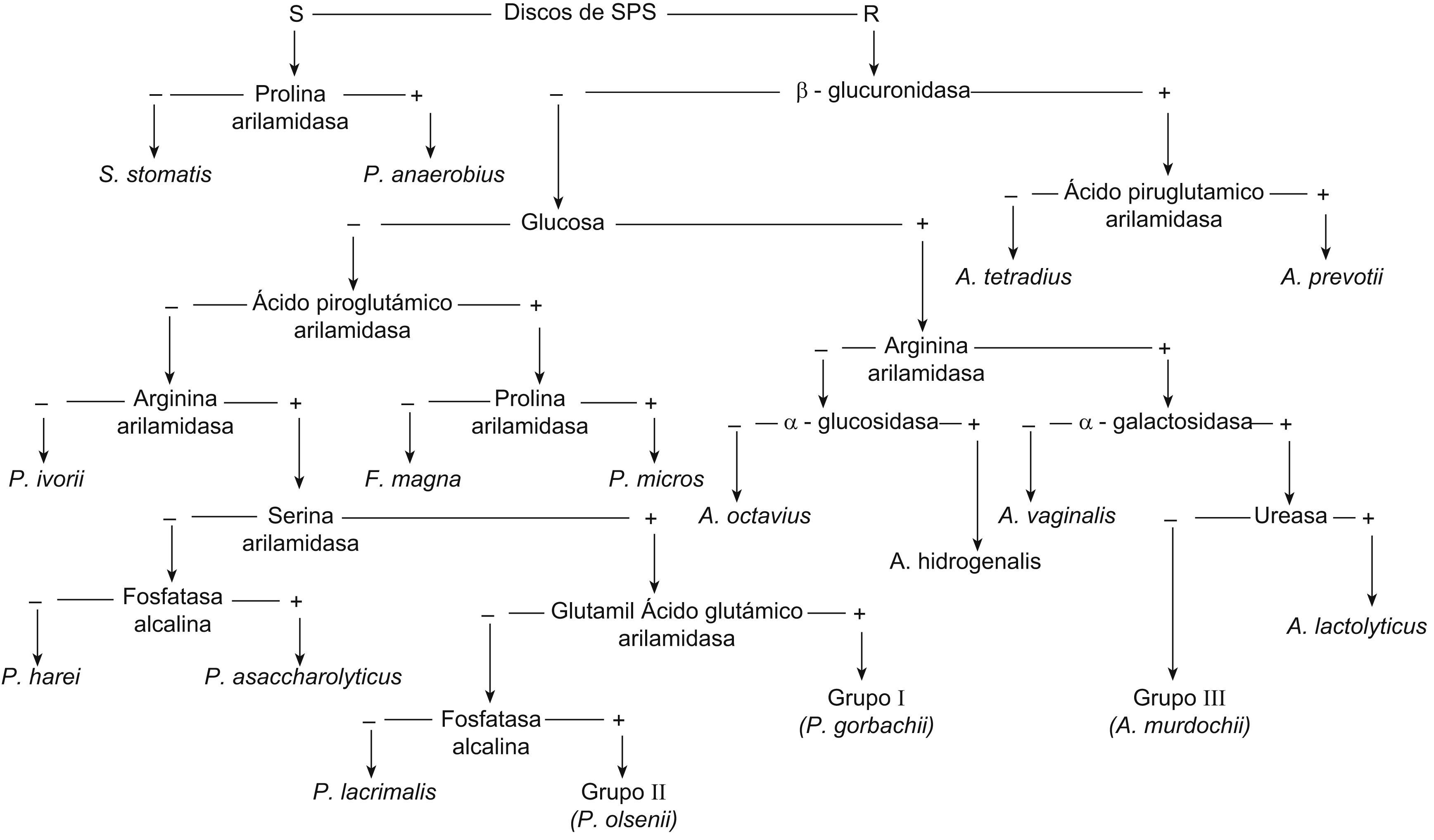
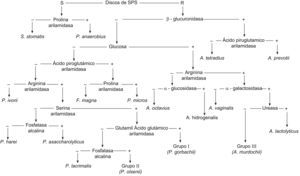
Tras los Bacteroides, los cocos son las bacterias anaerobias más frecuentemente aisladas en clínica. La mayoría pertenece a los géneros Anaerococcus, Finegoldia, Peptoniphilus y Peptostreptococcus. Son difíciles de identificar sobre la base de las propiedades bioquímicas, aunque se han desarrollado algoritmos que facilitan este proceso (fig. 1)3. Desde 2005 se han descrito 5 nuevas especies no esporuladas e inmóviles relacionadas con la clínica y se ha reclasificado una.
Género AnaerococcusEngloba a cocos grampositivos que producen ácido butírico y son sacarolíticos. Pertenecen a la familia Peptostreptococcaceae y se relacionan con los clostridios (sublínea del grupo XIII). Incluye a Anaerococcus prevotii, Anaerococcus tetradius, Anaerococcus lactolyticus, Anaerococcus hydrogenalis, Anaerococcus vaginalis y Anaero octavius4.
Anaerococcus murdochii
Descrita en 2007, debe su nombre a D. A. Murdoch, un investigador en cocos anaerobios. Es de tamaño pequeño (⩾0,7μm) y de crecimiento lento. A los 5 días de incubación da lugar a la aparición de colonias grises de 1 a 2mm. Sus propiedades bioquímicas se reflejan en la tabla 2. Además, fermenta glucosa y maltosa. No produce catalasa ni coagulasa. Produce ácidos acético y butírico como productos finales del metabolismo. Ha sido caracterizada por secuenciación del ARNr 16S. Se ha aislado de infecciones mixtas. La descripción se realizó con 6 cepas recuperadas de diversas infecciones: pie diabético (3 casos), herida esternal, tejido blando cervical y absceso de pulgar. Fueron sensibles a la asociación de betalactámico inhibidor de betalactamasas, cefazolina, cefoxitina, imipenem, metronidazol y vancomicina. Dos cepas tuvieron sensibilidad intermedia a penicilina y una fue resistente; otra fue resistente a clindamicina. La cepa tipo es la WAL 17230T (=CCUG 53340T=ATCC BAA-1385T). La secuencia del ARNr 16S depositada en GenBank es la DQ9112435.
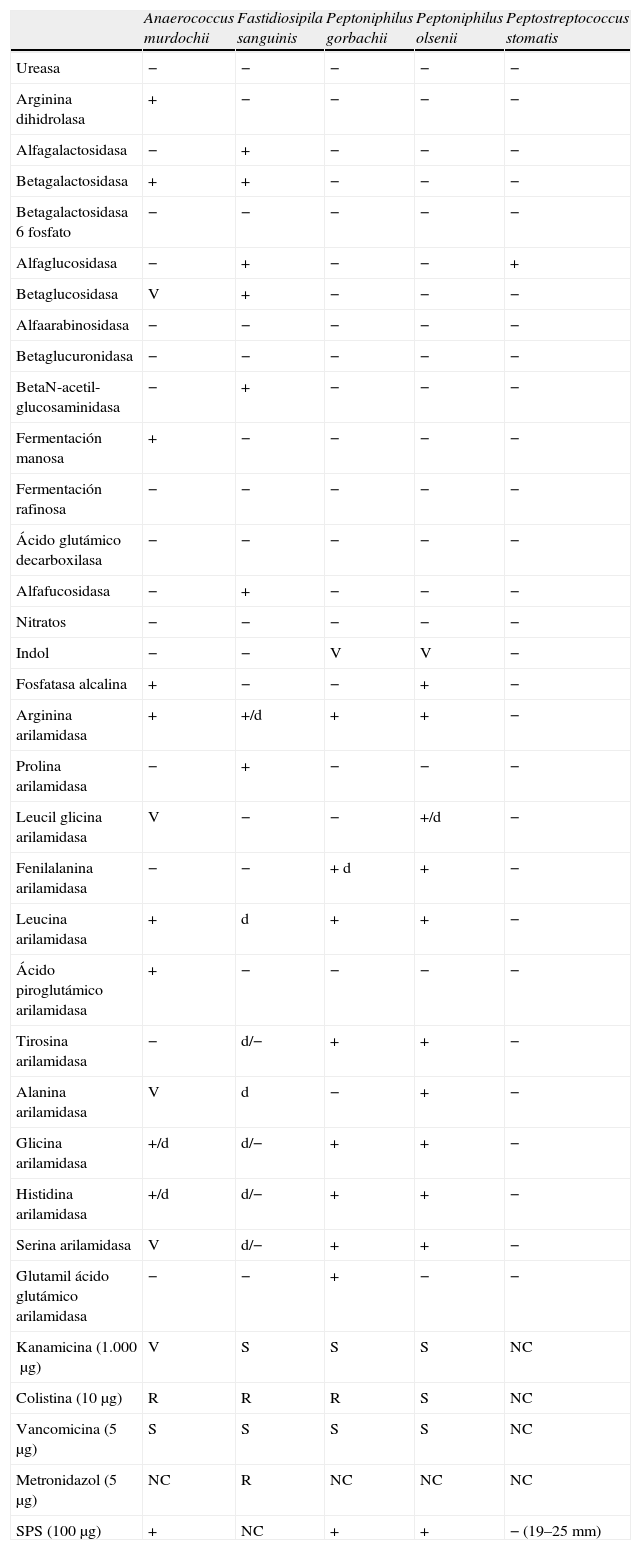
Características bioquímicas obtenidas mediante el sistema Rapid ID 32A y la utilización de discos diagnósticos de kanamicina, colistina, vancomicina, metronidazol y polianetol solfonato sódico de los cocos grampositivos anaerobios estrictos
| Anaerococcus murdochii | Fastidiosipila sanguinis | Peptoniphilus gorbachii | Peptoniphilus olsenii | Peptostreptococcus stomatis | |
| Ureasa | − | − | − | − | − |
| Arginina dihidrolasa | + | − | − | − | − |
| Alfagalactosidasa | − | + | − | − | − |
| Betagalactosidasa | + | + | − | − | − |
| Betagalactosidasa 6 fosfato | − | − | − | − | − |
| Alfaglucosidasa | − | + | − | − | + |
| Betaglucosidasa | V | + | − | − | − |
| Alfaarabinosidasa | − | − | − | − | − |
| Betaglucuronidasa | − | − | − | − | − |
| BetaN-acetil-glucosaminidasa | − | + | − | − | − |
| Fermentación manosa | + | − | − | − | − |
| Fermentación rafinosa | − | − | − | − | − |
| Ácido glutámico decarboxilasa | − | − | − | − | − |
| Alfafucosidasa | − | + | − | − | − |
| Nitratos | − | − | − | − | − |
| Indol | − | − | V | V | − |
| Fosfatasa alcalina | + | − | − | + | − |
| Arginina arilamidasa | + | +/d | + | + | − |
| Prolina arilamidasa | − | + | − | − | − |
| Leucil glicina arilamidasa | V | − | − | +/d | − |
| Fenilalanina arilamidasa | − | − | + d | + | − |
| Leucina arilamidasa | + | d | + | + | − |
| Ácido piroglutámico arilamidasa | + | − | − | − | − |
| Tirosina arilamidasa | − | d/− | + | + | − |
| Alanina arilamidasa | V | d | − | + | − |
| Glicina arilamidasa | +/d | d/− | + | + | − |
| Histidina arilamidasa | +/d | d/− | + | + | − |
| Serina arilamidasa | V | d/− | + | + | − |
| Glutamil ácido glutámico arilamidasa | − | − | + | − | − |
| Kanamicina (1.000μg) | V | S | S | S | NC |
| Colistina (10μg) | R | R | R | S | NC |
| Vancomicina (5μg) | S | S | S | S | NC |
| Metronidazol (5μg) | NC | R | NC | NC | NC |
| SPS (100μg) | + | NC | + | + | − (19–25mm) |
+: positivo; −: negativo; d: débil; NC: no comunicado; R: resistente; S: sensible; SPS: polianetol sulfonato sódico; V: variable.
Es un género establecido en 2007 que debe su nombre a sus exigencias nutricionales (fastidiosus) y a su forma (pila, ‘coco’). Pertenece a la clase Clostridia pero su posicionamiento en ella está por definirse, aunque se lo relaciona con el grupo III de Clostridium6.
Fastidiosipila sanguinis
De tamaño pequeño (0,5μm) y anaerobio estricto, aunque crece con un 2 y un 6% de oxígeno. A las 48h de incubación se observan colonias puntiformes y grises. Es bastante inactiva, no produce catalasa, lecitinasa, lipasa, gelatinasa y es asacarolítica (tabla 2). Produce pequeñas cantidades de ácidos acético y butírico en Fastidious Anaerobe Broth con granos de carne. La descripción se basó en 2 aislamientos procedentes de hemocultivos. Se desconoce su hábitat. No se han comunicado datos de sensibilidad a los antibióticos. La cepa tipo es la CCUG 47711T (=CIP 108292T). La secuencia del ARNr 16S depositada en GenBank es la AJ575187. El contenido de guanina (G) + citosina (C) en ADN es de 32,9 moles%6.
Género ParvimonasCreado para reclasificar a Micromonas, nombre ilegítimo pues ya estaba dado a un grupo de algas. Micromonas micros, antiguo Peptostreptococcus micros, se denomina actualmente Parvimonas micra (tabla 1)7.
Género PeptoniphilusSe caracteriza por producir ácido butírico y ser asacarolítico. Usa peptonas y aminoácidos como fuente de energía principal. Pertenece a la familia Peptostreptococcaceae y se relaciona con los clostridios (sublínea dentro del grupo XIII). Incluye a Peptostreptococcus asaccharolyticus, Peptostreptococcus lacrimaris, Peptostreptococcus harei, Peptostreptococcus indolicus y Peptostreptococcus ivorii4.
Peptoniphilus gorbachii
Comunicada en 2007, debe su nombre a S. Gorbach, “anaerobista” americano. Incluye a cocos pequeños (⩾0,7μm) de crecimiento lento y asacarolíticos. A los 5 días de incubación da lugar a la aparición de colonias grises, opacas y de 1 a 2mm de diámetro. No produce ni catalasa ni coagulasa (tabla 2). El producto final principal del metabolismo es el ácido acético; además, produce cantidades moderadas de ácido butírico y trazas de ácido propiónico. Se ha caracterizado genéticamente por secuenciación del ARNr 16S. Se aisló de infecciones mixtas. La descripción se realizó con 6 cepas: en 5 se disponía de datos clínicos, procedían de sujetos diabéticos, uno con gangrena seca infectada, 2 de celulitis (uno con absceso y otro con osteomielitis) y 2 de infección de pie diabético. Fueron sensibles a la asociación de betalactámico inhibidor de betalactamasas, cefazolina, cefoxitina, imipenem, metronidazol y vancomicina. Una cepa tuvo sensibilidad intermedia a penicilina y 2 cepas fueron resistentes a clindamicina. La cepa tipo es la WAL 10418T (=CCUG 53341T=ATCC BAA-1383T). La secuencia del ARNr 16S depositada en GenBank es la DQ9112415.
Peptoniphilus olsenii
Referida en 2007, debe su nombre a I. Olsen, “anaerobista” americano. Engloba a cocos pequeños (⩾0,7μm) y de crecimiento lento. A los 5 días de incubación da lugar a la aparición de colonias grises con el centro más claro, opacas y de 2 a 3mm de diámetro. Es asacarolítica y no produce catalasa ni coagulasa (tabla 2). El producto final principal del metabolismo es el ácido acético; además, produce cantidades moderadas de ácido butírico y trazas de ácido propiónico. Aislada de infecciones mixtas, se ha caracterizado genéticamente por secuenciación del ARNr 16S. La descripción se realizó con 4 cepas procedentes de diferentes infecciones: osteomielitis en un diabético, gangrena seca de pierna, úlcera de pie diabético y un absceso de un dedo del pie. Son sensibles a la asociación de betalactámico inhibidor de betalactamasas, cefazolina, cefoxitina, imipenem, metronidazol y vancomicina. La cepa tipo es la WAL 12922T (=CCUG 53342T=ATCC BAA-1384T). La secuencia del ARNr 16S depositada en GenBank es la DQ9112425.
Género PeptostreptococcusContiene a cocobacilos y a cocos que producen ácido acético, ácido butírico, ácido isobutírico, ácido isovalérico y ácido isocaproico y son moderadamente sacarolíticos. Pertenece a la familia Peptostreptococcaceae y está relacionado con los clostridios. Incluye a Peptrostreptococcus anaerobius8.
Peptostreptococcus stomatis
Fue descrita en 2006 y debe su nombre a la boca (stoma) que se consideró su hábitat. Incluye a cocos (0,8×0,8–0,9μm) agrupados en parejas o cadenas cortas y de crecimiento lento. Tras 5 días de incubación aparecen colonias opacas, brillantes, de color crema o blanquecinas y de 0,8 a 1,8mm. Es bastante inactiva (tabla 2), débilmente sacarolítica (fructosa, glucosa y maltosa), no hidroliza gelatina ni esculina y no produce catalasa. El perfil obtenido mediante la utilización del sistema Rapid ID 32A es 0400 0000 00. Alrededor de los discos de SPS dan halos de inhibición de 19 a 25mm frente a los 15 a 17mm de P. anaerobius que, además, en el Rapid ID 32A produce prolina ariamidasa y da un perfil de 0400 0200 009. Hay sondas fluorescentes de P. anaerobius y P. stomatis (Pana134) frente al ARNr 16S que permiten la identificación fiable y la hibridación con todos los aislamientos10. Produce ácido acético, ácido butírico, ácido isobutírico, ácido isovalérico e ácido isocaproico como productos finales del metabolismo. Se ha caracterizado genéticamente por hibridación ADN-ADN y secuenciación del ARNr 16S. Al describirla se pensó que su hábitat era orofaríngeo (a diferencia de P. anaerobius, que estaría en otras localizaciones)9, pero ulteriormente también se ha aislado de heces11. La descripción se realizó con 7 cepas, 6 de estas aisladas de infecciones orales: 3 casos de abscesos dentoalveolares, 2 casos de infecciones endodónticas y un caso de pericoronitis9. Además, con posterioridad se ha aislado de muestras apendiculares y sangre11. En una bacteriemia tras una aspiración con aguja fina guiada por ultrasonidos de una lesión del tracto abdominal bajo, se consideró contaminante12. Son sensibles a amoxicilina, a la asociación de amoxicilina más ácido clavulánico, a cefoxitina, ertapenem, metronidazol y moxifloxacino. La mayoría son sensibles, también, a clindamicina y seguramente a azitromicina, aunque no hay puntos de corte para anaerobios11. La cepa tipo es la W2278T (=DSM 17678T=CCUG 51858T). La secuencia del ARNr 16S está depositada en GenBank con el número de acceso DQ160208. El contenido de G+C del ADN es de 36 moles%9.
Bacilos grampositivos no esporulados anaerobios estrictosLa mayoría de las infecciones en las que están implicados se producen por especies de Actinomyces, Bifidobacterium, Eggerthella, Eubacterium, Lactobacillus y Propionibacterium. En 2007 se describieron 2 nuevos géneros implicados en clínica.
Género CatabacterDebe su nombre a que sus miembros producen catalasa. Engloba a cocobacilos móviles por un penacho lateral de flagelos. Está relacionado filogenéticamente con los grupos I, III, y XIVb de Clostridium. Se ha situado en una nueva familia: Catabacteriaceae13.
Catabacter hongkongensis
Crece en agar sangre y da colonias puntiformes a las 48h. Es relativamente aerotolerante y sensible a la bilis. No es hemolítica. Fermenta arabinosa, glucosa, manosa y xilosa. Los perfiles obtenidos con el sistema API 20A son el 40415042 y el 40414052 y el perfil obtenido con el sistema Rapid ID 32A es el 0012000010 (tabla 3). La producción de catalasa y la movilidad indican su identificación. La especie se ha caracterizado genéticamente por secuenciación del ARNr 16S a partir de 4 aislamientos realizados en Hong Kong y Canadá. Probablemente forma parte de la flora fecal. Produce bacteriemias de origen abdominal, causa de fallecimiento en un paciente. Las cepas fueron sensibles a metronidazol y vancomicina y resistentes a cefotaxima; 2 fueron sensibles a penicilina y 2 fueron resistentes (concentración mínima inhibitoria [CMI] de 4μg/ml). La cepa tipo es la HKU16T. La secuencia del ARNr 16S depositada en GenBank es la AY574991. El contenido de G+C del ADN es de 40,2 ± 2,2 moles%13.
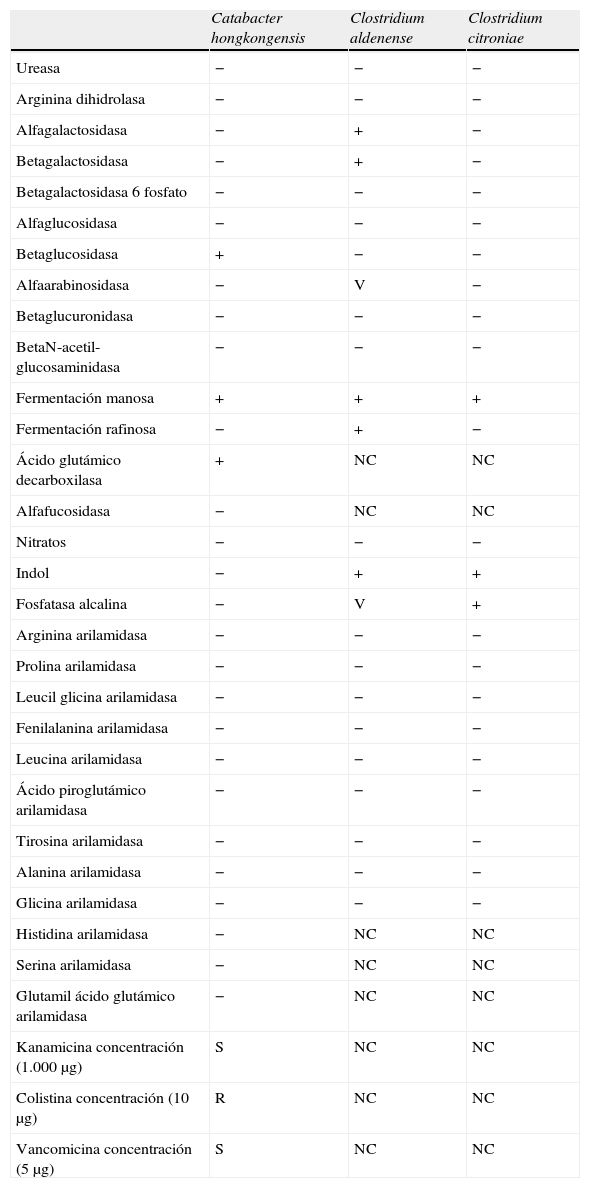
Características bioquímicas obtenidas mediante el sistema Rapid ID 32A y la utilización de discos diagnósticos de kanamicina, colistina y vancomicina de las nuevas especies de bacilos grampositivos anaerobios
| Catabacter hongkongensis | Clostridium aldenense | Clostridium citroniae | |
| Ureasa | − | − | − |
| Arginina dihidrolasa | − | − | − |
| Alfagalactosidasa | − | + | − |
| Betagalactosidasa | − | + | − |
| Betagalactosidasa 6 fosfato | − | − | − |
| Alfaglucosidasa | − | − | − |
| Betaglucosidasa | + | − | − |
| Alfaarabinosidasa | − | V | − |
| Betaglucuronidasa | − | − | − |
| BetaN-acetil-glucosaminidasa | − | − | − |
| Fermentación manosa | + | + | + |
| Fermentación rafinosa | − | + | − |
| Ácido glutámico decarboxilasa | + | NC | NC |
| Alfafucosidasa | − | NC | NC |
| Nitratos | − | − | − |
| Indol | − | + | + |
| Fosfatasa alcalina | − | V | + |
| Arginina arilamidasa | − | − | − |
| Prolina arilamidasa | − | − | − |
| Leucil glicina arilamidasa | − | − | − |
| Fenilalanina arilamidasa | − | − | − |
| Leucina arilamidasa | − | − | − |
| Ácido piroglutámico arilamidasa | − | − | − |
| Tirosina arilamidasa | − | − | − |
| Alanina arilamidasa | − | − | − |
| Glicina arilamidasa | − | − | − |
| Histidina arilamidasa | − | NC | NC |
| Serina arilamidasa | − | NC | NC |
| Glutamil ácido glutámico arilamidasa | − | NC | NC |
| Kanamicina concentración (1.000 μg) | S | NC | NC |
| Colistina concentración (10 μg) | R | NC | NC |
| Vancomicina concentración (5μg) | S | NC | NC |
+: positivo; −: negativo; NC: no comunicado; R: resistente; S: sensible; V: variable.
Debe su nombre a F. Mory, microbióloga francesa. Se relaciona con el grupo XVa de Clostridium. Pertenece a la familia Lachnospiraceae.
Moryella indoligenes
Su nombre específico refleja su capacidad de producir indol. Bacilo (0,8–1,7×0,5–0,6) gramvariable (estructuralmente grampositivo), elongado, a veces deforme, de extremos afilados, generalmente aislado o en parejas, a veces en cadena e inmóvil. Tras 24 a 48h de incubación da lugar a la aparición de colonias de 0,5 a 1mm de diámetro. Es débilmente sacarolítico; la glucosa, galactosa, maltosa y ribosa se fermentan variablemente. No produce catalasa y no hidroliza la gelatina ni la esculina; tampoco hay reducción de los nitratos. Es sensible a kanamicina, colistina, vancomicina, metronidazol y bilis. Produce ácidos acético, butírico y láctico como productos finales principales del metabolismo. La especie se ha caracterizado genéticamente por secuenciación del ARNr 16S. Las 3 cepas en las que se basa la descripción se aislaron de abscesos: glúteo, abdomen y muslo (en éste, junto a otras bacterias). Su hábitat es presumiblemente abdominal. La cepa tipo es sensible a penicilina, aminopenicilinas, cefalosporinas, imipenem y metronidazol; es intermedia a tetraciclinas y resistente a trimetoprim más sulfametoxazol, eritromicina y rifampicina. La cepa tipo es la AIP 220.04T (=CIP 109174T=CCUG 52648T). La secuencia del ARNr 16S está depositada en GenBank con el número de acceso DQ377947. El contenido de G+C del ADN es de 50,2 moles%14.
Género SolobacteriumAunque la única especie que alberga se asemeja a las eubacterias está lejana de éstas pues pertenece a la familia Erysipelotrichaceae.
Solobacterium moorei
Fue descrita en el año 2000 a partir de 3 cepas aisladas de heces15. Recientemente se han comunicado casos que demuestran claramente su implicación clínica. El primer caso fue una bacteriemia en un paciente con mieloma múltiple, con probable origen en un absceso dentoalveolar que se trató con éxito con cefepima16. El segundo caso fue también una bacteriemia, en esta ocasión en una paciente con una proctitis aguda posradioterapia por un carcinoma de cérvix que respondió al tratamiento con piperacilina más tazobactam17. El tercer caso corresponde a una tromboflebitis séptica femoral con embolismo pulmonar en un adicto a drogas parenterales. En los hemocultivos se aisló junto a Fusobacterium nucleatum y Bacteroides ureolyticus, y respondió satisfactoriamente a un tratamiento con penicilina y metronidazol18. En los 3 casos la bacteria fue identificada por secuenciación del ARNr 16S y en los 2 primeros las características fenotípicas de los aislamientos no coincidieron exactamente con las descritas previamente (gelatina, esculina, galactosa, glucosa y sacarosa). Los aislamientos fueron sensibles a penicilina, vancomicina, metronidazol y cefotaxima17. Recientemente, se aisló en todos los pacientes estudiados y en ninguno de los controles en un estudio etiológico sobre halitosis19.
Bacilos grampositivos esporulados anaerobios estrictosHay más de 150 especies dentro del género Clostridium (familia Clostridiaceae), muchas de ellas producen infecciones en el hombre. Desde 2003 se ha implicado a 4 especies en clínica: 2 nuevas y 2 descritas anteriormente.
Clostridium aldenense
Especie caracterizada en 2006 y que debe su nombre al R. M. Alden Research Laboratory y a R. M. Alden Goldstein. Bacilo (0,8–1,1×2–5μm) gramnegativo (grampositivo genéticamente) de esporulación infrecuente como la mayoría de las especies del grupo de Clostridium clostridioforme al que pertenece. Las colonias, de 1 a 2mm de diámetro son de opacas a blancas y no hemolíticas. Se diferencia de Clostridium bolteae, C. clostridioforme y Clostridium hathewayi por producir indol, y se diferencia de Clostridium citroniae (que produce indol) por fermentar la rafinosa y no la ramnosa y por presentar actividad alfagalactosidasa y betagalactosidasa. Es sacarolítico, fermenta la glucosa, maltosa, manosa, rafinosa, sacarosa y xilosa, y variablemente arabinosa, lactosa, salicina y trealosa. No hidrolizan almidón ni gelatina (tabla 3). Se ha caracterizado genéticamente por secuenciación del ARNr 16S. Esta especie se ha aislado fundamentalmente de infecciones intraabdominales. Es sensible a penicilina, a la asociación de betalactámicos inhibidores de betalactamasas, a ertapenem, metronidazol y clindamicina y resistente a moxifloxacino y levofloxacino. La cepa tipo es la RMA 9741T (=ATCC BAA-1318 T=CCUG 52204 T). La secuencia del ARNr 16S está depositada en GenBank con el número de acceso DQ27973620.
Clostridium amygdalinum
Fue descrita en 2003 como bacteria ambiental. Sus componentes son bacilos ovales o largos (0,5–1,0×0,5–10μm), aislados, en parejas o en cadenas y aerotolerantes. Son móviles por un flagelo polar en los cultivos jóvenes; termófilos moderados (45°C) y sacarolíticos. Fermentan arabinosa, celobiosa, galactosa, glucosa, fructosa, maltosa, manitol, melibiosa, rafinosa, ribosa, sacarosa, trealosa y xilosa. No producen lipasa ni lecitinasa, y pueden producir indol. No hidrolizan la gelatina. No producen catalasa y sí hidrolizan la esculina. Producen ácido acético, ácido propiónico, ácido butírico y ácido valérico como productos finales principales del metabolismo. Pueden identificarse por sus propiedades fermentativas21,22. Se han aislado de una osteomielitis crónica. Son sensibles a penicilina, ampicilina, amoxicilina, a la asociación de amoxicilina con ácido clavulánico, a cefalotina, cefoxitina, imipenem, metronidazol, tetraciclina y rifampicina22. La cepa tipo es la BR-10T (=ATCC BAA-501T= DSM 12857T). La secuencia del ARNr 16S está depositada en GenBank con el número de acceso AY35395721. La cepa aislada en clínica es la AIP 321.05 (= CIP 109224) y la secuencia de ARNr 16S tiene como número de acceso DQ50724522.
Clostridium citroniae
Especie caracterizada en 2006 que debe su nombre a D. M. Citron, “anaerobista” americana. Bacilo (0,8–1,1×2–5μm) gramnegativo (grampositivo genéticamente) de esporulación difícil. Las colonias son de opacas a blancas, no hemolíticas y de uno a 2mm de diámetro. El ser gramnegativa y esporular difícilmente, la asemeja a otros miembros del grupo de C. clostridioforme al que pertenece y de los que se diferencia por producir indol como C. aldenense, pero de éste se diferencia por fermentar la ramnosa y no la rafinosa y por no presentar actividad alfagalactosidasa y betagalactosidasa. Es sacarolítica, fermenta glucosa, maltosa, manosa, ramnosa, sacarosa, trealosa y xilosa y variablemente arabinosa, lactosa, salicina y trealosa. No hidroliza esculina ni almidón ni gelatina (tabla 3). Se ha caracterizado genéticamente por secuenciación del ARNr 16S. Se aísla fundamentalmente de infecciones intraabdominales. Es sensible a penicilina, a la asociación de betalactámicos inhibidores de betalactamasas, a ertapenem, metronidazol y clindamicina, y es resistente a moxifloxacino y levofloxacino. La cepa tipo es la RMA 16102T (=ATCC BAA-1317T= CCUG 52203T). La secuencia del ARNr 16S está depositada en GenBank con el número de acceso DQ27973720.
Clostridium glycolicum
Aunque se describió en 1963 y se cita en algunos textos como productor de infecciones, el primer caso clínico publicado es el de una bacteriemia con shock séptico en una mujer que recibió trasplante de médula ósea debido a una recaída de una enfermedad de Hodgkin. Se aisló en 3 hemocultivos (en 2 de ellos junto a un Enterococcus spp.), por lo que su patogénesis es incierta. Se trató a la paciente con éxito durante 2 semanas con ampicilina, gentamicina y metronidazol. La especie es sensible a penicilina G, clindamicina y metronidazol. La secuencia de ARNr 16S de esta cepa tiene como número de acceso DQ986354 en GenBank23.
Cocos gramnegativos anaerobios estrictosLa familia Acidaminococceae se relaciona con los clostridios y en 4 de sus géneros (Acidaminococcus, Dialister, Megasphaera y Veillonella) hay especies que se han aislado de infecciones humanas. Son inmóviles, no esporuladas, bastante inactivas, de tal forma que puede ser necesario recurrir a procedimientos genéticos o de quimiotaxonomía para identificarlas. El género Veillonella, a diferencia de los otros 3, reduce los nitratos, Acidaminococcus produce ácido butírico, Megasphaera produce ácido caproico y Dialister produce ácido acético y ácido propiónico, pero no ácido butírico ni ácido caproico. En Acidaminococcus y Dialister se han descrito nuevas especies a partir de muestras clínicas1,24.
Género AcidaminococcusAsacarolítico, bastante inactivo, produce ácido acético y ácido butírico y a veces ácido propiónico como productos finales del metabolismo. Ha sido raramente implicado en clínica. Incluía sólo a Acidaminococcus fermentans1,24.
Acidaminococcus intestini
Descrita en 2007, su nombre deriva de su hábitat. Incluye a cocos pequeños (0,5 a 0,6μm), aislados o en parejas, que producen colonias no pigmentadas ni hemolíticas de 0,3 a 0,5mm tras 2 días de incubación. En la tabla 4 se pueden observar sus propiedades bioquímicas. Su perfil con el sistema Rapid ID 32A es 0000016410. A diferencia de A. fermentans (perfil 0000012401), produce ácido propiónico y tiene actividad ácido glutámico arilamidasa y leucil glicina arilamidasa. Forma parte de la flora fecal normal. Se ha aislado en cultivo mixto en infecciones intraabdominales y abscesos, frecuentemente en las proximidades del abdomen. No se han comunicado datos de sensibilidad a los antimicrobianos. La cepa tipo es la ADV 255.99T (=AIP 283.01T= CIP 108586T=CCUG 50930T). La secuencia del ARNr 16S está depositada en GenBank/EMBL/DDBJ con el número de acceso AF473835. El contenido de C+G del ADN es de 49,3 moles%24.
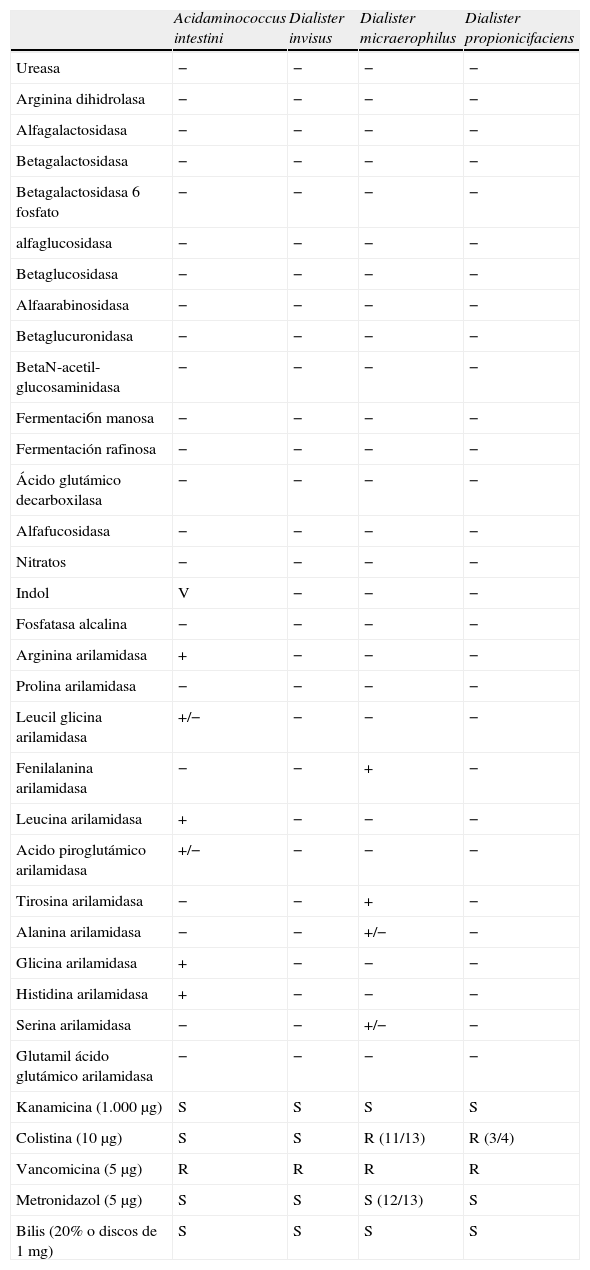
Características bioquímicas obtenidas mediante el sistema Rapid ID 32A y la utilización de discos diagnósticos de kanamicina, colistina, vancomicina y metronidazol y bilis de los cocos gramnegativos anaerobios estrictos
| Acidaminococcus intestini | Dialister invisus | Dialister micraerophilus | Dialister propionicifaciens | |
| Ureasa | − | − | − | − |
| Arginina dihidrolasa | − | − | − | − |
| Alfagalactosidasa | − | − | − | − |
| Betagalactosidasa | − | − | − | − |
| Betagalactosidasa 6 fosfato | − | − | − | − |
| alfaglucosidasa | − | − | − | − |
| Betaglucosidasa | − | − | − | − |
| Alfaarabinosidasa | − | − | − | − |
| Betaglucuronidasa | − | − | − | − |
| BetaN-acetil-glucosaminidasa | − | − | − | − |
| Fermentaci6n manosa | − | − | − | − |
| Fermentación rafinosa | − | − | − | − |
| Ácido glutámico decarboxilasa | − | − | − | − |
| Alfafucosidasa | − | − | − | − |
| Nitratos | − | − | − | − |
| Indol | V | − | − | − |
| Fosfatasa alcalina | − | − | − | − |
| Arginina arilamidasa | + | − | − | − |
| Prolina arilamidasa | − | − | − | − |
| Leucil glicina arilamidasa | +/− | − | − | − |
| Fenilalanina arilamidasa | − | − | + | − |
| Leucina arilamidasa | + | − | − | − |
| Acido piroglutámico arilamidasa | +/− | − | − | − |
| Tirosina arilamidasa | − | − | + | − |
| Alanina arilamidasa | − | − | +/− | − |
| Glicina arilamidasa | + | − | − | − |
| Histidina arilamidasa | + | − | − | − |
| Serina arilamidasa | − | − | +/− | − |
| Glutamil ácido glutámico arilamidasa | − | − | − | − |
| Kanamicina (1.000μg) | S | S | S | S |
| Colistina (10μg) | S | S | R (11/13) | R (3/4) |
| Vancomicina (5μg) | R | R | R | R |
| Metronidazol (5μg) | S | S | S (12/13) | S |
| Bilis (20% o discos de 1mg) | S | S | S | S |
+: positivo; −: negativo; R: resistente; S: sensible; V: variable.
Tradicionalmente se ha estudiado dentro de los bacilos gramnegativos y comprendía una sola especie, Dialister pneumosintes. Engloba a cocos y cocobacilos muy pequeños (0,2–0,4×0,3–0,6μm), inmóviles, no esporulados, no producen catalasa, son asacarolíticos y bastante inactivos y pueden ser microaerófilos. Crece mal en caldo. Produce ácido acético y ácido propiónico, a veces en pequeñas cantidades (tanto que pueden no detectarse), pero no produce ácido butírico ni ácido caproico25,26.
Dialister invisus
Descrita en 2003, su nombre hace referencia a la falta de turbidez en caldo. Sus miembros se presentan aislados, en parejas, en cadenas cortas y pequeños grupos. Las colonias son translúcidas o transparentes de 0,5 a 0,7mm tras 7 días de incubación. Inactiva, su perfil con el sistema Rapid ID 32A es el 0000 0000 00 frente al obtenido en D. pneumosintes, que es el 0000 0100 00 (tabla 4). Produce trazas de ácido acético y ácido propiónico. La descripción se realizó con 6 cepas: 5 aisladas de infecciones endodónticas y una aislada de placa subgingival25. Ulteriormente, se ha detectado en orina en pacientes que han recibido trasplante renal27, y se ha aislado de infecciones de piel y tejidos blandos, intraabdominales y respiratorias. Es sensible a amoxicilina, a la asociación de amoxicilina con ácido clavulánico, a piperacilina, cefoxitina, imipenem, eritromicina, clindamicina, ciprofloxacino, moxifloxacino, rifampicina, cloranfenicol y metronidazol28. La cepa tipo es E7.25T (=CCUG 47026T=DSM 15470T). La secuencia del ARNr 16S está depositada en GenBank con el número de acceso AY162469. El valor de C+G en el ADN es de 45 moles%25.
Dialister micraerophilus
Comunicada en 200329 y descrita en 2005, su nombre específico proviene de su capacidad de crecer en microaerofilia. Se presenta como elementos aislados, en parejas o en grupo que dan colonias translúcidas y muy pequeñas en agar sangre (0,5mm). En el sistema Rapid ID 32A es característica su actividad fenilalanina arilamidasa y tirosina arilamidasa (tabla 4). No se detectan ácidos grasos como productos finales de su metabolismo. Se ha sido caracterizado por secuenciación del ARNr 16S y dnaK, y por los ácidos grasos celulares. La descripción se realizó a partir de 24 cepas recuperadas: 9 recuperadas de infecciones de tejidos blandos, 9 recuperadas de hemocultivos, 4 recuperadas de muestras ginecológicas, una recuperada de muestras óseas y una recuperada de muestras renales26. Es sensible a amoxicilina, a la asociación de amoxicilina con ácido clavulánico, a piperacilina, cefoxitina, imipenem, ertapenem, eritromicina, clindamicina, ciprofloxacino, levofloxacino, moxifloxacino, rifampicina, doxiciclina, cloranfenicol y metronidazol, aunque las CMI de este último pueden ser altas28. La cepa tipo es la ADV 04.01T (=AIP 25.04T=CIP 108278T=CCUG 48837T). La secuencia del ARNr 16S y dnaK están depositadas en GenBank/EMBL/DDBJ con los números de acceso AF473837 y AY851488. El G+C ADN es de 36,3 moles%26.
Dialister propionicifaciens
Descrita en 2005, debe su nombre a su capacidad de producir ácido propiónico. Sus características son idénticas a las referidas para D. micraerophilus (tabla 4) pero es más inactiva. Produce pequeñas cantidades de ácido acético, ácido propiónico y ácido láctico. Produce ácido propiónico a partir del succinato sódico que estimula su crecimiento. Por esta propiedad se diferencia de las otras especies del género, además de por la secuenciación del ARNR 16S y dnaK. La descripción se realizó a partir de 4 cepas recuperadas: 3 recuperadas de infecciones de tejidos blandos y una recuperada de semen26. Es sensible a amoxicilina, a la asociación de amoxicilina con ácido clavulánico, a piperacilina, cefoxitina, imipenem, ertapenem, eritromicina, clindamicina, ciprofloxacino, levofloxacino, moxifloxacino, rifampicina, doxiciclina, cloranfenicol y metronidazol28. La cepa tipo es la ADV 1053.03T (=AIP 26.04T=CIP 108336T=CCUG 49291T) La secuencia del ARNr 16S y dnaK están depositadas en GenBank/EMBL/DDBJ con los números de acceso AY850119 y AY851500. El porcentaje de C+G del ADN no ha podido determinarse26.
Bacilos gramnegativos anaerobios estrictos no esporuladosGénero AlistipesDescrito en 2003 —su nombre significa “otros bacilos”— para incluir a Bacteroides putredinis (reclasificado como Alistipes putredinis) y a Alistipes finegoldii. Incluye a bacilos rectos o ligeramente curvos, inmóviles y de crecimiento rápido (4 días) similares al grupo de Bacteroides fragilis, que pueden resistir a un 20% de bilis (no es el caso de A. putredinis) y son sacarolíticos (no es el caso de A. putredinis). A diferencia de éstos, producen colonias pigmentadas de negro en medios con sangre (no es el caso de A. putredinis) que no producen fluorescencia con luz ultravioleta. La pigmentación aparece tras incubaciones prolongadas (más de 4 días) en cultivos en agar sangre de conejo lacada. Crecen mal en caldo. Producen ácido succínico como elemento final principal de su metabolismo y, a diferencia de Porphyromonas, no produce ácido butírico30,31. Pertenece a la familia Rikenellaceae.
Alistipes finegoldii
Comunicada en 199732 y denominada y situada taxonómicamente en 2003, debe su nombre a S. Finegold, “anaerobista” americano. Incluye a bacilos de 0,2×0,8–2μm, aislados y a veces forma filamentos que dan colonias débilmente betahemolíticas de aproximadamente 0,7mm. No produce catalasa, hidroliza gelatina y la hidrólisis de esculina es variable. Fermenta glucosa y lactosa (tabla 5). Forma parte de la flora fecal y se ha aislado de apendicitis, abscesos perirrectales, abscesos cerebrales30,32, generalmente en el marco de una infección mixta y en bacteriemias en pacientes con cáncer de colon33. Puede producir betalactamasas, por lo que su sensibilidad a penicilina es variable. Se ha caracterizado por hibridación ADN-ADN y secuenciación de ARNr 16S y por los ácidos grasos celulares. La cepa tipo es la CCUG 46020T (=AHN243T)30,32. La secuencia del ARNr 16S está depositada en GenBank con el número de acceso AJ518874. El valor de C+G en ADN es de 57 moles%.
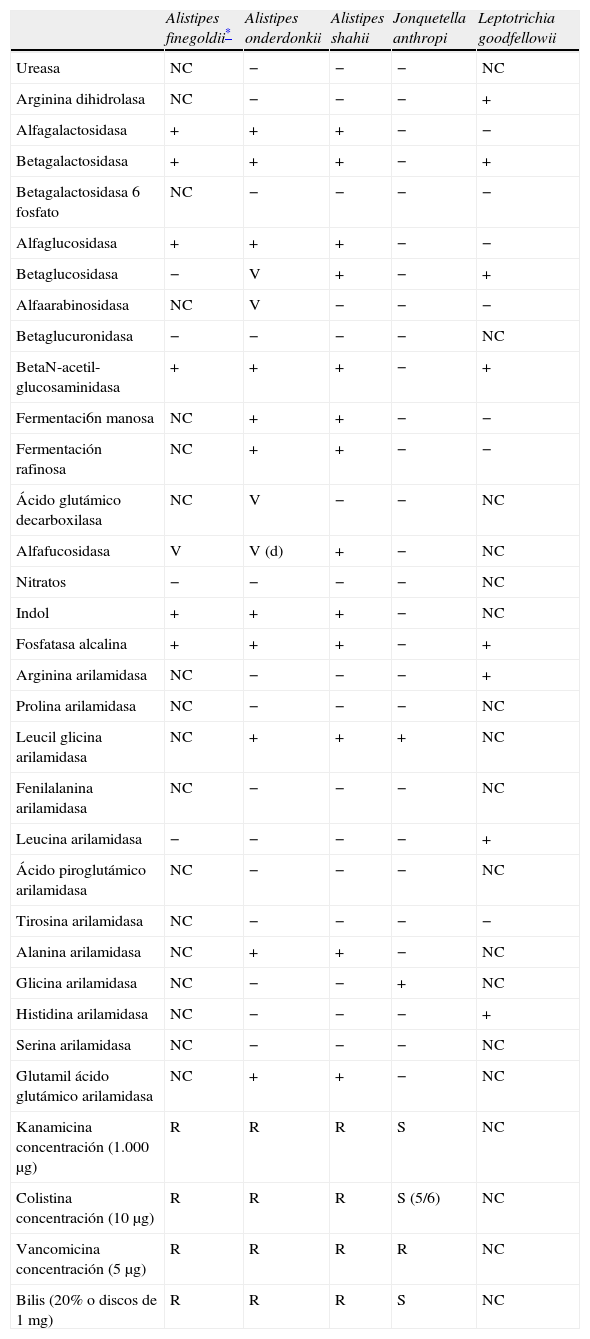
Características bioquímicas obtenidas mediante el sistema Rapid ID 32A, API ZYM, la utilización de discos diagnósticos de kanamicina, colistina y vancomicina y bilis de los nuevos bacilos gramnegativos anaerobios no esporulados (no es el caso de Prevotella spp.)
| Alistipes finegoldii* | Alistipes onderdonkii | Alistipes shahii | Jonquetella anthropi | Leptotrichia goodfellowii | |
| Ureasa | NC | − | − | − | NC |
| Arginina dihidrolasa | NC | − | − | − | + |
| Alfagalactosidasa | + | + | + | − | − |
| Betagalactosidasa | + | + | + | − | + |
| Betagalactosidasa 6 fosfato | NC | − | − | − | − |
| Alfaglucosidasa | + | + | + | − | − |
| Betaglucosidasa | − | V | + | − | + |
| Alfaarabinosidasa | NC | V | − | − | − |
| Betaglucuronidasa | − | − | − | − | NC |
| BetaN-acetil-glucosaminidasa | + | + | + | − | + |
| Fermentaci6n manosa | NC | + | + | − | − |
| Fermentación rafinosa | NC | + | + | − | − |
| Ácido glutámico decarboxilasa | NC | V | − | − | NC |
| Alfafucosidasa | V | V (d) | + | − | NC |
| Nitratos | − | − | − | − | NC |
| Indol | + | + | + | − | NC |
| Fosfatasa alcalina | + | + | + | − | + |
| Arginina arilamidasa | NC | − | − | − | + |
| Prolina arilamidasa | NC | − | − | − | NC |
| Leucil glicina arilamidasa | NC | + | + | + | NC |
| Fenilalanina arilamidasa | NC | − | − | − | NC |
| Leucina arilamidasa | − | − | − | − | + |
| Ácido piroglutámico arilamidasa | NC | − | − | − | NC |
| Tirosina arilamidasa | NC | − | − | − | − |
| Alanina arilamidasa | NC | + | + | − | NC |
| Glicina arilamidasa | NC | − | − | + | NC |
| Histidina arilamidasa | NC | − | − | − | + |
| Serina arilamidasa | NC | − | − | − | NC |
| Glutamil ácido glutámico arilamidasa | NC | + | + | − | NC |
| Kanamicina concentración (1.000μg) | R | R | R | S | NC |
| Colistina concentración (10μg) | R | R | R | S (5/6) | NC |
| Vancomicina concentración (5μg) | R | R | R | R | NC |
| Bilis (20% o discos de 1mg) | R | R | R | S | NC |
+: positivo; −: negativo; d: débil; NC: no comunicado; S: sensible; R: resistente; V: variable.
Alistipes onderdonkii
Comunicada en 2006, su nombre se debe a A. B. Onderdonk, microbiólogo americano. Bacilo de 0,2–0,5×0,5–3μm que puede caracterizarse por pruebas bioquímicas convencionales o automatizadas (tabla 5). A las 48h de incubación las colonias son grises, opacas, débilmente betahemolíticas y de 0,5 a 0,8mm de diámetro. A los 4 días comienzan a pigmentarse. La betaglucosidasa y la alfafucosidasa la diferencian de Alistipes shahii y de A. finegoldii mediante el sistema API ZYM. No produce catalasa y la hidrólisis de esculina es variable. Se ha caracterizado por secuenciación del ARNr 16S y por los ácidos grasos celulares. Probablemente forma parte de la flora fecal y se asocia a infecciones polimicrobianas y mixtas. Se ha aislado de tejido apendicular, de absceso abdominal y de orina. Es sensible a metronidazol, a clindamicina, a la asociación de betalactámico inhibidor de betalactamasas y a ertapenem. Puede producir betalactamasas. La cepa tipo es la WAL 8169T (=CCUG 48946T=ATTC BAA-1178T). La secuencia del ARNr 16S está depositada en GenBank/EMBL/DDBJ con el número de acceso AY974071. El contenido de G+C del ADN es de 58 moles%31.
Alistipes shahii
Notificada en 2006, su nombre deriva de H. N. Shah, microbiólogo americano. Bacilos (0,2–0,5×0,5–3μm) con características fenotípicas (tabla 5) y sensibilidad a los antibióticos similares a las comentadas para A. onderdonkii. A las 48h de incubación las colonias son grises, opacas, débilmente betahemolíticas y de 0,5 a 1mm de diámetro. A los 4 días comienzan a pigmentarse. No produce catalasa y sí hidroliza esculina. Probablemente forma parte de la flora fecal y se ha aislado de tejido apendicular y de líquidos peritoneales. La cepa tipo es la WAL 8301T (=CCUG 48947T=ATTC BAA-1179T). La secuencia del ARNr 16S está depositada en GenBank/EMBL/DDBJ con el número de acceso AY974072. El contenido de G+C del ADN es de 56 moles%31.
Género JonquetellaEn honor a Jonquet, el clínico que identificó la primera infección por este género. No pertenecen a ningún filo bacteriano conocido y se ha propuesto incluirlo en uno nuevo, el de los “Sinergistetes”. Creado para albergar la especie que se describe a continuación34.
Jonquetella anthropi
Descrita en 2007, su denominación hace referencia a su origen humano. Incluye a bacilos (0,8–0,9×1,4–1,7μm) rectos, gramnegativos, inmóviles y no esporulados, anaerobios estrictos, con requerimientos nutricionales complejos y de crecimiento relativamente lento (3 a 5 días); da colonias muy pequeñas (0,5mm), no hemolíticas, circulares, grises y brillantes. El cloruro sódico estimula su crecimiento en medio Dethiosulfovibrio peptidovorans (DP)35. No produce oxidasa ni catalasa. Es sensible a la bilis, es asacarolítica y no reactiva en otras pruebas fenotípicas habituales. Con el sistema Rapid ID 32 A muestra actividad leucil glicina y glicina ariamidasa (código 0000040400) (tabla 5). Los productos finales principales de su metabolismo son ácido acético, ácido láctico, ácido succínico y ácido isovalérico. Se ha caracterizado por secuenciación del ARNr 23S y por los ácidos grasos celulares. Las 6 cepas estudiadas se aislaron de muestras clínicas: una en cultivo puro de un líquido peritoneal, 4 en cultivo mixto de absceso de mama, quiste sebáceo, herida y absceso pélvico, y una sexta de colección procedente de una úlcera sacra. No se han comunicado datos de sensibilidad a los antimicrobianos. La cepa tipo es la ADV 126T (=AIP 136.05T=CIP 109408T=CCUG 53819T). La secuencia del ARNr 16S está depositada en GenBank/EMBL/DDBJ con el número de acceso EF436500. El contenido de G+C del ADN es de 59,4 moles%34.
Género LeptotrichiaEngloba a bacilos fusiformes, inmóviles, que requieren medios con sangre para anaerobios en los que crecen lentamente y producen ácido láctico como producto final de su metabolismo1. Pertenece a la familia Fusobacteriaceae. Forma parte de la flora oral y genitourinaria. Son patógenos ocasionales, con frecuencia en inmunodeprimidos. Con procedimientos polifásicos (fenotípicos, quimiotaxonómicos y genéticos) se han caracterizado nuevas especies, algunas implicadas en enfermedades infecciosas36.
Leptotrichia amnionii
Fue descrita en 2002, su nombre hace referencia a su origen. Esta especie, que aún no ha sido admitida, parece encajar más en el género Sneathia que en el género Leptotrichia36. Comprende a bacilos pleomorfos, de cocobacilos a formas largas y fusiformes que a veces se unen por el extremo y dan la sensación de filamentos largos. En agar sangre, en cultivo primario, crecen en 3 días pero no son subcultivables en este medio37. Se han logrado subcultivar en agar chocolate (Nippon Becton Dickinson, Tokio, Japón) en una atmósfera anaerobia con el 10% de dióxido de carbono (CO2) (no con un 5%)38. Se identifica por secuenciación del ARNr 16S37. Es parte de la flora normal vaginal, al menos en la embarazada39. Prácticamente la totalidad de los casos en los que se ha implicado (por cultivo o detección genética) ha sido en mujeres, especialmente en el parto o en las proximidades: muerte fetal intrauterina37, bacteriemias durante el parto40, piosalpinx41, aborto42, vaginosis bacteriana43, aborto con corioamnoiditis y absceso renal44 e infección urinaria en trasplante renal27. Se ha implicado en un caso de artritis en el varón38. Es sensible a amoxicilina, ceftriaxona, meropenem y ciprofloxacino38. La secuencia del ARNr 16S está depositada en GenBank con el número de acceso AY07842537.
Leptotrichia goodfellowii
Descrita en 2004, su denominación es en honor a M. Goodfellow por sus aportaciones en sistemática microbiana. Incluye a bacilos (2–4×0,3–0,6μm) agrupados en parejas o en cadenas, unidos por extremos aplanados, algunos son ligeramente curvos. Es un anaerobio poco estricto, pues puede crecer en aerobiosis. No crece ni a 25°C ni a 42°C. Las colonias (0,8 a 2,0mm) son grisáceas y ligeramente marrones, brillantes, opacas, secas y betahemolíticas. Como otras especies del género, produce catalasa e hidroliza la esculina. En el sistema Rapid ID 32A da un perfil 2111412001. A causa de la arginina dihidrolasa, betagalactosidasa, N-acetil- glucosaminidasa, arginina arilamidasa, leucina arilamidasa e histidina arilamidasa (que son positivas) y a la alfaglucosidasa (que es negativa), se diferencia de otras especies de Leptotrichia (tabla 5). A pesar de esto, los aislamientos clínicos se han identificado por secuenciación del ARNr 16S. Se ha caracterizado por secuenciación completa del ADNr 16S, hibridación ADN-ADN, análisis de RAPD, electroforesis de las proteínas de células enteras en SDS-PAGE, análisis de los ácidos grasos celulares y propiedades bioquímicas y enzimáticas. Su hábitat es presumiblemente oral. Descrita sobre 3 aislamientos: uno de bacteriemia36 y posteriormente 2 casos de endocarditis, de los que se la consideró causante. Uno fue de válvula mitral y otro fue de la válvula aórtica protésica, y se sospechó que el origen de la bacteria fue gástrico y oral (periodontitis), respectivamente. Se trataron con éxito mediante piperacilina más tazobactam y ceftriaxona más metronidazol, aunque ulteriormente requirieron tratamiento quirúrgico45. La cepa tipo es la LB 57T (=CCUG 32286T =CIP 107915T). La secuencia del ARNr 16S está depositada en GenBank con el número de acceso AY02980736.
Leptotrichia trevisanii
Descrita en 2001, su denominación es en honor a V. Trevisan, creador del nombre del género. Incluye a bacilos largos (0,8–0,9×6–13μm), fusiformes e inmóviles agrupados en parejas o en cadenas, y con los extremos unidos. Es aerotolerante, crece con un 5% de CO2. Tras 5 días de incubación produce colonias de aproximadamente 2mm, grisáceas y lisas. Produce catalasa y ureasa, y no produce indol ni oxidasa. Se identificó bioquímicamente mediante el sistema Rapid ANA II en el que proporciona un perfil 064160. Fermenta la glucosa y produce ácido láctico como producto final. Se aisló de una bacteriemia en un paciente con una leucemia mieloide aguda que se trató con éxito con ceftriaxona, gentamicina y metronidazol. Es sensible a penicilina, a la asociación de amoxicilina con ácido clavulánico, a imipenem, metronidazol y a cloranfenicol; de sensibilidad intermedia a clindamicina y resistente a eritromicina. La cepa tipo es la ATCC 700907T. La secuencia del ARNr 16S está depositada en GenBank con el número de acceso AF20630546.
Género OdoribacterEl Bacteroides splanchnicus, un patógeno humano ocasional relacionado con el género Porphyromonas, ha sido transferido al género Odoribacter (de olor y bacteria) como Odoribacter splanchnicus (tabla 1)47. Su descripción corresponde a la descrita originalmente por Werner et al48.
Género ParabacteroidesAlgunas especies del género Bacteroides, a pesar de presentar características fenotípicas propias de éste (producción de ácido acético y ácido succínico, resistencia a la bilis y poder sacarolítico), de acuerdo a datos obtenidos por estudios genéticos y a la composición de menoquinonas, han sido incluidos en el nuevo género Parabacteroides (“al lado de Bacteroides”) junto con Parabacteroides johnsonii49, en concreto: Bacteroides distasonis, Bacteroides goldsteinii y Bacteroides merdae y han pasado a denominarse Parabacteroides distasonis, Parabacteroides goldsteinii y Parabacteroides merdae50 (tabla 1). Tanto B. distasonis como B. goldsteinii están implicados en infecciones humanas51. Para profundizar en estas especies es útil consultar la revisión de Wexler52.
Género PrevotellaIncluye a bacilos gramnegativos, anaerobios estrictos, sensibles a la bilis (20%), débilmente sacarolíticos, que producen ácido acético y ácido succínico. Algunas especies dan colonias negras. Tienen su hábitat en la cavidad oral y en la vagina1. Es el género en el que se han comunicado recientemente más especies relacionadas tanto con infecciones humanas como con componentes de la flora normal.
Prevotella amnii
Descrita en 2008, debe su nombre al líquido amniótico. Formada por bacilos (0,8–3,0×0,5–1.5μm) inmóviles, relativamente aerotolerantes (6h), que producen colonias no pigmentadas. A las 48h de incubación son blancas, lisas y brillantes, de menos de 1mm de diámetro. Moderadamente sacarolítica (glucosa, lactosa, maltosa), hidroliza la esculina pero no la gelatina. Las pruebas bioquímicas obtenidas mediante el sistema Rapid ID 32A se pueden observar en la tabla 6. Está relacionada genética y fenotípicamente con Prevotella bivia, de la que se diferencia porque no produce alfafucosidasa y sí produce betagalactosidasa-6-fosfato. Además, esta propiedad la separa de Prevotella disiens, Prevotella intermedia, Prevotella nigrescens y Prevotella pallens, que carecen de ella. Se ha caracterizado por secuenciación del ARNr 16S y por los ácidos grasos celulares. Las 2 cepas empleadas para su descripción se aislaron de líquido amniótico. En uno de los casos era turbio y maloliente. No se han comunicado datos de sensibilidad a los antimicrobianos. La cepa tipo es la CCUG 53648T (=JCM 14753T). La secuencia del ARNr 16S depositada en GenBank/EMBL/DDBJ es la AM42212553.
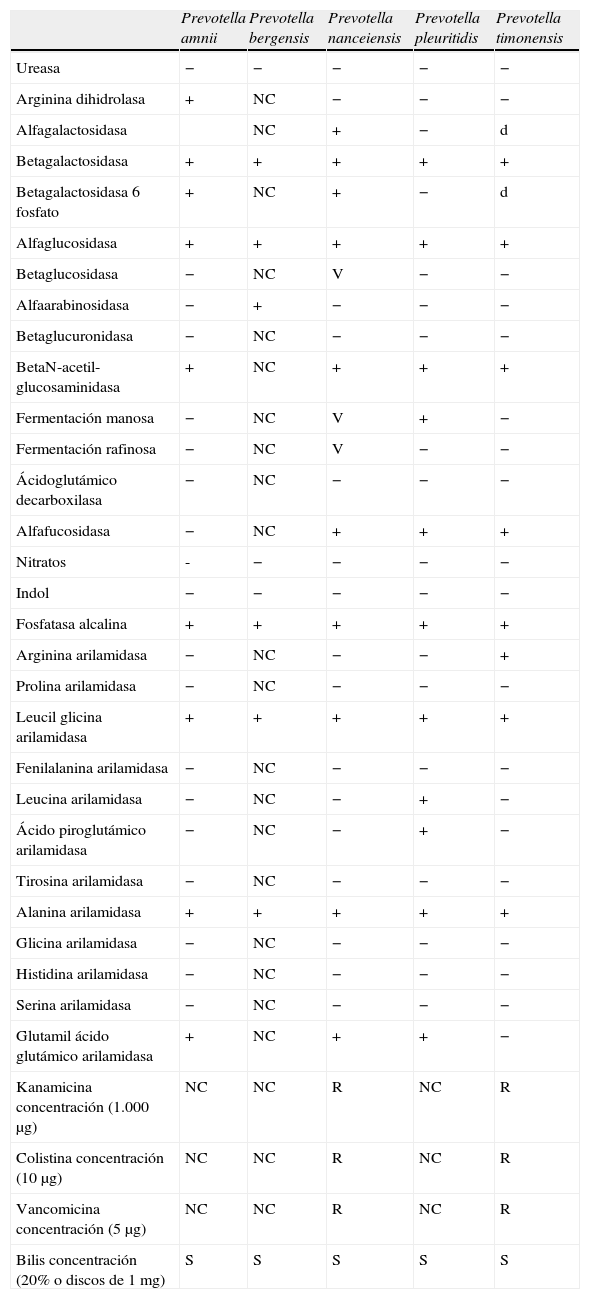
Características bioquímicas obtenidas mediante el sistema Rapid ID 32A y la utilización de discos diagnósticos de kanamicina, colistina y vancomicina y bilis de los nuevos bacilos gramnegativos anaerobios pertenecientes al género Prevotella
| Prevotella amnii | Prevotella bergensis | Prevotella nanceiensis | Prevotella pleuritidis | Prevotella timonensis | |
| Ureasa | − | − | − | − | − |
| Arginina dihidrolasa | + | NC | − | − | − |
| Alfagalactosidasa | NC | + | − | d | |
| Betagalactosidasa | + | + | + | + | + |
| Betagalactosidasa 6 fosfato | + | NC | + | − | d |
| Alfaglucosidasa | + | + | + | + | + |
| Betaglucosidasa | − | NC | V | − | − |
| Alfaarabinosidasa | − | + | − | − | − |
| Betaglucuronidasa | − | NC | − | − | − |
| BetaN-acetil-glucosaminidasa | + | NC | + | + | + |
| Fermentación manosa | − | NC | V | + | − |
| Fermentación rafinosa | − | NC | V | − | − |
| Ácidoglutámico decarboxilasa | − | NC | − | − | − |
| Alfafucosidasa | − | NC | + | + | + |
| Nitratos | - | − | − | − | − |
| Indol | − | − | − | − | − |
| Fosfatasa alcalina | + | + | + | + | + |
| Arginina arilamidasa | − | NC | − | − | + |
| Prolina arilamidasa | − | NC | − | − | − |
| Leucil glicina arilamidasa | + | + | + | + | + |
| Fenilalanina arilamidasa | − | NC | − | − | − |
| Leucina arilamidasa | − | NC | − | + | − |
| Ácido piroglutámico arilamidasa | − | NC | − | + | − |
| Tirosina arilamidasa | − | NC | − | − | − |
| Alanina arilamidasa | + | + | + | + | + |
| Glicina arilamidasa | − | NC | − | − | − |
| Histidina arilamidasa | − | NC | − | − | − |
| Serina arilamidasa | − | NC | − | − | − |
| Glutamil ácido glutámico arilamidasa | + | NC | + | + | − |
| Kanamicina concentración (1.000μg) | NC | NC | R | NC | R |
| Colistina concentración (10μg) | NC | NC | R | NC | R |
| Vancomicina concentración (5μg) | NC | NC | R | NC | R |
| Bilis concentración (20% o discos de 1mg) | S | S | S | S | S |
+: positivo; −: negativo; d: débil; NC: no comunicado; R: resistente; S: sensible; V: variable.
Prevotella bergensis
Caracterizada en 2006, debe su nombre a la ciudad de Bergen. Bacilo (0,7–0,8×0,8–6μm), inmóvil, capsulado, de crecimiento moderadamente lento (4 días), que produce colonias no pigmentadas de 0,6 a 0,8mm, opacas y de coloración gris a blanquecina. Sacarolítica, fermenta celobiosa, fructosa, glucosa, lactosa, maltosa, manosa, ramnosa, salicina y xilosa y además, como Prevotella buccae, Prevotella dentalis, Prevotella oris y Prevotella salivae, fermenta arabinosa, a diferencia de otras prevotelas54. Prevotella maculosa, recientemente comunicada a partir de muestras dentales, es también arabinosa positiva y, como las anteriores, sacarosa positiva a diferencia de P. bergensis55. Hidroliza la esculina pero no la gelatina y no produce catalasa. Las pruebas bioquímicas obtenidas en el sistema Rapid ID 32A se pueden observar en la tabla 6. Las que no se reflejan son débiles y variables. Produce ácido acético, ácido succínico y trazas de ácido isovalérico como productos finales del metabolismo. Se ha caracterizado por secuenciación del ARNr 16S y por los ácidos grasos celulares. Las cepas empleadas para su descripción se aislaron de infecciones de piel y de tejidos blandos: 2 de abscesos cutáneos, 3 de abscesos de tejidos blandos, herida y úlcera de presión. No se han comunicado datos de sensibilidad. La cepa tipo es la CCUG 94067913T (=CCUG 51224T=DSM 17361T=JCM 13869T). El contenido de G+C del ADN es de 48 moles%. La secuencia del ARNr 16S depositada en GenBank es la AY35061354.
Prevotella massiliensis
Comunicada en 2005, debe su nombre a Massilia, nombre romano de Marsella. Bacilo inmóvil que a los 4 días (crecimiento relativamente lento) produce colonias de aproximadamente 1mm, no pigmentadas, brillantes, lisas y no hemolíticas. Produce indol y oxidasa, no hidroliza la gelatina ni la esculina y es asacarolítica, lo que se observa utilizando el sistema API 20A. Estas características la diferencian claramente de otras especies del género. Es sensible a kanamicina (otras especies de Prevotella son resistentes) y es resistente a vancomicina. Se ha caracterizado por secuenciación del ARNr 16S y rpoB y por los ácidos grasos celulares. La cepa en la que se basa la descripción se aisló de un hemocultivo de un paciente febril, politraumatizado, que recibía ventilación mecánica y que había ingresado en la unidad de cuidados intensivos. Es sensible a penicilina, imipenem, claritromicina, clindamicina y metronidazol. La cepa tipo es la CIP 107630T. La secuencia del ARNr 16S depositada en GenBank es la AF487886 y la secuencia de la rpoB depositada es la DQ00548056.
Prevotella nanceiensis
Caracterizada en 2007, su nombre deriva de Nanceiensis, antiguo nombre de Nancy, ciudad en la que se aislaron las cepas. Incluye a bacilos cocoides o cortos (0,4–0,5×0,5–1μm), inmóviles, de crecimiento moderadamente lento (4 días), que producen colonias pequeñas de 0,5 a 1mm, blancas, lisas, no pigmentadas y rodeadas de una zona de betahemólisis. Sacarolítica, fermenta glucosa, lactosa, maltosa, manosa, rafinosa y sacarosa y variablemente celobiosa. Hidroliza la esculina pero no la gelatina; no produce catalasa (sistema API 20A). El perfil de 10 dígitos obtenido mediante la utilización del sistema Rapid ID 32A es 47(º/1) (1/5/7) 440222 y refleja la variabilidad de las pruebas incluidas en los bloques 2 y 3, entre las diferentes cepas estudiadas (tabla 6). Similar a P. buccalis y Prevotella veroralis, de las que se diferencia respectivamente por la ausencia de producción de arginina arilamidasa y de fermentación del xilano. Produce ácido acético, ácido láctico y ácido succínico como productos finales del metabolismo. Se ha caracterizado por secuenciación del ARNr 16S y rpoB y por los ácidos grasos celulares. Dos de las cepas empleadas para su descripción se aislaron en cultivo puro de un hemocultivo y de un absceso de pulmón. La otra cepa se aisló de un cultivo mixto de un lavado broncoalveolar. No se han comunicado datos de sensibilidad a los antimicrobianos. La cepa tipo es la LBN 293T (=AIP 261.03T=CIP 108993T=CCUG 54409T). La secuencia del ARNr 16S depositada en GenBank/EMBL/DDBJ es la AY957555. El contenido de G+C del ADN es de 39,4 moles%57.
Prevotella pleuritidis
Comunicada en 2007, debe su nombre a su origen. Bacilo (0,8×1,7–4,2μm) aislado e inmóvil. Produce colonias de 0,5 a 1,5mm, grises o blancogrisáceas, lisas, brillantes y no pigmentadas. Moderadamente sacarolítica, fermenta glucosa, lactosa, maltosa y manosa. Hidroliza la gelatina pero no la esculina y produce catalasa. Las propiedades obtenidas con el sistema Rapid ID 32A se pueden observar en la tabla 6. Es similar a Prevotella enoeca, de la que se diferencia porque ésta no produce leucina arilamidasa y glutamil glutámico arilamidasa. Se ha caracterizado por secuenciación del ARNr 16S, por los ácidos grasos celulares y por las menaquinonas. La cepa empleada para su descripción se aisló del líquido pleural de una pleuritis supurada. No se han comunicado datos de sensibilidad a antimicrobianos. La cepa tipo es la GTC 3021T (=JCM 14110T=CCUG 54350T). La secuencia del ARNr 16S depositada en GenBank/EMBL/DDBJ es la AB278593. El contenido de G+C del ADN es de 45,4 moles%58.
Prevotella timonensis
Notificada en 2007, debe su nombre al Hospital de la Timone (Marsella, Francia) donde se aisló la cepa tipo. Bacilo (0,3–0,5×0,8–1,4) inmóvil. Tras 72h de incubación en agar sangre da lugar a la aparición de colonias lisas, brillantes, blancogrisáceas, no pigmentadas y no hemolíticas. Crece entre 25 y 37°C con una temperatura óptima de crecimiento de 37°C. Moderadamente sacarolítica, fermenta glucosa, lactosa y maltosa. Hidroliza gelatina pero no la esculina y no produce catalasa. Las propiedades obtenidas mediante el sistema Rapid ID 32A se pueden observar en la tabla 6. A diferencia de P. buccalis, P. shahii y P. oralis, (especies similares) no fermenta ni la manosa ni la rafinosa. Se ha caracterizado por secuenciación del ARNr 16S y por los ácidos grasos celulares. Se aisló de un absceso mamario. Es sensible a la asociación de betalactámico inhibidor de betalactamasas, imipenem y metronidazol. La cepa tipo es la 4401737T (=CIP 108522T=CCUG 50105T). La secuencia del ARNr 16S depositada en GenBank/EMBL/DDBJ es la DQ51891959.