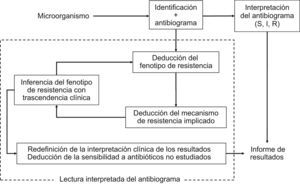
La lectura interpretada del antibiograma es una práctica habitual en el laboratorio de microbiología como complemento de la interpretación o de la categorización clínica de los resultados de sensibilidad. Consiste en el reconocimiento fenotípico de los mecanismos de resistencia y permite, a partir de éste, la inferencia de fenotipo inicial. Asimismo, condiciona la modificación de las categorías clínicas y la deducción de los valores de sensibilidad de antimicrobianos no incluidos en el antibiograma. Es útil en el control de calidad y en la validación de los resultados y se facilita con los sistemas automáticos de estudio de sensibilidad y paquetes informáticos asociados. La actual revisión de los puntos de corte con criterios farmacocinética/farmacodinamia y de correlación de los valores de concentración mínima inhibitoria con el resultado clínico puede hacer innecesaria que en la lectura interpretada se modifiquen las categorías clínicas. Es una herramienta imprescindible para establecer medidas epidemiológicas, adecuación de los tratamientos y aplicación de políticas de antimicrobianos. La lectura interpretada del antibiograma trasciende la vertiente clínica del microbiólogo y es útil en la toma de decisiones.
The interpretive reading of the antibiogram is routinely performed in the microbiology laboratory as a complement of the clinical categorization of antimicrobial susceptibility testing results. This process consists in the phenotypic recognition of resistance mechanisms allowing the inference of the initial resistance phenotype. Moreover, it assists in the modification of clinical categories and in the deduction of susceptibility of antimicrobials not included in the antibiogram. It is also useful in the quality control and validation of susceptibility results. This process is facilitated with the automatic susceptibility testing devices and the corresponding software. The current review of breakpoints with Pk/Pd (pharmacokinetics/pharmacodynamics) criteria and correlation of MIC values with clinical outcomes will make clinical category modifications unnecessary. It is an essential tool to establish epidemiological measures, correct antimicrobial treatments and implementation of antimicrobial policies. The interpretive reading of the antibiogram transcends the microbiologist's clinical role and is essential for clinical decisions.
En el año 2002 planteábamos en esta misma sección de Formación Continuada la lectura interpretada del antibiograma como parte inherente de la labor del microbiólogo en su trabajo diario en el laboratorio y como una necesidad clínica más allá de un mero ejercicio intelectual1. Ocho años después esta afirmación sigue vigente. Este proceso se ha incorporado plenamente en su actividad clínica y refuerza su participación en la toma de decisiones. Esta actitud es relevante en la elección del tratamiento antimicrobiano y en el conocimiento de la epidemiología de los mecanismos de resistencia y se ha convertido en una herramienta imprescindible para establecer medidas epidemiológicas en el control de las infecciones producidas por las bacterias resistentes y en la aplicación de las políticas de antimicrobianos, en particular, en las denominadas instituciones de cuidados de salud2,3.
En estos 8 años se ha producido un avance espectacular en el conocimiento de los mecanismos de resistencia, esencialmente de sus bases genéticas y en el de los determinantes asociados a su dispersión. Este hecho ha estado en parte influenciado por la introducción de la lectura interpretada del antibiograma en los laboratorios de microbiología, primer escalón en el reconocimiento de los mecanismos de resistencia, y también por la popularización de las técnicas de microbiología molecular. Asimismo, otros facultativos, con responsabilidad directa sobre el paciente, como infectólogos, pediatras o intensivistas, han comprendido la importancia de este proceso, han asumido cierto aprendizaje en este terreno y han demandado al microbiólogo información complementaria en los informes de sensibilidad.
Por otra parte, durante este tiempo también se ha acumulado un enorme conocimiento en el terreno de la farmacodinamia (Pd) de los antimicrobianos que ha influido de manera decisiva en la definición de los índices farmacocinética (Pk)/Pd como elementos predictivos de la eficacia clínica de estos compuestos. Para algunos antimicrobianos, estos índices han contribuido de manera decisiva en la consideración de las categorías clínicas utilizadas en la interpretación del antibiograma4,5. El proceso de armonización en Europa de los puntos críticos de sensibilidad y de resistencia liderado por el grupo European Committe of Antimicrobial Susceptibility Testing (EUCAST) y la revisión de los establecidos por el Clinical Laboratory Stardards Institute (CLSI) en los Estados Unidos han estado, en parte, guiados por este conocimiento6,7. Además, el grupo EUCAST ha establecido los denominados puntos de epidemiological cut-offs (ECOFF, ‘corte epidemiológicos’) y ha enunciado reglas de experto de los resultados de sensibilidad8.
La lectura interpretada del antibiograma no puede ser ajena a todos estos avances y debe incorporar aquellos conocimientos que permitan una mayor eficiencia en su ejercicio para aumentar el valor clínico de las pruebas de sensibilidad que diariamente se realizan en los laboratorios de Microbiología Clínica. Este hecho cobra una mayor importancia en momentos en los que es necesario reforzar las áreas de conocimiento y las competencias del microbiólogo clínico ante las tendencias de troncalidad de las especialidades de laboratorio de diagnóstico clínico y externalización de laboratorios9. La lectura interpretada, necesaria en la actividad clínica, cumple estos objetivos y trasciende la vertiente clínica del microbiólogo.
En este trabajo actualizaremos la información recogida en el año 2002 en relación con la lectura interpretada del antibiograma1 y revisaremos la influencia que pueden tener en este proceso los nuevos puntos de corte.
Pruebas de sensibilidad, puntos de corte y categorización clínica de los resultadosEl antibiograma tiene como objetivo evaluar en el laboratorio la respuesta de un microorganismo a uno o a varios antimicrobianos, y traducir, en una primera aproximación, su resultado como factor predictivo de la eficacia clínica. Las primeras pruebas de sensibilidad se realizaron en la década de 1920 del siglo pasado ligadas al propio descubrimiento de los antimicrobianos10. Tal y como se conocen en la actualidad, basadas en la difusión o en el cálculo de la concentración mínima inhibitoria (CMI), no se generalizaron hasta bien entrada la década de 1960. Con posterioridad se identificaron las múltiples variables que afectaban a los resultados obtenidos, y comienzan a establecer durante las décadas de 1970 y de 1980 normas sobre las condiciones en las que debían realizarse los antibiogramas con el objetivo de asegurar su reproducibilidad. Asimismo, durante estos años se debaten los criterios que deben regir la interpretación de los resultados. Éstos hacen esencialmente referencia al análisis de las poblaciones microbianas en función de los valores de la CMI de los antimicrobianos, su relación con los mecanismos de resistencia, la Pk del antimicrobiano, en particular en el compartimento sérico, y la correlación entre el valor de la CMI y el posible éxito o fracaso terapéutico11,12. Con estos objetivos se crean diferentes comités nacionales y otros con vocación internacional cuya misión principal es establecer los denominados puntos de corte con que se diferencian las categorías clínicas de tratamiento (sensible, intermedio y resistente)13,14. A pesar de utilizar los distintos comités criterios y datos de sensibilidad muy similares, los puntos de corte definidos eran muy dispares y se constata la necesidad de llegar a consensos que unifiquen estos valores12,15. Aunque se han realizado progresos importantes, particularmente en Europa, estas diferencias persisten hasta nuestros días5,6. Asimismo, diferentes grupos han utilizado distintas definiciones de las categorías clínicas que aparecen en los informes de sensibilidad y no fue hasta hace pocos años en los que la International Organization for Standardization16 redefinió estas categorías con el objetivo de evitar la confusión existente hasta el momento, en particular con la categoría intermedia. Éstas han quedado definidas en función de la probabilidad del éxito o del fracaso terapéutico:
- •
Sensible: cuando un aislado bacteriano es inhibido in vitro por una concentración de un antimicrobiano que se asocia a una alta probabilidad con el éxito terapéutico.
- •
Intermedio: cuando un aislado bacteriano es inhibido in vitro por una concentración de un antimicrobiano que se asocia a un efecto terapéutico incierto.
- •
Resistente: cuando un aislado bacteriano es inhibido in vitro por una concentración de un antimicrobiano que se asocia a una alta probabilidad con el fracaso terapéutico.
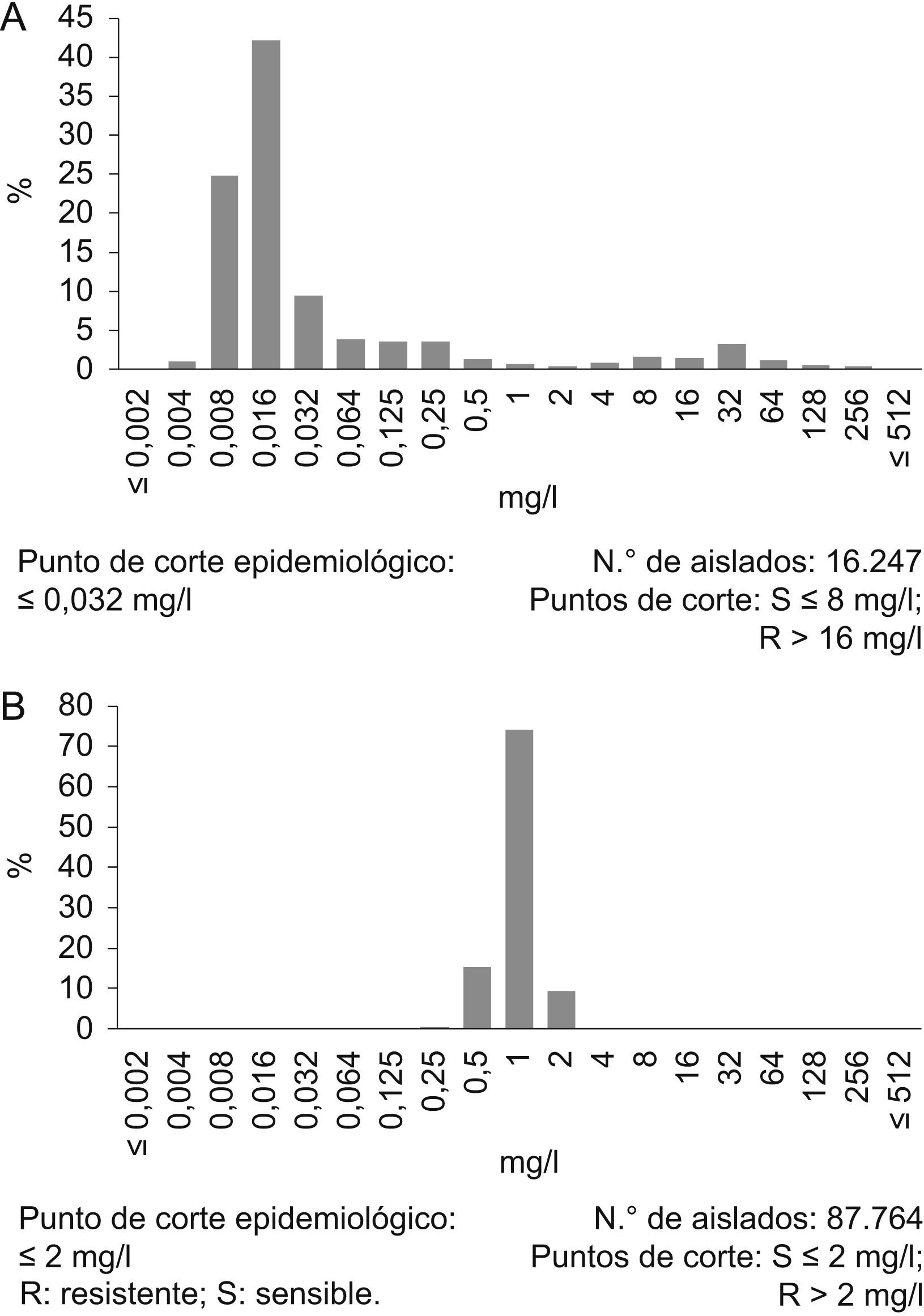
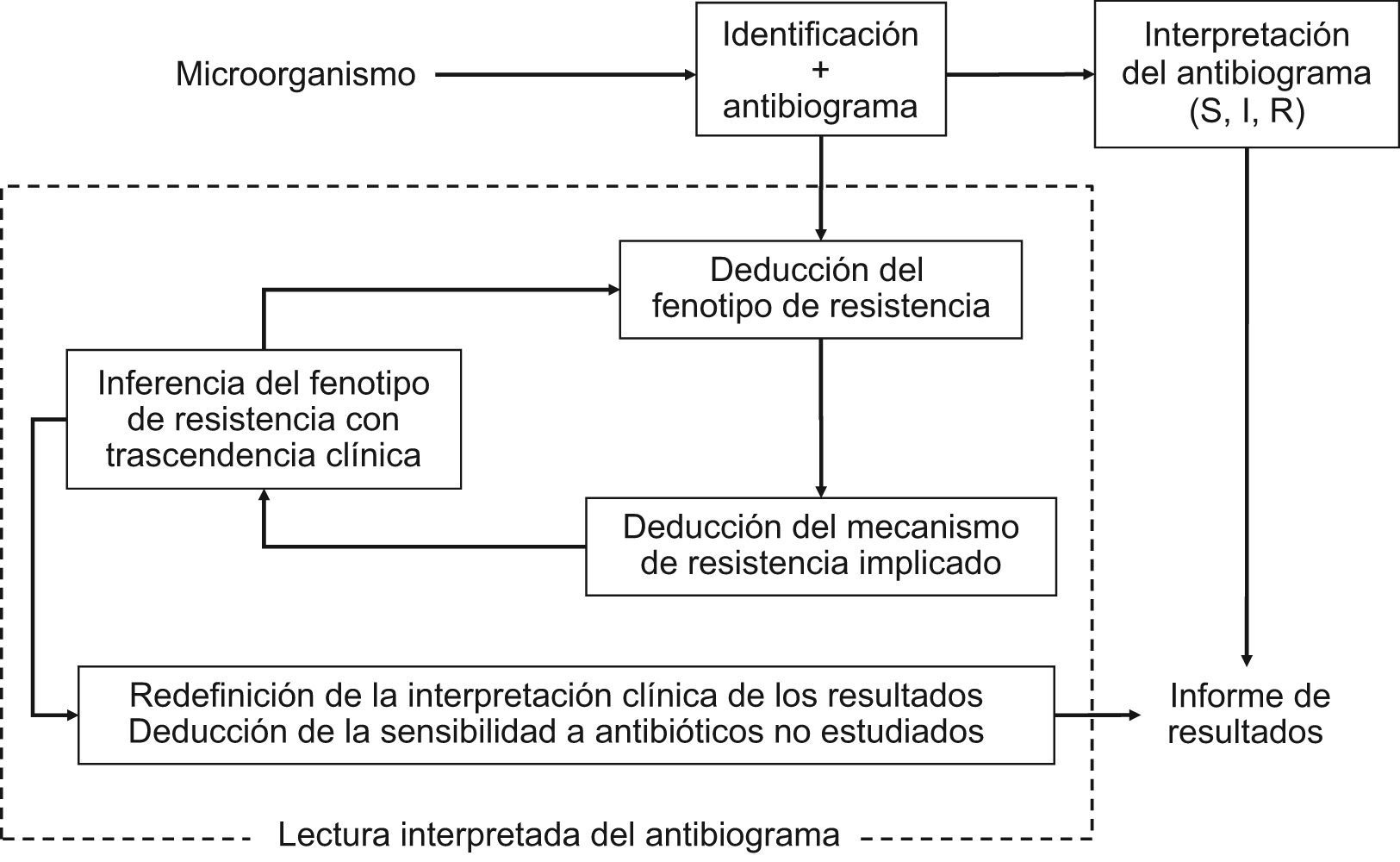
Los puntos de corte, bien en valores de halos de inhibición o de CMI, se utilizan para separar estas categorías. Tanto el CLSI como el grupo EUCAST establecen en los Estados Unidos y en Europa, respectivamente, estos puntos de corte y ambos comités tienen vocación internacional. En el primer caso se publican anualmente, mientras que en el segundo están permanentemente disponibles en su página web8. Este último grupo también ha establecido los denominados puntos de ECOFF que separan las poblaciones que carecen o no expresan mecanismos de resistencia de aquellas que los presentan y expresan. Los puntos de corte que definen la categoría clínica sensible no necesariamente han de coincidir con los ECOFF17. En la figura 1A se ilustra esta posibilidad para Escherichia coli y ciprofloxacino. Como se explicará más adelante, en estos casos, los ECOFF son más adecuados que los puntos de corte clínicos en el ejercicio de la lectura interpretada del antibiograma. En otras ocasiones, el punto de corte de sensibilidad coincide con el ECOFF, como en Staphylococcus aureus y la vancomicina (fig. 1B).
A) Distribución de los valores de concentración mínima inhibitoria del ciprofloxacino R: resistente; S: sensible para Escherichia coli. B) Distribución de los valores de concentración mínima inhibitoria de la vancomicina y Staphylococcus aureus. (Datos tomados de la página web del grupo European Committe of Antimicrobial Susceptibility Testing [http://www.eucast.org/mic_distributions_of_wild_type_microorganisms/]). Se indican los puntos de corte clínicos y el punto de corte epidemiológico definido por el grupo European Committe of Antimicrobial Susceptibility Testing. R: resistente; S: sensible.
La lectura interpretada del antibiograma no debe confundirse con el proceso de interpretación de los resultados de las pruebas de sensibilidad. Este último consiste en la categorización clínica de los resultados, es decir, en la traducción por medio de los puntos de corte clínicos, los halos de inhibición o los valores de CMI en las categorías clínicas sensible, intermedio o resistente, definidas en el apartado anterior. Por el contrario, la lectura interpretada realiza un análisis fenotípico de los resultados de las pruebas de sensibilidad y se fundamenta en el conocimiento de los mecanismos de resistencia y en su expresión fenotípica. Su objetivo principal es evitar el posible fracaso terapéutico derivado del uso antimicrobiano cuando se expresan estos mecanismos de resistencia en la bacteria estudiada en el antibiograma1,18,19. Esta actitud es complementaria a la categorización clínica. Microbiológicamente, con la lectura interpretada del antibiograma se facilita poder establecer su epidemiología con independencia de la propia caracterización fenotípica del mecanismo de resistencia2,20. Este hecho redunda en una mejor información para una correcta utilización dirigida y empírica de los antimicrobianos, por lo que puede influir en un mejor control de la resistencia. Incluso trasciende a un valor de microbiología de salud pública21.
El ejercicio de la lectura interpretada del antibiograma comenzó a desarrollarse a partir de la década de 1970. En esta década, y con mayor frecuencia en la década de 1980, muchos laboratorios de microbiología comenzaron a analizar habitualmente los datos de sensibilidad y trataron de asimilar sus resultados con los posibles mecanismos de resistencia22,23. Esta actitud permitió detectar e identificar rápidamente mecanismos de resistencia emergentes, incluso antes de que éstos tuvieran verdadera importancia clínica24. Este proceso se fundamentó en el conocimiento molecular de los mecanismos de resistencia y en su expresión fenotípica. Patrice Courvalin en el año 199218 recogió y explicó los conceptos y los objetivos de la lectura interpretada del antibiograma al enunciar los 3 pilares básicos o pasos en los que se fundamenta: a) caracterización del fenotipo de resistencia a partir del estudio de sensibilidad de un microorganismo previamente identificado frente a grupos de antibióticos pertenecientes a una misma familia o relacionados por mecanismos de resistencia comunes; b) deducción a partir del fenotipo de resistencia del correspondiente mecanismo bioquímico implicado, y c) inferencia, y modificación si es necesario, del fenotipo previamente establecido a partir del mecanismo de resistencia deducido.
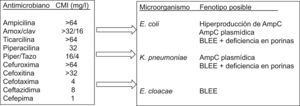
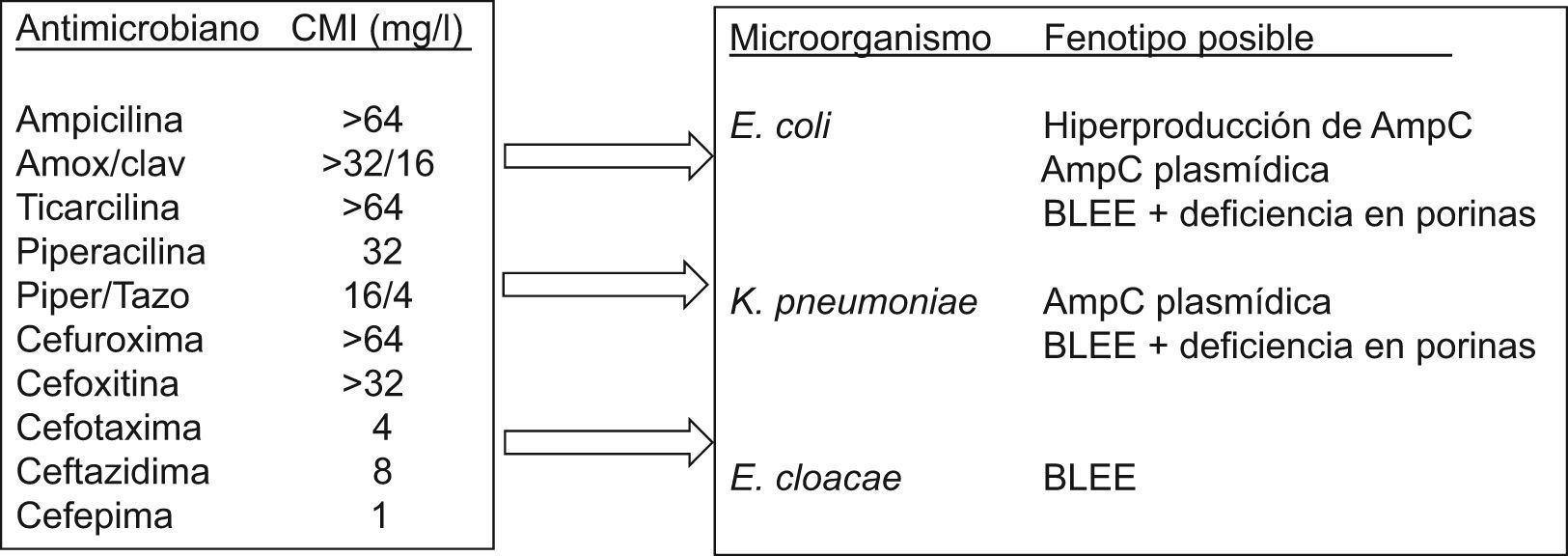
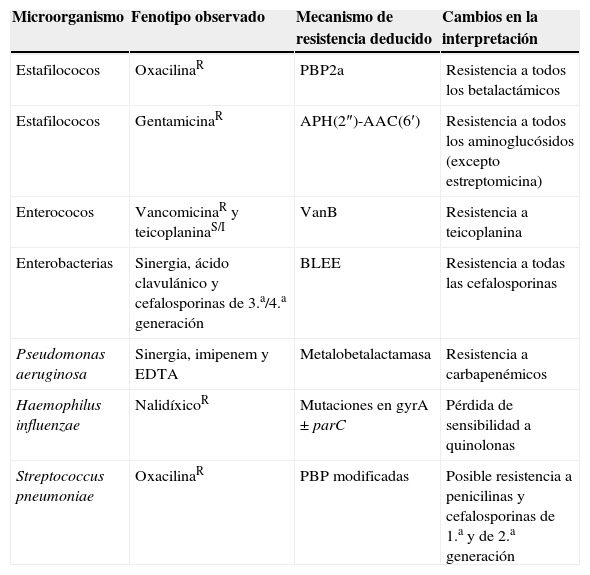
Durante el proceso de la lectura interpretada del antibiograma puede inferirse la sensibilidad de antibióticos no incluidos en el antibiograma. Esta práctica atiende esencialmente a la posible resistencia de clase, como es el caso de la resistencia a la oxacilina en S. aureus que implica resistencia a todos los antibióticos betalactámicos comercializados hasta la fecha o la resistencia al ciprofloxacino en Streptococcus pneumoniae cuando se detecta resistencia a levofloxacino o moxifloxacino25–27. En la figura 2 se indica esquemáticamente el proceso de interpretación del antibiograma y el de la lectura interpretada del antibiograma1 y en la tabla 1 se ofrecen algunos ejemplos clásicos.
Ejemplos de actuación en la lectura interpretada del antibiograma
| Microorganismo | Fenotipo observado | Mecanismo de resistencia deducido | Cambios en la interpretación |
| Estafilococos | OxacilinaR | PBP2a | Resistencia a todos los betalactámicos |
| Estafilococos | GentamicinaR | APH(2″)-AAC(6′) | Resistencia a todos los aminoglucósidos (excepto estreptomicina) |
| Enterococos | VancomicinaR y teicoplaninaS/I | VanB | Resistencia a teicoplanina |
| Enterobacterias | Sinergia, ácido clavulánico y cefalosporinas de 3.a/4.a generación | BLEE | Resistencia a todas las cefalosporinas |
| Pseudomonas aeruginosa | Sinergia, imipenem y EDTA | Metalobetalactamasa | Resistencia a carbapenémicos |
| Haemophilus influenzae | NalidíxicoR | Mutaciones en gyrA ± parC | Pérdida de sensibilidad a quinolonas |
| Streptococcus pneumoniae | OxacilinaR | PBP modificadas | Posible resistencia a penicilinas y cefalosporinas de 1.a y de 2.a generación |
BLEE: betalactamasas de espectro extendido; I: intermedio; R: resistente; S: sensible.
Tradicionalmente, en el ejercicio de la lectura interpretada del antibiograma se han modificado las categorías clínicas, esencialmente la de aquellos antibióticos escasamente afectados por los mecanismos de resistencia que en las pruebas de sensibilidad se informarían como sensible y se traducirían como resistentes1,18,19. Como ejemplo clásico se considera a los aislados de Salmonella resistentes al ciprofloxacino y a la levofloxacino (aun cuando los valores de las CMI sean sensibles), si estos aislados se muestran resistentes al ácido nalidíxico28–30. También, la resistencia a la amicacina y la tobramicina en S. aureus cuando éste es resistente a la gentamicina31,32. No obstante, el proceso de convergencia entre los diferentes comités que establecen los puntos de corte clínicos y la definición de valores cercanos o similares a los ECOFF pueden hacer innecesaria en un futuro esta modificación. Con esto se reafirma el valor predictivo de la CMI en la categorización clínica de los resultados de las pruebas de sensibilidad. En este caso, la lectura interpretada del antibiograma perdería valor en cuanto a su utilidad en la modificación de las categorías clínicas, pero mantendría el objetivo esencial de la detección fenotípica de los mecanismos de resistencia que podría utilizarse con posterioridad en el control epidemiológico de los microorganismos resistentes y en la adecuación de las políticas de antimicrobianos.
Ejemplo de esta nueva tendencia serían los criterios del CLSI publicados en 201033: se han reducido sustancialmente los puntos de corte clínicos de las cefalosporinas de tercera y de cuarta generación y se ha dejado de recomendar el cribado de los microorganismos potencialmente productores de betalactamasas de espectro extendido (BLEE) y, con esto, la modificación de las categorías clínicas cuando las cefalosporinas de amplio espectro aparecen como sensibles. Igualmente, en el caso del grupo EUCAST, con puntos de corte de resistencia aún más bajos para estas cefalosporinas, particularmente para ceftazidima y cefepima, se recomienda informar estos antimicrobianos (sensible, intermedio o resistente) tal y como se obtienen después de aplicar los puntos de corte, con independencia del posible mecanismo de resistencia implicado, incluidos los BLEE o las enzimas plasmídicas de tipo AmpC8. Se recomienda expresamente realizar la inferencia de la presencia de estas betalactamasas con un motivo epidemiológico y de control de infección. Este importante cambio de actitud ha venido impulsado por los datos de Pk/Pd en diferentes modelos, como las simulaciones de Montecarlo o en animal de experimentación, y en la información que demuestra el éxito terapéutico en el tratamiento de las infecciones producidas por enterobacterias con BLEE cuando las CMI para las cefalosporinas son iguales o inferiores al punto de corte de sensibilidad8,34–36.
Por otra parte, como un paso más en la lectura interpretada del antibiograma, el grupo EUCAST ha recogido de manera sistemática y ha publicado en su página web8 un conjunto de reglas o acciones (reglas de experto o expert rules), basadas en evidencias clínicas o microbiológicas, que deben llevarse a cabo como respuesta ante un resultado en una prueba de sensibilidad. Estas reglas están basadas en evidencias clínicas y microbiológicas e implican, con anterioridad a su enunciación, una lectura interpretada del antibiograma, y, posteriormente, un cambio de la categoría clínica19,37,38. Las evidencias clínicas y su traducción en reglas de experto podrían en un futuro hacer que se modifiquen nuevamente las interpretaciones de CMI categorizadas como sensibles por puntos de corte en microorganismos que además pertenecen a la población salvaje (CMI menor del punto crítico de sensibilidad y del ECOFF). Un ejemplo de actualidad y que cuenta con un número creciente de publicaciones que lo avalan es la situación producida por S. aureus, la vancomicina y los valores de CMI de 2 mg/l. A pesar de estar incluidos estos aislados dentro de la población salvaje (por debajo del punto de ECOFF), su aislamiento se ha asociado a una mayor probabilidad de fracaso terapéutico que la de aquellas cepas para las que la vancomicina tiene una CMI menor o igual a 1,5 mg/l. En esta situación se requeriría un cambio en la interpretación de la categoría clínica o en el punto de corte o una nota adicional en el antibiograma que alerte de esta situación39–41.
La lectura interpretada del antibiograma también puede hacer que se modifiquen los puntos de corte y las reglas de experto; como ejemplo, la posible extensión a todas las enterobacterias de la regla aplicada en Salmonella en relación con la resistencia al ácido nalidíxico y sensibilidad al ciprofloxacino que conlleva el cambio de ésta a resistente42,43. Este proceso, realizado por algunos laboratorios, se producirá de manera más evidente en los aislados que presentan resistencia al ácido nalidíxico y sensibilidad disminuida al ciprofloxacino por mutaciones en las topoisomerasas44. En este caso sería también importante considerar el tipo de infección, ya que tendría mayor importancia en la bacteriemia que en la infección urinaria, dado que estos compuestos se eliminan mayoritariamente por la orina45. Este cambio en la interpretación sería menos evidente en los aislados sensibles o con sensibilidad disminuida al ácido nalidíxico y con sensibilidad disminuida al ciprofloxacino, fenotipo relacionado con la presencia de genes transferibles de tipo qnr46. No obstante, a este respecto existe alguna evidencia clínica que avala la importancia clínica de este mecanismo47,48. Estos cambios en la interpretación podrían ser innecesarios con puntos de corte clínicos más ajustados y la lectura interpretada se realizaría con el objetivo de conocer el mecanismo de resistencia y, en su caso, establecer medidas epidemiológicas o de políticas de antimicrobianos.
Lectura interpretada, fenotipos de resistencia, sistemas automáticos y sistemas expertosEn la lectura interpretada del antibiograma debe procesarse la información en función de los fenotipos obtenidos con el objetivo final de la detección del mecanismo de resistencia. El fenotipo de sensibilidad o de resistencia se define como el conjunto de datos obtenidos en el antibiograma para antibióticos de la misma familia o relacionados por mecanismos de actuación comunes o mecanismos de resistencia compartidos. Se clasifican en habituales, raros e imposibles18. Los primeros, fenotipos habituales, recogen los aislamientos con mecanismos de resistencia cuya presencia es epidemiológicamente normal en el medio donde se realiza el estudio de sensibilidad. Como ejemplo clásico incluiríamos la resistencia a la penicilina, la sensibilidad a la oxacilina en S. aureus por producción de penicilinasa, la resistencia al ácido nalidíxico en las enterobacterias por alteración de la subunidad GyrA de la topoisomerasa ii o la resistencia a la eritromicina y clindamicina con sensibilidad a la estreptogramina A en S. pneumoniae por producción de una metilasa que afecta la afinidad de los macrólidos por el ribosoma. Al contrario de éstos, los fenotipos raros son consecuencia de la expresión de mecanismos de resistencia poco habituales, recientemente caracterizados o cuya dimensión epidemiológica es por el momento poco relevante en el área geográfica de estudio. En nuestro medio, entre los fenotipos raros se encontraría la resistencia a la vancomicina en Enterococcus spp.49, la resistencia a la clindamicina en ausencia de resistencia a la eritromicina en Staphylococcus spp.50, la resistencia a la quinupristina-dalfopristina en S. pneumoniae16, la resistencia al imipenem en Enterobacter cloacae51 o la resistencia al linezolid en estafilococo por la presencia del gen transferible cfr52,53.
Todos estos fenotipos deben definirse a nivel local ya que en algunos lugares pueden ser muy comunes, mientras que en otros pueden ser excepcionales o muy raros. En este sentido, la resistencia a carbapenémicos en Klebsiella pneumoniae es habitual en Grecia y es excepcional, por el momento, en nuestro país54,55.
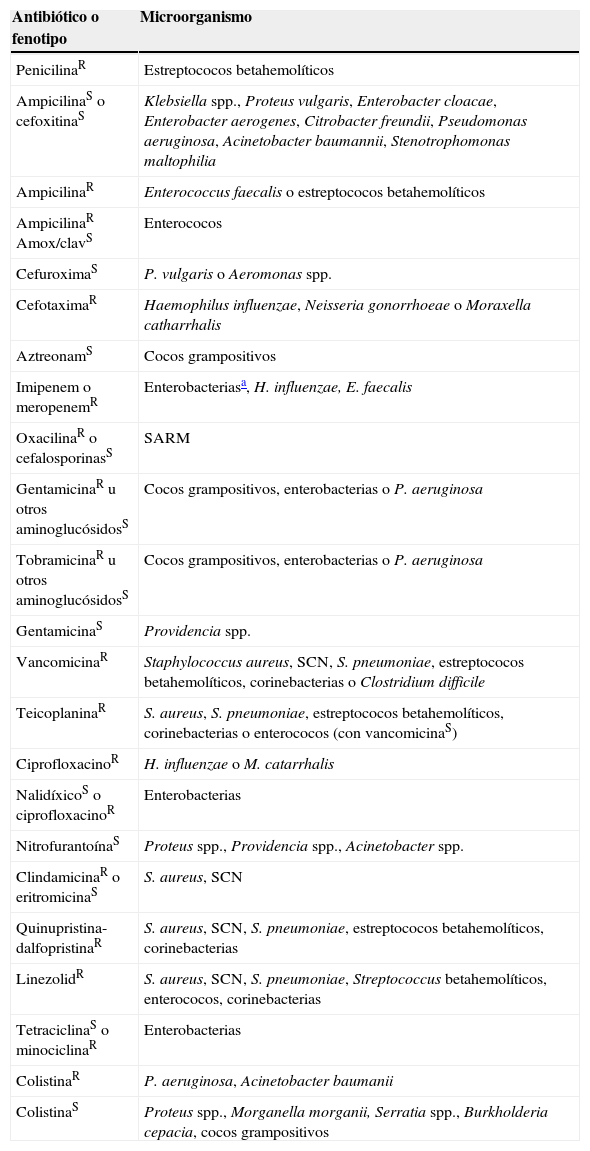
Los denominados fenotipos imposibles son particularmente interesantes en el proceso de la lectura interpretada del antibiograma. No responden a mecanismos de resistencia conocidos y, en la mayoría de los casos, no se confirman con un nuevo estudio de sensibilidad y suelen representar problemas técnicos derivados de la realización de las pruebas de sensibilidad o fallos en la identificación del microorganismo en el que se realiza el estudio. No obstante, la reiteración de este fenotipo en bacterias correctamente identificadas puede suponer un nuevo mecanismo de resistencia. Ante esta situación, debe caracterizarse el mecanismo implicado y, en su caso, enviar el microorganismo a un centro de referencia. En este epígrafe también se deben incluir aquellos microorganismos con resistencias naturales que presenten en el antibiograma fenotipos sensibles a los antibióticos teóricamente afectados por estos mecanismos. En la tabla 2 se indican algunos ejemplos de fenotipos que requieren su confirmación, bien por su rareza o por considerarse actualmente imposibles. Esta lista debe revisarse continuamente ya que pueden emerger nuevos mecanismos de resistencia, como S. aureus con resistencia a la vancomicina, descrita de forma esporádica en los Estados Unidos56.
Ejemplos de fenotipos raros e imposibles y que requieren confirmación de la identificación o del fenotipo
| Antibiótico o fenotipo | Microorganismo |
| PenicilinaR | Estreptococos betahemolíticos |
| AmpicilinaS o cefoxitinaS | Klebsiella spp., Proteus vulgaris, Enterobacter cloacae, Enterobacter aerogenes, Citrobacter freundii, Pseudomonas aeruginosa, Acinetobacter baumannii, Stenotrophomonas maltophilia |
| AmpicilinaR | Enterococcus faecalis o estreptococos betahemolíticos |
| AmpicilinaR Amox/clavS | Enterococos |
| CefuroximaS | P. vulgaris o Aeromonas spp. |
| CefotaximaR | Haemophilus influenzae, Neisseria gonorrhoeae o Moraxella catharrhalis |
| AztreonamS | Cocos grampositivos |
| Imipenem o meropenemR | Enterobacteriasa, H. influenzae, E. faecalis |
| OxacilinaR o cefalosporinasS | SARM |
| GentamicinaR u otros aminoglucósidosS | Cocos grampositivos, enterobacterias o P. aeruginosa |
| TobramicinaR u otros aminoglucósidosS | Cocos grampositivos, enterobacterias o P. aeruginosa |
| GentamicinaS | Providencia spp. |
| VancomicinaR | Staphylococcus aureus, SCN, S. pneumoniae, estreptococos betahemolíticos, corinebacterias o Clostridium difficile |
| TeicoplaninaR | S. aureus, S. pneumoniae, estreptococos betahemolíticos, corinebacterias o enterococos (con vancomicinaS) |
| CiprofloxacinoR | H. influenzae o M. catarrhalis |
| NalidíxicoS o ciprofloxacinoR | Enterobacterias |
| NitrofurantoínaS | Proteus spp., Providencia spp., Acinetobacter spp. |
| ClindamicinaR o eritromicinaS | S. aureus, SCN |
| Quinupristina-dalfopristinaR | S. aureus, SCN, S. pneumoniae, estreptococos betahemolíticos, corinebacterias |
| LinezolidR | S. aureus, SCN, S. pneumoniae, Streptococcus betahemolíticos, enterococos, corinebacterias |
| TetraciclinaS o minociclinaR | Enterobacterias |
| ColistinaR | P. aeruginosa, Acinetobacter baumanii |
| ColistinaS | Proteus spp., Morganella morganii, Serratia spp., Burkholderia cepacia, cocos grampositivos |
R: resistente; S: sensible; SARM: S. aureus resistentes a la meticilina; SCN: stafilococo coagulasa negativa.
El avance de la robótica y de la informática ha facilitado el desarrollo de aparatos automáticos o semiautomáticos para el estudio de la sensibilidad con el objetivo de simplificar el proceso. La mayoría de estos sistemas utilizan un sistema de microdilución que permite el cálculo del valor de la CMI, aunque también existen en el mercado sistemas que realizan la lectura de los halos de inhibición. Asimismo, suelen incluir programas informáticos, denominados expertos o lógicos-expertos, que además de categorizar los resultados en función de los valores de CMI o halos de inhibición, ofrecen información en la inferencia de los mecanismos de resistencia57. Como se indicará más adelante, es esencial un buen diseño de los paneles (antimicrobianos y concentraciones) para cumplir con los objetivos de la lectura interpretada del antibiograma58. También, los programas informáticos deben actualizarse con relativa periodicidad para incorporar los mecanismos de resistencia detectados recientemente y evitar que queden obsoletos en poco tiempo. Sería deseable que tuviesen en cuenta la epidemiología a nivel local. Hoy en día se han convertido en una parte esencial de los laboratorios de microbiología y aseguran una mayor calidad de los datos ofrecidos59.
Requisitos necesarios para la lectura interpretada del antibiogramaLa aplicación de los principios básicos que rigen la lectura interpretada del antibiograma precisa de un conocimiento previo de los mecanismos de resistencia y de una valoración adecuada de su expresión fenotípica. Además, son necesarios unos requisitos mínimos, resumidos a continuación. Sin estos en su ejecución puede ser inadecuada o no podrían obtenerse todos los beneficios que de ésta se derivan.
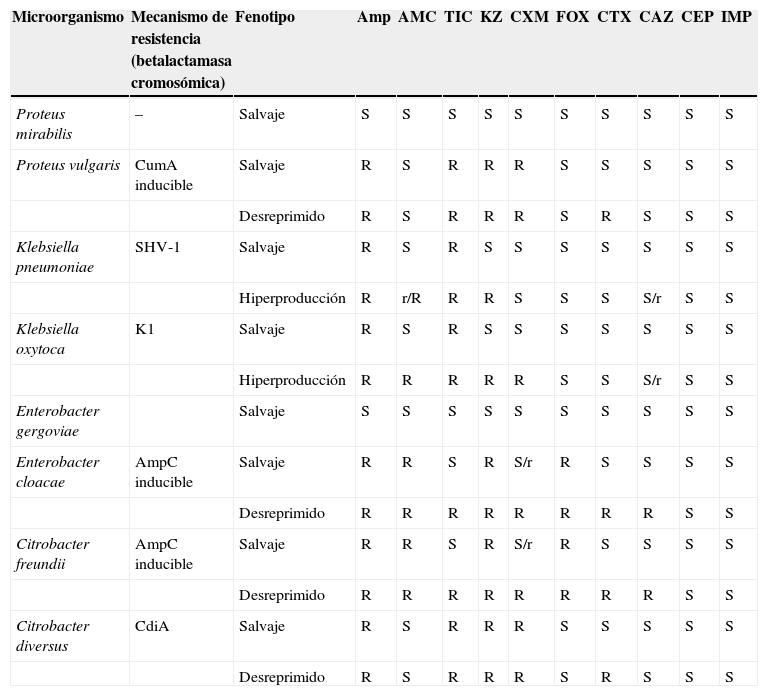
Identificación del microorganismo estudiadoDebe realizarse al mismo tiempo que el cálculo del valor de la CMI. Sin ésta, la aplicación del conocimiento interpretativo puede llevar a conclusiones erróneas y, por tanto, a una utilización incorrecta de los antimicrobianos o a la implantación de medidas de política de antimicrobianos erróneas. En la figura 3 se recoge como ejercicio un fenotipo de sensibilidad y las inferencias que pueden realizarse sin conocer previamente la identificación. Esta identificación debe realizarse al nivel de especie, ya que con sólo la identificación del género el análisis fenotípico podría conducir a varias posibilidades. La tabla 3 recoge algunos microorganismos de igual género, pero de diferente especie que presentan mecanismos de resistencia distintos. Los sistemas automáticos o semiautomáticos suelen cumplir con este requerimiento. Asimismo, la introducción en los laboratorios de sistemas de identificación basados en técnicas moleculares o de espectrometría de masas también facilita la diferenciación al nivel de especie.
Microorganismos que pertenecen a especies diferentes e igual género con mecanismos de resistencia que afectan a la lectura interpretada del antibiograma
| Microorganismo | Mecanismo de resistencia (betalactamasa cromosómica) | Fenotipo | Amp | AMC | TIC | KZ | CXM | FOX | CTX | CAZ | CEP | IMP |
| Proteus mirabilis | – | Salvaje | S | S | S | S | S | S | S | S | S | S |
| Proteus vulgaris | CumA inducible | Salvaje | R | S | R | R | R | S | S | S | S | S |
| Desreprimido | R | S | R | R | R | S | R | S | S | S | ||
| Klebsiella pneumoniae | SHV-1 | Salvaje | R | S | R | S | S | S | S | S | S | S |
| Hiperproducción | R | r/R | R | R | S | S | S | S/r | S | S | ||
| Klebsiella oxytoca | K1 | Salvaje | R | S | R | S | S | S | S | S | S | S |
| Hiperproducción | R | R | R | R | R | S | S | S/r | S | S | ||
| Enterobacter gergoviae | Salvaje | S | S | S | S | S | S | S | S | S | S | |
| Enterobacter cloacae | AmpC inducible | Salvaje | R | R | S | R | S/r | R | S | S | S | S |
| Desreprimido | R | R | R | R | R | R | R | R | S | S | ||
| Citrobacter freundii | AmpC inducible | Salvaje | R | R | S | R | S/r | R | S | S | S | S |
| Desreprimido | R | R | R | R | R | R | R | R | S | S | ||
| Citrobacter diversus | CdiA | Salvaje | R | S | R | R | R | S | S | S | S | S |
| Desreprimido | R | S | R | R | R | S | R | S | S | S |
AMC: amoxicilina/clavulánico; Amp: ampicilina; CAZ: ceftazidima; CEP: cefepima; CTX: cefotaxima; CXM: cefuroxima; FOX: cefoxitina; IMP: imipenem; KZ; cefazolina; R: resistente; r: sensibilidad disminuida; S: sensible; TIC: ticarcilina.
La información obtenida con el estudio de sensibilidad de un solo antibiótico es muy limitada y no ofrece habitualmente elementos válidos para la deducción de los mecanismos de resistencia implicados. Los resultados de sensibilidad deben considerarse en su conjunto y deben analizarse grupos de antibióticos pertenecientes a una misma familia o de diferentes familias, pero relacionados por un mismo mecanismo de resistencia.
Los ejemplos más sencillos son los de los betalactámicos o de los aminoglucósidos, ya que el análisis del fenotipo de resistencia permite, respectivamente, inferir la presencia de betalactamasas o de enzimas modificantes de aminoglucósidos e incluso facilitar su identificación presuntiva60,61. En el caso de los macrólidos, el fenotipo de resistencia debe partir del estudio de antibióticos de diferentes familias (macrólidos, lincosamidas y estreptograminas), pero relacionados entre sí por presentar dianas comunes de actuación50. En la detección genotípica del gen cfr en S. aureus que confiere resistencia al linezolid, puede ayudar la resistencia simultánea a otros antibióticos, incluidas, entre otras, la resistencia al cloranfenicol, a las lincosamidas y a la estreptogramina A53. En el caso de asociarse la resistencia al linezolid a mutaciones ribosómicas, este perfil de multirresistencia estaría ausente. Finalmente, para antibióticos que tienen dianas diferentes, pero que se afectan por un mecanismo de resistencia común (mecanismos pleiotrópicos de resistencia asociados a sistemas de expulsión), es necesario el estudio de betalactámicos, quinolonas, tetraciclinas, cloranfenicol, macrólidos y, en ocasiones, aminoglucósidos para deducir su presencia62.
Utilización de antibióticos marcadores o indicadores de la presencia de los mecanismos de resistenciaEn ocasiones, los antibióticos utilizados como marcadores han dejado de tener vigencia en la clínica, pero son fundamentales para inferir los mecanismos de resistencia. Como ejemplos, destacamos el ácido nalidíxico y la kanamicina, utilizados respectivamente para detectar con mayor eficiencia las enterobacterias resistentes a las quinolonas por mutaciones en la girasa63 y los estafilococos que producen APH(3′) y ANT(4′) que pueden aparecer falsamente sensibles a la amicacina en las pruebas habituales de sensibilidad64. Otro ejemplo clásico sería el de la oxacilina para predecir la resistencia de los estafilocococos a los betalactámicos o de S. pneumoniae a la penicilina65. En este apartado debe mencionarse la utilidad de algunos antibióticos marcadores en la identificación de los microorganismos, como la colistina, y que también suelen incluirse en los estudios de sensibilidad con fines taxonómicos18.
Estudio de combinaciones entre antimicrobianos e inhibidores de mecanismos de resistenciaEn la mayoría de las ocasiones las combinaciones empleadas no se utilizan en la clínica o los compuestos inhibidores se utilizan con otros fines. Entre éstos destacan los inhibidores de betalactamasas, como el ácido clavulánico, que asociado a la ceftazidima o a la cefotaxima permite deducir la presencia de BLEE65,66, o el EDTA, que asociado al imipenem o a la ceftazidima facilita el reconocimiento de determinadas carbapenemasas en las enterobacterias67. Recientemente se ha introducido el empleo del ácido borónico como inhibidor de betalactamasas de clase A, útil en la discriminación de carbapenemasas de tipo KPC en las enterobacterias68, se ha propuesto el ácido dipicolínico como alternativa al EDTA en la detección de las metalobetalactamasas69 y existen esquemas de utilización de cloxacilina para la detección de aislados con hiperproducción de betalactamasa cromosómica de tipo AmpC70. También se han utilizado clásicamente la reserpina o el CCCP en la caracterizaión de aislados con mecanismos de resistencia asociados a sistemas de eflujo71,72.
Estudio cuantitativo de sensibilidad con un amplio rango de concentracionesLa utilización exclusiva de las categorías clínicas (sensible, intermedia o resistente) puede limitar la lectura interpretada del antibiograma, en particular si los puntos de corte clínicos no son adecuados o están alejados de los puntos de ECOFF. Esta circunstancia se agrava cuando se consideran mecanismos de resistencia de baja expresión que implican valores de CMI o halos de inhibición que apenas superan los puntos de corte de sensibilidad establecidos.
El análisis interpretativo debe utilizar un amplio número de concentraciones y hacer especial énfasis en incluir concentraciones por debajo del punto de corte de sensibilidad con el objetivo de valorar cualquier desvío de los valores normales o habituales. Además, el estudio cualitativo y su análisis soslayan las discrepancias de criterios que puedan existir en la definición de las categorías clínicas entre los distintos comités. El ejemplo típico es el de los BLEE. Su presencia puede no incrementar significativamente los valores de CMI o reducir solo ligeramente los halos de inhibición de las cefalosporinas de espectro expandido o los del aztreonam, y permanecer, aun cuando se producen estas enzimas, dentro de la categoría «sensible». También sucede con otros antimicrobianos, como las fluoroquinolonas, la eritromicina o el linezolid, que pueden afectarse por mecanismos de resistencia con baja expresión73. En España existe un consenso de cuáles deben ser los antimicrobianos y las concentraciones para incluir en los paneles de sensibilidad de los sistemas automáticos, con el objetivo, entre otros, de facilitar la lectura interpretada58.
Estudio con inóculos elevados o sistemas que incrementan el valor de la concentración mínima inhibitoriaLa detección de mecanismos de resistencia con inóculos elevados puede facilitar el reconocimiento de determinados mecanismos de resistencia, entre éstos los asociados a bajo nivel de expresión o aquellos que se producen en poblaciones heterogéneas que no dan una uniformidad en el estudio de sensibilidad. Entre los ejemplos más representativos destacaríamos las carbapenemasas en Enterobacteriaceae y el de S. aureus con sensibilidad disminuida a los glucopéptidos74,75. En estos casos, el aumento del inóculo facilita su expresión y, por tanto, su inferencia.
Parecido al efecto del incremento del inóculo bacteriano estaría la utilización de sistemas que tienden a aumentar ligeramente el valor de la CMI. Para algunos antibióticos, como la vancomicina, el Etest ha demostrado valores ligeramente más elevados que los que se obtienen con los sistemas automáticos de microdilución. Por este motivo, cuando se compara la pérdida de eficacia de la vancomicina en pacientes con aislados de S. aureus resistentes a la meticilina (SARM) con CMI superiores a 1 mg/l con la que se produce en aquellos que tienen valores de CMI inferiores o iguales a 1, la correlación con el Etest es mejor que con los métodos automáticos76.
Conocimiento de la epidemiología local de la resistencia a los antimicrobianosPuesto que el análisis de los fenotipos de resistencia permite su clasificación en fenotipos habituales, raros e imposibles18, es también imprescindible conocer cuál es la situación epidemiológica de los mecanismos de resistencia en el área geográfica en la que se esté aplicando la lectura interpretada del antibiograma. Con esto puede mejorarse el reconocimiento de los mecanismos de resistencia habituales y la detección fenotípica de los nuevos.
La resistencia a la eritromicina en S. pneumoniae en los Estados Unidos y en Canadá está más frecuentemente asociada a los sistemas de expulsión, mientras que en Europa suele deberse a la producción de una metilasa que altera la conformación del ribosoma50. El primer mecanismo no afecta a la clindamicina (fenotipo M), mientras que en el segundo se elevan simultáneamente los valores de CMI de eritromicina y clindamicina (fenotipo MLSB). Asimismo, en nuestro hospital la detección de carbapenemasas (metalobetalactamasa) en enterobacterias parte de la premisa de su escasa presencia, por lo que todos los aislados con sensibilidad disminuida a los carbapenémicos deben estudiarse como posibles productores de estas enzimas54.
Disponibilidad de técnicas de referenciaLa mayoría de los laboratorios de microbiología en España utilizan sistemas automáticos o semiautomáticos para la determinación de la sensibilidad a los antimicrobianos. En algunas ocasiones, estos sistemas pueden ofrecer resultados anómalos y es difícil discernir si estos errores están relacionados con problemas técnicos o con la aparición de nuevos mecanismos de resistencia. La utilización de técnicas de referencia o el envío de los aislamientos a otro centro para su estudio molecular debe ser una práctica habitual que profundice en el conocimiento de los mecanismos de resistencia.
Beneficios de la lectura interpretadaLos beneficios derivados de la lectura interpretada del antibiograma son evidentes y redundan en valores añadidos a las pruebas de sensibilidad. Tanto es así, que hoy en día no debe entenderse el estudio de sensibilidad sin llevar asociado este proceso. Estos beneficios pueden agruparse en la detección de nuevos mecanismos, en el conocimiento de la epidemiología de la resistencia, en la mejor adecuación del tratamiento antimicrobiano y en la mejora de la calidad y la gestión de los resultados.
Detección de nuevos mecanismos de resistencia y predicción de la aparición de resistenciasLa constatación de un fenotipo inesperado puede representar la aparición de un nuevo mecanismo de resistencia. Este debe comprobarse mediante la utilización de métodos de referencia y, en su caso, enviarse a un centro de referencia para su caracterización. Este proceso es esencial en la detección de los mecanismos de resistencia de bajo nivel de expresión y que pueden derivar en otros con mayor nivel y de gran trascendencia clínica73.
Por otra parte, el ejercicio de la lectura interpretativa a partir del estudio de sensibilidad de mutantes o construcciones in vitro permite anticipar el fenotipo de resistencia; como ejemplos, el estudio de mutantes con expresión establemente desreprimida de su betalactamasa cromosómica, AmpC en Citrobacter freundii o en Pseudomonas aeruginosa, de variantes de betalactamasas en bacterias en las que se han transferido los genes correspondientes con el objetivo de conocer su expresión77. Con el análisis interpretativo contribuiremos a su identificación y al establecimiento de medidas de control.
Análisis de la epidemiología de la resistenciaLa lectura interpretada del antibiograma permite definir los perfiles epidemiológicos de los distintos mecanismos de resistencia, y conseguir aumentar la información generada por los antibiogramas y la posición del laboratorio de microbiología en el control epidemiológico de la resistencia2,20,78. A modo de ejemplo, la resistencia de E. coli a la ampicilina en nuestro medio se sitúa en torno al 60%. Esta cifra, por sí misma importante, adquiere mayor trascendencia si decimos que se debe a la suma de diferentes mecanismos de resistencia, incluidas las betalactamasas de amplio espectro de tipo TEM, SHV u OXA, las hiperproductoras de betalactamasas de amplio espectro, las betalactamasas con resistencia a inhibidores de betalactamasas (enzimas IRT), las BLEE, la hiperproducción de AmpC o cefamicinasas, y por alteración de la permeabilidad. La lectura interpretada permite estudiar la presencia de los diferentes mecanismos de resistencia que afectan a un antibiótico determinado y permite analizar las diferencias entre pacientes, unidades, áreas o países y su evolución temporal. Un ejemplo ilustrativo es el cambio epidemiológico de SARM y de los microorganismos productores de BLEE. En ambos casos se encuentran crecientemente en el ámbito extrahospitalario79,80. La lectura interpretada podría ser una herramienta útil para el reconocimiento de los clones de alto riesgo que presentan elevada epidemicidad y que pueden permanecer durante largos períodos de tiempo en el ambiente hospitalario81,82. Con esta práctica se pueden modificar actuaciones epidemiológicas ante situaciones de brotes.
Adecuación de los tratamientos antimicrobianos y control de la política de antimicrobianosEl análisis fenotípico permite adecuar los tratamientos antimicrobianos a los perfiles de sensibilidad y de resistencia ofrecidos en los informes de sensibilidad, sobre todo en aquellos casos en los que los puntos de corte clínicos fuesen inadecuados (tabla 1). Esta práctica es ya antigua y arrancaría de la constatación de la ineficacia del tratamiento con cualquier antibiótico betalactámico en pacientes con infecciones producidas por aislamientos de SARM83. Ejemplos más cercanos serían el riesgo de selección de mutantes establemente desreprimidos con la utilización de cefalosporinas de tercera generación en el tratamiento de infecciones producidas por Enterobacter spp. o C. freundii84, la ineficacia de la teicoplanina en el tratamiento de las infecciones producidas por Enterococcus spp. con fenotipo VanB (resistentes a la vancomicina y aparentemente sensible a la teicoplanina)85 y la no recomendación con puntos de corte antiguos del uso de cualquier cefalosporina ante el aislamiento de enterobacterias con BLEE65.
La lectura interpretada del antibiograma puede utilizarse también en la restricción de la información, práctica habitual en la aplicación de la política de antibióticos. Con esta estrategia se mejora la selección de las opciones terapéuticas y se adecuan los tratamientos a los posibles mecanismos de resistencia. Asimismo, la lectura interpretada puede ser una buena herramienta para establecer cambios en las políticas de antibióticos, ya que puede alertar de la aparición de perfiles de resistencia que afectan a los tratamientos recomendados y permite establecer fundamentos en los ciclos de utilización de antimicrobianos86. Un buen ejemplo sería los sucesivos cambios en la política de antimicrobianos que se ha llevado a cabo con los aminoglucósidos o con los betalactámicos. En el primer caso, la rotación de los aminoglucósidos se fundamentó en los perfiles de resistencia observados, y se reintrodujeron o se eliminaron del formulario los diferentes aminoglucósidos87. Con los betalactámicos la mejor experiencia contrastada fue la de una institución en la que el sucesivo cambio de política de antibióticos condujo a la emergencia de aislados con diferentes mecanismos de resistencia, primero de aislamientos con BLEE y posteriormente de Acinetobacter baumannii multirresistente88.
Mejora de la calidadLa lectura interpretada del antibiograma puede servir como control de la validación de los propios resultados de sensibilidad. Durante este proceso se integran los valores de sensibilidad de cada uno de los antimicrobianos con el conjunto de éstos, y pueden detectarse anomalías o inconsistencias en la identificación de los microorganismos o problemas derivados de la aplicación de las técnicas de sensibilidad. Este aspecto es de gran importancia con los aparatos automáticos de sensibilidad, que presentan sistemas expertos asociados que suelen alertar de posibles anomalías y mejoran la calidad de los resultados obtenidos57.
Asimismo, con la lectura interpretada se incrementa la información generada en el antibiograma. Aparte de la corrección de los resultados obtenidos (fig. 2), es posible reducir el número de antibióticos por estudiar sin disminuir la información ofrecida, ya que tras la inferencia del fenotipo podemos deducir la sensibilidad de antimicrobianos no estudiados57. Entre los ejemplos típicos podemos citar el de las quinolonas o el de las cefalosporinas orales de primera generación. Aunque estos compuestos presentan algunas diferencias en cuanto a su actividad intrínseca, incluso cuando existen mecanismos de resistencia, el estudio de la sensibilidad de uno de éstos puede servir, respectivamente, como guía de la sensibilidad del resto de los antibióticos de su grupo, ya que la resistencia es cruzada.
Limitaciones de lectura interpretada del antibiogramaLa lectura interpretada del antibiograma requiere la aplicación de numerosos conocimientos que puede limitar su proceso. Estos conocimientos están fundamentalmente asociados a: i) la propia naturaleza de los mecanismos de resistencia, sus bases genéticas, expresión y epidemiología; ii) los antimicrobianos y su farmacología, incluidas la Pk, la Pd y la relación Pk/Pd, y iii) la relación entre la utilización clínica de los antimicrobianos y el posible éxito o fracaso terapéutico, sobre todo en las infecciones producidas por bacterias resistentes.
Una de las limitaciones más importantes de la lectura interpretada del antibiograma deriva de la propia complejidad de los mecanismos de resistencia, sobre todo en aquellas bacterias multirresistentes que presentan varios mecanismos de resistencia. También esta limitación está motivada por la existencia de un mayor número de mecanismos que son capaces de afectar a un mismo antimicrobiano o a varios de la misma familia. Este fenómeno de multirresistencia también se ha denominado capitalismo genético, que incide en el hecho de que una bacteria resistente tiene mayor probabilidad de acumular mayor número de mecanismos de resistencia89. Se ha constatado con frecuencia que las enterobacterias o los aislados de P. aeruginosa que son resistentes a los aminoglucósidos presentan más de una enzima modificante61: que las cepas de K. pneumoniae con BLEE suelen tener deficiencias en sus porinas88; que las enterobacterias con BLEE también producen resistencia a los aminoglucósidos por producción de metilasas o enzimas modificantes de aminoglucósidos y a quinolonas asociadas a genes transferibles en plásmidos, como qnr, aac[6’]I-cr o qepA46 ; la presencia simultánea de varias BLEE en el mismo aislado90; la producción de BLEE en enterobacterias con metalobetalactamasas54,91; que los aislados de P. aeruginosa resistentes a los carbapenémicos lo son por la presencia de varios mecanismos de resistencia60, y que cada vez son más frecuentes los aislamientos de S. pneumoniae, S. pyogenes o estafilococos que presentan varios mecanismos capaces de afectar independientemente a los macrólidos50,92,93.
Por otra parte, la presencia de mecanismos de resistencia intrínsecos puede enmascarar la adquisición de otros mecanismos, tal y como sucede en los bacilos gramnegativos no fermentadores, como P. aeruginosa en la que es difícil inferir a priori la presencia de los BLEE94. El conocimiento genético de los mecanismos asociados a la multirresistencia y la corresistencia (transposones, integrones, secuencias de inserción, procesos de conjugación y recombinación) puede explicar su fundamento, pero no da respuestas interpretativas en el antibiograma. En un futuro, la aplicación de las técnicas moleculares podrá parcialmente resolver este problema, aunque debe recordarse que la presencia de un determinante de resistencia no implica por sí mismo su expresión y, por tanto, la resistencia fenotípica.
Las limitaciones de la lectura interpretada pueden también agravarse por la necesidad de que varios mecanismos estén presentes en una misma bacteria para que se manifieste fenotípicamente la resistencia, como es el caso de la resistencia a quinupristina-dalfopristina en los estafilococos92 o la resistencia a los carbapenémicos en las enterobacterias no productoras de carbapenamasas95,96. Igualmente, la expresión de los mecanismos de resistencia puede variar drásticamente conforme la bacteria en la que se presente, como el caso de las carbapenemasas según se presenten en enterobacterias, P. aeruginosa o en A. baumanni97. Estos hechos pueden impedir el reconocimiento de determinados mecanismos y redundar en su mayor diseminación.
La diferente validez de algunas de las técnicas de sensibilidad también puede afectar los resultados obtenidos. En este sentido, es bien conocido que el estudio de los halos de inhibición del ciprofloxacino en S. pneumoniae puede dar lugar a falsas conclusiones aun cuando la cepa presente mutaciones en parC o en gyrA, la ausencia de correlación entre la producción de betalactamasa y la resistencia a la penicilina en S. aureus, la ineficacia de la técnica de difusión para detectar aislados de S. aureus con resistencia a los glucopéptidos o la escasa afectación de la ampicilina en las cepas de enterococo productoras de betalactamasa. Nuevamente, esta ineficacia suele producirse con los mecanismos de resistencia con bajo nivel de expresión.
Por último, debemos destacar que la simplificación en el análisis interpretativo puede limitar la identificación de nuevos mecanismos, ya que puede darse por supuesta la presencia de un determinado mecanismo de resistencia ante un fenotipo concreto y no explorarse otras posibilidades.
Aspectos de futuro y conclusionesLa revisión de los puntos de corte mediante la utilización de criterios Pk/Pd y de correlación de los valores de CMI con el resultado clínico puede hacer innecesaria que en la lectura interpretada del antibiograma se modifiquen en un futuro las categorías clínicas. Este proceso reforzaría el valor de la CMI como valor predictivo de la eficacia clínica en la utilización de los antimicrobianos y el análisis conjunto de los valores de CMI en el antibiograma tendría gran relevancia en la detección fenotípica de los mecanismos de resistencia. Los sistemas automáticos y semiautomáticos para el estudio de sensibilidad se han incorporado plenamente a los laboratorios de microbiología, se han convertido en herramientas de gran utilidad en el proceso de la lectura interpretada del antibiograma y han facilitado enormemente esta labor al microbiólogo clínico. La lectura interpretada del antibiograma sigue siendo un proceso necesario en el estudio de la sensibilidad a los antimicrobianos. Ésta es complementaria a la categorización clínica de los resultados de sensibilidad, permite una mejor gestión de éstos, tiene aplicaciones importantes para el tratamiento antimicrobiano y el control de las enfermedades infecciosas, y facilita la participación del microbiólogo en la toma de decisiones clínicas.
En la actualidad, la lectura interpretada del antibiograma se ha convertido en uno de los primeros escalones en el estudio de la resistencia a los antimicrobianos. Precisa una aplicación posterior de técnicas de microbiología molecular para la caracterización definitiva del mecanismo de resistencia. Aunque se han producido avances importantes en los últimos años, la automatización de las técnicas de microbiología molecular en el terreno de la resistencia a los antimicrobianos está aún por desarrollarse plenamente. Estas técnicas tienen numerosas ventajas, pero también presentan limitaciones, como las relacionadas con su coste económico, la posible ausencia de correlación entre la presencia de los genes de resistencia y su expresión fenotípica o la hipotética diferencia entre la expresión in vitro e in vivo. La irrupción de las técnicas y las plataformas de alto rendimiento (high throughput) en el ámbito del diagnóstico puede facilitar esta labor, incluso aplicada sobre las muestras de los pacientes y no exclusivamente sobre los microorganismos aislados. El microbiólogo clínico no es ajeno a este avance en el contexto de su actividad en el laboratorio y en la importancia de su labor en la toma de decisiones. La lectura interpretada del antibiograma continúa siendo de plena actualidad y es necesaria en su actividad clínica.





![A) Distribución de los valores de concentración mínima inhibitoria del ciprofloxacino R: resistente; S: sensible para Escherichia coli. B) Distribución de los valores de concentración mínima inhibitoria de la vancomicina y Staphylococcus aureus. (Datos tomados de la página web del grupo European Committe of Antimicrobial Susceptibility Testing [http://www.eucast.org/mic_distributions_of_wild_type_microorganisms/]). Se indican los puntos de corte clínicos y el punto de corte epidemiológico definido por el grupo European Committe of Antimicrobial Susceptibility Testing. R: resistente; S: sensible. A) Distribución de los valores de concentración mínima inhibitoria del ciprofloxacino R: resistente; S: sensible para Escherichia coli. B) Distribución de los valores de concentración mínima inhibitoria de la vancomicina y Staphylococcus aureus. (Datos tomados de la página web del grupo European Committe of Antimicrobial Susceptibility Testing [http://www.eucast.org/mic_distributions_of_wild_type_microorganisms/]). Se indican los puntos de corte clínicos y el punto de corte epidemiológico definido por el grupo European Committe of Antimicrobial Susceptibility Testing. R: resistente; S: sensible.](https://static.elsevier.es/multimedia/0213005X/0000002800000006/v2_201308271827/S0213005X1000087X/v2_201308271827/es/main.assets/thumbnail/gr1.jpeg?xkr=ue/ImdikoIMrsJoerZ+w96p5LBcBpyJTqfwgorxm+Ow=)