Introducción
Durante la última década, el siempre perseguido diagnóstico rápido, sensible y fiable de los agentes causales de las enfermedades infecciosas ha empezado a experimentar una revolucionaria transformación. La aplicación de técnicas microbiológicas convencionales, que requieren el crecimiento de los microorganismos para su posterior análisis fenotípico y bioquímico, ha pasado a ser complementada con la aplicación de métodos moleculares de diagnóstico. En la actualidad, de entre tales metodologías, la reacción en cadena de la polimerasa (PCR) es la más sensible y la que se aplica más satisfactoriamente en microbiología clínica. En particular, en el caso de patógenos que son difíciles de crecer in vitro o que presentan crecimiento lento, así como en el de todas aquellas infecciones clínicamente atribuibles a diferentes agentes, la PCR ha aportado un gran valor diagnóstico. En esta revisión, para evitar una exposición demasiado compleja, sólo se van a considerar las PCR múltiples desarrolladas para el diagnóstico de bacterias y no se incluirá el diagnóstico de hongos, virus y parásitos. A partir de 1990 se han desarrollado múltiples protocolos que permiten identificar de forma específica y rápida bacterias de diagnóstico difícil hasta ahora, como Mycobacterium spp., Chlamydia spp., etc., así como detectar la presencia de genes de relevancia clínica, como los que codifican resistencia a antibióticos o los que confieren mayor virulencia a los organismos que los portan1-3. Una vez optimizadas tales aplicaciones individuales de la PCR y comprobada su utilidad clínica, durante los últimos años ha cobrado gran interés el desarrollo de las denominadas PCR múltiples, reacciones que consiguen detectar de forma simultánea y en un único tubo diferentes secuencias diana, permitiendo la detección e identificación simultánea de distintos genes de interés.
En la actualidad, las posibles aportaciones de la PCR múltiple a la microbiología clínica son tantas y tan variadas que sería demasiado extenso introducirlas todas en una misma revisión del tema, por lo que en este capítulo no se ha intentado más que reflejar cómo la aplicación de esta tecnología puede ser de gran ayuda en la detección e identificación de bacterias de interés clínico y de sus genes de resistencia y/o virulencia. En concreto, se abordará su aplicabilidad en el diagnóstico de: Escherichia coli, otras enterobacterias, Pseudomonas aeruginosa, estafilococos, enterococos, Campylobacter spp. Mycobacterium spp., agentes causales de la meningitis bacteriana, de las infecciones genitourinarias, de la neumonía, de otitis media.
Por otro lado, durante los últimos años se han empezado a desarrollar protocolos de PCR múltiple muy prometedores mediante la utilización de PCR en tiempo real. Esta es una nueva metodología que combina la PCR con el uso de marcadores fluorescentes de forma que se pueda monitorizar la cinética de la reacción, conocer la cantidad de ADN molde y detectar la presencia de variaciones genéticas. En este capítulo no se profundizará en los protocolos de PCR múltiple en tiempo real, que se abordarán en un próximo número de Enfermedades Infecciosas y Microbiología Clínica.
La PCR múltiple: ventajas y dificultades
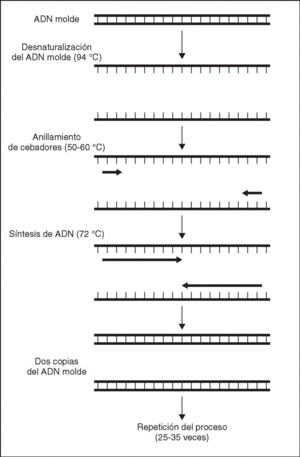
La PCR es un método engañosamente simple, pero muy versátil, que tiene aplicación en todas las áreas donde se haga uso de la biología molecular. Expresado de una manera simple, todo lo que la PCR consigue es fabricar múltiples copias de una secuencia diana de ADN mediante un proceso de amplificación que llega a permitir la obtención de microgramos de ADN a partir de cada molécula inicial. Cualquier segmento de ADN o de ARN puede ser amplificado siempre que se conozcan las secuencias flanqueantes de la región diana o, lo que es lo mismo, podemos amplificar cualquier porción de ácido nucleico, conocido o no, siempre que quede comprendido entre dos secuencias conocidas con las que podrán hibridar oligonucleótidos complementarios que actuarán como cebadores para que la polimerasa pueda copiar las hebras molde (fig. 1). Por tanto, para que la reacción tenga lugar, los componentes básicos e indispensables de la misma son: ADN molde que se desea amplificar, ADN polimerasa, cebadores de secuencia específica, nucleótidos para la síntesis de nuevo ADN y tampón de reacción que incluye las distintas sales requeridas por la enzima.
Figura 1. Representación esquemática de los distintos pasos de una reacción de PCR.
La PCR es una reacción que ocurre en un único tubo tras mezclar en él los componentes necesarios e incubarlos en un termociclador, aparato que permite variar la temperatura de incubación a lo largo del tiempo de forma programada. Los protocolos básicos de PCR constan de los siguientes pasos fundamentales:
1. Desnaturalización térmica del ADN que se va a usar como molde.
2. A una temperatura menor que la de desnaturalización, anillamiento de cebadores sintéticos cuya secuencia les permite hibridar uno a cada lado de la secuencia diana.
3. Extensión por parte de la ADN polimerasa de los oligonucleótidos anillados que actúan como cebadores.
4. Este proceso de tres pasos es entonces repetido un número determinado de veces (25-35), duplicándose cada vez el número de moléculas de producto. Esta amplificación exponencial tiene como resultado un gran número de copias de la secuencia de ADN flanqueada por el par de oligonucleótidos. De esta manera, la reacción permite obtener una gran sensibilidad al detectar muy bajas cantidades de ADN molde y especificidad al detectar únicamente la secuencia de ácido nucleico para la que han sido diseñados los cebadores.
Sin embargo, en este proceso teórico que hemos descrito pueden influir numerosos factores que hagan que la reacción no sea realmente específica o que su eficiencia no sea la adecuada, de manera que la amplificación no ocurra en la progresión esperada y, por tanto, no sea sensible. Entre los muchos factores que pueden influir en el resultado final de la PCR, son destacables el diseño apropiado de los cebadores, la calidad y concentración del ADN, la concentración de magnesio, el tipo y la cantidad de ADN polimerasa y el programa de amplificación.
En el caso de una PCR múltiple, lo que se persigue es amplificar simultáneamente en un único tubo distintas secuencias específicas, lo cual necesariamente implica que los reactivos mezclados y el programa utilizado sean suficientes y adecuados para permitir la detección de cada diana y no inhibir la de las demás. Con este fin, algunos parámetros, como la concentración de magnesio y de cebadores, y el tipo y la cantidad de ADN polimerasa, pueden ajustarse experimentalmente. Para otros, en cambio, debe realizarse un diseño exhaustivo previo. En el caso de los cebadores y el programa de temperaturas utilizado, es necesario tener en cuenta varias premisas: a) escoger o diseñar oligonucleótidos que no interaccionen entre sí, es decir, que no formen oligómeros; b) que tengan temperaturas de anillamiento similares; c) que cada pareja amplifique una única secuencia diana, y d) que generen amplicones de tamaño suficientemente diferente como para poder ser separados y diferenciados tras la amplificación. En cuanto a la calidad y cantidad de ADN molde, por supuesto debemos intentar partir de la concentración menor posible y con ausencia de sustancias inhibidoras que puedan interferir en la reacción. Para ello, los protocolos de purificación variarán en función del tipo de muestra clínica de partida.
Durante los últimos años, se han publicado numerosos trabajos en los que se describen nuevos protocolos para la extracción de ADN en cantidad y calidad adecuadas para la PCR o para la realización de reacciones de amplificación sensibles y específicas directamente a partir de muestras clínicas. Por supuesto, por su rapidez y sencillez, esta última opción es la más deseable, aunque también la más difícil de lograr. Afortunadamente, disponemos ya de métodos que permiten la detección directa de microorganismos a partir de: colonia de cultivo, cultivos líquidos, sangre, suero, orina, líquido pleural, heces, esputo, líquido cefalorraquídeo, lavado broncoalveolar, aspirados nasofaríngeos y traqueales, aspirado transtorácico, aspirado de nódulo linfático, aspirado de médula ósea, muestras serosas de oído, tejido pulmonar fijado, biopsias de piel, cerebral, hepática, ósea, pulmonar, de bazo, de tejido vertebral, etc.2-5
Por otro lado, también ha de tenerse en cuenta que, en los casos en que se detectan genes que codifican toxinas u otros factores de virulencia, la presencia del gen no implica que la proteína esté siendo producida, por lo que la PCR múltiple debería aplicarse, siempre que sea posible, en conjunción con métodos bioquímicos y/o inmunológicos. No obstante, tampoco se debe dejar de considerar la gran utilidad que puede tener una detección precoz de información genética que puede ser peligrosa si llega a ser expresada, ya que permite la ejecución de medidas preventivas, tanto de tratamiento como de control. Por último, es conveniente recordar que, tal y como se ha mencionado antes, la PCR es una técnica que, por su aparente simplicidad y su alta sensibilidad, es muy susceptible de errores de ejecución y de contaminaciones, por lo que la introducción de la PCR múltiple en los laboratorios de microbiología clínica requiere una formación especializada previa del personal que ejecute los análisis.
Escherichia coli
E. coli constituye el microorganismo más abundante de la microbiota gastrointestinal humana, desempeñando un importante papel en el funcionamiento normal del sistema digestivo. Sin embargo, distintas cepas de E. coli son capaces de causar un amplio rango de enfermedades, y presentan además una alta capacidad de dispersión vía cadena alimentaria o por transmisión hídrica. Las infecciones por E. coli pueden afectar al tracto gastrointestinal (este microorganismo es una de las causas principales de brotes de diarrea, colitis hemorrágica y síndrome hemolítico-urémico) o pueden localizarse a nivel extraintestinal, causando, entre otros cuadros, meningitis, septicemia o infecciones del tracto urinario. Las cepas de E. coli que causan patología intestinal pueden ser agrupadas en distintos fenotipos: enterotoxigénicas (ETEC), enteropatogénicas (EPEC), enteroinvasivas (EIEC), enterohemorrágicas (EHEC) y enteroagregativas (EaggEC). De especial relevancia clínica son las cepas portadoras de la información genética necesaria para la producción de toxinas Shiga (cepas STEC), de hemolisina entérica, del factor de adhesión "intimina", del factor 1 necrótico citotóxico (CNF1), o de diferentes factores de virulencia como sistemas de captación de hierro (p. ej., aerobactina o enterobactina) o diversos antígenos flagelares, fimbrilares o de superficie. La detección rápida y precisa de estas variantes génicas de E. coli es fundamental para evitar el riesgo asociado a tratamientos tardíos y para prevenir la posible existencia de brotes o epidemias comunitarios o nosocomiales.
Durante la última década, se ha prestado gran atención al desarrollo de métodos inmunológicos y moleculares rápidos, sensibles y específicos para la detección e identificación de cepas patógenas de E. coli. Con este objetivo, en el año 2002 se publicaron tres nuevos protocolos de PCR múltiple que permiten la detección simultánea de numerosos genes de relevancia clínica como son aquellos que codifican toxinas Shiga, la hemolisina entérica, la intimina, el serotipo de superficie O157 y el serotipo flagelar H7 (tabla 1)6-8. Estos protocolos no sólo posibilitan la detección de los genes de virulencia y toxicidad más importantes asociados a E. coli, sino que además permiten diferenciar las variedades más patógenas (p. ej., los serotipos O157:H7). Asimismo, en el año 2003 otros dos nuevos protocolos de PCR múltiple aportan importantes avances en la detección de cepas EPEC, STEC, EIEC, ETEC y EaggEC. En el primero de estos protocolos, Cerna et al9 utilizaron la detección simultánea de tres genes codificados en plásmidos que confieren la denominada Adherencia de Agregación (AA) a las variantes enteroagregativas. En la segunda de estas PCR múltiples, Toma et al10 optimizaron un ensayo que permite la identificación y clasificación simultánea de los distintos tipos de E. coli causales de diarrea mediante la detección de genes específicos de cepas enteropatogénicas, de cepas productoras de toxinas Shiga, de variantes enterotoxigénicas, de cepas enteroinvasivas y de variedades enteroagregativas (tabla 1). Estos protocolos para diferenciar tipos patógenos pueden, además, ser complementados con otros protocolos de PCR múltiple que permiten subtipificar las variantes toxigénicas (p. ej., subtipos de Shiga toxin 2), o variantes virulentas (p. ej., distintos operones para adhesinas)11-13.
Otras enterobacterias
Aunque las infecciones causadas por E. coli son las que mayor atención han recibido a la hora de establecerse nuevos métodos de diagnóstico molecular, debido, probablemente, a la gran cantidad de población mundial que se ve afectada por ellas, muchas otras Enterobacteriaceae spp. tienen un gran impacto sobre la salud humana y requieren un diagnóstico rápido, sensible y eficaz. De entre la gran variedad de protocolos de PCR múltiple que han sido desarrollados para el diagnóstico de estas especies, sólo mencionaremos a continuación algunos ejemplos que hemos seleccionado por su posible aplicación clínica.
Otras enterobacterias que afectan directamente al hombre son las pertenecientes al género Salmonella, especialmente S. enterica. Ésta constituye uno de los grupos más importantes de patógenos con origen en la cadena alimentaria14. Estas enfermedades llevan a un gran número de hospitalizaciones anuales y pueden llegar a ser fatales. Por tanto, el control de Salmonella es necesario a lo largo de toda la cadena de producción de alimentos y en todos los casos de enfermedad relacionados con el género. Los métodos tradicionales de detección de Salmonella están basados en cultivos en medios selectivos y posterior análisis mediante pruebas bioquímicas y serológicas. Con el objetivo de dar mayor rapidez al proceso de detección, se han optimizado distintos protocolos de PCR. En el año 2002, se publicó un nuevo protocolo de PCR múltiple que permite simultáneamente la detección de Salmonella spp. y la identificación específica de variedades responsables de casos esporádicos o brotes de enteritis14.
Por otro lado, aunque ya erradicada en gran parte de Occidente, Yersinia pestis sigue siendo un importante problema de salud en muchos países15. Esto, unido a la alta posibilidad de diseminación actual a través de viajeros a áreas contaminadas, lo convierte en un asunto de interés para la Salud Pública Mundial. Por ello, una vez más, es necesario disponer de métodos rápidos y precisos que posibiliten una intervención rápida durante posibles brotes y/o diseminación. Con tal fin, Leal y Almeida desarrollaron en 1999 un protocolo de PCR múltiple que permite la identificación simultánea de distintos marcadores de cepas virulentas de Y. pestis15. En concreto, el método consiste en la detección de los genes de virulencia más característicos de la especie presentes en tres plásmidos de virulencia y en una isla de patogenicidad cromosómica.
Por otro lado, las bacterias pertenecientes al género Klebsiella son la causa de un importante porcentaje de infecciones hospitalarias, particularmente de los tractos respiratorio y urinario. Recientemente, el género ha sido reanalizado con respecto a su estructura filogenética y todos los estudios realizados mostraron su heterogeneidad taxonómica16. Así, se ha sugerido que el género Klebsiella se divida en dos géneros, Klebsiella y Raoultella, y que K. oxytoca quede considerado como un taxón monofilético. Los datos de secuencias disponibles permiten distinguir entre estos grupos de bacterias, pero es muy difícil diferenciar bioquímicamente los aislados del género Klebsiella, particularmente K. oxytoca, de los del género Raoultella. Por estos motivos, Granier et al16 publicaron en el año 2003 un nuevo protocolo de PCR que permite diferenciar Raoultella sp. de K. oxytoca. Asimismo, van der Zee et al17 desarrollaron otra PCR múltiple de gran utilidad para el seguimiento de los brotes de K. pneumoniae y K. oxytoca.
Un problema adicional a la hora de luchar contra infecciones causadas por bacterias gramnegativas, entre ellas las enterobacterias, es la creciente diseminación de cepas multirresistentes a antibióticos. En concreto, son muy limitadas las opciones terapéuticas en el caso de infecciones causadas por bacterias que expresan betalactamasas tipo AmpC codificadas en plásmidos, ya que estos microorganismos, normalmente, son resistentes a la mayoría de los betalactámicos, y aunque habitualmente son sensibles a cefalosporinas de cuarta generación (cefepima, cefpiroma, etc.) y a carbapenems18, ya se han descrito cepas que también son resistentes a ambos grupos. El seguimiento y control de estas cepas se ve dificultado por el hecho de que se han descrito varias familias de betalactamasas AmpC codificadas en plásmidos, las cuales no pueden diferenciarse fenotípicamente y porque su fenotipo de resistencia puede ser similar al de cepas productoras de betalactamasas de espectro extendido con alteraciones en la permeabilidad. Con el fin de ayudar a subsanar esta limitación, Pérez-Pérez y Hanson18 desarrollaron una PCR múltiple que permite detectar e identificar las distintas familias de genes ampC.
Pseudomonas spp.
Pseudomonas aeruginosa ha emergido como una de las bacterias gramnegativas actualmente más problemáticas a nivel hospitalario, presentando altos grados de morbilidad y mortalidad. Los pacientes quemados, aquellos que reciben ventilación mecánica y los enfermos de fibrosis quística son particularmente susceptibles a la infección por esta bacteria. El incremento progresivo de cepas multirresistentes a antibióticos y la gravedad que pueden presentar las infecciones causadas por estas bacterias, han hecho muy importante y necesario el desarrollo de métodos de diagnóstico rápidos, sensibles y específicos. Como ejemplo se puede citar la PCR múltiple desarrollada por de Vos et al19 que, gracias a la detección de genes que codifican lipoproteínas de la membrana externa, permite simultáneamente identificar P. aeruginosa y detectar el grupo de pseudomonas fluorescentes como P. aeruginosa, P. fluorescens, P. putida, P. stutzeri, etc.
Enterococos
El género Enterococcus es particularmente relevante desde el punto de vista clínico debido al incremento en su incidencia como agentes responsables de infecciones, principalmente en el ámbito hospitalario, como endocarditis, bacteriemia e infecciones del tracto urinario. Además, la aparición de cepas resistentes a la mayoría de antibióticos disponibles hace que las terapias actualmente empleadas puedan resultar ineficaces. Especialmente relevante es el incremento de aislados resistentes a glucopéptidos, de los que se han descrito siete genotipos de resistencia. Dentro de este género hay un gran número de especies, de las cuales Enterococcus faecalis y E. faecium representan aproximadamente el 90% de los aislamientos. La incidencia de otras especies, como E. gallinarum y E. casseliflavus podría estar infraestimada debido a errores en la identificación. Por lo tanto, la identificación correcta a nivel de especie dentro de este género bacteriano, así como la detección de los genes que confieren resistencias, es de vital importancia en la práctica clínica para llevar a cabo el tratamiento más apropiado en cada caso.
Se han desarrollado protocolos de PCR múltiple rápidos y fiables que permiten la detección de los enterococos en pocas horas. En 1999, Ke et al20 diseñaron un protocolo para la detección específica de todos los enterococos de importancia clínica a nivel de género. Además, la PCR múltiple se ha empleado para discriminar entre diferentes tipos de genes de resistencia a glucopéptidos en estas bacterias. Uno de estos métodos permite la detección simultánea de los genes vanA, vanB y vanC1, además de la identificación a nivel de especie de E. faecium, E. faecalis, E. gallinarum y E. casseliflavus21. En el año 2002, Pérez-Hernández et al22 desarrollaron un protocolo de PCR múltiple a partir de colonia que permite la identificación de los enterococos a nivel de género y la detección de los genes de resistencia a vancomicina más frecuentes (vanA, vanB, vanC1 y vanC2/C3).
PCR múltiple en el diagnóstico de estafilococos
Aunque dentro del género Staphylococcus la especie S. aureus es la causante de un mayor número de patologías, diversas especies de estafilococos coagulasa negativos como S. epidermidis, S. haemolyticus, S. hominis, etc., se han asociado a un número creciente de infecciones hospitalarias. S. saprophyticus es una causa muy frecuente de las infecciones del tracto urinario en mujeres jóvenes sexualmente activas.
Cada vez es más frecuente la aparición de cepas de S. aureus resistentes a los antibióticos, lo cual ha complicado enormemente el tratamiento de las infecciones producidas por este agente. Tal y como hemos descrito para otros grupos, el desarrollo de métodos de PCR múltiple específicos, sensibles y rápidos para la identificación de S. aureus y de sus genes de resistencia ha tenido gran relevancia.
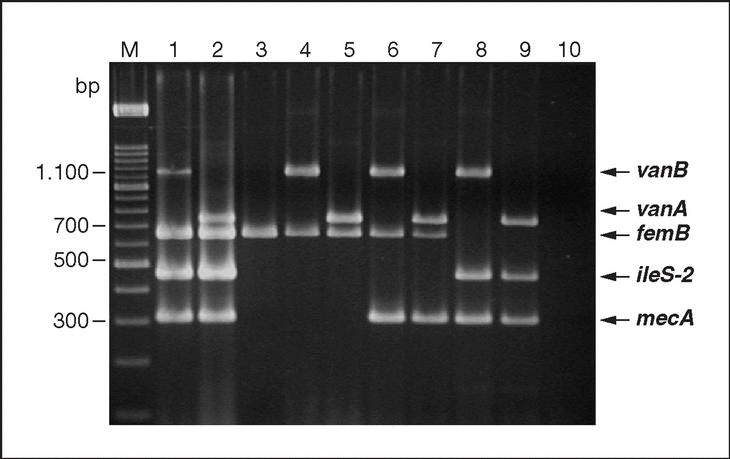
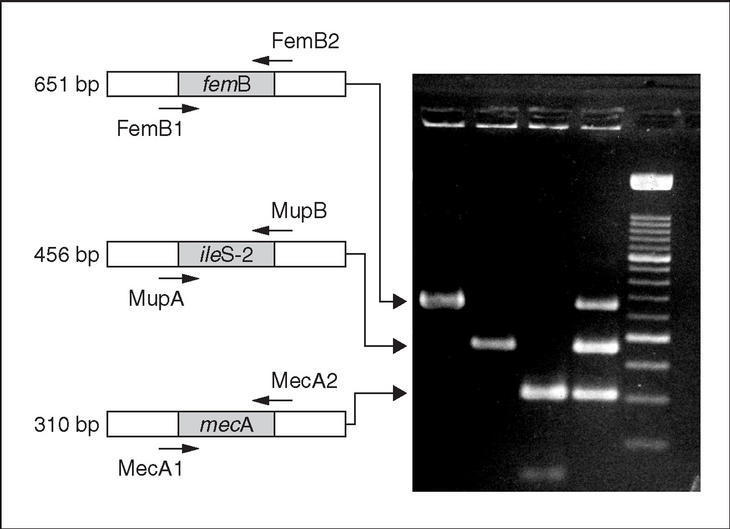
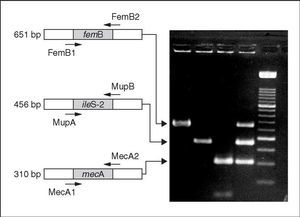
Recientemente, se ha publicado un ensayo que permite la detección simultánea de genes de resistencia clínicamente relevantes en S. aureus23. Especial atención merecen aquellos protocolos que permiten la discriminación entre S. aureus y estafilococos coagulasa negativos, simultáneamente a la detección del gen mecA, y, en ocasiones, de otros genes de resistencia. Estos protocolos normalmente parten del ADN extraído a partir de colonias bacterianas o de cultivos líquidos puros24 (fig. 2). Sin embargo, actualmente el objetivo es realizar la identificación directamente a partir de las muestras clínicas, con lo que el diagnóstico sería mucho más rápido25.
Figura 2. Gel de electroforesis en el que se observan los fragmentos generados mediante PCR múltiple para la identificación de S. aureus mediante la detección del gen femB y la detección simultánea de los genes de resistencia a meticilina (gen mecA) y alta resistencia a mupirocina (gen ileS-2). Carriles: 1-3, Productos de amplificación a partir de los genes femB, ileS-2 y mecA, respectivamente. La electroforesis fue corrida en gel al 2% de agarosa y, posteriormente, teñido con bromuro de etidio. Esta figura fue previamente publicada por Pérez-Roth et al24.
La capacidad de muchas cepas de S. aureus de producir exotoxinas incrementa su poder patogénico. Dentro de este grupo se incluyen las enterotoxinas (SE), causantes de intoxicaciones alimentarias, las toxinas exfoliativas (ET), responsables del síndrome de piel escaldada, y la toxina 1 del síndrome del shock tóxico (TSST-1), implicada en el síndrome del shock tóxico estafilocócico. La detección de estas toxinas aún se basa principalmente en métodos inmunológicos tediosos, y que dependen de la cantidad de toxina que se produce. Aunque estos métodos son necesarios para verificar la producción de las mismas, en los últimos años se ha considerado esencial, desde el punto de vista epidemiológico y de diagnóstico, la detección por PCR de los genes que codifican estas toxinas. De este modo, se han desarrollado varios protocolos de PCR múltiple que permiten la detección rápida, específica y especialmente, simultánea, de muchos de estos genes. Un ejemplo reciente es el método publicado en el año 2002 mediante el que se detecta la presencia de 8 genes que codifican para enterotoxinas en S. aureus aislados de humanos implicados en brotes de intoxicación alimentaria, humanos sanos, vacas con mastitis, y leche cruda de vaca26. Otro protocolo de diagnóstico que merece una atención especial fue publicado en el año 2000 por Mehrotra et al27; en el que, además de detectar ocho de los genes que codifican para las principales toxinas estafilocócicas, detectan el gen mecA y un fragmento del gen femA que permite identificar de manera específica a S. aureus.
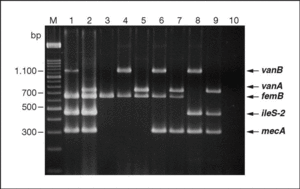
Por otro lado, la reciente detección en Estados Unidos de dos aislados clínicos de S. aureus portadores de genes de origen enterocócico y que confieren alta resistencia a vancomicina, ha llevado a considerar de gran interés el desarrollo de una PCR múltiple que permite simultáneamente la identificación S. aureus y la detección de los genes enterocócicos vanA y vanB y de los genes estafilocócicos de resistencia a meticilina y a mupirocina28 (fig. 3).
Figura 3. Gel de electroforesis en el que se observan los fragmentos generados mediante PCR múltiple para la detección simultánea de los genes enterocócicos vanA y vanB y estafilocócicos femB, ileS-2 y mecA a partir de distintas mezclas de Enterococcus sp. y Staphylococcus sp. Carriles: M, Marcador de Peso Molecular XIV (Roche Diagnostics); 1, Enterococcus faecalis V 583 (vanB)/ Staphylococcus aureus 242 (femB, ileS-2 y mecA); 2, Enterococcus faecium BM 4147 (vanA)/S. aureus 242 (femB, ileS-2 y mecA); 3, E. faecalis ATCC 29212/ S. aureus ATCC 29213 (femB); 4, E. faecalis V 583 (vanB)/S. aureus ATCC 29213 (femB); 5, E. faecium BM 4147 (vanA)/ S. aureus ATCC 29213 (femB); 6, E. faecalis V 583 (vanB)/S. aureus SEIMC (femB, mecA); 7, E. faecium BM 4147 (vanA)/ S. aureus SEIMC (femB, mecA); 8, E. faecalis V 583 (vanB)/S. epidermidis (ileS-2, mecA); 9, E. faecium BM 4147 (vanA)/ S. epidermidis (ileS-2, mecA); 10, control sin ADN molde. La electroforesis fue corrida en gel al 2% de agarosa y, posteriormente, teñido con bromuro de etidio. Esta figura fue previamente publicada por Ramos-Trujillo et al28.
Campylobacter spp.
Las infecciones causadas por distintas especies del género Campylobacter son la causa de numerosas enfermedades con origen en la cadena alimentaria. Las especies patógenas pertenecientes al género presentan requerimientos de crecimiento "fastidiosos", haciendo problemáticas la detección y la identificación de estas bacterias. Como consecuencia, el desarrollo de métodos rápidos y fiables de detección ha tomado gran importancia. Se están usando ya distintos métodos de detección genética de especies de Campylobacter a partir de muestras clínicas, ambientales y de alimentos. A finales del año 2002, se publicó un nuevo protocolo de PCR múltiple para la detección simultánea de las cinco especies clínicamente más relevantes del género: C. jejuni, C. coli, C. lari, C. upsaliensis y C. fetus subsp. fetus29. El método es rápido y tiene alta sensibilidad y especificidad para la detección de las distintas especies, incluso a partir de cultivos mixtos, por lo que puede ser una alternativa efectiva a los métodos bioquímicos tradicionales de identificación de tales bacterias.
Mycobacterium
El diagnóstico clínico de enfermedades causadas por especies pertenecientes al género Mycobacterium pasa en primer lugar por la diferenciación del "complejo" de M. tuberculosis (MTC) de micobacterias atípicas. Esa primera distinción sirve para confirmar el diagnóstico inicial y el tratamiento que va a ser administrado. Pero, al mismo tiempo, conlleva el problema de que los distintos miembros del MTC no pueden diferenciarse con facilidad, lo cual dificulta la realización de estudios epidemiológicos detallados que sirvan para prevenir brotes e identificar orígenes de infección. La identificación tradicional recae en una batería de pruebas bioquímicas, las cuales son lentas. Asimismo, existen pruebas moleculares comerciales para el análisis de cultivos, pero, normalmente, son específicas para una especie determinada y no son capaces de diferenciar entre miembros del MTC. Por tanto, se ha percibido la necesidad de desarrollar nuevos métodos sensibles y eficaces. Como complemento para el diagnóstico de Mycobacterium spp. se han planteado varios protocolos diferentes de PCR múltiple. Con el primero de ellos, si bien no quedan perfectamente diferenciados los distintos miembros del MTC, Kox et al4 consiguieron optimizar la identificación de múltiples especies del género: M. tuberculosis, M. avium, M. kansasii, M. chelonae, M. genavense, M. bovis BCG, M. xenopi y M. marinum. Por el contrario, en el segundo, Yeboah-Manu et al30 optimizaron un método que permite identificar un número menor de especies, pero que distingue el MTC de las micobacterias no tuberculosas y diferencia los distintos miembros del MTC: M. tuberculosis, M. bovis, M. bovis BCG y M. africanum subtipos I y II. Este protocolo queda a su vez complementado con otro, recientemente desarrollado por Motiwala et al31, que permite la detección de dianas específicas de M. avium.
Infecciones genitourinarias
La uretritis se puede subclasificar en uretritis gonocócica y uretritis no gonocócica dependiendo en la presencia o ausencia de Neisseria. El control de estas infecciones es especialmente relevante en el caso de mujeres, en las que pueden llegar a causar enfermedad pélvica inflamatoria, embarazos ectópicos e, incluso, infertilidad. En la uretritis no gonocócica, la infección es atribuida a Chlamydia trachomatis, o a otros patógenos, como micoplasmas o ureaplasmas. Como para el resto de enfermedades, la necesidad de una correcta asignación del agente causal ha llevado al desarrollo de métodos sensibles y específicos entre los que se encuentran varias PCR múltiples. En el año 2002, Gaydos et al32 publicaron un protocolo que permite una efectiva detección e identificación de N. gonorrhoeae y de C. trachomatis a partir de exudados vaginales. Un año más tarde, el desarrollo de una nueva PCR múltiple permite la detección e identificación de cuatro importantes agentes implicados en la uretritis no gonocócica32 como Mycoplasma genitalium, M. hominis, Ureaplasma parvum y U. urealyticum33.
Asimismo, la PCR múltiple también ha resultado una herramienta muy útil en la identificación del agente etiológico en casos de úlceras genitales y de sífilis. En concreto, es destacable el protocolo desarrollado por Orle et al34 que permite la detección específica y sensible de Haemophilus ducreyi, Treponema pallidum y herpes simple tipos 1 y 2. Posteriormente, en el año 2001, este protocolo fue complementado por otro que permite la detección inequívoca de subespecies patogénicas de T. pallidum aprovechando características génicas únicas en su gen codificante de la ADN polimerasa I, lo cual permite diferenciarlas de las no patogénicas o de cualquier otra especie35.
PCR múltiple en el diagnóstico de la meningitis bacteriana
Los tres agentes causales de la mayor parte de casos de meningitis bacteriana son Neisseria meningitidis, Haemophilus influenzae y Streptococcus pneumoniae. A ellos hay que sumar, además, otras bacterias tan diversas como Listeria monocytogenes, Streptococcus agalactiae, Staphylococcus spp., enterobacterias, P. aeruginosa, Mycobacterium tuberculosis, Borrelia burgdorferi, Leptospira interrogans, algunas de las cuales causar cuadros clínicamente no distinguibles de las meningitis asépticas y encefalitis producidas por virus. Los métodos de diagnóstico tradicionales, dependientes en gran medida de cultivo, para la identificación del agente causal de la meningitis suelen requerir un período de tiempo igual o superior a 36 h. Además, se ha observado la existencia de un cierto grado de discrepancia entre el número de casos clínicamente sospechosos de meningitis bacteriana y de septicemia y el número de casos confirmados por cultivo. Con el objetivo de solucionar estos problemas, se ha trabajado mucho en la puesta a punto de métodos moleculares no dependientes de cultivo y, durante los últimos años, se ha obtenido resultados muy satisfactorios con el uso de distintos protocolos de PCR múltiple. Dado que constituyen un amplio número de métodos, y sería largo y poco clarificador mencionarlos todos, a continuación se introducen simplemente aquellos protocolos que, en su conjunto, permiten descartar un mayor número de agentes causales.
En el año 2001, Corless et al36 desarrollaron un protocolo de detección múltiple que permite la identificación simultánea de N. meningitidis, H. influenzae y S. pneumoniae a partir de distintos tipos de muestras clínicas, concretamente, líquido cefalorraquídeo (LCR), plasma, suero y sangre. La aplicación de este protocolo a un amplio conjunto de muestras, previamente catalogadas como negativas en virtud de la ausencia de crecimiento el cultivo, permitió detectar alguna de esas tres bacterias. Si bien éste no es un protocolo de PCR convencional sino de PCR en tiempo real, su gran aplicabilidad clínica para el diagnóstico preciso de las afecciones que se discuten en este apartado nos hace creer necesario el mencionarlo. Un año antes, en 2000, Chesky et al37 desarrollaron un método que es destacable por la cantidad y diversidad de agentes infecciosos que permite detectar simultáneamente. En concreto, a partir de muestras de LCR permitió identificar L. monocytogenes, Mycobacterium spp., citomegalovirus, enterovirus, virus de Epstein-Barr, virus del herpes simple, herpesvirus humano tipo 6, virus JC, virus de la varicela-zóster y Toxoplasma gondii.
Neumonía
Entrados ya en el siglo xxi, las afecciones respiratorias continúan siendo un problema de salud mundial tanto en niños como en adultos. El problema es tan relevante que las enfermedades respiratorias han sustituido a las afecciones diarreicas como la causa mayor de muerte en niños menores de 5 años en todo el mundo. Uno de los patógenos más importantes implicados en esta situación es S. pneumoniae, pero no es el único. Distintos agentes bacterianos pueden desencadenar neumonía, siendo los más relevantes entre ellos Legionella pneumophila, Chlamydia pneumoniae, Mycoplasma pneumoniae, K. pneumoniae38. A todos ellos se unen, además, diversos grupos de virus que pueden causar complicaciones respiratorias similares. Una vez más, la multiplicidad de posibles agentes causales y la gran similitud de sintomatologías clínicas, han hecho necesaria la búsqueda de nuevos métodos de diagnóstico que permitan la identificación sensible, eficaz y lo más rápida posible del agente infeccioso. Con tal fin, se han optimizado distintos protocolos de PCR múltiple.
En el año 2003, se han desarrollado dos nuevos protocolos para la determinación de serogrupos y serotipos de S. pneumoniae que pueden tener gran utilidad en los laboratorios de diagnóstico. A nivel mundial, S. pneumoniae es uno de los principales agentes causales de mortalidad y morbilidad asociadas a neumonía en niños y ancianos. La capacidad de los neumococos de producir enfermedad está directamente relacionada con la producción de cápsula, recubrimiento polisacarídico que confiere resistencia a la fagocitosis y facilita la evasión del sistema inmunitario. Se han identificado al menos 90 tipos capsulares inmunológicamente diferentes, pero la mayoría de los cuadros infecciosos están producidos por un reducido número de serotipos. Los métodos recientemente descritos por Brito et al39 y Lawrence et al40 permiten determinar mediante PCR múltiple los serotipos y serogrupos más relevantes, de manera que, si bien no sustituyen completamente los métodos serológicos, sí reducen drásticamente el número de cepas que tienen que ser analizadas inmunológicamente para poder clasificarlas con fines clinicoepidemiológicos.
Las infecciones causadas por distintas especies del género Streptococcus (p. ej., S. pneumoniae, S. pyogenes y Streptococcus del grupo viridans) pueden ser muy complicadas de erradicar cuando son producidas por cepas multirresistentes a antibióticos. Por tal motivo, en los últimos años también se han optimizado distintos protocolos de PCR múltiple para la detección de distintos genes de resistencia a macrólidos, codificando bien bombas de expulsión activa (genes mef), bien metilasas que modifican la diana del antibiótico (genes erm)41,42.
Distintos estudios han mostrado la aplicabilidad de la PCR múltiple en la identificación precisa del agente causal de la neumonía cuando se sospecha que se trate de C. pneumoniae, C. psittaci o M. pneumoniae43. Además, otra aportación derivada de estos métodos ha sido la optimización de la detección de secuencias de interés a partir de muestras clínicas como esputos, exudados faríngeos, aspirados nasofaríngeos y traqueales, lavados broncoalveolares, aspirados transtorácicos, tejidos pulmonares fijados y biopsias pulmonares.
Patógenos del oído medio
En pacientes con otitis media la identificación de los agentes causales a partir de muestras clínicas empleando métodos tradicionales es difícil, laborioso y lento. Por ese motivo, en la última década fueron optimizados distintos protocolos de PCR para la detección individual de los agentes más habituales. La utilidad clínica de estos métodos quedó reducida por la laboriosidad de aplicar una detección para cada posible bacteria. Sin embargo, Hendolin et al44 desarrollaron una PCR múltiple que podría tener una mayor utilidad en el diagnóstico rutinario, ya que permite detectar e identificar simultáneamente a partir de muestras serosas los patógenos más frecuentemente aislados a partir de muestras del oído medio: S. pneumoniae, H. influenzae, Moraxella catarrhalis y Alloiococcus otitidis.
Conclusiones y perspectivas
Durante las últimas décadas del siglo xx, cuando se planteaban los posibles problemas de salud pública para el siglo venidero, enfermedades como el cáncer, el sida o los accidentes cardiovasculares aparecían como los grandes problemas contra los que luchar. Por su parte, los éxitos terapéuticos obtenidos contra las enfermedades infecciosas después de mitad de siglo gracias al uso de antibióticos, hicieron pensar que las infecciones bacterianas serían un problema, a lo sumo, menor. Sin embargo, la creciente aparición y diseminación de estirpes bacterianas virulentas y de mecanismos de resistencia frente a todos los antibióticos disponibles han hecho incierta aquella predicción. Por el contrario, adentrados ya en el siglo xxi, tal y como ya indicaba la Organización Mundial de la Salud en 1999 (Informe WHO/CDS/99.1), las enfermedades infecciosas son una de las cargas más pesadas para la población, y muchas de estas infecciones son originadas o complicadas por agentes bacterianos.
Por lo tanto, al igual que antes de 1950, la humanidad se encuentra en una situación en la que es prioritaria la optimización de la lucha contra las enfermedades infecciosas. Como parte de este gran objetivo global, la microbiología clínica necesita optimizar a nivel de especificidad, sensibilidad y rapidez, la detección de agentes infecciosos y de sus genes de virulencia y de resistencia, lo cual permitirá ejecutar programas adecuados de prevención y tratamiento de estas enfermedades. Para lograr este propósito, la constante optimización de la PCR múltiple se plantea como una gran alternativa que permitirá la detección simultánea de un número cada vez mayor de dianas, a partir de un número también creciente de tipos de muestras clínicas y necesitando una cantidad menor de ácido nucleico molde. Además, la reciente introducción de la PCR en tiempo real en el diagnóstico clínico, permitirá el desarrollo de nuevos y eficientes protocolos de PCR múltiple.
ANEXO 1. La PCR múltiple en la microbiología clínica
1. Los protocolos básicos de PCR constan de:
a)Dos pasos fundamentales: desnaturalización y finalización.
b)Dos pasos básicos: amplificación y visualización.
c)Tres pasos fundamentales: desnaturalización, anillamiento de cebadores y extensión por parte de la polimerasa.
d)Tres pasos fundamentales: extracción, anillamiento de cebadores y extensión por parte de la polimerasa.
e)La PCR es una reacción que ocurre en un único tubo y un solo paso.
2. Entre los muchos factores que pueden influir en el resultado final de la PCR:
a)El diseño o elección de los cebadores no es especialmente crítico.
b) La cantidad de ADN molde utilizada no tiene relevancia.
c)Es muy importante la concentración de cebadores utilizada, pero no lo es la de sales.
d)Son importantes, entre otros, el diseño apropiado de los cebadores, la concentración de magnesio, la calidad del ADN molde y el programa de amplificación.
e)Normalmente, es independiente del programa de amplificación.
3. En el caso de los cebadores y el programa de temperaturas a utilizar:
a)Hay que escoger cebadores que tengan temperaturas de anillamiento bien diferenciadas.
b) Escoger parejas de cebadores que muestren una buena interacción entre ellos.
c)Para asegurar la amplificación, que cada pareja amplifique más de una secuencia diana.
d)Diseñar oligonucleótidos que no interaccionen entre sí y que tengan temperaturas de anillamiento similares.
e)Es indiferente el tamaño de los amplicones que se generen.
4. La PCR múltiple:
a)Permite la amplificación de genes de resistencia, pero no simultáneamente de genes que codifican factores de virulencia.
b) La optimización no requiere más esfuerzo que una PCR sencilla, ya que no hay más factores importantes que se deben considerar.
c)Es aplicable en la detección/amplificación de cualquier gen, con independencia de lo que codifique.
d)Supone un gran avance en el diagnóstico de enfermedades infecciosas, pero su aplicabilidad clínica es imposible.
e)Es sustitutiva de los métodos de diagnóstico convencionales.
5. En el caso de enterobacterias:
a) Es posible la detección mediante PCR múltiple de Salmonella spp., pero no la identificación de variedades responsables de enteritis.
b) Dado que Yersinia pestis ya ha sido erradicada en gran parte de Occidente, no ha sido considerado de interés el desarrollo de PCR múltiple para su identificación.
c) Raoultella sp. no puede ser distinguido de Klebsiella oxytoca mediante ningún protocolo de PCR múltiple.
d)Mediante de PCR múltiple pueden distinguirse los distintos tipos de genes ampC codificados por plásmidos.
e) No se ha descrito, hasta el momento, ningún protocolo de PCR múltiple que permita diferenciar las distintas familias de genes ampC.
6.En el caso de infecciones causadas por Streptococcus pneumoniae:
a)La identificación simultánea de Neisseria meningitidis, Haemophilus influenzae y Streptococcus pneumoniae mediante PCR múltiple aún no es posible.
b) Los nuevos protocolos de PCR múltiple permiten identificar la bacteria, pero no detectar genes de resistencia a macrólidos en muestras clínicas.
c)Los nuevos protocolos de PCR múltiple, permiten identificar la bacteria a nivel de especie, pero no determinar los serotipos y serogrupos más relevantes.
d)Los nuevos protocolos de PCR múltiple, sustituyen completamente los métodos serológicos.
e)Los nuevos protocolos de PCR múltiple permiten detectar en muestras clínicas cepas portadoras de distintos genes de resistencia a macrólidos.
7.Los distintos miembros del "complejo" de Mycobacterium tuberculosis (MTC):
a)Son claramente distinguibles en base a pruebas fenotípicas.
b) Su distinción fenotípica no es posible, pero su identificación bioquímica es rápida e inequívoca.
c)Existen pruebas moleculares comerciales que permiten tanto la identificación específica a nivel de especie como la diferenciación entre miembros del grupo MTC.
d)Mediante un protocolo único de PCR múltiple se puede identificar a nivel de especie y diferencia entre miembros del grupo MTC.
e)La aplicación combinada de varios protocolos de PCR múltiple puede identificar a nivel de especie y diferenciar entre miembros del grupo MTC.
8.En el caso de Escherichia coli:
a)Las variantes patogénicas quedan incluidas en un único grupo que ha de ser distinguido de las no patogénicas mediante PCR múltiple.
b) La información genética necesaria para la producción de toxinas Shiga y/o de hemolisina entérica no puede ser detectada mediante PCR múltiple.
c)Se ha logrado optimizar la detección rápida y precisa de variantes génicas productoras de factores de virulencia, pero aún no se ha conseguido mediante PCR.
d)Hasta ahora, no se ha considerado prioritario el desarrollo de sistemas de detección múltiple.
e)Distintos protocolos de PCR múltiple permiten la detección simultánea, rápida, específica y sensible de numerosos genes de relevancia clínica.
9.En lo que se refiere a Staphylococcus aureus:
a)La detección molecular no se considera de interés dada la casi ausencia de infecciones causadas por esta especie.
b) Su presencia habitual en individuos sanos ha hecho imposible el diseño de pruebas moleculares para detectar cepas virulentas.
c)Su escasa adquisición de resistencias a antibióticos ha hecho poco relevante la posible detección múltiple de resistencias.
d)Recientemente, se ha dado a conocer una prueba que permite la detección simultánea de hasta nueve genes de resistencia.
e)Recientemente, ha sido dado a conocer una prueba que permite la detección simultánea de hasta nueve genes específicos de especie.
10.En el caso de bacterias pertenecientes al género Enterococcus:
a)El bajo número de infecciones que producen hace escasa la relevancia clínica de su detección.
b) La clara diferenciación entre especies permite seguir considerando óptimos los análisis fenotípicos.
c)Se han descrito métodos de PCR múltiple que permiten la detección simultánea de distintos genes de resistencia a glucopéptidos.
d)Los métodos convencionales de identificación son óptimos por lo que ha sido considerado innecesario el desarrollo de métodos moleculares.
e)Hay métodos de PCR múltiple para la detección simultánea de distintos genes de resistencia a glucopéptidos, pero no para la identificación a nivel de género.
NOTA
Los artículos publicados en la sección "Formación Médica Continuada" forman parte de grupos temáticos específicos (antibiograma, antimicrobianos, etc.). Una vez finalizada la publicación de cada tema, se irán presentando al Sistema Español de Acreditación de la Formación Médica Continuada (SEAFORMEC) para la obtención de créditos.
Una vez concedida la acreditación, ésta se anunciará oportunamente en la revista y se abrirá un período de inscripción gratuito durante 3 meses para los socios de la SEIMC y suscriptores de la Revista, al cabo del cual se iniciará la evaluación, durante 1 mes, que se realizará a través de la web de Ediciones Doyma.