Introducción
En los últimos años se han producido cambios muy significativos en el tratamiento de los pacientes infectados por el virus de la inmunodeficiencia humana (VIH). Los tests de resistencia del VIH se han considerado importantes armas en la práctica clínica en el manejo de pacientes en fracaso terapéutico. La utilidad de estos tests en la prescripción de nuevos regímenes terapéuticos en pacientes con una amplia historia de tratamiento antirretroviral previa se ha demostrado en varios estudios como VIRADAPT, GART y Vira 30011-3. En el último documento de consenso de la Sociedad Internacional de Sida se amplían las situaciones clínicas en las cuales se recomienda la realización de estudios de resistencia incluyéndose los casos de infección aguda o reciente y previo a la inicialización del tratamiento antirretroviral en infección VIH ya establecida4. Los métodos que actualmente se emplean en el estudio de resistencia del VIH incluyen los tests fenotípicos, que determinan en medios de cultivo la sensibilidad a los diferentes fármacos5,6, y los tests genotípicos, que determinan la secuencia del gen de la transcriptasa inversa y de la proteasa del virus, detectando cambios en la secuencia de la población viral del paciente al compararla con la de una cepa salvaje del virus7,8. Los estudios fenotípicos resultan más fáciles de interpretar, pero no detectan cambios pequeños en la sensibilidad del virus, son más laboriosos y costosos. Los estudios genotípicos se usan con más frecuencia en la práctica clínica diaria debido a su mayor capacidad de detectar pequeños cambios en la sensibilidad, son más sencillos de realizar, los resultados se emiten en un menor tiempo y con un menor coste. La mayor limitación de los tests genotípicos es su interpretación, debido al elevado número de mutaciones de resistencia que actualmente se conocen y la interacción que existen entre ellas. Recientemente la compañía Virco ha introducido el fenotipo virtual® que combina las características de la genotipificación y fenotipificación comparando la información genética del virus del paciente con los genotipos y fenotipos incluidos en su base de datos. La interpretación de los resultados genotípicos pueden realizarse empleando diferentes algoritmos lo que genera aún más confusión. En este trabajo se recogen los resultados obtenidos en el Hospital Universitario de Valme, Sevilla, desde que se iniciaron los estudios de resistencia en el año 2001. Con estos datos se pretende realizar una descriptiva del grupo de pacientes que han requerido este tipo de estudio en función de los tratamientos que han recibido, así como la prevalencia de mutaciones en el área sanitaria que atendemos. Hemos estudiado también la concordancia existente entre dos algoritmos de interpretación genotípica, Visible Genetics versión 5 y 6 y Retrogram® 1.4 y el recientemente introducido, fenotipo virtual.
Métodos
Pacientes y muestras
En el período comprendido entre junio de 2001 y mayo de 2003, el Hospital Universitario de Valme (Sevilla) ha secuenciado los genes de la proteasa (PR) y de la transcriptasa inversa (TI) de 397 muestras pertenecientes a pacientes infectados por el VIH. Para el estudio de concordancia entre los diferentes sistemas de interpretación se incluyeron 105 del total de muestras secuenciadas, a las cuales por su patrón complejo de mutaciones (acumulación de NAMS para inhibidores de la transcriptasa inversa análogos de nucleósidos [ITIAN] y de mutaciones secundarias para inhibidores de la proteasa [IP]) se les realizó estudio del fenotipo virtual. Los pacientes estudiados pertenecían a las categorías: pacientes no tratados previamente, primer fracaso terapéutico, segundo fracaso o multifracaso.
Extracción del ARN
El ARN viral fue extraído de 140 ml de suero empleando el kit Quiagen (TRUPREP® Extraction kit, Visible Genetics) siguiendo las instrucciones del fabricante.
Análisis de la secuencia de la PR y de la TI
El análisis de las muestras se realizó empleando el kit TRUGENE HIV-1 Genotyping, kit, siguiendo las recomendaciones del fabricante (Visible Genetics). Los primers empleados en este kit amplifican la proteasa entera y los primeros 250 codones de la retrotranscriptasa. Las secuencias obtenidas se compararon con una secuencia de referencia (VIH-1LAV-1) empleando la base de datos de Gene Objects (Visible Genetics Inc.) para determinar la presencia de mutaciones7.
Algoritmos
Para la interpretación genotípica se emplearon dos algoritmos, Visible Genetics versión 5 y 6 (Toronto, Ontario, Canadá) (VG) y Retrogram® 1.4. (RG). Para interpretar el patrón de mutaciones, también utilizamos el fenotipo virtual® (FV) el cual compara la información genética del virus del paciente con los genotipos y fenotipos incluidos en la base de datos de Virco. El software proporciona un "fenotipo virtual" que muestra en valor porcentual el número de cepas sensibles y resistentes para cada fármaco además del valor del IC50. La interpretación de los resultados es distinta en función del algoritmo utilizado; VG informa de tres niveles de resistencia: susceptible o no evidencia de resistencia (S), resistente (R) y un nivel intermedio (I) descrito como posible resistencia. El RG informa de cuatro niveles de resistencia, clase A a D, y el FV considera susceptible cuando el 50% o más de las cepas de la base de datos que casan con el genotipo del paciente están dentro del rango susceptible y resistente cuando menos del 50% de las cepas de la base de datos está por debajo del rango de susceptible. Para poder comparar los resultados obtenidos por los tres sistemas, la interpretación "S" de VG se ha comparado con la clase "A" de RG, la interpretación "R" de VG ha sido comparada con la clase "D" de RG y la interpretación "I" de VG se ha comparado con las clases "B" y "C" de RG. Para comparar con el FV, se consideró un rango de susceptible mayor del 60%, resistente menor de 40% e intermedio cuando entre el 40-60% de las cepas encontradas en la base de datos se encontraba dentro del rango susceptible.
Antirretrovirales
Se han estudiado 16 antirretrovirales pertenecientes a tres familias: inhibidores de la transcriptasa inversa análogos de nucleósidos y nucleótidos (ITIANt): zidovudina (AZT), lamivudina (3TC), estavudina (d4T), didanosina (ddI), zalcitabina (ddC), abacavir (ABC) y tenofovir (TDF); inhibidores de la transcriptasa inversa no análogos de nucleósidos (ITINN): delavirdina (DLV), nevirapina (NVP) y efavirenz (EFV); e inhibidores de la proteasa (IP): saquinavir (SQV), indinavir (IDV), ritonavir (RTV), nelfinavir (NFV), amprenavir (AMP) y lopinavir (LPV).
Análisis
La concordancia entre los diferentes sistemas de interpretación se estudió mediante el análisis estadístico del índice kappa de Cohen (k > 0,75 concordancia excelente; k 0,4-0,75 concordancia moderada; k < 0,4 concordancia deficiente) utilizando el programa estadístico SPSS (versión 11.5, Año 2003, Statistical Product and Service Solutions Iberica. S.L.U., Madrid, España). En el análisis hemos empleado tres términos: concordancia cuando todos los algoritmos dan el mismo nivel de resistencia (S, I o R) a una secuencia para un fármaco en particular; discordancia menor cuando los algoritmos asignan diferentes categorías (S/I o I/R), y discordancia mayor cuando un algoritmo asigna una S y otro una R para un fármaco en particular.
Mutaciones
Las mutaciones de resistencia se han definido como mutaciones asociadas con la resistencia a los diferentes antirretrovirales. El término NAMS (mutaciones asociadas a los análogos de nucleósidos) se ha usado para hacer referencia a las mutaciones comunes de la RT: M4IL, D67N, K70R, L210W, T215Y/F y K219Q/E.
Resultados
Hemos realizado el estudio de resistencia genotípica a 397 muestras de pacientes con carga viral comprendida entre 963 y 4.400.000 copias/ml (Roche Amplicor HIV-1 Monitor Assay).
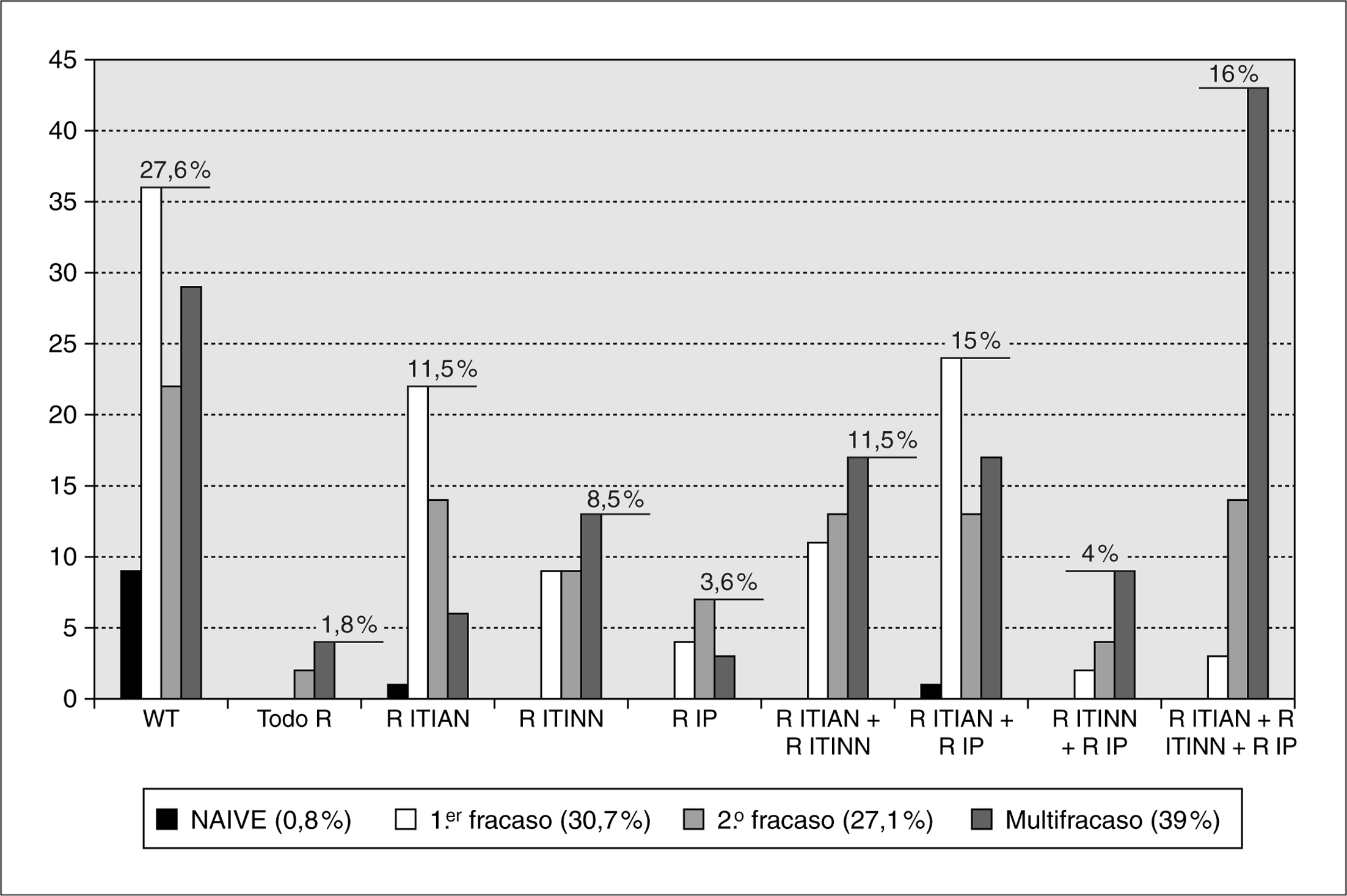
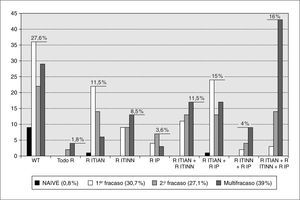
En la figura 1 se representa la distribución del tipo de pacientes implicados en el estudio y su relación con el porcentaje de cepas salvajes y cepas resistentes tanto a uno como a más fármacos de una o de más familias de antirretrovirales. El mayor porcentaje de pacientes correspondió a multifracaso (39%) seguidos de pacientes en segundo (30,7%) y en primer fracaso (27,1%), encontrándose un bajo porcentaje de pacientes no tratados previamente (0,8%). Encontramos que el 27,6% de las muestras eran cepas salvajes, estando aquí representados todos los grupos de pacientes. El 1,8% de las muestras fue resistente a todos los antirretrovirales correspondiendo principalmente a pacientes en multifracaso. La resistencia a una única familia de antirretrovirales fue mayor para los ITIAN (11,5%) principalmente en pacientes en primer y segundo fracaso, seguida por la de los ITINN (8,5%), sobre todo en pacientes en multifracaso, siendo menor la resistencia para la familia de los IP (3,6%). En cuanto a la resistencia a varias familias de antirretrovirales, el mayor porcentaje lo encontramos para ITIAN e IP (15%) en mayor medida en pacientes en primer fracaso, seguido de resistencia a algún fármaco de la familia ITIAN e ITINN (11,5%), siendo el grupo menos representado el que mostraba resistencia conjunta para los ITINN e IP (4%). En un 16% de las muestras estudiadas se encontró resistencia a alguno de los fármacos pertenecientes a las tres familias de antirretrovirales, principalmente en pacientes en multifracaso, seguidas de pacientes en segundo fracaso terapéutico.
Figura 1. Resistencias asociadas al tipo de fracaso. WT: Wild Type (Cepa salvaje); Todo R: resistencia a todos los fármacos pertenecientes a todas las familias de antirretrovirales. El resto se refiere a resistencia a algún o algunos antirretrovirales de la familia de análogos de nucleósidos/nucleótidos (ITIAN), no análogos (ITINN) e inhibidores de la proteasa (IP).
Cuando se analiza el número de mutaciones, se encuentra que para la TI el 64% de las cepas presentó de 0 a 3 mutaciones y el 35,9% más de 3 mutaciones. En cuanto a las mutaciones NAMS, más del 50% de nuestras muestras presentaron más de tres y como era de esperar el número de NAMS aumentaba proporcionalmente con el número de fracasos.
Para la PR, el 65,9% de las cepas mostraron de 0 a 3 mutaciones y el 33,6% más de 3 mutaciones. De ellas, el 40,8% de las cepas presentaron alguna mutación primaria y el resto (21,6%) sólo mutaciones secundarias (99,7% entre 0 y 3).
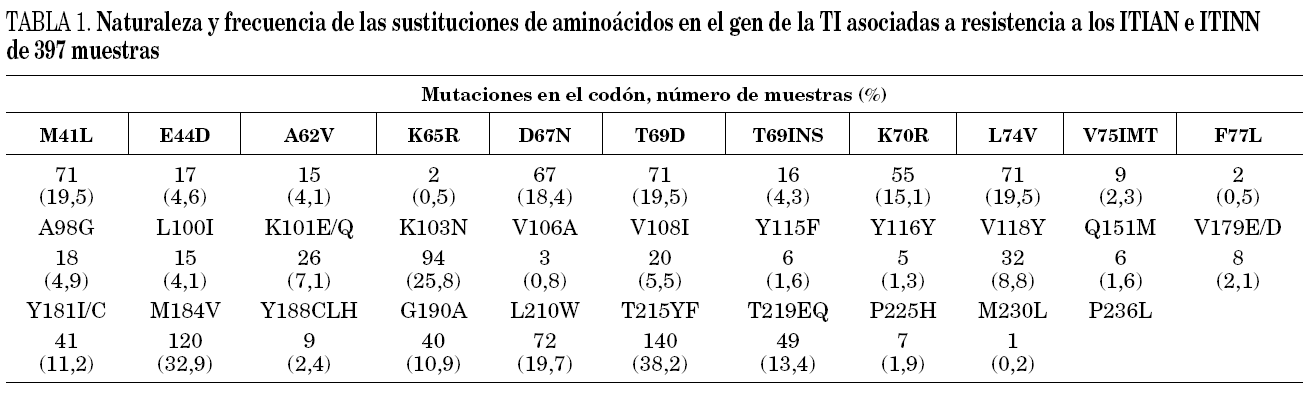
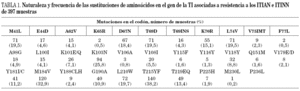
En la tabla 1 vienen representados los porcentajes de las diferentes mutaciones de resistencia para la familia de los ITIAN/ITINN. En cuanto a las mutaciones asociadas a los ITIAN la mutación más frecuente fue la T215Y/F (37,2%) y la M184V (32,9%), seguida por las mutaciones NAMS, entre las cuales, además de la 215, las más representadas han sido las mutaciones L210W (19,7%), K70R (15,1%) y la K219Q/E (13,4%). Las mutaciones responsables de fenotipos de multirresistencia, inserción en el codón 69 y complejo Q151M tuvieron una baja representación en esta serie (1,5%). Para los ITINN la mutación K103N es la que mayor representación ha tenido en nuestra serie (25,8%), seguida por la mutación Y181C (11,2%) y G190A (10,9%). El resto de mutaciones se encontraban en un porcentaje inferior al 10%.
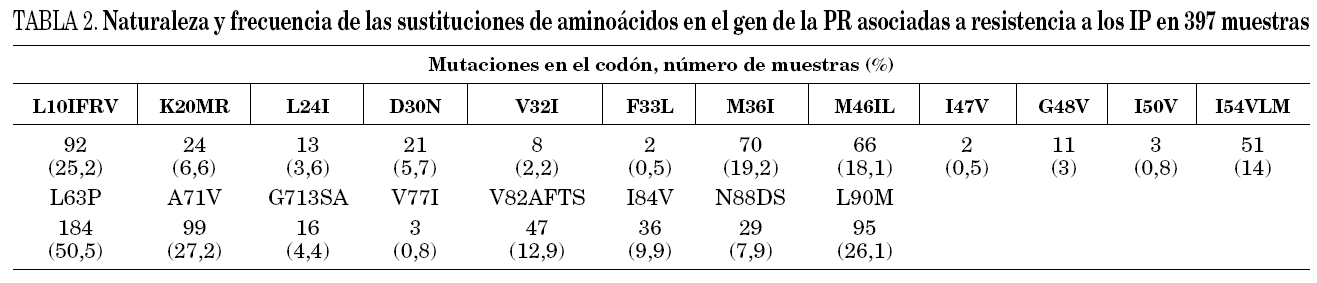
En la tabla 2 se recogen las mutaciones asociadas a la PR. En cuanto a las mutaciones primarias que han aparecido con mayor frecuencia son la L90M (26,1%), M46I (18,1%) y V82A/F/T/S (12,9%), seguidas de las mutaciones en los codones 30, 48, 50 y 84 (< 10%). Las mutaciones secundarias con más representación en esta serie coinciden con los cambios en sitios polimórficas, L63P (50,5%), A71V (27,2%), L10I (25,2%) y M36I (19,2%) seguida por la mutación en el codón VI54V (14%), el resto de mutaciones secundarias han aparecido en un porcentaje menor al 10%.
En el estudio de correlación entre los diferentes algoritmos, VG y RG asignaron 1.679 interpretaciones de resistencia a las 105 secuencias analizadas. El FV no asignó 128 interpretaciones, ya que para algunos antirretrovirales de secuencias determinadas no se pudo establecer comparación con su base de datos. Se encontró coincidencia total entre los tres sistemas para todos los antirretrovirales estudiados en 902/1.541 (58,5%) de las muestras: en el 27,5% de los casos los algoritmos asignaron S, en el 29,1% R y en el 1,8% I. El algoritmo que asignó más interpretaciones S fue el FV (50,3%), seguido de VG (37,7%) y RG (30,5%). VG fue el que asignó más interpretaciones R (47,5%), seguido de FV (40,8%) y RG (35,2%). La interpretación I fue más prevalente en RG (34,2%) y VG (18,7%), siendo el FV el algoritmo que asignó menos interpretaciones I (9,1%).
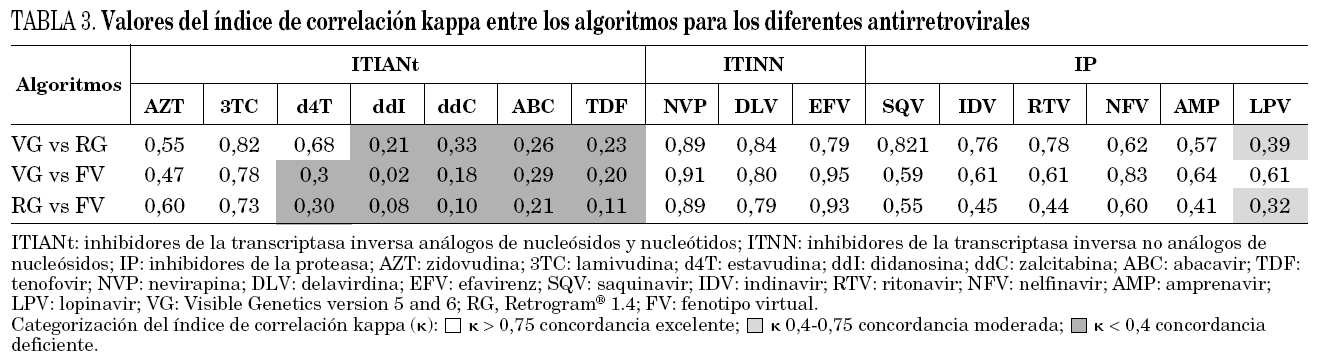
La tabla 3 muestra la cuantificación de la concordancia interalgoritmo empleando el índice k. Para los ITIAN se obtuvo una buena concordancia entre los sistemas estudiados para el 3TC (k = 0,73-0,82), y moderada para el AZT (k = 0,47-0,60); sin embargo, para el resto de fármacos del grupo la concordancia fue inferior con un índice de kappa menor de 0,3. Para el d4T se encontró una concordancia moderada cuando se compararon VG-RG (0,68) pero no cuando se compararon los resultados de estos algoritmos genotípicos con el FV (k = 0,3). Se encontraron resultados discordantes entre RG-FV y VG-RG en el 50% de las muestras para ddI, ddC, ABC y TDF, el nivel de discordancia entre VG-FV superó el 50% para el ddI y el ddC.
Para los ITINN la concordancia hallada fue excelente (k = 0,79-0,95) con independencia de los sistemas comparados, encontrándose resultados discordantes únicamente en el 10% de los casos.
Para los IP la concordancia entre algoritmos varió según el fármaco analizado. Para SQV, IDV y RTV, la mejor concordancia se obtuvo entre VG-RG (k = 0,70-0,82). Para NFV, APV y LPV la mejor concordancia se observó entre VG-FV (k = 0,61-0,83). Para todos los IP se obtuvo una peor concordancia cuando los algoritmos comparados fueron RG-FV (k = 0,38-0,60).
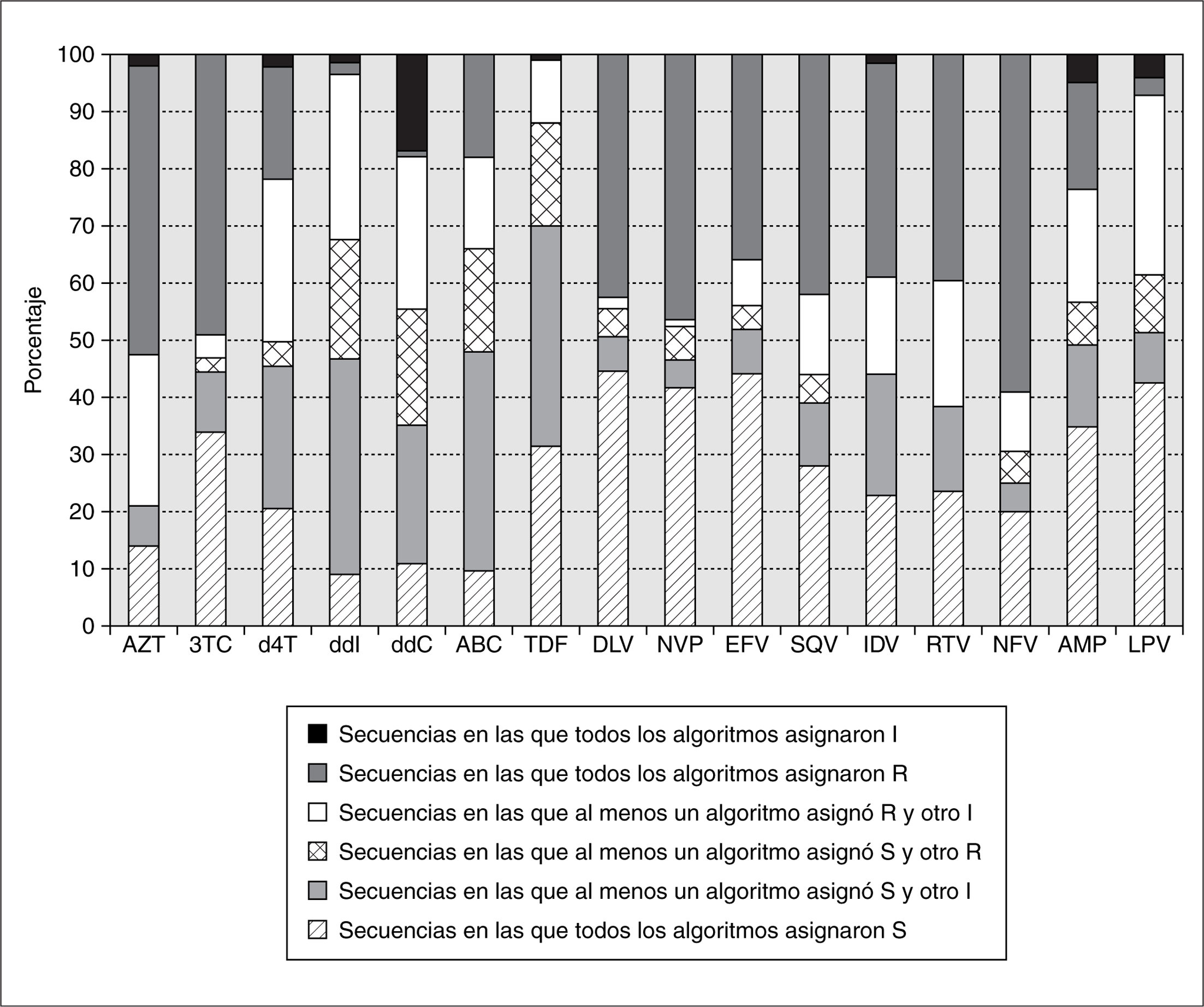
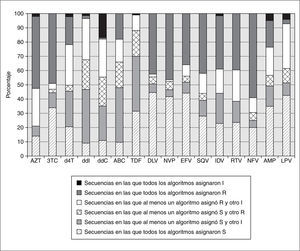
En la figura 2 se representan las naturalezas de las discordancias entre los diferentes algoritmos de interpretación. Se observaron discrepancias mayores (resultados S/R) en el 8,3% de los resultados. Estas discrepancias se debieron principalmente a la interpretación de los ITIAN (5,7%), sobre todo para el ddC, ddI, ABC y TDF, seguida de la interpretación de los IP (1,7%) principalmente para LPV y APV, siendo los ITINN los antirretrovirales que mostraron menor número de estas discrepancias (0,99%). En la mayoría de los casos las discrepancias mayores se debieron a una mayor interpretación como R por parte de los sistemas genotípicos en comparación con el FV. En cuanto al porcentaje de discrepancias menores (resultados R/I y S/I) fue de 32,5%. Los ITIAN fueron los antirretrovirales que presentaron un mayor número de ellas (18,8%) principalmente ABC, d4T, ddC, ddI y TDF, seguidos de los IP (11,8%) principalmente APV, IDV, LPV y RTV, y los ITINN (1,96%), principalmente el EFV.
Figura 2. Naturaleza de las discordancias halladas en 1.541 interpretaciones entre Visible Genetics, Retrogram® y fenotipo virtual.
Discusión
Las recomendaciones referentes al estudio de resistencia del VIH establecieron su empleo en situaciones concretas9,10. En el último documento de consenso publicado en el año 2003 se aumenta el número de situaciones clínicas en las que se recomiendan los estudios de resistencia. Éstas incluyen: infección VIH aguda o reciente, por la posibilidad de transmisión de una cepa resistente; estudio previo a la instauración de terapia en pacientes con infección por el VIH establecida, basándose en la perdurabilidad de las mutaciones inducidas por un régimen terapéutico incluso más de 2 años tras la supresión del mismo11; en primer y multifracaso terapéutico y en el embarazo4.
Uno de los objetivos de este trabajo ha sido analizar en qué situaciones clínicas se solicita los estudios de resistencia, así como realizar una descripción del patrón de mutaciones que presentan nuestro grupo de pacientes. Hemos observado que los cambios en las recomendaciones para la realización de estudios de resistencia son confirmados por nuestro trabajo, ya que el porcentaje de estudios de resistencia solicitados a pacientes en multifracaso fue muy similar al de pacientes en primer y segundo fracaso terapéutico. Hemos encontrado una tasa de resistencia algo menor para la familia de los ITIAN (53%), con respecto a los ITINN e IP (40%). Observamos cómo la resistencia a varios grupos aparece en mayor medida en pacientes en segundo y multifracaso, debido al mayor empleo de combinaciones de antirretrovirales. Cabe destacar que los pacientes en primer fracaso son los que presentan mayor resistencia aislada a los fármacos de la familia ITIAN y de las familias ITIAN + IP. Esto posiblemente se debe a que generalmente en la terapia inicial de tratamiento están incluidas ambas familias de antirretrovirales. Por otra parte hemos encontrado un elevado número de pacientes en fracaso a más de una línea de tratamiento que presentaban aislamientos de virus salvaje, lo que demuestra posiblemente la falta de adherencia de los pacientes de esta serie a la terapia antirretroviral.
Con respecto a las mutaciones detectadas, observamos cómo la frecuencia de mutaciones asociadas a los ITIAN concuerdan con los resultados publicados por otros autores12-14. La mutación que ha aparecido con mayor frecuencia es la T215Y seguida por la mutación M184V la cual confiere alta resistencia al 3TC y baja al ABC. Se ha visto que esta mutación revierte la resistencia a la AZT mediada por la mutación T215Y15, lo que explica la lenta evolución de resistencia fenotípica a la AZT en pacientes tratados con AZT + 3TC, aunque este efecto se ve neutralizado ante la presencia de cuatro o más mutaciones de resistencia al AZT. El 17% de nuestros pacientes recibían tratamiento combinado con ambos antirretrovirales, de los cuales sólo el 3% presentaba ambas mutaciones y el 48% presentaba la mutación M184V en ausencia de la T215Y. En frecuencia de aparición la mutación M184V ha sido seguida por las mutaciones NAMS, más del 50% de nuestras muestras presentaron más de tres y, como era de esperar, el número de NAMS aumentaba proporcionalmente con el número de fracasos. Varios trabajos han demostrado recientemente que a medida que se acumulan estas mutaciones en la TI se produce un incremento del nivel resistencia fenotípica en todos los ITIAN incluido el TDF16,17.
Otra mutación que ha aparecido con bastante frecuencia en esta serie es la L74V, mutación seleccionada más frecuentemente por ddI y que confiere un moderado incremento en su IC50, pudiendo conferir resistencia cruzada con el ddC. En cuanto a las mutaciones responsables de fenotipos de multirresistencia, inserción en el codón 69 y complejo Q151M, han obtenido una baja representación en este grupo, lo que coincide con estudios previos15,18.
En cuanto a los ITINN los resultados que hemos obtenidos concuerdan con los aportados por otros autores19. La mutación K103N es la que aparece con mayor frecuencia, y causa por sí sola una resistencia de 20-50 veces a todos los no análogos. Dentro de las mutaciones que causan alto nivel de resistencia encontramos una representación notable de la Y181C (NVP y DLV) y la G190A/S (NVP y EFV), apareciendo con mucha menor frecuencia la mutación Y188C/L/H (alto nivel de resistencia para NVP y EFV, intermedio para DLV).
La resistencia del VIH a los IP se debe a la adquisición por el virus de una serie de mutaciones en la región del genoma que codifica la proteasa viral. Dichas mutaciones se han denominado primarias o principales y secundarias o accesorias. En cuanto a las mutaciones primarias, las que han aparecido con mayor frecuencia son L90M, V82A/F/T/S y I84V, respectivamente, como ya han descrito otros autores19. En cuanto a las mutaciones fármaco-específicas, existe una presencia moderada de la D30N que aparece en pacientes en tratamiento con NFV, seguida por la G48V que confiere resistencia a SQV y con una frecuencia mucho menor la mutación I50V implicada en la resistencia a amprenavir. Para la PR se han descrito siete posiciones polimórficas, que incluyen los codones 10, 20, 36, 63, 71, 77 y 93, que por sí mismas no causan resistencia pero que en presencia de otras mutaciones contribuyen de manera importante a la adquisición de ésta. En pacientes muy tratados, la prevalencia de mutaciones en estas posiciones aumenta dramáticamente20,21. En nuestra serie todos estos polimorfismos se encuentran representados de forma importante, muchos de ellos incluso en mayor porcentaje que las mutaciones primarias. Uno de los objetivos prioritarios de este estudio ha sido analizar la concordancia existente entre dos algoritmos de interpretación genotípica y el fenotipo virtual.
El análisis de concordancia que se presenta muestra la concordancia que existe entre los tres sistemas comparados para todas las familias de antirretrovirales, encontrándose concordancia entre ellos en el 58,5% de las interpretaciones. Existen diferencias de concordancia según la comparación que se realice, sistema genotipo/genotipo o genotipo/FV. Para los ITIAN, la concordancia entre el genotipo y el FV es muy inferior a la obtenida entre VG y RG; en todos los casos la mayor concordancia fue la obtenida para el 3TC y AZT. Para los IP la concordancia entre algoritmos varió según el fármaco analizado, obteniéndose una mejor correlación para SQV, IDV y RTV cuando se compararon los sistemas basados en reglas (RG frente a VG), mientras que para NFV, APV y LPV la mejor concordancia se observó entre VG y el FV. Se observa para la familia de los ITINN una excelente concordancia con independencia de los sistemas comparados.
Igualmente Torti et al22 obtienen mejores resultados de concordancia entre VG y RG principalmente para los ITINN y los IP, detectando una menor concordancia para la mayoría de los ITIAN cuando comparan con el FV. Kijak et al23 han publicado resultados similares que incluyen también la comparación con el sistema de interpretación de la base de datos de Stanford. Puchhammer et al24 obtienen similares conclusiones cuando comparan esta guía con el FV.
En las discordancias entre los diferentes sistemas se pone de manifiesto el hecho de que el FV es el sistema que asigna más interpretaciones "S". Estudios previos apuntan que la información de la resistencia basada en el genotipo es un mejor predictor de la respuesta clínica1,22,25,26.
Hemos observado que para los ITINN, en las discrepancias mayores estaba principalmente implicada la mutación K103N acompañada o no de la mutación V108I. Para los ITIAN, como era de esperar, el grupo de mutaciones NAMS estaba presente en la mayoría de las discrepancias entre algoritmos. En cuanto a los IP se observan patrones muy diversos.
En conclusión, estos resultados reflejan que el número de pacientes a los que se les solicita estudio de resistencia en nuestro área sanitaria, es muy similar para los tres grupos de fracasos, siendo muy pocas las peticiones a pacientes no tratados previamente, debido a la escasa presencia de este tipo de pacientes en la actualidad. Nos parece importante destacar que muchos de los fracasos terapéuticos que hemos estudiado no se deben a la adquisición de mutaciones de resistencia, sino posiblemente a una falta de adherencia al tratamiento ya que hemos detectado un gran número de pacientes en segundo fracaso y multifracaso que no presentaban ninguna mutación que respaldara dicho fracaso virológico. Sería deseable poder evaluar de manera fiable el grado de adherencia al tratamiento en estos pacientes y poder optimizar así las peticiones de los estudios de resistencia. Por otra parte, el patrón de mutaciones encontradas en este estudio es el que puede esperarse en este momento con los tratamientos antirretrovirales de gran actividad (TARGA) que se instauran en la actualidad, utilizando un método de resistencia.
Este estudio pone de manifiesto que los antirretrovirales que presentan patrones de resistencia complejos (IP, ITIAN), hacen la interpretación más complicada, tanto para los sistemas de interpretación genotípicos como fenotípicos, obteniéndose por tanto peores índices de correlación entre los sistemas de interpretación, por lo que sería necesario llegar a un consenso entre expertos de tal forma que se elaborara un algoritmo de interpretación consensuado y avalado por estudios prospectivos de eficacia clínica del mismo.