La tuberculosis continúa siendo hoy en día un grave problema de salud pública con 10,8 millones de casos nuevos y 1,8 millones de muertes a nivel mundial en el año 2015. La diversidad existente entre los miembros del complejo Mycobacterium tuberculosis que producen la tuberculosis permiten el diseño de métodos para su rápido diagnóstico. Así mismo, las mutaciones en los genes implicados en los mecanismos de resistencia permiten escapar a la bacteria del tratamiento. Hemos revisado los métodos de diagnóstico rápido de M. tuberculosis complex y de la detección de la sensibilidad/resistencia a los fármacos; ambas cosas son necesarias para detener la aparición de nuevas resistencias e instaurar un tratamiento precoz y correcto.
Tuberculosis is still a serious public health problem, with 10.8 million new cases and 1.8 million deaths worldwide in 2015. The diversity among members of the Mycobacterium tuberculosis complex, the causal agent of tuberculosis, is conducive to the design of different methods for rapid diagnosis. Mutations in the genes involved in resistance mechanisms enable the bacteria to elude the treatment. We have reviewed the methods for the rapid diagnosis of M. tuberculosis complex and the detection of susceptibility to drugs, both of which are necessary to prevent the onset of new resistance and to establish early, appropriate treatment.
Los primeros indicios de la enfermedad tuberculosa se han hallado en momias de 5.000 años antes de la era actual y pese a que han transcurrido 135 años desde que Robert Koch descubriera el bacilo productor de la «tisis blanca», la tuberculosis (TB) continúa siendo hoy en día un grave problema de salud pública a nivel mundial. Según los últimos datos comunicados por la OMS, en el año 2015 se produjeron 10,8 millones de casos nuevos y 1,8 millones de muertes a nivel mundial debidas a esta enfermedad de transmisión respiratoria1. En ese mismo año en España se comunicaron 4.191 casos nuevos (12/100.000 habitantes).
Desde el descubrimiento de los primeros fármacos, la estreptomicina (SM) en 1943, la isoniazida (INH) en 1952, y la rifampicina (Rif) en 1959, comenzaron a surgir cepas resistentes, lo que hizo necesario tratar la enfermedad con una combinación de fármacos. El tratamiento insuficiente o interrumpido genera la aparición de cepas resistentes a algún fármaco o a varios, apareciendo las cepas multirresistentes (MDR), definidas como resistentes al menos a la INH y a la Rif. Cuando las resistencias no se identifican antes del inicio del tratamiento, la situación se vuelve particularmente peligrosa, ya que los pacientes infectados con cepas monorresistentes tienen un alto riesgo de desarrollar resistencias adicionales a otros fármacos si reciben la terapia estándar. Una mala administración de los fármacos de segunda línea, menos eficaces y con efectos secundarios más graves, puede conducir a la aparición de cepas extensamente resistentes (XDR), que son cepas MDR con mecanismos de resistencia adicionales frente al menos a uno de los 3 fármacos inyectables: kanamicina (KM), amicacina (AK) y capreomicina (CPM), y a cualquier fluoroquinolona empleada frente a Mycobacterium tuberculosis (MTB)1. Los casos de TB-XDR en la práctica son casi imposibles de tratar. Se han notificado casos con cepas XDR que son incluso resistentes a delamanid y bedaquilina, fármacos antituberculosos introducidos recientemente para tratar TB-XDR2. Es por tanto crucial el diagnóstico temprano de la TB, la obtención de resultados de sensibilidad rápidos, y una correcta prescripción de los fármacos, para un tratamiento y control adecuados de la enfermedad.
El género Mycobacterium es el único de la familia Mycobacteriaceae, del orden de los actynomicetales, caracterizados por tener un elevado contenido en guanina y citosina (G+C). Apareció como lo conocemos hoy en día hace unos 15.000 años. Existen más de 170 especies descritas, la mayoría de ellas pertenecen al grupo de micobacterias no tuberculosas (MNT) y solo unas pocas, que comparten un 99,9% de homología en sus genomas, pertenecen al complejo M. tuberculosis (MTBc), todas ellas productoras de TB, en el hombre o en diferentes especies animales. El MTBc incluye distintos linajes que han evolucionado genotípica y fenotípicamente tras su expansión clonal desde un progenitor común adaptándose a distintos huéspedes.
En el momento de tener una sospecha clínica de TB, el primer paso es confirmar el diagnóstico microbiológicamente. La baciloscopia o visualización de los microorganismos a partir de una extensión del producto patológico teñido con tinción de Ziehl-Neelsen continúa siendo el método más rápido, barato y accesible para diagnosticar los casos bacilíferos, si bien su sensibilidad comparada con el cultivo oscila entre el 25 y el 90%, dependiendo de la muestra que estemos estudiando y de la carga bacteriana de dicha muestra, así como de la calidad del observador. El cultivo en medio sólido, Lowenstein-Jensen y Coletsos, tiene el inconveniente de que MTBc tarda más de 15 días en crecer. El primer avance en cuanto al diagnóstico rápido se debe a S. Siddiqi, quien diseñó a finales de los 70 el sistema radiométrico Bactec 460TB, con el que se acortaba el tiempo hasta la obtención del cultivo positivo y se aumentaba la sensibilidad3. Además aportó la ventaja adicional de que se podía realizar el antibiograma disminuyendo considerablemente el tiempo para la obtención del resultado en comparación con el método de las proporciones de Canetti4. La identificación por métodos fenotípicos: temperatura óptima de crecimiento, pigmentación de las colonias y características bioquímicas, es lenta y a veces compleja, sobre todo en muchas de las especies de MNT. En la década de los 90 aparecieron las primeras técnicas de identificación genética, logrando nuevamente una mayor rapidez y precisión en el diagnóstico micobacteriológico5. La necesidad de disponer en el momento actual de un diagnóstico rápido es quizá el aspecto más importante para el control de la enfermedad tuberculosa. Ello ha impulsado en las últimas décadas la investigación y el desarrollo de nuevos métodos diagnósticos basados en la biología molecular. De manera ideal deberíamos tener una técnica sensible, rápida, razonablemente económica y que no necesitase medios técnicos complejos ya que la mayoría de casos se dan en países poco desarrollados y con recursos económicos limitados.
Con este trabajo no se pretende realizar una descripción exhaustiva de toda la metodología disponible o en desarrollo en el momento actual, para lo cual remitimos al lector interesado a la literatura más especializada6,7, sino revisar las distintas aproximaciones existentes al diagnóstico rápido y microbiológico de la enfermedad tuberculosa.
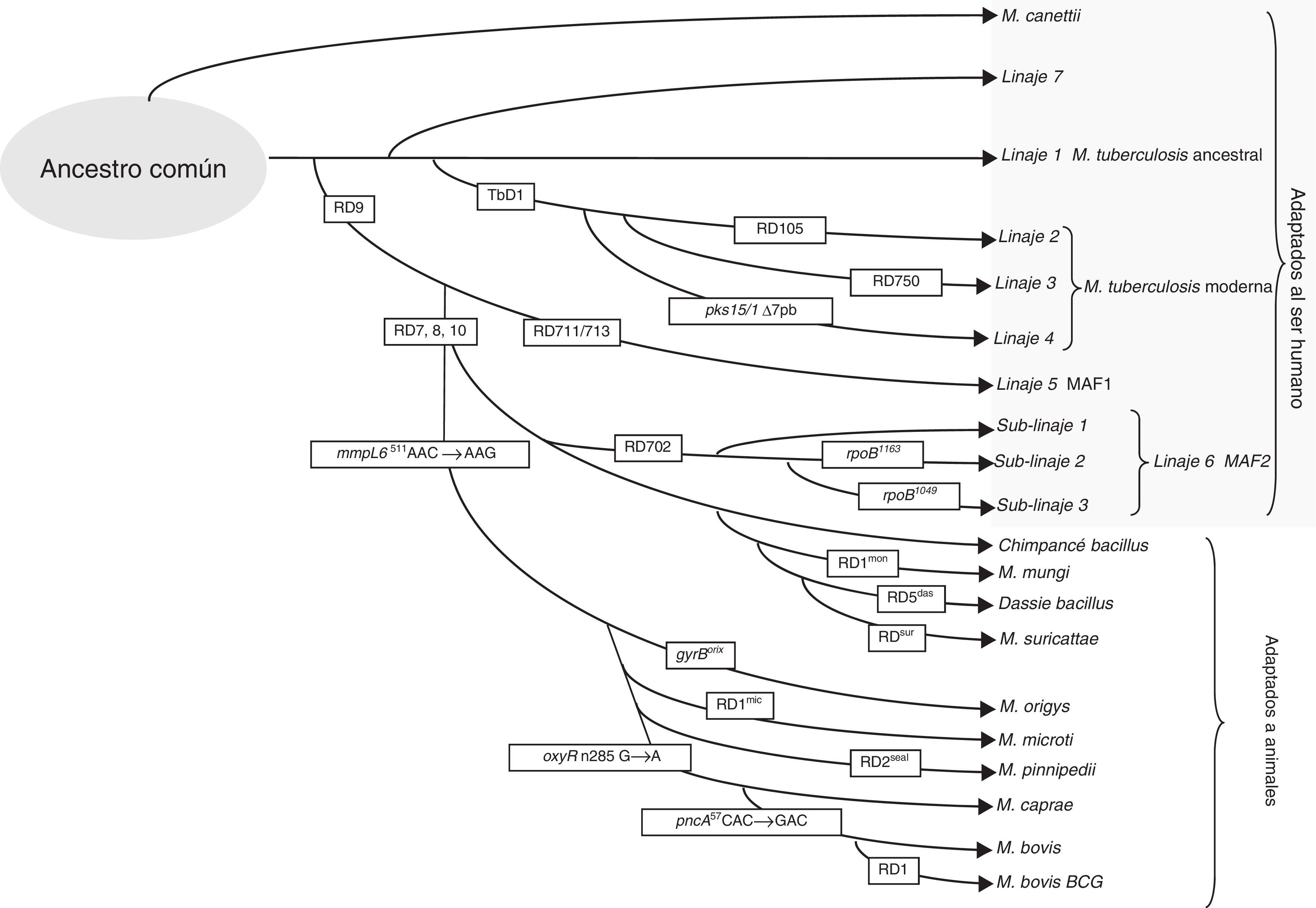
Diferencias geonómicas de los miembros del complejo Mycobacterium tuberculosisSe han detectado polimorfismos en sus genomas debidos a pérdidas de secuencias largas de nucleótidos (LSP), mutaciones afectando a un nucleótido (SNP) o variaciones de los elementos repetitivos en su genoma. Las deleciones específicas en el genoma de los diferentes miembros del complejo se han llamado regiones diferenciales (RDs)8 y pueden utilizarse para identificarlos y clasificarlos en 8 linajes diferentes (fig. 1). MTB incluye los linajes 1 a 4 y se ha adaptado de forma selectiva al ser humano. A pesar de infectar a una tercera parte de la población mundial, no ha pasado a establecerse en el mundo animal12. La presencia o ausencia de la región llamada TbD1 diferencia entre MTB ancestral (L1) y moderna8. Otras diferencias de SNP permiten clasificar a los aislados en sublinajes dentro de las cepas de MTB modernas: el L2 que incluye a la familia Beijing, L3 que incluye la familia CAS y L4 que incluye la familia Latinoamericana, la más presente en Europa. Para diferenciar los distintos linajes y sublinajes o familias se han propuesto diferentes técnicas mediante multiplex PCR, pirosecuenciación, SNAPshot y secuenciación de grupos de genes13-15. La pérdida de una región llamada RD9 separaría MTB de Mycobacterium africanum, asociados también al huésped humano (L5 y L6), y del resto de linajes del complejo asociados con huésped animal, linaje 816. Un grupo de cepas que circula entre humanos con aparentemente menor virulencia es el relacionado con el linaje 6, o también llamado M. africanum West African 2 (MAF2). Estas cepas han perdido las regiones RD7, RD8, RD9 y RD10, como ocurre en las cepas asociadas a animales, que incluyen Mycobacterium bovis, M. bovis bacillus Calmette-Guerin (BCG), Mycobacterium caprae, Mycobacterium orix, Mycobacterium pinnipedii, y los descritos más recientemente, el llamado chimpanzee bacillus, dassie bacillus, Mycobacterium mungi, y Mycobacterium suricattae, los cuales se han aislado de chimpancé, damán roquero, mangosta rayada y suricata, respectivamente17.
Diagnóstico microbiológico rápidoLa necesidad de un diagnóstico temprano cobra especial sentido si tenemos en cuenta que casi un 20% de las transmisiones son debidas a pacientes con baciloscopia negativa y cultivo positivo9, y al creciente aumento de los aislamientos con resistencias a distintos fármacos.
A pesar de que la microscopia continúa siendo el método más extendido y económico para el diagnóstico inmediato, sus inconvenientes —ya mencionados— han impulsado la investigación y el desarrollo de nuevos métodos, entre los cuales los de mayor implantación, sin lugar a dudas, han sido los basados en la biología molecular.
BiomarcadoresUn método rápido teórico para detectar infecciones activas es la detección de biomarcadores a partir de muestras mínimamente o no invasivas; sin embargo, a pesar de que hay un cierto desarrollo en esta línea, hasta el momento no se dispone de técnicas que puedan cumplir con las necesidades requeridas.
Serología. Al igual que en numerosas enfermedades infecciosas, con la TB se ha ensayado la detección de anticuerpos circulantes. Sin embargo, debido a su baja sensibilidad y a las reacciones cruzadas, la OMS ha desaconsejado formalmente su uso.
Detección de antígeno. Otra posibilidad es la búsqueda de antígeno de MTB en productos del paciente. A este respecto, existen comercializadas o en estudio algunas pruebas.
La detección en la orina de los pacientes del lipoarabinomanano, lipopolisacárido presente en la pared micobacteriana, ha sido desarrollada por Alere (LF-LAM, Alere DetermineTM TB LAM Ag, Alere Inc., Waltham, MA, EE.UU.). Se trata de una inmunocromatografía de flujo lateral para realizar a pie de cama (point-of-care) que solo requiere una mínima cantidad de orina, 25min de incubación, y es muy económica. Un inconveniente de esta técnica es que este lipopolisacárido no se encuentra exclusivamente en MTB, sino también en otras micobacterias. Tanto el informe de la OMS18 como una reciente revisión de la Biblioteca Cochrane19 concluyen que su sensibilidad, tanto en pacientes infectados por VIH como en convivientes, es baja, no recomendando su uso como cribado ni como diagnóstico, aunque podría ser de utilidad en pacientes con menos de 50CD4/μl, donde aumenta su sensibilidad.
Compuestos volátiles orgánicos. La detección de distintos componentes en el aire espirado del paciente constituye un área de interés creciente. Varios dispositivos con filosofía point-of-care están en fase de desarrollo o evaluación. Se pide al paciente que exhale el aire o tosa en un dispositivo (tras nebulización con suero fisiológico) y se analizan compuestos volátiles orgánicos o la presencia de antígeno de MTB. Se están llevando a cabo estudios con resultados prometedores7.
TAM-TB. Otro biomarcador sería el marcador de activación de células T (TAM), descrito en 201420, que mide por citometría de flujo la expresión de una proteína de membrana (CD127) en las células T CD4 expuestas a antígenos de MTB. En los ensayos preliminares se notificó una sensibilidad del 83%, con una especificidad del 96,6%. Próximamente se dispondrá de una versión comercial de esta técnica.
Detección de Mycobacterium tuberculosis en muestra clínicaEn los últimos 20 años se ha trabajado intensamente para conseguir un diagnóstico directo fiable a partir de muestra clínica. Fruto de ese trabajo son las múltiples técnicas que se han puesto en práctica y se han comercializado.
Micobacteriófagos. La utilización de fagos de micobacterias para su detección en muestra directa viene de la segunda mitad del sigloXX, cuando los micobacteriófagos fueron descubiertos. Desde entonces, se han ensayado diversos tipos de fagos y de técnicas. Las 2 técnicas más importantes son: Phague Amplified Biologically Assay, la primera que se utilizó, se basa en la capacidad del fago en lisar las células de un cultivo de Mycobacterium smegmatis, utilizado como control. Si el esputo del paciente contiene MTB viables, una vez que los fagos han sido liberados por lisis, se replican y crean «agujeros» en el agar del control. La segunda, más reciente, el Luciferase Reporter Phague Assay, consiste en un fago al que se le ha incorporado el gen de la luciferasa. En presencia del substrato luciferina, si hay micobacterias en la muestra, las infecta y se produce una emisión de luz que puede ser medida en un luminómetro. Estos test se realizan en 2-3 días, son sencillos y económicos y tienen buena especificidad, pero una sensibilidad variable que disminuye en las muestras con baja carga bacteriana21.
Métodos moleculares. Los mayores avances de los últimos años en el diagnóstico rápido de las enfermedades infecciosas, incluyendo la TB, se han producido sin lugar a dudas en el campo de la biología molecular. Los diferentes kits comerciales se basan en los polimorfismos genéticos que diferencian a los linajes con mayor repercusión clínica: MTB, M. bovis, M. bovis BCG y M. africanum. La sensibilidad, reproducibilidad, rapidez de resultados, la cada vez mayor automatización y el abaratamiento de costes han convertido estos métodos en trabajo de rutina en los laboratorios de microbiología.
Los test clásicos para amplificación de ácidos nucleicos, con diferentes tecnologías, se basan en la amplificación de secuencias diana y su posterior detección, ya sea por quimioluminiscencia, colorimetría o fluorometría22. Entre ellos:
Amplified M. tuberculosis Direct Test (AMTD, Gen-Probe Inc., EE.UU.). Amplifica el ARNr 16S mediante una reacción isotérmica y luego lo hibrida con sondas específicas de MTB que detecta mediante quimioluminiscencia. Fue el primer test aprobado por la Food and Drug Administration (FDA) en 1995 para muestras con baciloscopia positiva (>90% de sensibilidad), y en el 2000 para baciloscopia negativa (menor sensibilidad, 65-93%). Los principales inconvenientes son la ausencia de control interno y que no está automatizado.
Cobas Amplicor M. tuberculosis test (Roche Diagnostic System Inc., Suiza). Amplifica el ARNr 16S mediante PCR, hibrida con sondas y realiza la detección por colorimetría. También cuenta con la aprobación de la FDA desde 1996. Tiene una sensibilidad similar al anterior en baciloscopia positiva, y algo inferior si es negativa, manteniendo una muy buena especificidad (91-100%). A diferencia del AMTD, puede automatizarse y cuenta con control interno.
BD Probe Tec ET Direct TB System (DTB, Becton Dickinson). Amplifica 2 dianas, el ARNr 16S y el IS6110 mediante la técnica SDA (strand displacement amplification), que es una reacción enzimática isotérmica, detectando el producto final por fluorescencia. Su sensibilidad es mayor del 90% en muestras con baciloscopia positiva, y desciende considerablemente, con un rango variable en distintos estudios, en las negativas.
TB-LAMP (Eiken Chemical Company Ltd, Japón). Es un test basado en una técnica bastante reciente denominada LAMP (loop-mediated isothermal amplification), que amplifica ADN de forma muy rápida (menos de una hora), con lectura del resultado a simple vista por turbidez, color o fluorescencia, y con muy pocos requerimientos de infraestructura. Sus resultados son prometedores, y en una reciente revisión para muestras de esputo23 la OMS concluye que puede ser una alternativa en lugares con recursos escasos.
Otros métodos que detectan el producto amplificado por una hibridación en fase sólida consisten en una tira de nitrocelulosa que lleva fijadas las sondas específicas. Entre ellos, relativamente reciente (2010), está el método GenoQuick MTB (Hain Lifescience), que tiene como diana la secuencia de inserción específica para el MTBc, IS6110, y realiza la detección mediante hibridación en flujo lateral. La técnica se realiza en 3h, la fase postamplificación es manual y tiene una alta sensibilidad.
Existen otros 2 ensayos de hibridación en fase sólida comercializados INNO-LiPA Rif. TB (Innogenetics, Bélgica) y GenoType MTBDRplus (Hain Lifescience) que, además de identificar MTBc, son capaces de detectar mutaciones de resistencia, de las que hablaremos más adelante. Son de muy fácil interpretación y pueden utilizarse tanto en muestra directa como en cultivos, ambos con sensibilidades muy altas, y en ambos la fase postamplificación puede hacerse de forma manual o automatizada.
PCR a tiempo real. La PCR a tiempo real realiza simultáneamente la amplificación de las dianas y la detección mediante sondas fluorescentes tipo TaqMan o Molecular Beacons generalmente. Estas sondas incorporan un fluorocromo y un inhibidor que se encuentran muy próximos, de manera que cuando hay unión con la diana amplificada se produce la separación y se emite una luz que puede ser detectada y que es proporcional a la cantidad de microorganismo que había en la muestra. La irrupción de la PCR a tiempo real ha supuesto un gran avance en el diagnóstico microbiológico, incluidas las micobacterias, reduciendo los problemas de contaminación al realizarse todo el ensayo en un solo tubo que no hay que manipular, aumentando la sensibilidad y con la ventaja de poder obtener una cuantificación del microorganismo estudiado en la muestra.
Cobas Taqman MTB (Roche Diagnostics) es una adaptación del Cobas Amplicor MTB de la misma casa, visto anteriormente (amplifica ARNr 16S), que parece haber aumentado su especificidad con esta técnica.
Fluorotype MTB (Hain Lifescience), aparecido en 2011, tiene como diana a IS6110, puede usarse también en muestras extrapulmonares y el resultado es analizado por el programa que evalúa la fluorescencia emitida a distintas temperaturas, reconociendo la correspondiente a MTB. Varios estudios le han reconocido una sensibilidad del 100% en muestras baciloscopia positiva y entre el 55-85% en baciloscopia negativa24.
También utilizan como diana IS6110 el Abbott Real Time MTB, con igual sensibilidad que el anterior en baciloscopia positiva y algo superior en baciloscopia negativa, si bien solo está indicado en muestras respiratorias. Está disponible otro test de la misma marca (Abbott RealTime MTB RIF/INH Resistance), que también detecta resistencia a INH y Rif.
Anyplex plus MTB/NTM/DR-TB (Seegene Inc., Corea), no solo diferencia entre MTB y MNT, ya que, si es positivo, en un segundo ensayo (de solo 40min), puede detectar resistencias a INH y Rif.
Por último cabe mencionar el Xpert MTB/RIF (Cepheid), que es un sistema totalmente automatizado, sin manipulación, salvo la introducción de la muestra en el cartucho, y que detecta simultáneamente la presencia de MTB y la resistencia a Rif. Como inconveniente cabe destacar que es más caro que otros sistemas, si bien tiene un acuerdo de bajo precio para países con recursos limitados. En el año 2011, tras una evaluación de su efectividad, la OMS lo recomendó como primer paso diagnóstico en países con alta incidencia de TB-MDR25.
Identificación a partir de cultivos positivosLa rapidez en la identificación de las cepas que se obtienen por cultivo es igualmente importante para discriminar MTB de las que no lo son. Actualmente disponemos de 2 métodos muy rápidos no basados en amplificación molecular:
Inmunocromatografía. Hay varias pruebas comerciales basadas en la detección del antígeno MPT64 que es una proteína excretada por la casi totalidad de MTBc (excepto si presentan mutaciones del gen mpt64), y salvo algunos M. bovis BCG. La prueba puede realizarse a partir de medio líquido directamente, o a partir de medio sólido utilizando un tampón de extracción. El dispositivo, tipo «jabonera», lleva fijados anticuerpos anti-MPT64 y aparece una línea de color en menos de 15min a partir de añadir la suspensión bacteriana. La sensibilidad y especificidad están muy cercanas al 100%.
Espectrometría de masas. En los últimos años, la identificación de los cultivos microbiológicos en general ha experimentado una auténtica revolución, pasando de los tradicionales métodos bioquímicos a la espectrometría de masas mediante el MALDI-TOF (matrix-assisted laser desorption/ionization-time of flight). En el campo de las micobacterias, se ha tardado algo más en incorporar esta técnica debido a los distintos protocolos no estandarizados, y a los peores resultados. Sin embargo, poco a poco se ha producido una mejora, tanto en la técnica como en las bibliotecas de bases de datos de los sistemas, que están haciendo que cada vez se empleen con mayor frecuencia, y estudios recientes indican que su concordancia con los métodos genéticos es muy alta26. Con seguridad, en poco tiempo será uno de los métodos más utilizados para identificar micobacterias de forma rápida y fiable en aquellos laboratorios que puedan disponer del espectrómetro.
Técnicas molecularesSondas de ácidos nucleicos. Fue la primera prueba basada en detección de ácidos nucleicos de la que se dispuso. Consiste en diseñar sondas fluorescentes de ADN complementario al ARNr de la micobacteria que queramos identificar, y al realizarse la unión, se produce una reacción quimioluminiscente. Se puede utilizar tanto para medio líquido como sólido, y su sensibilidad y especificidad son muy buenas. No se realiza amplificación genética ya que el ARN es obtenido sonicando la suspensión bacteriana, por lo que no necesita medios técnicos complejos y el resultado se obtiene en menos de 2h.
Existen sondas para MTBc y para las MNT más importantes en la clínica como Mycobacterium avium-intracellulare, Mycobacterium kansasi, Mycobacterium gordonae, etc., y su mayor inconveniente es que es necesario tener una orientación de la micobacteria de la que se trata, para poder elegir la sonda adecuada, y en caso de que el resultado sea negativo, hay que probar con otra.
Amplificación genética. Estas pruebas consisten en amplificar los ácidos nucleicos de determinadas regiones genéticas y analizarlos posteriormente. La amplificación se realiza mediante PCR del extracto de ADN obtenido del cultivo sólido o líquido, y el producto final puede ser estudiado de diferentes maneras:
PRA (PCR-RFLP del gen hsp65). Es una técnica relativamente sencilla, económica y rápida, basada en la amplificación de dicho gen, cuya secuencia presenta zonas conservadas y otras variables, con lo que pueden diferenciarse distintas especies. Una vez amplificado, se somete a una digestión enzimática y posterior electroforesis, analizándose el patrón de bandas. El patrón de bandas resultante es característico para las diferentes especies de micobacterias.
Secuenciación ADNr 16S. La secuenciación del ADNr del 16S, tras amplificación por PCR, ha supuesto un vuelco en el sistema de taxonomía bacteriana en los últimos años, permitiendo establecer las relaciones filogenéticas que existen entre los distintos microorganismos, dando lugar a un nuevo sistema de clasificación, y a la posibilidad de una identificación absolutamente precisa de los mismos. Es una técnica compleja y cara, por lo que su uso está limitado a laboratorios especializados, aunque se está trabajando en aplicaciones más rápidas y económicas como la pirosecuenciación, que hace esta técnica mucho más asequible.
Hibridación en fase sólida. Como ya hemos mencionado anteriormente, las técnicas de hibridación en fase sólida enfrentan el producto de la amplificación genética a unas tiras de nitrocelulosa en las que están fijadas unas sondas específicas marcadas. Tras el revelado por reacción enzimática, obtenemos un patrón de bandas que nos indica a qué especie micobacteriana corresponde. Existen 2 sistemas comerciales en el mercado: el INNO-LIPA MYCOBACTERIA (Innogenetics, Bélgica) y el Geno-Type (GT) Mycobacterium (Hain Lifescience, Alemania). En el primero de ellos, la amplificación se realiza sobre el espacio intergénico 16S-23S, y la tira de nitrocelulosa tiene 22 sondas que permiten identificar el complejo MTB y otras 16 MNT, de relevancia clínica. El GT Mycobacterium amplifica el gen 23SrRNA y existen 3 diferentes formatos. El GT MTBC identifica las subespecies del MTBc, el GT CM identifica MTBc y 13 especies de MNT, y el GT AS identifica 16 especies más. Aunque estos ensayos están validados para estudio de cepas, es posible realizarlos en muestra directa con baciloscopia positiva.
Existen también técnicas de hibridación en fase sólida en pocillos (microarrays), que permiten un número mayor de sondas, pero en la actualidad no están suficientemente desarrollados para su comercialización en la clínica.
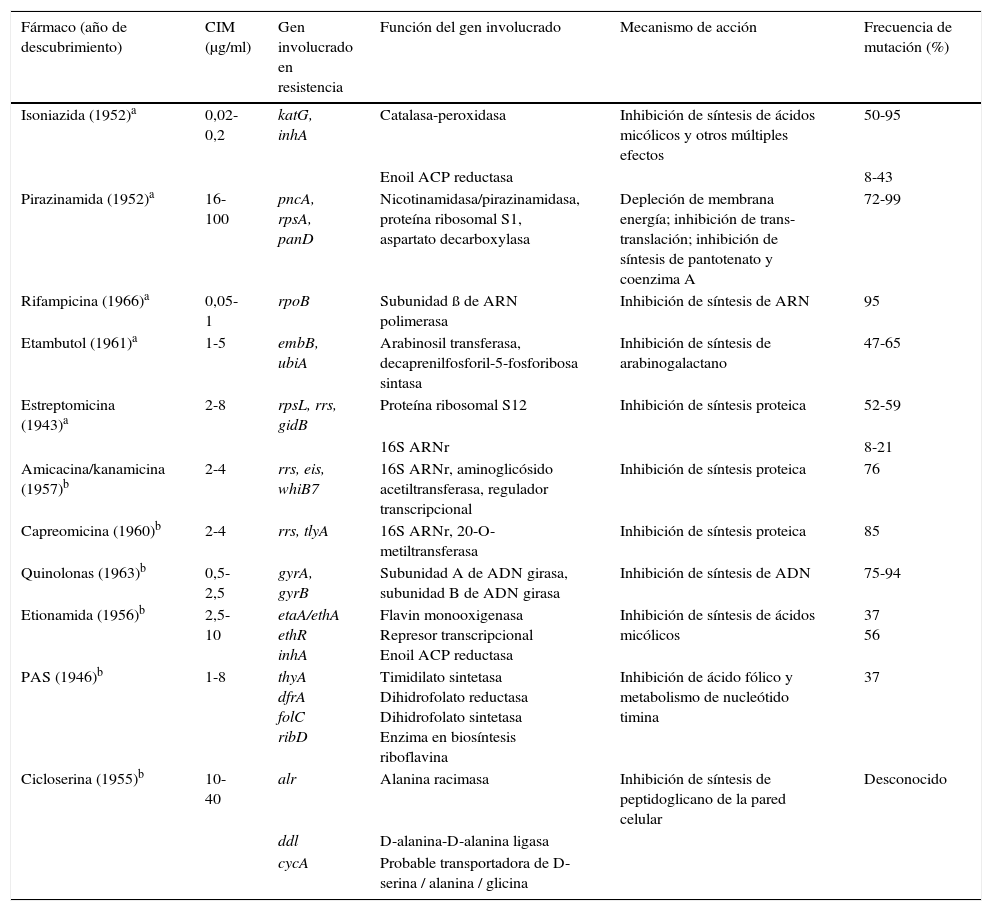
Estudio de los mecanismos que confieren resistenciaExisten algunos métodos para determinar la sensibilidad de las micobacterias que son más rápidos que el tradicional método de las proporciones, y que están recomendados por la OMS en aquellos lugares que no pueden disponer de la tecnología necesaria, como son la observación microscópica directa de la sensibilidad a los fármacos (MODS, por su acrónimo en inglés), las pruebas con indicador colorimétrico redox (CRI) o el ensayo de la nitrato reductasa (NRA). Sin embargo, el mayor impulso en este campo ha sido, sin lugar a dudas, el desarrollo de la detección de los mecanismos de resistencia mediante técnicas moleculares recomendadas por la OMS de manera universal. En esta sección resumimos los eventos moleculares que se han seleccionado en la evolución de las cepas clínicas de MTB para la adquisición de resistencias en el curso de las infecciones y brotes. El mecanismo de resistencia a fármacos de novo entre los miembros del MTBc se debe a la adquisición de mutaciones individuales, a diferencia de otras bacterias en las que el mecanismo de adquisición de resistencia es principalmente por transferencia horizontal de casetes con genes de resistencia. Desde hace varios años se está trabajando para determinar las mutaciones que causan la resistencia a los fármacos antituberculosos. La tabla 1 resume las bases genéticas de la resistencia de MTBc frente a los fármacos de primera y segunda línea.
Mecanismos de resistencia en Mycobacterium tuberculosis frente a fármacos de primera y segunda línea
| Fármaco (año de descubrimiento) | CIM (μg/ml) | Gen involucrado en resistencia | Función del gen involucrado | Mecanismo de acción | Frecuencia de mutación (%) |
|---|---|---|---|---|---|
| Isoniazida (1952)a | 0,02-0,2 | katG, inhA | Catalasa-peroxidasa | Inhibición de síntesis de ácidos micólicos y otros múltiples efectos | 50-95 |
| Enoil ACP reductasa | 8-43 | ||||
| Pirazinamida (1952)a | 16-100 | pncA, rpsA, panD | Nicotinamidasa/pirazinamidasa, proteína ribosomal S1, aspartato decarboxylasa | Depleción de membrana energía; inhibición de trans-translación; inhibición de síntesis de pantotenato y coenzima A | 72-99 |
| Rifampicina (1966)a | 0,05-1 | rpoB | Subunidad ß de ARN polimerasa | Inhibición de síntesis de ARN | 95 |
| Etambutol (1961)a | 1-5 | embB, ubiA | Arabinosil transferasa, decaprenilfosforil-5-fosforibosa sintasa | Inhibición de síntesis de arabinogalactano | 47-65 |
| Estreptomicina (1943)a | 2-8 | rpsL, rrs, gidB | Proteína ribosomal S12 | Inhibición de síntesis proteica | 52-59 |
| 16S ARNr | 8-21 | ||||
| Amicacina/kanamicina (1957)b | 2-4 | rrs, eis, whiB7 | 16S ARNr, aminoglicósido acetiltransferasa, regulador transcripcional | Inhibición de síntesis proteica | 76 |
| Capreomicina (1960)b | 2-4 | rrs, tlyA | 16S ARNr, 20-O-metiltransferasa | Inhibición de síntesis proteica | 85 |
| Quinolonas (1963)b | 0,5-2,5 | gyrA, gyrB | Subunidad A de ADN girasa, subunidad B de ADN girasa | Inhibición de síntesis de ADN | 75-94 |
| Etionamida (1956)b | 2,5-10 | etaA/ethA ethR inhA | Flavin monooxigenasa Represor transcripcional Enoil ACP reductasa | Inhibición de síntesis de ácidos micólicos | 37 56 |
| PAS (1946)b | 1-8 | thyA dfrA folC ribD | Timidilato sintetasa Dihidrofolato reductasa Dihidrofolato sintetasa Enzima en biosíntesis riboflavina | Inhibición de ácido fólico y metabolismo de nucleótido timina | 37 |
| Cicloserina (1955)b | 10-40 | alr | Alanina racimasa | Inhibición de síntesis de peptidoglicano de la pared celular | Desconocido |
| ddl | D-alanina-D-alanina ligasa | ||||
| cycA | Probable transportadora de D-serina / alanina / glicina |
ACP: proteína transportadora de acilo; CIM: concentración mínima inhibitoria; PAS: ácido para-aminosalicílico.
Fuente: Zhang y Yew27.
La rifampicina es un gran bactericida que, además de interferir con la síntesis del ARN por unirse a la subunidad β de la ARN polimerasa, se ha descrito que induce la formación de radicales hidroxilo en las cepas sensibles, contribuyendo a aumentar su poder bactericida28. La resistencia a Rif en más de un 96% está causada por mutaciones en una región de 81-pb del gen rpoB (RRDR, codones 428 a 456 o 507 a 533 referidos a H37Rv o Escherichia coli respectivamente), que codifica la subunidad β de la ARN polimerasa29. Estas mutaciones en la proteína esencial RpoB provocarían una reducción del fitness y se asociarían a una menor transmisibilidad de la bacteria de un huésped a otro. Sin embargo, se sugiere que una cepa resistente a Rif puede tener éxito en su transmisión, así como una concentración mínima inhibitoria (CIM) alta si es capaz de generar mutaciones compensatorias eficientes que le permitieran eludir o sortear este coste, lo que explicaría que algunas cepas MDR se transmiten mejor que otras30. Casali et al. encontraron que el 97% de las cepas Beijing que producían los casos de TB-MDR en la población rusa tenían la mutación más frecuente en rpoB, S450L (S531L referido a E. coli), y tenían mutaciones compensatorias en los genes rpoA o rpoC, o en el propio gen rpoB31. Meftahi et al. también lo describen para una cepa MDR Haarlem responsable de un brote en Túnez, que tenía una segunda mutación en codón V534M confirmando con pruebas fenotípicas que esta mutación compensaba el fitness perdido además de aumentar el nivel de resistencia a Rif32. Mutaciones en rpoB generalmente se asocian a resistencia a todas las rifamicinas, incluida la rifabutina, sin embargo, las mutaciones 514, D516V y S522L se han descrito asociadas a resistencia a Rif y sensibilidad a rifabutina. Así mismo se han encontrado mutaciones en cepas sensibles, por ejemplo 510H, L511P, D516Y, N518D, H526N y L533P33,34, lo que podría causar errores en las técnicas moleculares si no se consideran. La monorresistencia a Rif es rara, generalmente se asocia a resistencia a INH; este hecho, junto con el alto rendimiento de su detección molecular, hace que se utilice como un buen marcador de TB-MDR1. La tabla 2 muestra las mutaciones más frecuentes y consistentes con resistencia para los fármacos de primera línea.
Resistencia a fármacos de primera línea
| Fármaco | Nom. gen | Codón afectado |
|---|---|---|
| Isoniazida | katG | Ser315Thr/Asn/Ile/Gly |
| Trp300Gly | ||
| Thr275Pro | ||
| fabG1 | -15 c-t | |
| -8 t-a / t-c | ||
| inhA | Ser94Ala | |
| Rifampicina | rpoB | Val170Phe |
| Gln432Leu | ||
| Asp435Tyr/Gly/Val | ||
| His445Tyr/Asn/Asp/Pro/Arg | ||
| Ser450Leu/Trp | ||
| Leu452Pro | ||
| Ile491Phe | ||
| Pirazinamida | pncA | Más de 100 posiciones diferentes incluida la región promotora |
| Etambutol | embB | Met306Val/Leu/Thr/Ile |
| Gly406Ser/Cys/Ala/asp | ||
| Gln497Arg | ||
| Estreptomicina | rpsL | Lys43Arg |
| Lys88Arg |
En negrita se señalan las mutaciones que se presentan con mayor frecuencia asociadas a la resistencia del correspondiente fármaco.
La resistencia de alto nivel a isoniacida se debe principalmente a mutaciones en el gen catalasa-peroxidasa KatG, que cataliza la oxidación del profármaco a su forma activa35. La resistencia de bajo nivel se asocia con mutaciones en el gen inhA, que codifica una enoil-acil-portador-proteína reductasa NADH-dependiente, diana del fármaco, involucrada en la síntesis de ácidos micólicos36. Se han descrito otros genes relacionados con la resistencia a INH, como ahpC, y mutaciones que podrían ser compensatorias a la pérdida de la actividad catalasa37.
La pirazinamida (PZ) es un importante fármaco esterilizante que acorta la terapia de la TB. Sin embargo, el mecanismo de acción de la PZ es poco conocido. El ácido pirazinoico, principio activo de la PZ, altera la energía de la membrana e inhibe la función de transporte de la membrana en MTB. La actividad preferencial de la PZ frente a bacilos en estado no replicativo se correlaciona con su bajo potencial de membrana y la disrupción del potencial de membrana por el ácido pirazinoico y el pH ácido38. Mutaciones a lo largo del gen pncA se han descrito como causa de resistencia a PZ. M. bovis se caracteriza por una mutación en el aminoácido 57 del gen pncA, que además le convierte en resistente a PZ de forma natural. También podrían estar involucrados el gen rpsA en casos de bajo nivel de resistencia y panD. De hecho, Mycobacterium canettii presenta una mutación en panD que parece ser le confiere resistencia intrínseca a PZ27.
El etambutol (EMB) interfiere con la síntesis del arabinogalactano de la pared celular. Tiene como diana la enzima arabinosil transferasa, y mutaciones en el operón que la codifica embCAB pueden causar resistencia a EMB. La mutación más frecuente se da en el codón 306 de embB, llegando hasta el 68% de los casos39.
Entre los aminoglucósidos utilizados se encuentran SM, AK, KM y CPM. La SM inhibe la síntesis proteica uniéndose a la subunidad ribosomal 30S y causando así alteraciones en la lectura del ARN mensajero. La resistencia está causada por mutaciones en el gen rpsL que codifica para la proteína 12S y en rrs que codifica para ARNr 16S, que suponen el 50 y el 20% de las resistencias a SM respectivamente. Se han descrito mutaciones en gidB en cepas resistentes a SM y se ha visto resistencia cruzada con SM y KM en la región promotora de whyB7. Mutaciones en la posición 1400 de rrs se han descrito como causantes de resistencia a KM y AK. Mutaciones en la región promotora de eis causan bajo nivel de resistencia a KM pero no a AK27. Mutaciones en tlyA y en rrs (A1401G y G1484T) están relacionadas con resistencia a CPM y KM. Múltiples mutaciones pueden darse en el gen rrs y producir resistencia entre estos fármacos.
El mecanismo de acción de las fluoroquinolonas es la inhibición de la topoisomerasa II (ADN girasa), codificada por los genes gyrA y gyrB. Mutaciones en el gen gyrA, con mayor frecuencia en los codones 94, 90 y 88, están asociadas a resistencia. Mutaciones en gyrB pueden dar resistencia de bajo nivel, y mutaciones dobles en gyrA y gyrB pueden dar un mayor nivel de resistencia. Un hallazgo interesante es que la concurrencia de 2 mutaciones T80A y A90G en gyrA hacen a la cepa hipersensible a la mayoría de quinolonas40, lo que puede hacer más complejo el estudio de estas resistencias41.
La cicloserina actúa inhibiendo la síntesis del peptidoglicano de la pared celular, si bien los mecanismos de resistencia todavía no están bien definidos.
El ácido para-aminosalicílico tiene un mecanismo de acción no bien conocido, que podría interferir con la biosíntesis del ácido fólico. Se han descrito mutaciones en los genes thyA en el 40% de los casos y también en los genes folC y dfrA42.
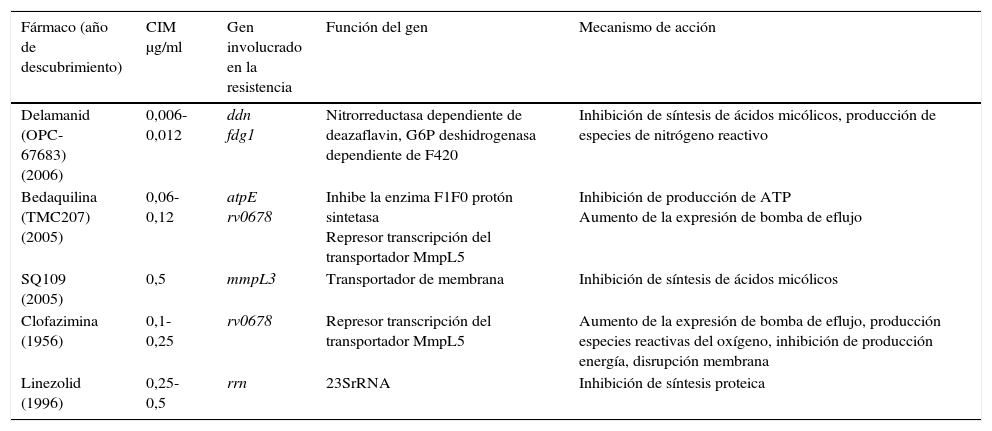
Entre los nuevos fármacos propuestos para el tratamiento de TB, la bedaquilina inhibe la adenosín trifosfato (ATP) sintasa, siendo activa frente a población en crecimiento y no crecimiento. En el modelo de ratón, parece tener efecto sinérgico con la PZ. Las resistencias que se han descrito estarían en la atpE y en el gen Rv0678 que sobreexpresaría la bomba de eflujo MmpL5 provocando resistencia cruzada de clofacimina y bedaquilina. El delamanid inhibe la síntesis de ácidos micólicos, debe ser activado por una enzima nitrorreductasa, Ddn, y tiene actividad frente a los bacilos en estado no replicativo. Se ha asociado su resistencia a mutaciones en los genes fgd1, ddn, fbiA, fbiB y fbiC. Las oxazolidonas, y entre ellas el linezolid, inhiben la síntesis proteica uniéndose a la subunidad 50S del ribosoma y la resistencia se asocia principalmente a mutaciones en el gen 23SrRNA. SQ109 es un análogo del EMB pero con un mecanismo de acción diferente, que tiene como diana la proteína MmpL3, imposibilitando la síntesis de ácidos micólicos de la pared celular. Mutaciones en el gen mmpL3 se han asociado con resistencia a SQ10927 (tabla 3).
Mecanismos de resistencia de los nuevos fármacos o agentes propuestos frente a Mycobacterium tuberculosis
| Fármaco (año de descubrimiento) | CIM μg/ml | Gen involucrado en la resistencia | Función del gen | Mecanismo de acción |
|---|---|---|---|---|
| Delamanid (OPC-67683) (2006) | 0,006-0,012 | ddn fdg1 | Nitrorreductasa dependiente de deazaflavin, G6P deshidrogenasa dependiente de F420 | Inhibición de síntesis de ácidos micólicos, producción de especies de nitrógeno reactivo |
| Bedaquilina (TMC207) (2005) | 0,06-0,12 | atpE rv0678 | Inhibe la enzima F1F0 protón sintetasa Represor transcripción del transportador MmpL5 | Inhibición de producción de ATP Aumento de la expresión de bomba de eflujo |
| SQ109 (2005) | 0,5 | mmpL3 | Transportador de membrana | Inhibición de síntesis de ácidos micólicos |
| Clofazimina (1956) | 0,1-0,25 | rv0678 | Represor transcripción del transportador MmpL5 | Aumento de la expresión de bomba de eflujo, producción especies reactivas del oxígeno, inhibición de producción energía, disrupción membrana |
| Linezolid (1996) | 0,25-0,5 | rrn | 23SrRNA | Inhibición de síntesis proteica |
ATP: adenosín trifosfato; CIM: concentración mínima inhibitoria.
Fuente: Zhang y Yew27.
Existen varias bases de datos de libre acceso que recopilan los polimorfismos asociados a resistencia a fármacos de primera y segunda línea, como son https://tbdreamdb.ki.se/Info/43 y https://umr5558-bibiserv.univ-lyon1.fr/mubii/mubii-select.cgi.
Detección rápida de los genes implicados en resistenciaLos métodos de detección son de 2 tipos fundamentalmente, los basados en amplificación mediante PCR e hibridación en fase sólida, y los basados en PCR a tiempo real.
Hibridación en fase sólida. Como ya mencionamos en la identificación de muestra directa, hay 2 pruebas disponibles, el INNO-LIPA1 Rif.TB y el Genotype, del que tenemos 3 versiones: MTBDRplus, MTBDRsl y NTM-DR44. El INNO-LIPA consta de 10 sondas en su tira, una de ellas identifica MTBc, 5 ponen de manifiesto regiones específicas del gen rpoB presentes en las cepas sensibles, y 4 detectan las mutaciones. Tiene una sensibilidad muy alta en cultivos, y, como siempre, menor en muestra directa. Las mutaciones que se pueden poner de manifiesto cubren aproximadamente el 95% de las resistencias confirmadas por antibiograma, y sirve como marcador de multirresistencia (TB-MDR)45 ya que la resistencia aislada a Rif es bastante rara (alrededor de un 5%). El MTBDRplus es similar al anterior, pero también detecta la resistencia a INH mediante el estudio de los genes inhA (que codifica resistencia de bajo nivel) y katG (que codifica resistencia de alto nivel). Tiene, al igual que antes, sondas para las regiones salvajes (sensibles) y sus correspondientes mutaciones, cubriendo el 80-85% de las resistencias a INH. Está recomendado por la OMS desde el año 2008 y puede realizarse tanto sobre muestra directa como sobre cultivo, con magnífica sensibilidad y especificidad.
De más reciente aparición son el MTBDRsl, que busca detectar resistencias a fármacos de segunda línea como CP, AK y KM (gen rrs), fluoroquinolonas (gen gyrA) y a EMB (gen embB), con buena sensibilidad para quinolonas, algo peor para aminoglucósidos y más baja aún para EMB. Existe una segunda versión que incluye el estudio del gen eis (que mejora el rendimiento de la KM, aunque empeora la especificidad de los inyectables), y el del gen gyrB para las quinolonas, desapareciendo el EMB.
El NTM-DR identifica algunas especies de MNT (M. avium, M. intracellulare, Mycobacterium quimaera, Mycobacterium chelonae y Mycobacterium abscessus) y detecta mutaciones en los genes rrs para aminoglucósidos, y rrl para macrólidos, así como la presencia del gen erm en M. abscessus (resistencia inducible a macrólidos).
PCR a tiempo real. Como ya se ha comentado, la PCR a tiempo real tiene grandes ventajas, por lo que también está irrumpiendo en la detección de resistencias de las micobacterias. Ya hemos visto que el test Anyplex plus MTB/NTM/DR-TB, en un segundo paso, una vez que ha resultado positivo para MTB, puede detectar mutaciones de resistencia. En concreto estudia 4 mutaciones para el gen katG y 3 para el inhA (resistencia a INH), 18 mutaciones del gen rpoB (resistencia a Rif), 7 mutaciones del gyrA (quinolonas) y 6 mutaciones para fármacos inyectables (3 para el gen rrs y 3 para el promotor eis). Su sensibilidad y especificidad ha sido recientemente comparada en cultivos con MTBDRplus y MTBDRsl siendo equiparable, salvo para aminoglucósidos, que ha sido ligeramente superior46. El Abbott RealTime MTB RIF/INH también detecta las resistencias mediadas por los mismos genes (katG e inhA para INH y rpoB para Rif) con muy buena sensibilidad y especificidad, habiendo recibido recientemente el marcado CE-IVD.
Por último hay que mencionar el Xpert MTB/RIF, que como vimos ha sido recomendado por la OMS en 2010. Estudia las mutaciones del gen rpoB, y según la revisión Cochrane47 tiene una sensibilidad acumulada del 95% y una especificidad del 98%. Su rapidez y extrema sencillez, al estar todo el proceso integrado en un cartucho que no requiere manipulación, hace que sea muy atractivo, habiendo sido impulsado por la OMS como herramienta para diagnosticar rápidamente tanto la enfermedad como las cepas resistentes en las zonas con altas tasas de ambas. Gracias a un acuerdo comercial con los países con bajos ingresos (la mayoría de los que tienen esas altas incidencias), se dispensa a un precio reducido, habiéndose distribuido millones de cartuchos. En los países desarrollados, el mayor inconveniente es el precio, aunque en un estudio muy reciente se expone que su uso en todos los pacientes resulta coste-efectivo48.
La secuenciación del genoma completo (WGS) de MTB para la detección de resistencia a los fármacos antituberculosos de primera y segunda línea se ha utilizado ya en distintas ocasiones. En una revisión reciente sobre el tema, se ha determinado la sensibilidad, la especificidad, el valor predictivo positivo y el valor predictivo negativo de WGS utilizando los métodos de ensayo de sensibilidad a fármacos fenotípicos como patrón de referencia. Concluyendo, WGS puede considerarse una alternativa prometedora a los métodos de ensayo de sensibilidad fenotípica y molecular existentes para Rif e INH en espera de la estandarización de los procedimientos analíticos49. También recientemente se ha publicado una biblioteca con 1.325 polimorfismos basados en la secuenciación genómica que serían predictivos de resistencia a AM, CP, EMB, etionamida, INH, KM, moxifloxacino, ofloxacina, PZ, Rif y estreptomicina50.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.