Introducción
Los macrólidos, las lincosamidas y las estreptograminas de tipo B (antibióticos MLSB) son antibióticos químicamente distintos pero que muestran un mecanismo de acción similar, inhibiendo la síntesis proteica de las bacterias al unirse al sitio P en la subunidad 50S del ribosoma bacteriano y se usan habitualmente en el tratamiento de las infecciones estafilocócicas 1,2.
La resistencia de los estafilococos a estos antibióticos, se debe principalmente a dos mecanismos: bombas de expulsión activa del antibiótico y modificación del sitio de unión del antibiótico por metilación 1. En el caso de las bombas de expulsión, existen dos tipos (ABC y MSF) localizadas en la superficie citosólica de la membrana, el sistema de expulsión es específico para macrólidos de 14-15 átomos y estreptograminas tipo B. La clindamicina no es sustrato de la expulsión por estas bombas. Las cepas con este mecanismo presentarían bajos niveles de resistencia a eritromicina y estreptogramina del tipo B pero no a clindamicina. Este es el llamado fenotipo MSB y está codificado por el gen msrA 2.
La modificación ribosómica, también llamada resistencia MLSB, se debe a la acción de una enzima metilasa codificada por una variedad de genes erm, siendo éste el mecanismo de resistencia más común y que además puede conducir a resistencia cruzada a macrólidos, lincosamidas y estreptograminas B 1,2. La resistencia MLSB puede ser constitutiva, cMLSB, con alto nivel de resistencia cruzada a todos estos agentes, o inducible, iMLSB, con resistencia a los macrólidos de 14 y 15 átomos (p. ej., eritromicina) y sensibilidad in vitro a macrólidos de 16 átomos, lincosamidas y estreptograminas tipo B, pero in vivo el uso de clindamicina como tratamiento puede seleccionar mutantes con resistencia constitutiva 1,3,4.
En la mayoría de los laboratorios de microbiología clínica la sensibilidad a los antibióticos está semiautomatizada basándose en métodos de microdilución en caldo que no permiten detectar la resistencia inducible a clindamicina 5.
El Clinical and Laboratory Standards Institute (CLSI) recomienda el método de difusión con doble disco para la detección de la resistencia inducible a clindamicina en Staphylococcus spp. 6,7, debido a que la utilización de clindamicina en pacientes con infección por estafilococos con este tipo de resistencia podría conducir a un posible fracaso terapéutico 4,8,9 por la aparición de resistencia a clindamicina durante el tratamiento 10.
La clindamicina es un antibiótico de elección para el tratamiento de las infecciones de piel y tejidos blandos por estafilococos debido a su buena absorción oral, excelente penetración y a que no necesita ajustarse la dosis en insuficiencia renal. Es una opción terapéutica por vía oral en pacientes ambulatorios así como para la continuación de una terapia intravenosa. Así mismo, la clindamicina es el tratamiento alternativo en pacientes alérgicos a penicilina 3,8,10.
Nuestro objetivo fue determinar la incidencia de la resistencia a macrólidos y lincosamidas en aislados clínicos de estafilococos procedentes de muestras cutáneas mediante métodos fenotípicos y genotípicos y correlacionar los niveles de resistencia antibiótica con los mecanismos de resistencia genéticos.
Métodos
Cepas
Se estudiaron 117 estafilococos resistentes a eritromicina [57 Staphylococcus aureus y 60 estafilococos coagulasa negativos (ECN)] procedentes de muestras cutáneas que fueron seleccionados a partir de 536 aislados clínicos de estafilococos (401 S. aureus y 135 ECN) aislados entre diciembre de 2003 y junio de 2004, en los Hospitales Universitarios Virgen del Rocío de Sevilla. La caracterización fenotípica de los aislados se realizó mediante la tinción de Gram, la actividad de la catalasa y la prueba de la coagulasa, determinada esta última por una prueba de aglutinación con látex con el sistema Pastorex Staph Plus (Sanofi Diagnostic Pasteur, Marnes la Coquette, Francia). La identificación y pruebas de sensibilidad se hicieron con el sistema semiautomatizado Walk-Away Microscan® (Dade-Behring).
Se estudió un único aislado por paciente. Todos los aislados fueron congelados a 70 °C para los estudios genotípicos.
Técnica de difusión por doble disco
La técnica fue realizada siguiendo las recomendaciones del CLSI 6,7. Dicha técnica consiste en colocar un disco de eritromicina (15 μg) y otro de clindamicina (2 μg) separados por una distancia de 15 mm de borde a borde en una placa de agar Mueller-Hinton (Becton Dickinson Microbiology Systems, Cockeysville, Md.) que ha sido previamente inoculada con una suspensión (0,5 McFarland) del microorganismo 6,7. Después de 16-18 h de incubación a 35 °C, el achatamiento en la zona de inhibición de la clindamicina próxima al disco de eritromicina (efecto zona D) indica un fenotipo de resistencia inducible (Cr-ind), la resistencia a eritromicina y a clindamicina indica un fenotipo de resistencia constitutivo (Cr-const) y la sensibilidad a clindamicina fue definida por la ausencia de inducción de resistencia a clindamicina en la zona próxima al disco de eritromicina (CS). Se realizó una segunda lectura a las 24 h de incubación usando luz transmitida en todos los aislados que mostraron un fenotipo de resistencia inducible para distinguir los diferentes fenotipos de resistencia inducible descritos recientemente por Steward et al 11.
Determinación de la CIM
La concentración inhibitoria mínima (CIM) a eritromicina y clindamicina frente a los estafilococos en estudio y de la cepa control S. aureus ATCC 29213 se determinó por el método de microdilución en caldo de forma manual siguiendo las recomendaciones del CLSI 6,7.
Genotipado
La determinación de la presencia de los genes de resistencia a eritromicina se realizó mediante la reacción en cadena de la polimerasa (PCR) en tiempo real con cebadores específicos para los genes ermA, ermB, ermC y msrA descritos por Martineau et al 12 (tabla 1), usando Faststart DNA MasterPlus SYBR Green I. Para lisar las células y extraer el ADN, se resuspendieron 1 o 2 colonias aisladas en placas de Columbia Agar Sangre en 50 μl de agua destilada estéril, la suspensión se calentó durante 10 min a 95 °C y se centrifugó (14.000 g, 10 min, 4 °C) para eliminar los restos celulares. Una alícuota de 2 μl de este extracto se usó directamente para la realización de la PCR en un volumen final de 20 μl. Las condiciones de la reacción fueron: un primer ciclo de desnaturalización de 95 °C durante 10 min seguido de un protocolo de 30 ciclos de desnaturalización a 95 °C durante 5 s, hibridación a 60 °C durante 5 s y extensión a 72 °C durante 10 s, con monitorización de la fluorescencia al final de la fase de extensión con la polimerasa. A continuación se realizó la curva de fusión de los productos amplificados para determinar su Tm.
Resultados
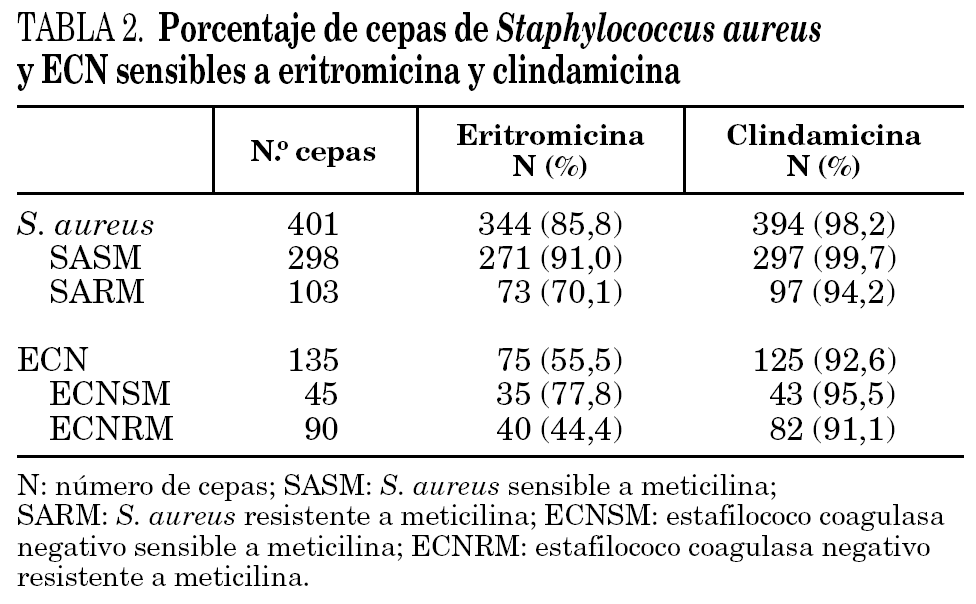
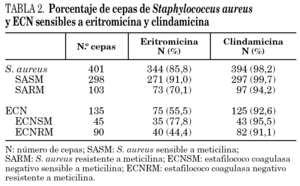
Se estudiaron 536 (100%) aislados clínicos de estafilococos, 401 (74,8%) cepas de S. aureus y 135 (25,2%) de ECN. Un total de 57 (14,2%) cepas de S. aureus fueron resistentes a eritromicina y 7 (1,7%) a clindamicina. Sesenta (44,4%) cepas de ECN fueron resistentes a eritromicina y 10 (7,4%) a clindamicina (tabla 2).
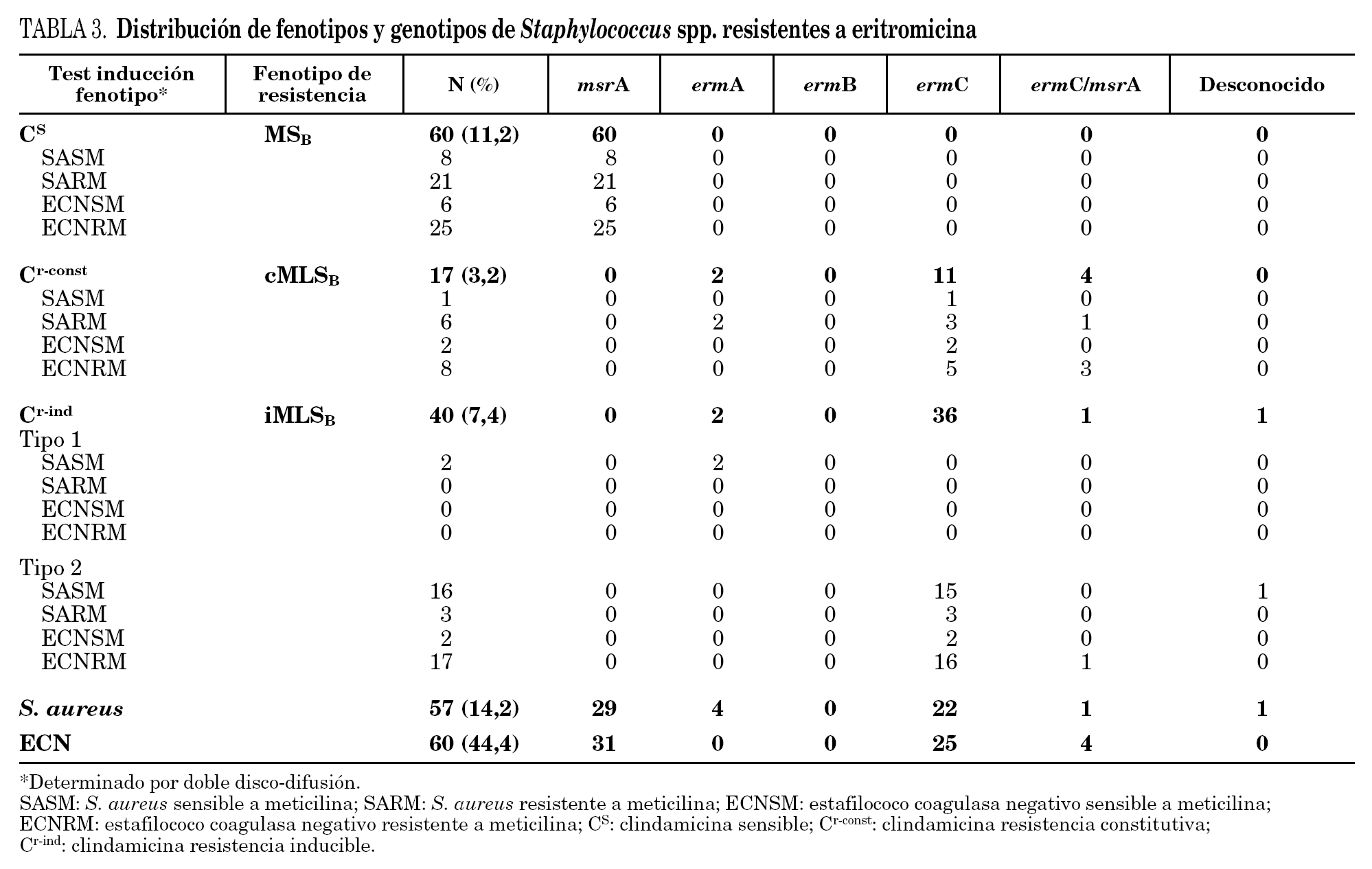
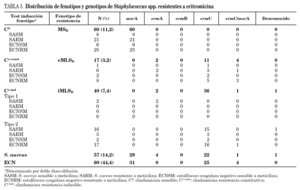
En la tabla 3 se detallan los resultados de las 57 cepas de S. aureus y 60 cepas de ECN resistentes a eritromicina estudiadas. El fenotipo MSB fue el más frecuente en las cepas resistentes a meticilina de S. aureus, y el fenotipo MLSB inducible en sensibles a meticilina (SASM). Se observaron dos tipos diferentes de fenotipos en las cepas de S. aureus con resistencia inducible a clindamicina. El fenotipo tipo 2, caracterizado por la presencia de pequeñas colonias dentro de la zona de inhibición, se observó en 19 cepas de S. aureus. Sólo se encontró en 2 cepas de SASM el fenotipo tipo 1, caracterizado por la ausencia de colonias dentro de la zona de inhibición.
El fenotipo MSB fue el más común en las cepas de ECN, tanto en las cepas sensibles como en las resistentes a meticilina.
En el 99,2% de las 117 cepas estudiadas se detectó la presencia de algunos de los genes implicados en la resistencia a macrólidos. El gen mrsA se detectó en todos los aislados con fenotipo de bomba de expulsión (MSB). En el resto de las cepas, con fenotipo MLSB (constitutivo o inducible), fue el gen ermC el más frecuentemente detectado tanto en las cepas de S. aureus como en las cepas de ECN.
La mayoría de los aislamientos de estafilococos poseían un solo mecanismo de resistencia, excepto en 5 cepas en las cuales se detectaron dos mecanismos codificados por los genes msrA y ermC.
En un aislado de S. aureus no se obtuvo producto de amplificación con ninguno de los genes estudiados.
La CIM90 a eritromicina frente a las cepas de S. aureus y ECN con fenotipo Cs fue de 64 μg/ml, un nivel de resistencia menor en comparación con la CIM90 / 4 1.024 μg/ml, indicativa de un alto nivel de resistencia, encontrada en la cepas de S. aureus y SCN con fenotipo Cr-ind. La CIM90 a clindamicina frente a los aislados de ECN y S. aureus con fenotipo de resistencia Cs, fue de 0,25 y 0,125 μg/ml, respectivamente. Son destacables los bajos valores de CIM90 a clindamicina encontrados en los aislados de ECN y S. aureus con el fenotipo Cr-ind, que en todos los casos fue inferior o igual a 0,5 μg/ml.
Los aislados con fenotipo de resistencia Cr-const, mostraron un alto nivel de resistencia CIM90 de 1.024 mg/ml tanto a eritromicina como a clindamicina.
Discusión
La prevalencia de la resistencia a macrólidos y lincosamidas varía ampliamente entre distintos hospitales y áreas geográficas 5,13,14. Los resultados obtenidos en nuestro estudio presentan diferencias con los resultados de otros autores 15,16. Hamilton-Miller et al 16, en Londres, estudiaron 540 cepas de estafilococos (210 S. aureus y 330 ECN) procedentes de distintas muestras clínicas y encontraron que el fenotipo más común fue el iMLSB, mientras que el fenotipo MSB solamente estuvo presente en el 1% de los S. aureus y en el 13% ECN de los aislados estudiados, a diferencia del los resultados obtenidos en nuestro estudio, en el que el fenotipo MSB fue el más frecuente. Además, en nuestro estudio, el fenotipo iMLSB, se encontró con mayor frecuencia que el fenotipo cMLSB (tabla 3).
Recientemente, en Turquía, Delialioglu et al 15 estudiaron 521 cepas de estafilococos (230 S. aureus y 291 ECN) procedentes de distintas muestras clínicas y encontraron que el 24,3% de los S. aureus y el 40,2% de los ECN estudiados presentaban el fenotipo cMLSB, mientras que el 7,8% de los S. aureus y 14,7% de los ECN tenían un fenotipo iMLSB y que ninguna de las cepas de S. aureus y un 18,2% de las cepas de ECN mostraba el fenotipo MSB, siendo la frecuencia de los fenotipos totalmente opuesto al encontrado en nuestro estudio.
Jenssen et al 17 describieron en 1987 dos tipos distintos de fenotipos de resistencia inducible MLSB y encontraron el fenotipo tipo 1 en el 14,6% de S. aureus y en el 4% de los ECN resistentes a eritromicina estudiados, correlacionando este fenotipo con la presencia del gen ermA 17,18. Nuestro estudio demostró una tasa baja de cepas con el fenotipo tipo 1. Ninguna de las 19 cepas de ECN y solamente 2 de las cepas de S. aureus con el fenotipo de resistencia iMLSB presentaban el fenotipo tipo 1, detectando el gen ermA en estas 2 cepas. Recientemente, Steward et al 11 denominaron al tipo 1 "fenotipo D", lo encontraron en el 21,6% de los S. aureus resistentes a eritromicina estudiados y todas estas cepas contenían el gen ermA.
Hemos encontrado que el 14,1% de los ECN y en el 4,7% de los S. aureus con fenotipo iMLSB, presentaban el fenotipo tipo 2 y la mayoría de estas cepas contenían el gen ermC coincidiendo con los resultados previamente descritos 10,17. Desde el punto de vista clínico, no existen diferencias entre los fenotipos tipo 1 y tipo 2, ya que ambos deben ser considerados e informados como fenotipo de resistencia inducible MLSB; sin embargo, la detección de estos fenotipos puede ser útil en estudios epidemiológicos.
El gen más frecuentemente detectado en las cepas resistentes a clindamicina, tanto de S. aureus como en los ECN, fue el ermC, a diferencia de los resultados obtenidos en otros estudios 14,19, como un estudio hecho por Lina et al 19 en Francia con 294 cepas de estafilococos resistentes a eritromicina, donde el gen ermC fue detectado sólo en un 25% de las cepas con fenotipo cMLSB.
Hemos detectado cepas que poseían los 2 genes ermC y msrA, siendo este hecho más frecuente en las cepas de estafilococo coagulasa negativo (6, 6%), y en un porcentaje superior a lo previamente descrito 10,19.
Ninguno de nuestros aislados poseía el gen ermB. Este gen ha sido detectado en un pequeño porcentaje de cepas de estafilococos y además exclusivamente en aislados de procedencia animal 16,19,20.
En una cepa con fenotipo iMLSB no se obtuvo producto de amplificación con ninguno de los genes estudiados. Esta cepa podría poseer un mecanismo distinto de resistencia a eritromicina, como una mutación en el ARN 23 S ya descrito in Streptococcus pneumoniae 21.
Muchos autores sugieren que la clindamicina no debiera utilizarse en el tratamiento de las infecciones por estafilococos con resistencia inducible a clindamicina, ya que pueden aparecer mutantes con resistencia constitutiva que determinan el fracaso terapéutico 4,8,9,15.
Actualmente, el CLSI recomienda usar la técnica de difusión por doble disco para detectar la resistencia inducible y sugiere que los aislamientos con este tipo de resistencia deberían ser informados como resistentes a clindamicina o bien con un comentario indicando la resistencia inducible de la cepa y la posibilidad de fracaso terapéutico que puede existir si se usa clindamicina en el tratamiento.
La buena correlación entre los métodos fenotípicos (difusión con discos) y genotípicos permiten inferir el mecanismo de resistencia a eritromicina y clindamicina, instaurar el tratamiento antimicrobiano más adecuado, así como apreciar las diferencias epidemiológicas en su distribución 5,11,13,17.
Correspondencia:
Dr. J. Aznar-Martín.
Servicio de Microbiología. Hospitales Universitarios Virgen del Rocío.
Avda. Manuel Siurot, s/n. 41013 Sevilla. España.
Correo electrónico: javier.aznar.sspa@juntadeandalucia.es
Manuscrito recibido el 23-1-2006; aceptado el 4-7-2006.