La resistencia a linezolid se produce generalmente por mutaciones en el ARNr 23S. El objetivo de este estudio fue caracterizar cepas de Staphylococcus epidermidis (SE) y S. haemolyticus (SH) resistentes a linezolid y meticilina (SE-LMR y SH-LMR, respectivamente) detectadas en un hospital español.
MétodosSe estudiaron todas las cepas SE-LMR y SH-LMR aisladas en el periodo de junio 2009 a agosto 2011, en un hospital de segundo nivel, así como las características epidemiológicas de los pacientes. Se tiparon las cepas (25 SE-LMR y 2 SH-LMR, procedentes de 20 pacientes) y se determinó su fenotipo y genotipo de resistencia y la presencia de genes de virulencia.
ResultadosEn todas las cepas analizadas se detectó la mutación G2603T en el ARNr 23S y también cambios aminoacídicos en las proteínas ribosomales L3 y L4. Las 25 cepas SE-LMR pertenecieron a la secuencia tipo ST2, su SCCmec fue el tipoIII y presentaron 2 patrones diferentes de PFGE. El SCCmec de las 2 cepas SH-LMR fue no tipable. Las cepas SE-LMR contenían los genes de resistencia aac(6’)-aph(2”) y dfrS1, y las cepas SH-LMR poseían además el gen erm(C). No se detectaron genes de resistencia a lincomicina en las cepas SE-LMR a pesar de mostrar sensibilidad disminuida a clindamicina y resistencia a lincomicina.
ConclusionesLa resistencia a linezolid en el ámbito hospitalario es preocupante y requiere una continua vigilancia. Esta resistencia se asoció a la mutación G2603T en el ARNr 23S y a la presencia de cambios aminoacídicos en L3 y/o L4.
Linezolid resistance is mainly due to mutations in the 23S rRNA target. The aim of this study was to characterize linezolid and methicillin resistant Staphylococcus epidermidis (SE-LMR) and S. haemolyticus (SH-LMR) strains detected in a Spanish hospital.
MethodsSE-LMR and SH-LMR strains obtained in the period June 2009-August 2011 in a second level hospital were recorded along with the epidemiological characteristics of the patients. These strains were typed, and their resistance, phenotype, genotype and the factors determining their virulence were analysed.
ResultsLinezolid resistance was explained by the presence of G2603T mutation (23S rRNA) and aminoacid changes in L3 and L4 ribosomal proteins. The 25 SE-LMR strains belonged to sequence type ST2, presented SCCmec typeIII, and two different PFGE patterns. The two SH-LMR strains showed non-typeable SCCmec. SE-LMR strains harboured the resistance genes aac(6’)-aph(2”), and dfrS1. SH-LMR strains contained these genes and the gene erm(C). No lincomycin resistance mechanism was identified in SE-LMR strains regardless of showing lincomycin resistance and diminished susceptibility to clindamycin.
ConclusionsLinezolid resistance is of concern in hospitals, and requires continued vigilance. Several linezolid resistance mechanisms (mutation in 23S RNAr and amino acid changes in L3 and L4) were identified in this study.
Linezolid es el primer antibiótico comercializado del grupo de las oxazolidinonas. Este compuesto se utiliza principalmente para el tratamiento de infecciones graves causadas por patógenos gram positivos que son resistentes a varios grupos de antibióticos, incluyendo cepas de Staphylococcus aureus resistentes a meticilina (SARM). Su mecanismo de acción consiste en la inhibición de la síntesis de proteínas a través de la unión de N-formilmetionil-ARNt al ribosoma, afectando a la subunidad 50S. Desde su aprobación en el año 2000, la resistencia a este antibiótico ha aparecido raramente, siendo más común en estafilococos coagulasa-negativos (ECN)1. Esta resistencia se debe sobre todo a mutaciones en la diana (ARNr 23S)1, aunque también se ha descrito asociada a cambios aminoacídicos, inserciones y deleciones en las proteínas ribosomales L3, L4 y L222. Recientemente se ha reportado un gen (cfr) capaz de conferir resistencia a este antibiótico y a lincosamidas, estreptograminas, fenicoles, pleuromutilinas y oxazolidinonas3.
Los ECN son una causa importante de infecciones hospitalarias, que pueden estar implicados en casos graves y causar brotes nosocomiales, particularmente preocupantes si estos se producen por cepas que presentan resistencia a linezolid4-6. Entre los ECN, Staphylococcus epidermidis (SE) es el más frecuente y el de mayor relevancia y se asocia a infecciones principalmente en pacientes con catéteres intravenosos y otros dispositivos, debido a la capacidad de este microorganismo de producir biofilm7. En cuanto al resto de los ECN, Staphylococcus haemolyticus (SH) es uno de los más importantes en nuestros hospitales5. El objetivo de este estudio fue caracterizar todas las cepas clínicas de ECN resistentes a linezolid detectadas en un hospital español.
Material y métodosCepas estudiadasSe estudiaron todos los aislados de ECN resistentes a linezolid obtenidos en el Hospital Royo Villanova (HRV) de Zaragoza durante 2 años consecutivos (junio 2009-agosto 2011), comenzando con el primer aislado de estas características obtenido en dicho hospital. El HRV es un hospital español de segundo nivel, con 225 camas y una unidad de cuidados intensivos (UCI) polivalente con 10 camas. En este periodo se obtuvieron 25 aislados de SE y 2 de SH resistentes a linezolid, los cuales presentaron también resistencia a meticilina (denominados respectivamente SE-LMR y SH-LMR). Estos 27 aislados procedían de 20 pacientes no relacionados, y fueron caracterizados en este estudio.
Se recogieron los siguientes datos de los 20 pacientes: edad, sexo, servicio de procedencia, tratamiento previo con linezolid, tiempo de estancia previa al aislamiento de los ECN, relevancia clínica y evolución, así como los datos proporcionados por el servicio de Farmacia del consumo de linezolid en el periodo 2008-2011.
Fenotipo y genotipo de resistenciaLa sensibilidad a penicilina, oxacilina, cefoxitina, eritromicina, telitromicina, clindamicina, quinupristina-dalfopristina, gentamicina, tobramicina, tetraciclina, ciprofloxacina, levofloxacina, sulfametoxazol-trimetoprim, vancomicina, teicoplanina, mupirocina, linezolid, fosfomicina, nitrofurantoína, ácido fusídico y rifampicina se estudió por el sistema VITEK-2® (Bio-Mérieux); la sensibilidad a clindamicina, lincomicina, eritromicina y trimetoprim, por el método de dilución en agar8, y a linezolid, por el sistema E-test. Se estudió por PCR la presencia de los genes de resistencia mecA, lnu(A), lnu(B), vga(A), vga(B), vga(C), vga(E), cfr, lsa(B), aac(6’)-aph(2”), ant(4’)-Ia, aph(3’)-IIIa, dfrS1, dfrD, drfG y dfrK3,9. Se analizó la presencia y número de mutaciones en el gen ARNr 23S por PCR y secuenciación y por digestión con la enzima de restricción NheI10. Mediante PCR y secuenciación se estudiaron también las mutaciones que conducen a cambios aminoacídicos en las proteínas L3, L4, L22 (resistencia a linezolid) y GrlA y GyrA (resistencia a quinolonas)2,11.
Tipado molecularTodas las cepas fueron caracterizadas por el tipo de SCCmec (Staphylococcal Cassette Chromosome mec) por PCR múltiple12. Las cepas SE-LMR fueron, además, tipadas por la técnica del MLST (MultiLocus Sequence Typing) (http://sepidermidis.mlst.net/). Se llevó también a cabo en todas las cepas el tipado mediante la técnica de PFGE (Pulsed Field Gel Electrophoresis) y se comparó con el patrón obtenido en cepas previamente publicadas que se habían obtenido en otro hospital de la misma ciudad6.
Factores de virulencia y producción de biofilmSe analizó por PCR la presencia de los genes codificantes de la leucocidina de Panton-Valentine (PVL) lukF/lukS-PV, y de los genes relacionados con la formación de biofilm icaA, icaB e icaC6,9.
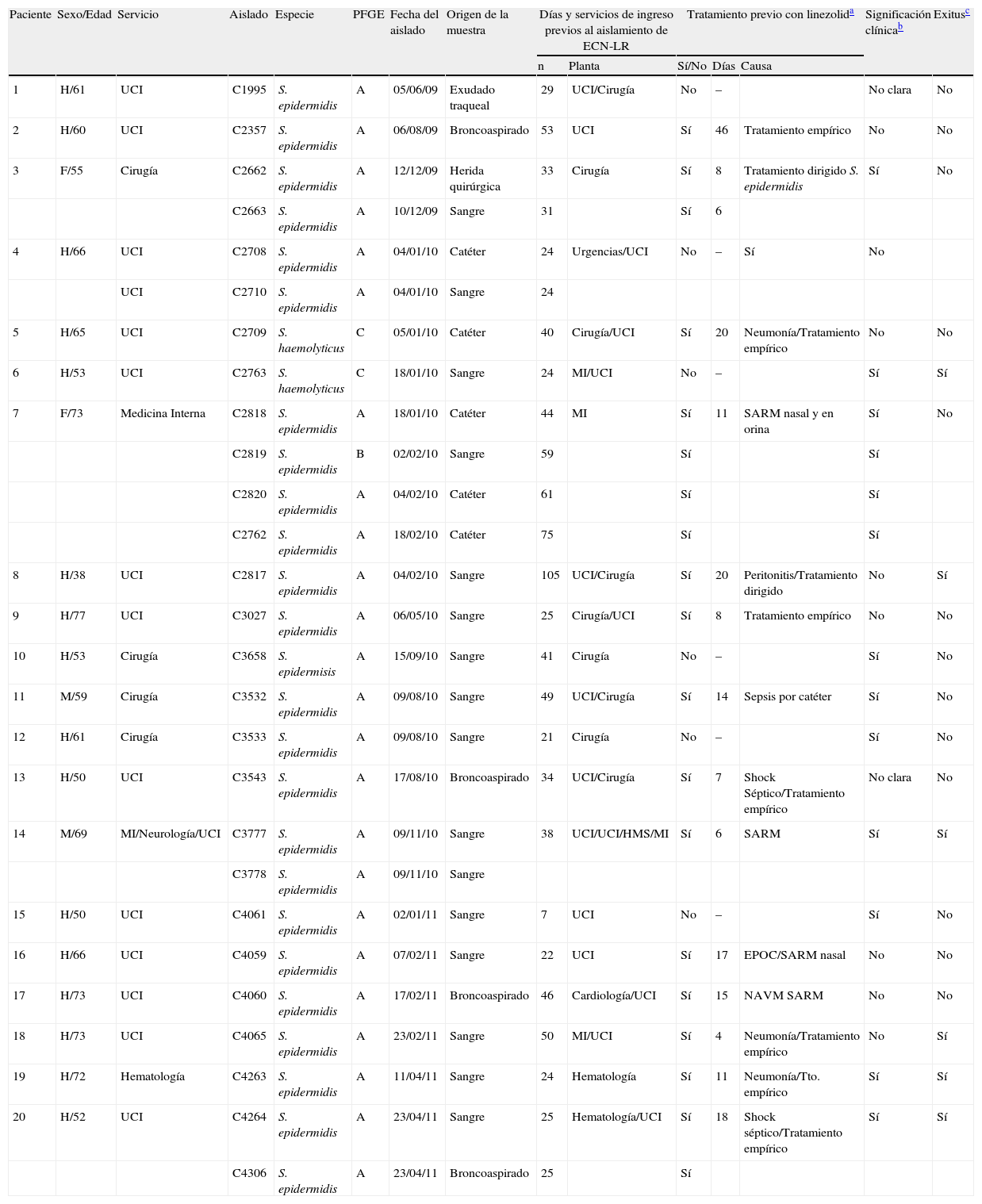
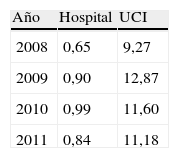
ResultadosEn la tabla 1 se muestra un resumen de los datos epidemiológicos de interés de los pacientes en los que se aislaron las cepas de ECN resistentes a linezolid. Las 27 cepas pertenecieron a 20 pacientes diferentes, y la UCI fue el servicio de procedencia de la mayoría de ellos (14 pacientes), seguido por Cirugía (4 pacientes), Medicina Interna (un paciente) y Hematología (un paciente). En cuanto al origen de los aislados, por orden de frecuencia, fue: sangre, catéteres, muestras respiratorias (broncoaspirado y aspirado traqueal) y heridas. Por otro lado, 14 de los 20 pacientes habían sido previamente tratados con linezolid, o estaban siéndolo en el momento del aislamiento de la cepa. El consumo de linezolid durante los años 2008-2011 en el HRV, tanto global como en la UCI, se refleja en la tabla 2.
Características clínicas de los 20 pacientes en los cuales se aislaron cepas de estafilococos coagulasa-negativos (ECN) resistentes a meticilina y linezolid
| Paciente | Sexo/Edad | Servicio | Aislado | Especie | PFGE | Fecha del aislado | Origen de la muestra | Días y servicios de ingreso previos al aislamiento de ECN-LR | Tratamiento previo con linezolida | Significación clínicab | Exitusc | |||
| n | Planta | Sí/No | Días | Causa | ||||||||||
| 1 | H/61 | UCI | C1995 | S. epidermidis | A | 05/06/09 | Exudado traqueal | 29 | UCI/Cirugía | No | – | No clara | No | |
| 2 | H/60 | UCI | C2357 | S. epidermidis | A | 06/08/09 | Broncoaspirado | 53 | UCI | Sí | 46 | Tratamiento empírico | No | No |
| 3 | F/55 | Cirugía | C2662 | S. epidermidis | A | 12/12/09 | Herida quirúrgica | 33 | Cirugía | Sí | 8 | Tratamiento dirigido S. epidermidis | Sí | No |
| C2663 | S. epidermidis | A | 10/12/09 | Sangre | 31 | Sí | 6 | |||||||
| 4 | H/66 | UCI | C2708 | S. epidermidis | A | 04/01/10 | Catéter | 24 | Urgencias/UCI | No | – | Sí | No | |
| UCI | C2710 | S. epidermidis | A | 04/01/10 | Sangre | 24 | ||||||||
| 5 | H/65 | UCI | C2709 | S. haemolyticus | C | 05/01/10 | Catéter | 40 | Cirugía/UCI | Sí | 20 | Neumonía/Tratamiento empírico | No | No |
| 6 | H/53 | UCI | C2763 | S. haemolyticus | C | 18/01/10 | Sangre | 24 | MI/UCI | No | – | Sí | Sí | |
| 7 | F/73 | Medicina Interna | C2818 | S. epidermidis | A | 18/01/10 | Catéter | 44 | MI | Sí | 11 | SARM nasal y en orina | Sí | No |
| C2819 | S. epidermidis | B | 02/02/10 | Sangre | 59 | Sí | Sí | |||||||
| C2820 | S. epidermidis | A | 04/02/10 | Catéter | 61 | Sí | Sí | |||||||
| C2762 | S. epidermidis | A | 18/02/10 | Catéter | 75 | Sí | Sí | |||||||
| 8 | H/38 | UCI | C2817 | S. epidermidis | A | 04/02/10 | Sangre | 105 | UCI/Cirugía | Sí | 20 | Peritonitis/Tratamiento dirigido | No | Sí |
| 9 | H/77 | UCI | C3027 | S. epidermidis | A | 06/05/10 | Sangre | 25 | Cirugía/UCI | Sí | 8 | Tratamiento empírico | No | No |
| 10 | H/53 | Cirugía | C3658 | S. epidermisis | A | 15/09/10 | Sangre | 41 | Cirugía | No | – | Sí | No | |
| 11 | M/59 | Cirugía | C3532 | S. epidermidis | A | 09/08/10 | Sangre | 49 | UCI/Cirugía | Sí | 14 | Sepsis por catéter | Sí | No |
| 12 | H/61 | Cirugía | C3533 | S. epidermidis | A | 09/08/10 | Sangre | 21 | Cirugía | No | – | Sí | No | |
| 13 | H/50 | UCI | C3543 | S. epidermidis | A | 17/08/10 | Broncoaspirado | 34 | UCI/Cirugía | Sí | 7 | Shock Séptico/Tratamiento empírico | No clara | No |
| 14 | M/69 | MI/Neurología/UCI | C3777 | S. epidermidis | A | 09/11/10 | Sangre | 38 | UCI/UCI/HMS/MI | Sí | 6 | SARM | Sí | Sí |
| C3778 | S. epidermidis | A | 09/11/10 | Sangre | ||||||||||
| 15 | H/50 | UCI | C4061 | S. epidermidis | A | 02/01/11 | Sangre | 7 | UCI | No | – | Sí | No | |
| 16 | H/66 | UCI | C4059 | S. epidermidis | A | 07/02/11 | Sangre | 22 | UCI | Sí | 17 | EPOC/SARM nasal | No | No |
| 17 | H/73 | UCI | C4060 | S. epidermidis | A | 17/02/11 | Broncoaspirado | 46 | Cardiología/UCI | Sí | 15 | NAVM SARM | No | No |
| 18 | H/73 | UCI | C4065 | S. epidermidis | A | 23/02/11 | Sangre | 50 | MI/UCI | Sí | 4 | Neumonía/Tratamiento empírico | No | Sí |
| 19 | H/72 | Hematología | C4263 | S. epidermidis | A | 11/04/11 | Sangre | 24 | Hematología | Sí | 11 | Neumonía/Tto. empírico | Sí | Sí |
| 20 | H/52 | UCI | C4264 | S. epidermidis | A | 23/04/11 | Sangre | 25 | Hematología/UCI | Sí | 18 | Shock séptico/Tratamiento empírico | Sí | Sí |
| C4306 | S. epidermidis | A | 23/04/11 | Broncoaspirado | 25 | Sí | ||||||||
Cambio vía: cambio de catéter; EPOC: enfermedad pulmonar obstructiva crónica; H: hombre; LZ: linezolid; M: mujer; MI: Medicina Interna; NAVM: neumonía asociada a ventilación mecánica; SARM nasal: colonización nasal por S. aureus resistente a meticilina (SARM); UCI: unidad de cuidados intensivos del Hospital Royo Villanova; UCI HMS: UCI del Hospital Miguel Servet de Zaragoza.
Días de tratamiento con linezolid antes del aislamiento de la cepa resistente y causa de dicho tratamiento.
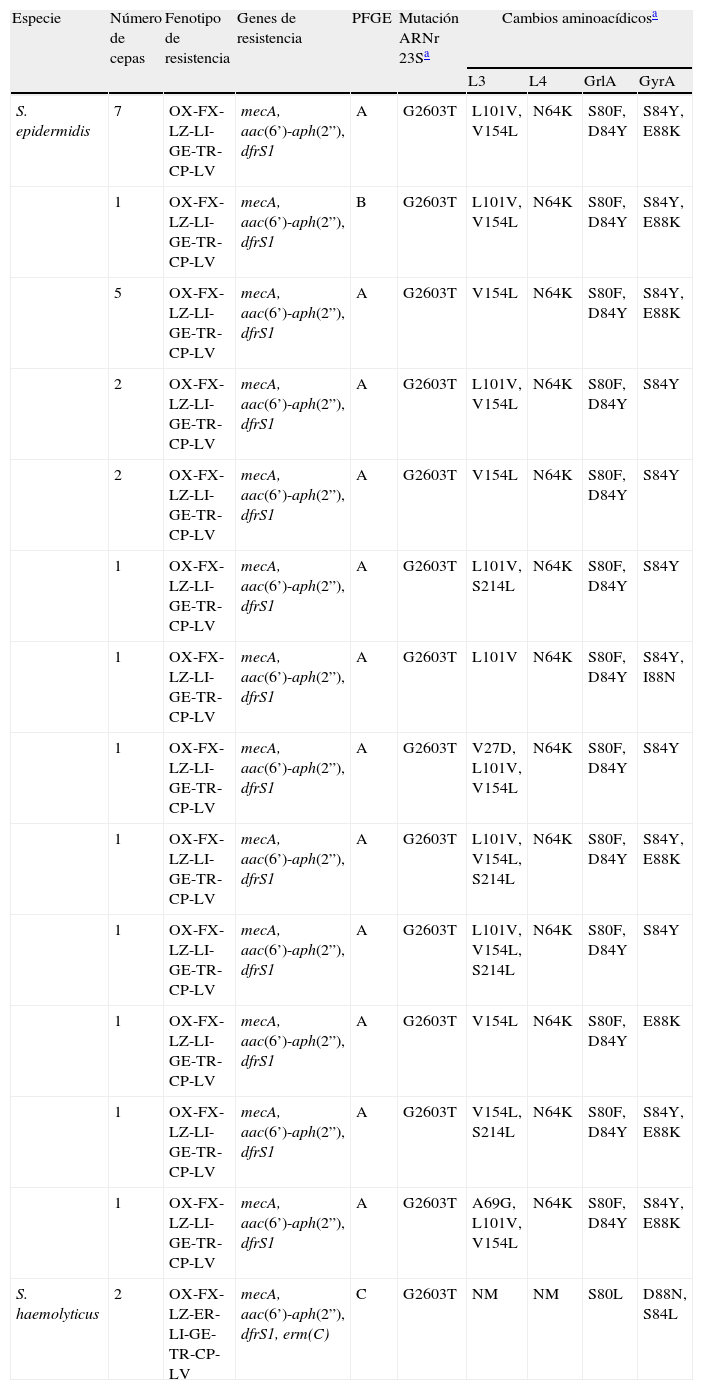
Las 25 cepas SE-LMR estudiadas pertenecieron a la secuencia tipo ST2 y presentaron el tipoIII de SCCmec. El SCCmec de las 2 cepas SH-LMR fue no-tipable con la metodología utilizada. Se analizaron las cepas por PFGE y se obtuvieron 3 patrones de campos pulsados, A y B (en las cepas SE) y C (en las cepas SH). El patrón A se obtuvo en todas las cepas SE-LMR, excepto en una de ellas (en la cual se obtuvo el patrón B).
Todas las cepas SE-LMR fueron resistentes, además de a linezolid (CMI>256mg/l), también a lincomicina (16mg/l), gentamicina, trimetoprim (64mg/l), ciprofloxacina y levofloxacina, y mostraron sensibilidad disminuida a clindamicina (1-2mg/l). Las dos cepas SH-LMR presentaron también un fenotipo de multirresistencia (tabla 3), detectándose los siguientes valores de CMI: linezolid (>256mg/l), eritromicina (32mg/l), lincomicina (16mg/l), clindamicina (16mg/l) y trimetoprim (128mg/l).
Mecanismos de resistencia detectados en las cepas de Staphylococcus epidermidis y S. haemolyticus resistentes a linezolid
| Especie | Número de cepas | Fenotipo de resistencia | Genes de resistencia | PFGE | Mutación ARNr 23Sa | Cambios aminoacídicosa | |||
| L3 | L4 | GrlA | GyrA | ||||||
| S. epidermidis | 7 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | L101V, V154L | N64K | S80F, D84Y | S84Y, E88K |
| 1 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | B | G2603T | L101V, V154L | N64K | S80F, D84Y | S84Y, E88K | |
| 5 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | V154L | N64K | S80F, D84Y | S84Y, E88K | |
| 2 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | L101V, V154L | N64K | S80F, D84Y | S84Y | |
| 2 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | V154L | N64K | S80F, D84Y | S84Y | |
| 1 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | L101V, S214L | N64K | S80F, D84Y | S84Y | |
| 1 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | L101V | N64K | S80F, D84Y | S84Y, I88N | |
| 1 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | V27D, L101V, V154L | N64K | S80F, D84Y | S84Y | |
| 1 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | L101V, V154L, S214L | N64K | S80F, D84Y | S84Y, E88K | |
| 1 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | L101V, V154L, S214L | N64K | S80F, D84Y | S84Y | |
| 1 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | V154L | N64K | S80F, D84Y | E88K | |
| 1 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | V154L, S214L | N64K | S80F, D84Y | S84Y, E88K | |
| 1 | OX-FX-LZ-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1 | A | G2603T | A69G, L101V, V154L | N64K | S80F, D84Y | S84Y, E88K | |
| S. haemolyticus | 2 | OX-FX-LZ-ER-LI-GE-TR-CP-LV | mecA, aac(6’)-aph(2”), dfrS1, erm(C) | C | G2603T | NM | NM | S80L | D88N, S84L |
CP, ciprofloxacina; ER, eritromicina; FX, cefoxitina; GE, gentamicina; LI, lincomicina; LV, levofloxacina; LZ, linezolid; NM, no se detectó ninguna mutación; OX, oxacilina; TR, trimetoprim.
Las cepas SE-LMR contenían los genes de resistencia aac(6’)-aph(2”) y dfrS1, y las cepas SH-LMR poseían además el gen erm(C) (tabla 3). No se detectaron genes de resistencia a lincomicina en las cepas SE-LMR. Se identificó, según la numeración de Staphylococcus, la mutación G2603T (G2576T según la numeración de Escherichia coli) en el gen ARNr 23S en todas las cepas. El análisis del cromatograma reveló la ausencia de dobles picos, lo que hizo pensar que todas las copias del gen se hallaban mutadas. Esto se corroboró tras la digestión con NheI, donde se obtuvo un único patrón consistente en 2 bandas (322 y 98pb). Además, en las cepas SE se detectaron las sustituciones V27D, A69G, L101V, V154L y S214L en la proteína L3 y N64K en la L4 (según numeración de Staphylococcus). No se detectó la presencia del gen cfr en ninguna de las cepas estudiadas. Se identificaron los siguientes cambios aminoacídicos en las dianas de quinolonas: S80F y/o D84Y (en SE) y S80L (en SH) en GrlA; y S84F y/o E88K (en SE) y S84L y D88N (en SH) en GyrA (tabla 3). Todas las cepas presentaron los genes icaA, icaB, icaC, y ninguna los genes lukF/lukS-PV.
Se realizó una comparación por PFGE de cepas SE-LMR de los patrones A y B de nuestro estudio con una cepa SE-LMR obtenida en otro hospital de la misma ciudad con pulsotipo 1b y caracterizada previamente6. El patrónA de nuestro estudio y el 1b del estudio referido fueron indistinguibles.
DiscusiónEste estudio describe las características moleculares de cepas SE-LMR y SH-LMR aisladas en un hospital español durante un periodo de 27 meses desde el aislamiento de la primera cepa con esa característica en junio de 2009. Linezolid es una opción terapéutica de relevancia para el tratamiento de infecciones producidas por estos microorganismos, aunque ya se han descrito con anterioridad cepas ECN resistentes a linezolid en otros estudios5,10. De forma similar a lo que se ha descrito en otros centros13, posiblemente la presión antibiótica en la UCI y el consumo especialmente elevado de linezolid en los años 2009 y 2010 en nuestro hospital han favorecido la diseminación clonal de una cepa SE-LMR. Se han descrito previamente situaciones epidémicas que han afectado a diferentes especies de ECN, como SE6,14-16, SH5 y Staphylococcus hominis17,18.
Hoy por hoy, tanto en España como en otros países, aunque la resistencia a linezolid continúa siendo poco común, el mecanismo de resistencia detectado más frecuentemente se debe a la presencia de mutaciones en la regiónV del gen ARNr 23S4. Hasta el momento se han descrito múltiples mutaciones en este gen que, siguiendo la numeración de Staphylococcus, corresponden a G2447T, G2474T, T2500A, T2530A, C2534T y G2603T1,15,17. De todas ellas, G2603T es la más frecuentemente identificada hasta el momento10, y se ha descrito en especies distintas de ECN procedentes de diferentes países. Este es el caso de cepas de SE en Brasil y en Grecia4,19, de cepas de S. hominis en el sur de España17 y en Mallorca18 y de cepas de Staphylococcus simulans en Italia20. La mutación G2603T se encontró en todas las cepas de este estudio, y se observó que todas las copias estaban mutadas. El valor de CMI a este antibiótico fue >256mg/l en todas las cepas estudiadas. La mutación G2603T se ha descrito en cepas de estafilococos proporcionando valores de CMI a linezolid de 16->256mg/l6,10,21. Las variaciones en los valores se deben a las especies en las cuales se han detectado, al número de copias mutadas y a la presencia o no de otros mecanismos asociados de resistencia a linezolid. Así, en nuestro caso, se encontraron también cambios aminoacídicos en las proteínas L3 y/o L4 que complementarían la resistencia a este antibiótico. En cualquier caso, se ha observado que cambios en L3 y L4 producen resistencia a linezolid, pero de bajo nivel2. Si analizamos los cambios aminoacídicos detectados en la proteína L3 vemos como L101V y V154L ya habían sido descritas en otros estudios2,22. Sin embargo, no existen datos sobre las modificaciones S214L, V27D y A69G. Tampoco existen publicaciones anteriores que indiquen la presencia de la mutación N64K en la proteína L4.
Ninguna de nuestras cepas presentó el gen cfr, el cual, aunque fue descrito inicialmente en cepas de origen animal3, ya ha sido reportado por otros autores en cepas clínicas tanto de S. aureus23 como de ECN20,24. En España se identificó el primer brote de SARM portador del gen cfr en el Hospital Clínico San Carlos de Madrid25. Desde entonces, este gen se ha hallado también en 2 casos de infecciones respiratorias en Mallorca causadas por SARM26, y en un caso muy grave producido por SE-LMR en Alicante27.
Por otro lado, la mutación G2603T ha sido detectada con anterioridad en cepas de SE pertenecientes a la ST26,16. Se ha sugerido, de hecho, que este clon podría tener facilidad para adquirir mutaciones en el ARNr 23S16. En un estudio llevado a cabo en un hospital de tercer nivel de la misma ciudad y unos meses antes que el nuestro (octubre 2008-agosto 2009)6, se identificaron cepas SE-LMR ST2. Las características de las cepas fueron similares a las de este trabajo, presentando el SCCmec tipoIII, el mismo perfil de resistencia, el mismo patrón de PFGE e identificándose también la mutación G2603T. Parece evidente, por tanto, que el mismo clon fue capaz de diseminarse en 2 hospitales de la misma ciudad. Este fenómeno ya había sido previamente descrito, tanto entre hospitales de la misma ciudad como entre diferentes países relacionados o no geográficamente, habiéndose aislado cepas SE resistentes a meticilina ST2 en más de 25 países de todo el mundo28.
Otro punto a destacar es que las cepas SE-LMR de nuestro estudio mostraron resistencia a lincomicina y sensibilidad disminuida a clindamicina. Se han hallado resultados similares en algunos de los estudios mencionados anteriormente6,18,19. En estos trabajos las cepas presentaban la misma mutación (G2603T) en el ARNr 23S y no se identificaron genes de resistencia a lincosamidas. Parece probable que esta mutación pueda estar implicada en dicho fenotipo, y estudios posteriores deberían esclarecer este punto. No obstante, aunque en nuestros aislados no se detectó ninguno de los genes vga descritos, en otro estudio llevado a cabo en Italia se hallaron cepas SE-LMR ST2 con la mutación G2603T y resistencia a lincomicina, que sí que contenían el gen vga20.
Es interesante reseñar también que todas las cepas del estudio presentaron los genes de patogenicidad icaA, icaB y icaC, los cuales se relacionan con la capacidad de formación de biofilm, favoreciendo la diseminación y la patogenicidad de las cepas en ambientes hospitalarios6. La presencia del operón ica en cepas SE ST2 no era de extrañar teniendo en cuenta los resultados obtenidos por otros autores6,29,30. Estos factores de virulencia tienen probablemente repercusión en la elevada frecuencia con que se asocian estos microorganismos con graves infecciones como las bacteriemias relacionadas con catéteres, y que en este, como en otros trabajos6, constituyen las infecciones más prevalentes.
En el presente estudio, además de la resistencia a linezolid, las cepas presentaron también resistencia a otros antimicrobianos. Así, en las cepas SE-LMR se identificaron los genes de resistencia aac(6’)-aph(2”) y dfrS1, y en las cepas SH-LMR se detectó también el gen erm(C). Todas las cepas fueron resistentes a quinolonas y se encontraron diferentes cambios aminoacídicos en las proteínas GyrA y ParC. Las modificaciones encontradas en estas proteínas en nuestras cepas SE-LMR y SH-LMR ya habían sido descritas previamente31,32.
Por otro lado, respecto al consumo de linezolid —que, como ya se ha comentado, se ha demostrado determinante en la aparición de estas cepas—, la tasa de ≥13DDDs/100 pacientes-día se ha considerado el umbral para la posible aparición de un brote12. Este fue el valor límite que se alcanzó en el año 2009 en la UCI de nuestro hospital (12,87DDD), y en junio de ese año se detectó la primera cepa resistente en ese servicio. En nuestro caso particular, la aparición de los primeros casos de ECN resistentes a linezolid alertaron sobre el peligro de un brote y se extremaron en la UCI tanto las precauciones de contacto como la vigilancia de su consumo, de modo que en años sucesivos (2010 y 2011) se frenó la tendencia ascendente, el consumo disminuyó ligeramente y se mantuvo por debajo de ese umbral, mientras que en el resto del hospital en el año 2010 el consumo aumentó ligeramente.
Dada la importante emergencia de este microorganismo en pacientes especialmente comprometidos, y con la cada vez más frecuente presencia de catéteres u otros dispositivos, la detección de SE multirresistente es altamente preocupante. Si a esto le unimos, además, la presencia de múltiples mecanismos de resistencia a linezolid, como es el caso de mutaciones en ARNr 23S y/o cambios aminoacídicos en las proteínas L3 y L4, es importante la rigurosa vigilancia de estas cepas para conocer más sobre su epidemiología, prevenir su aparición y eventual diseminación, y asegurar el éxito terapéutico.
FinanciaciónEste trabajo ha sido financiado en parte por el proyecto SAF2009-08570 del Ministerio de Ciencia e Innovación. Carmen Lozano tiene una beca FPU del Ministerio de Ciencia e Innovación de España y Elena Gómez-Sanz una beca FPI del Gobierno de La Rioja.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Nuestro agradecimiento a la Dra. Ohiana Horna, del Servicio de Farmacia del Hospital Royo Villanova, por proporcionar los datos de consumo de linezolid, y a la Dra. Ana Ezpeleta, médico de la UCI y miembro de la comisión de infecciones del HRV, por su apoyo en el desarrollo del trabajo.
Este trabajo ha sido presentado parcialmente en el XVI Congreso de la SEIMC, Bilbao 2012.