Los brotes detectados de campilobacteriosis son poco frecuentes y, por lo general, cursan con un bajo número de pacientes, aunque se estima que muchos más permanecerían sin diagnosticar. Las técnicas de investigación de brotes más exitosas en Campylobacter spp. (PFGE, MLST) tienen el inconveniente de ser laboriosas y no estar disponibles en muchos laboratorios.
MétodosDurante el año 2008 se recibieron 352 aislados de C. jejuni y C. coli procedentes de 16 hospitales. Todas las cepas fueron tipificadas genotípicamente mediante RFLP-PCR-flaA (tipo flaA) y fenotípicamente con su resistotipo. Se estableció que aquellas cepas de la misma especie procedentes del mismo hospital, aisladas en un periodo de hasta 11 días, con valores de CMI de±1 dilución y con el mismo tipo flaA, podrían pertenecer a un brote. Las cepas que cumpliesen con estos criterios serían posteriormente tipificadas mediante KpnI-PFGE y MLST.
ResultadosVeintitrés de los 352 aislados, en 10 grupos, cumplían con los criterios de pertenencia a posibles brotes no diagnosticados. En 8 grupos los pulsotipos (PFGE) de los aislados de cada grupo tenían una semejanza entre ellos mayor del 95%. En 7 de ellos los secuenciotipos (MLST) eran coincidentes.
ConclusionesEl uso de 2 marcadores sencillos (resistotipo y RFLP-PCR-flaA) puede detectar aislados que probablemente formen parte de un brote de campilobacteriosis no diagnosticado. Para su confirmación se requieren otros marcadores moleculares y los datos epidemiológicos de cada aislado. El estudio apunta a que, como en otros países, el número de brotes de campilobacteriosis en España está probablemente infraestimado.
Outbreaks of campylobacteriosis are infrequent and usually involve a low number of patients, although it is estimated that many more remain undiagnosed. The most successful techniques for outbreak investigation in Campylobacter spp. (PFGE, MLST) have the drawback of being laborious and not available in many laboratories.
MethodsDuring the year 2008, 352 isolates of C. jejuni and C. coli from 16 hospitals were received in our laboratory. All strains were genotyped by RFLP-PCR-flaA (flaA type) and phenotyped with their resistotype. It was established that the strains of the same species from the same hospital, isolated over a period of up to 11 days, with MIC values of±1 dilution with the same flaA type could belong to an outbreak. Strains that met these criteria would be later subtyped by KpnI-PFGE and MLST.
ResultsA total of 23 out of 352 isolates, distributed in 10 groups, met the criteria for being associated with putative undiagnosed outbreaks. The similarity of the PFGE-profiles in 8 groups was greater than 95% among the isolates from each group. In 7 of the groups, the sequence types (MLST) were coincident.
ConclusionsThe use of 2 easy markers (resistotype and RFLP-PCR-flaA) may detect isolates probably belonging to an undiagnosed outbreak of campylobacteriosis. Accurate diagnosis requires other molecular markers and epidemiological data of each isolate. The study suggests that, as in other countries, the number of outbreaks of campylobacteriosis in Spain is probably underestimated.
Las especies termofílicas del género Campylobacter (principalmente C. jejuni junto con C. coli) son la principal causa de gastroenteritis bacteriana a nivel mundial1. Se estima que debido a las características de la infección, con una sintomatología en ocasiones leve, la mayoría de los datos de campilobacteriosis están infraestimados2. El consumo o manipulación de la carne de pollo es contemplado como el principal factor de riesgo en la adquisición de la infección, aunque la mayoría de los casos son esporádicos3. A diferencia de otros patógenos alimentarios, como Salmonella enterica o Staphylococcus aureus, los brotes de campilobacteriosis se notifican de forma infrecuente y, por lo general, implicando un número reducido de casos4. Salvo algunos incidentes documentados en la literatura científica, los brotes con mayor número de casos se asocian principalmente al consumo de agua5, como por ejemplo 442 casos en Finlandia durante el verano del año 19986, 105 en Noruega7 o 71 en Japón8.
La investigación de brotes en patógenos alimentarios se basa en la comparación de diferentes marcadores fenotípicos y genotípicos, junto con los datos epidemiológicos preexistentes, para establecer si las cepas investigadas tienen un origen común. Generalmente se considera que la técnica de tipificación molecular de elección en investigación de brotes es la pulse field gel electrophoresis (PFGE, «electroforesis en gel de campo pulsante»)9,10, aunque en los últimos años se empiezan a utilizar otras como la tipificación por multi-locus sequence typing (MLST, «secuenciación de múltiples locus de genes constitutivos»)11. En Campylobacter la investigación de brotes puede ser complicada por la posible coexistencia de más de una cepa dentro de un brote individual12, esto es más evidente en los brotes originados por consumo de carne de pollo, frecuentemente colonizada por más de una cepa diferente13. Otra característica que dificulta el estudio de dichos brotes es la inestabilidad genética que se le atribuye a Campylobacter14, que hace que los perfiles moleculares puedan variar entre los aislados de un mismo brote.
Se estima que los brotes de campilobacteriosis deben ser en realidad mucho más frecuentes de lo informado, debido al bajo número de casos implicado3 y al diferente periodo de incubación que pueden tener los pacientes comprendidos en el brote, de hasta 11 días15.
El objetivo de nuestro trabajo fue investigar posibles brotes de campilobacteriosis no diagnosticados, basándonos en 2 métodos de tipificación sencillos como son el perfil de resistencia de las cepas frente a antimicrobianos (valores de las concentraciones mínimas inhibitorias [CMI]) y la determinación del tipo flaA mediante polimorfismos de los tamaños de los fragmentos de restricción del gen flaA amplificado por reacción en cadena de la polimerasa (RFLP-PCR-flaA). Los datos que proporcionan ambos marcadores permiten preseleccionar grupos de aislados que deben ser confirmados como parte de un mismo brote mediante la aplicación de otros marcadores complementarios, junto con los datos epidemiológicos existentes.
MétodosOrigen e identificación de los aisladosDurante el año 2008 se recibieron en el Laboratorio de Campylobacter, perteneciente al Centro Nacional de Microbiología, 352 aislados de Campylobacter spp. procedentes de 16 hospitales españoles. En este periodo solo se remitió de forma específica un brote que implicaba 8 aislados de C. jejuni, siendo la relación entre el resto de aislados desconocida, aunque teóricamente corresponderían a casos esporádicos. En todos los aislados recibidos se determinó la especie a la que pertenecían mediante técnicas de PCR16–18 o de secuenciación de diferentes genes diana (ADNr 16S, cpn60)19,20.
Test de sensibilidad a antimicrobianos y resistotipo de las cepasPara todos los aislados de Campylobacter recibidos en el laboratorio se realizaron tests de sensibilidad frente a los siguientes antimicrobianos: ciprofloxacino, eritromicina, gentamicina, amoxicilina, amoxicilina más ácido clavulánico, cloranfenicol y tetraciclina (codificación según WHONET)21 mediante tiras E-test (bioMérieux, España). Se siguieron las recomendaciones del Clinical and Laboratory Standards Institute tanto en la realización del método (turbidez del inóculo, medio de cultivo y cepa ATCC 33560 de C. jejuni como control de calidad), como en los puntos de corte de las CMI para diferenciar poblaciones sensibles y resistentes.
Puesto que en el Clinical and Laboratory Standards Institute solo están disponibles los puntos de corte para eritromicina, tetraciclina y ciprofloxacino22, para el resto de antimicrobianos los puntos de corte fueron seleccionados del European Committee on Antimicrobial Susceptibility Testing (disponibles en http://www.eucast.org/clinical_breakpoints/ (consultada 4/04/2013).
Siguiendo criterios ya publicados, consideramos a un aislado como multirresistente cuando era resistente frente a 3 o más grupos de antimicrobianos23.
Reacción en cadena de la polimerasa. Tipificación por polimorfismos de los tamaños de los fragmentos de restricción-reacción en cadena de la polimerasa-flaA y secuenciación de múltiples locus de genes constitutivos. Asignación de tipos flaA y secuenciotiposPara todas las PCR necesarias en el estudio se utilizó el kit de extracción de ADN genómico InstaGene Matrix (Bio-Rad, España) y el kit de amplificación de ADN Ready-To-Go System (GE Healthcare Life Sciences, Reino Unido).
El análisis de RFLP-PCR-flaA se llevó a cabo siguiendo la técnica descrita por Nachamkin et al.24. En resumen, se digieren 5μl del producto de amplificación obtenido mediante PCR con 6 unidades de enzima de restricción DdeI (FastDigest®, Fermentas GmBH, Madrid, España) a 37°C. Los fragmentos generados en la digestión se separaron en una electroforesis en agarosa (2,5% peso/volumen) tipo MS-8 (Pronadisa, España) en buffer TAE 1X.
De forma rutinaria, cada cepa de C. jejuni/C. coli que es remitida a nuestro laboratorio es tipificada mediante la técnica de RFLP-PCR-flaA. El patrón de bandas generado se compara con los ya existentes por medio del software InfoQuest FP Software (Bio-Rad, España) mediante el índice de Dice (optimización 1% y tolerancia 1%) y a cada patrón nuevo (al menos una banda de diferencia) se le asigna un nuevo tipo flaA.
Para la técnica de MLST se utilizaron los primers y el programa de PCR disponible en la página web: http://pubmlst.org/campylobacter/info/primers.shtml, amplificándose fragmentos de los 7 genes constitutivos seleccionados para C. jejuni/C. coli: aspA, glnA, gltA, glyA, pgm, tkt, uncA. Los productos de PCR se purificaron usando el kit GFX PCR DNA and Gel Band Purification Kit (GE Healthcare Life Sciences, Reino Unido) siguiendo las instrucciones del fabricante. La secuenciación de los fragmentos purificados se realizó con un kit comercial (Big Dye Ready Reaction Mix version 3.1, Applied Biosystems, EE. UU.). Se asignaron los tipos de alelos correspondientes a cada secuencia de cada gen por comparación con los de la base de datos de MLST, disponibles en: http://pubmlst.org/campylobacter/. Los secuenciotipos (ST), combinación de los 7 alelos de cada aislado, se asignaron de igual modo de acuerdo con los resultados obtenidos tras la comparación de estos en la base de datos de MLST.
Electroforesis en gel de campo pulsanteLa preparación del ADN genómico de cada aislado se realizó siguiendo el método estandarizado para Campylobacter25. La digestión del ADN genómico se hizo utilizando la enzima KpnI (FastDigest® Fermentas, ThermoFisher, España). Los geles de agarosa se prepararon al 0,9% de concentración (peso/volumen) en TBE 0,5X y las condiciones de electroforesis fueron las previamente establecidas por On et al. para la enzima KpnI26.
Los perfiles de bandas generados en la técnica de PFGE (pulsotipos) se analizan definiéndose patrones de bandas para cada aislado y se comparan con los ya existentes por medio del software InfoQuest FP Software (Bio-Rad, España), en las mismas condiciones (optimización y tolerancia) que en el caso de RFLP-PCR-flaA. Los árboles de semejanzas entre pulsotipos (dendrogramas) se construyen usando el método unweighted pair group method with arithmetic mean.
ResultadosDurante el año 2008, se recibieron 352 aislados del género Campylobacter, procedentes de 16 hospitales en España. De ellos, 282 fueron identificados como C. jejuni, y 52, como C. coli, mientras que en 12 aislados había coinfección de más de una especie. De forma minoritaria se identificaron otras especies: C. lari (n=1),C. fetus sbsp. fetus (n=4),C. curvus (n=1). A todos los aislados identificados como C. jejuni o C. coli (n=334) se les realizó un antibiograma frente a 7 antimicrobianos y se tipificaron molecularmente mediante la técnica RFLP-PCR-flaA. Esto generó una base de datos con los valores de CMI de cada cepa (y sus perfiles de resistencia) y el tipo de patrón de bandas que se generó por la digestión del fragmento amplificado del gen flaA (tipo flaA).
Del total de cepas recibidas en el año 2008, se encontraron 51 tipos flaA diferentes, destacando como más abundantes: tipo flaA 1 (39 cepas), tipo flaA 54 (27 cepas), tipo flaA 5 (20 cepas), tipo flaA 4 (18 cepas), tipo flaA 7 (18 cepas) y tipo flaA 11 (17 cepas). Aunque debemos resaltar que la mayoría de los tipos flaA descritos solo están representados por un número muy bajo de cepas e incluso en muchos casos, por una sola cepa.
En la base de datos se realizó una búsqueda de posibles brotes no diagnosticados en base al siguiente criterio: 2 o más aislados procedentes del mismo hospital, pertenecientes a la misma especie, en los que coincidiese el tipo flaA y el resistotipo (máximas variaciones permitidas en la CMI de±1dilución), y con fechas de aislamiento entre ellos de hasta 11 días. Siguiendo este criterio se seleccionaron 10 grupos de aislados (ver tabla 1), de los que 8 estaban compuestos por 2 aislados, mientras que los 2 restantes lo estaban por 3 y 4, respectivamente.
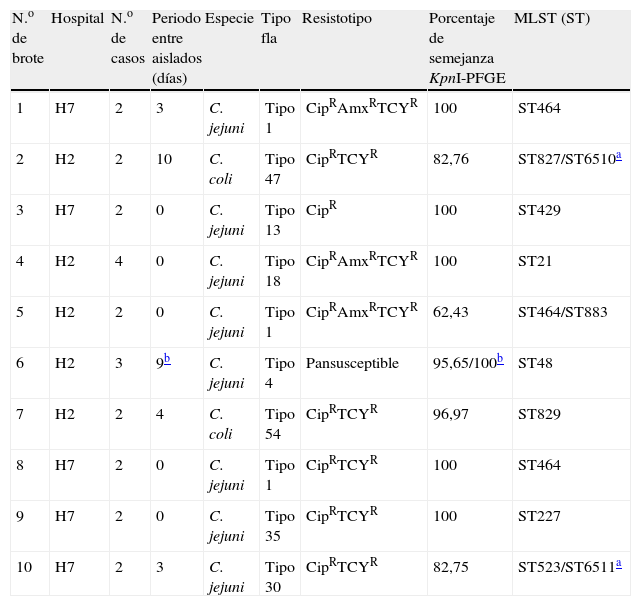
Tabla resumen con los datos epidemiológicos más relevantes de los aislados dentro de cada supuesto brote, los porcentajes de semejanza entre los pulsotipos obtenidos con la enzima KpnI, así como el secuenciotipo
| N.o de brote | Hospital | N.o de casos | Periodo entre aislados (días) | Especie | Tipo fla | Resistotipo | Porcentaje de semejanza KpnI-PFGE | MLST (ST) |
| 1 | H7 | 2 | 3 | C. jejuni | Tipo 1 | CipRAmxRTCYR | 100 | ST464 |
| 2 | H2 | 2 | 10 | C. coli | Tipo 47 | CipRTCYR | 82,76 | ST827/ST6510a |
| 3 | H7 | 2 | 0 | C. jejuni | Tipo 13 | CipR | 100 | ST429 |
| 4 | H2 | 4 | 0 | C. jejuni | Tipo 18 | CipRAmxRTCYR | 100 | ST21 |
| 5 | H2 | 2 | 0 | C. jejuni | Tipo 1 | CipRAmxRTCYR | 62,43 | ST464/ST883 |
| 6 | H2 | 3 | 9b | C. jejuni | Tipo 4 | Pansusceptible | 95,65/100b | ST48 |
| 7 | H2 | 2 | 4 | C. coli | Tipo 54 | CipRTCYR | 96,97 | ST829 |
| 8 | H7 | 2 | 0 | C. jejuni | Tipo 1 | CipRTCYR | 100 | ST464 |
| 9 | H7 | 2 | 0 | C. jejuni | Tipo 35 | CipRTCYR | 100 | ST227 |
| 10 | H7 | 2 | 3 | C. jejuni | Tipo 30 | CipRTCYR | 82,75 | ST523/ST6511a |
ST6510 y ST6511 han sido descritos por primera vez en este trabajo. Las secuencias y datos han sido depositados en la base de datos internacional de MLST de Campylobacter (http://pubmlst.org/campylobacter/).
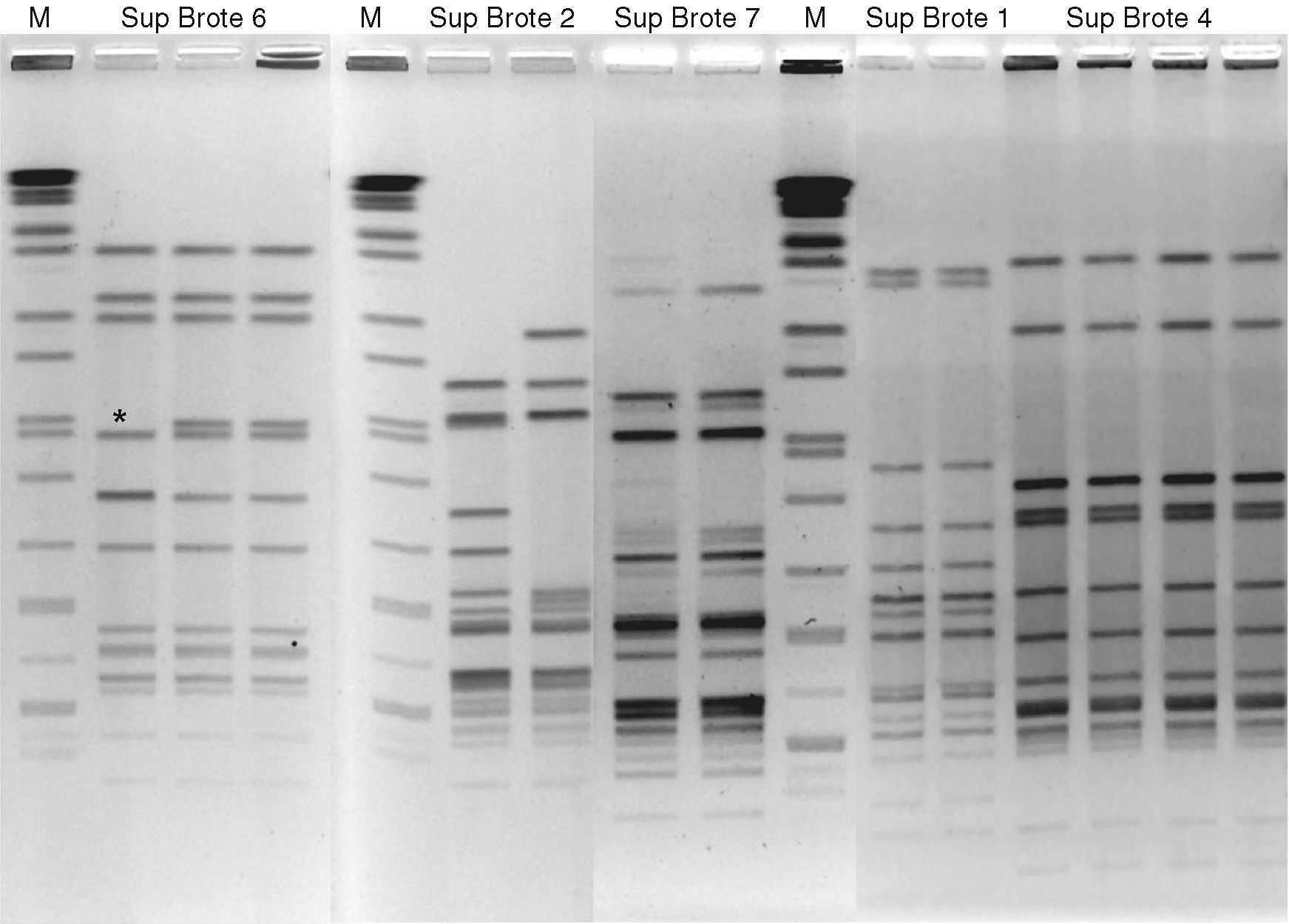
En nuestro estudio, el análisis de los perfiles de bandas generados mediante PFGE con la enzima KpnI (ejemplos de perfiles de bandas en la figura 1) reveló un porcentaje de semejanza entre aislados del 100% para los supuestos brotes 1, 3, 4, 6, 8 y 9, siendo en ellos el ST coincidente (ver tabla 1).
Composición a partir de 3 geles diferentes de electroforesis en gel de campo pulsante, donde podemos apreciar pulsotipos idénticos en algunas cepas (Supuestos Brotes 1 y 4), así como otros en los que hay variación en algunas de las bandas, como en el Supuesto Brote 5, donde se ha marcado con un asterisco la banda de diferencia. También se muestra alguno como el Supuesto Brote 6, en el que se aprecian diferencias en varias bandas.
M: marcador de peso molecular (Salmonella serotipo Braenderup H9812, digerido con XbaI).
En todos los casos con una semejanza mayor del 95% en el perfil de PFGE el ST también era coincidente, mientras que en el caso de valores inferiores al 95% había también variaciones en los alelos MLST (tabla 1).
DiscusiónDurante el periodo 1994-2003, en España se notificaron 36 brotes de campilobacteriosis en el Boletín Epidemiológico Semanal (http://www.isciii.es/htdocs/pdf/Informedebrotesalimentarios.pdf; consultada 8/04/2013), cifra muy baja en comparación con los 301 provocados por Staphylococcus aureus o los 2.568 por Salmonella enterica en el mismo periodo. En el año de estudio, 2008, en el Laboratorio de Campylobacter del Centro Nacional de Microbiología se recibió un solo brote de campilobacteriosis, causado por C. jejuni, con 8 casos, que fue confirmado mediante marcadores como el perfil de resistencia a antimicrobianos, RFLP-PCR-flaA, PFGE y MLST.
Como algún estudio ha apuntado15, es posible que haya muchos más brotes de campilobacteriosis sin diagnosticar que los notificados. Las técnicas moleculares recomendadas para la investigación de los supuestos brotes, como PFGE y MLST, son costosas y laboriosas. PFGE requiere un equipamiento que no se encuentra de forma rutinaria en los laboratorios de microbiología clínica, mientras que la técnica de MLST conlleva la realización de un alto número de PCR (7 por cepa) y de reacciones de secuenciación.
Nuestro objetivo fue intentar buscar en nuestra base de datos posibles brotes no diagnosticados, mediante el empleo de marcadores sencillos, utilizados en la mayoría de los laboratorios que trabajan con Campylobacter spp.
En la utilización del perfil de resistencias frente a una selección de antimicrobianos se ha tenido en cuenta que en el transcurso de un brote no debería haber variaciones en los valores de CMI entre los aislados, ya que la presión antibiótica habría sido igual para todos ellos. Aunque los pacientes hubieran recibido tratamiento antibiótico, teóricamente no habría el tiempo suficiente para que se desarrollara una resistencia de la bacteria frente al antibiótico o una variación significativa de los valores de CMI, si bien en el caso de las quinolonas, la resistencia se sabe que se desarrolla de forma muy rápida27.
Para Campylobacter, RFLP-PCR-flaA es un marcador con alto poder de discriminación, como evidencian los 51 tipos flaA diferentes identificados entre las cepas de Campylobacter spp. recibidas en el año 2008. Sin embargo, tanto nuestra experiencia, como la de otros autores28–30, indica que es un marcador que permanece invariable a lo largo de un brote de campilobacteriosis.
Con los criterios establecidos se seleccionaron 10 grupos de aislados que podrían pertenecer a brotes no diagnosticados. Para ratificar microbiológicamente si los aislados de un mismo grupo podían estar epidemiológicamente relacionados y formar parte de un brote, más allá de la coincidencia en sus valores de CMI y el tipo flaA, se estudiaron los marcadores complementarios: i) PFGE, que es considerada de elección en el estudio de brotes de patógenos alimentarios, y ii) MLST, que, a pesar de determinar secuencias de genes constitutivos muy conservados, tiene el suficiente poder de discriminación en Campylobacter. Estas técnicas, teóricamente, no deberían mostrar variación en las cepas que pertenezcan a un brote.
Sin embargo, en el caso de PFGE, la variabilidad intrínseca de Campylobacter hace que pueda haber con alguna frecuencia variaciones en este marcador. Un ejemplo concreto es la observación de la variación en una banda del perfil de PFGE de un brote por movilización de un profago presente en el genoma de la cepa causante del brote31.
En el estudio de brotes se han utilizado de forma tradicional para PFGE los criterios elaborados por Tenover et al. en 199510, en los que se considera que un solo evento genético (una mutación puntual) puede hacer que haya hasta 3 bandas de diferencia en el perfil de campo pulsado. Según estos criterios, dentro del contexto epidemiológico adecuado, aislados con una diferencia de hasta 6 bandas entre ellos podrían formar parte de un brote. Estos criterios deben ser considerados según las características propias del microorganismo que estemos estudiando.
En nuestro caso, siguiendo las recomendaciones de otros autores32, se ha usado la enzima KpnI, que genera un número de bandas adecuado para ser suficientemente discriminatorio (más de 8 bandas, ver figura 1) y atribuye con mayor consistencia las cepas a su posible origen.
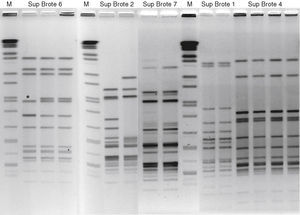
Como puede observarse en el dendrograma generado por la comparación de los pulsotipos entre los aislados del estudio (fig. 2), en 5 de los grupos la semejanza era del 100% entre los aislados de un mismo grupo, en 2 más era superior al 95%, mientras que en 3 de ellos era inferior al 85%. En los grupos 6 y 7 hay variaciones de tan solo una o 2 bandas (fig. 1), por lo que según los criterios de Tenover et al. para investigación de brotes, los aislados probablemente formaban parte de 2 brotes distintos. Incluso en el grupo 2 (fig. 1), en el que los 2 aislados tienen 3 bandas de diferencia, no se podría descartar que ambos aislados pertenecieran al mismo brote. Para confirmar y apoyar los datos obtenidos mediante PFGE se ha seguido la tendencia actual en el estudio de brotes de Campylobacter, que es utilizar más de una técnica de tipificación molecular además del PFGE33, es por ello que hemos utilizado una técnica basada en la secuenciación, como es la MLST.
Dendrograma obtenido con el software Infoquest mediante la comparación de los pulsotipos generados por la enzima KpnI en las distintas cepas de los supuestos brotes. A cada una de las cepas se le ha dado la nomenclatura POn-ISn, del inglés putative outbreak isolate, por ejemplo: «PO1-IS1: putative outbreak 1-isolate 1, es decir, posible brote 1-aislado 1». Los valores de semejanza entre cepas de un supuesto brote (diferentes al 100%) señalados en el dendrograma son A: 68,65% (posible brote 5), B: 82,75% (posible brote 10), C: 95,65% (posible brote 6), D: 82,76% (posible brote 2) y F: 96,97% (posible brote 7).
En todos los casos, los aislados con semejanzas en PFGE superiores al 95% poseían el mismo ST, encontrando solo variaciones entre alelos en aquellos con porcentajes inferiores. En el caso del grupo 2, ambos aislados pertenecían a diferentes ST, si bien se diferencian en un solo alelo (tkt), y las diferencias entre las secuencias de estos eran mínimas (un solo nucleótido). Estos datos, más la escasa diferencia entre bandas de sus pulsotipos, nos hace pensar que ambas cepas podrían pertenecer a un mismo brote.
En cualquier estudio de brotes es necesario disponer de datos epidemiológicos de los pacientes para su confirmación34. En nuestro caso, no disponer de estos datos para cada aislado, salvo los nombres de los pacientes, el hospital de procedencia y la fecha de aislamiento de los aislados, hace que no se pueda confirmar al 100% que las cepas de un mismo grupo pertenecían a un brote, más allá de que fueran indistinguibles o estuvieran estrechamente relacionadas desde el punto de vista microbiológico.
Sin embargo, en 2 de los supuestos brotes (3 y 9) los pacientes en cada uno de los grupos son hermanos, por lo que parece muy claro sospechar de un brote de tipo familiar. De hecho, se estima que dentro de los brotes de campilobacteriosis, los de tipo familiar serían los más frecuentes35.
Es destacable cómo 3 de los posibles brotes están constituidos por cepas multirresistentes (1, 4 y 5), lo cual es de preocupación en Salud Pública. Afortunadamente, ninguna de las cepas estudiadas era resistente a macrólidos, antibiótico de elección en las campilobacteriosis severas27. Con la excepción del brote 6, constituido por cepas pansusceptibles, todos los aislados eran resistentes al menos a ciprofloxacino, lo que resalta la idea del alto porcentaje de resistencia en las cepas patógenas aisladas en España y cómo se desaconseja el uso de ciprofloxacino u otras fluoroquinolonas en el tratamiento de las campilobacteriosis, como muestran varios autores36.
En conclusión, hemos visto cómo mediante el estudio de los datos obtenidos por 2 técnicas asequibles en laboratorio (resistotipo y tipo flaA) podemos sospechar de la presencia de posibles brotes no diagnosticados de Campylobacter. Más aún, estos resultados podrían ser obtenidos en poco tiempo (48h) tras el aislamiento de las cepas. Esta alerta temprana sería de gran ayuda para la investigación epidemiológica que permitiera hacer una búsqueda de la fuente de infección y/o de otros posibles casos implicados.
De los 10 posibles brotes encontrados, 8 parecen confirmarse como tales, aunque se requieren más datos epidemiológicos. Sin duda, como se muestra en otros países, el número de brotes de campilobacteriosis está también infraestimado en España. Finalmente, consideramos que este estudio puede ayudar a comprender mejor la epidemiología y características de 2 patógenos importantes en Salud Pública, como son C. jejuni y C. coli.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Queremos agradecer especialmente a los diferentes hospitales españoles que durante el año 2008 enviaron aislados de Campylobacter spp. al CNM. Esta publicación hizo uso de la página web de MLST de Campylobacter (http://pubmlst.org/campylobacter/) desarrollada por Keith Jolley y alojada en la Universidad de Oxford (Jolley & Maiden 2010, BMC Bioinformatics, 11:595). El desarrollo de dicho sitio ha sido financiado por el Wellcome Trust. Agradecemos a la Unidad de Genómica del CNM por llevar a cabo la secuenciación de los alelos de la técnica MLST. También agradecemos a los doctores Silvia Herrera-León y Sergio Sánchez Prieto, de la Unidad de Enterobacterias del CNM, por su ayuda en el análisis de las secuencias de los alelos en la técnica de MLST.
Actualmente, el primer autor posee una beca en la “Sección de Microbiología Alimentaria”. Agencia Española de Seguridad Alimentaria y Nutrición (AESAN). Madrid, España.