El hongo Conidiobolus coronatus (C. coronatus) tiene una extensa distribución de hábitats y organismos; se encuentra en forma saprófita, ataca insectos y mamíferos, incluido el humano, aunque de este los reportes son pocos y están restringidos a zonas tropicales. El objetivo de este trabajo fue determinar si existe o no variación genética entre aislamientos de C. coronatus provenientes de lesiones en humanos y otras fuentes.
MétodosOnce aislamientos de C. coronatus obtenidos de suelo, insectos y humanos se analizaron con las técnicas de polimorfismo derivado de la amplificación aleatoria de ADN (RAPD) y secuenciación del espaciador interno transcrito (ITS1-5.8S rADN-ITS2); para el análisis de las regiones ITS se usaron los métodos de máxima parsimonia, neighbour-joining (NJ) y mínima evolución.
ResultadosEl análisis de los polimorfismos de C. coronatus con las técnicas RAPD e ITS evidenció altos niveles de variación intraespecífica en los aislamientos evaluados. Los aislamientos obtenidos de lesiones en humanos presentan la mayor divergencia genética respecto de los obtenidos de otras fuentes; la mayor distancia genética entre grupos de aislamientos se da entre los provenientes de humanos y los tomados del insecto Lycoriella ingenua.
ConclusionesEste trabajo es el primero en evaluar y demostrar que existe variación intraespecífica a nivel molecular de C. coronatus que, a su vez, está relacionada con la fuente de la que se obtuvieron.
The fungus Conidiobolus coronatus (C. coronatus) has an extensive distribution of habitats and hosts. It is found saprophytically, and attacks insects and mammals, including humans. Although there are few reports on humans, and they are restricted to tropical areas. The aim of this work was to determine whether genetic variation exists between C. coronatus isolates coming from human lesions and other sources.
MethodsA total of 11C. coronatus isolates obtained from soil, insects and humans were analyzed with the random amplification of polymorphic ADN (RAPD) and internal transcribed spacer (ITS) techniques; the maximum parsimony, neighbour-joining and minimum evolution methods were used for the ITS analysis.
ResultsThe analysis of the C. coronatus polymorphisms showed high intra-species variation levels between the evaluated isolates. The isolates coming from human lesions showed the greatest genetic divergence compared with the remaining isolates. The greatest genetic distance between isolate groups was found between those coming from humans and those taken from the insect Lycoriella ingenua.
ConclusionsThis is the very first work evaluating and demonstrating that within species variation exists at molecular level in C. coronatus, and is related to the source where the isolates were taken from.
Conidiobolus coronatus (C. coronatus) (Entomophthorales) es un hongo cosmopolita y patógeno de insectos de diferentes órdenes1; se ha encontrado sobre madera en descomposición, hojarasca, suelo2 y también como patógeno de mamíferos, incluyendo al hombre, a quien produce rinoentomoftoromicosis, que afecta la superficie del tracto respiratorio, puede dañar la mucosa nasal y paranasal e incluso extenderse a la piel de la nariz, labio superior y área fronto-glabelar3.
El reporte de daño en humanos es poco frecuente y se restringe a regiones con climas tropicales y de alta humedad4. La extensa distribución de C. coronatus y los pocos casos de rinoentomoftoromicosis reportados sugieren que no todos los aislamientos son patógenos para el humano y animales superiores3,5.
En la planta de producción de champiñones (Agaricus bisporus) Hongos Rioxal (Perote, Veracruz, México) se detectó un brote epizoótico de C. coronatus sobre moscas de la especie Lycoriella ingenua (L. ingenua), considerada la principal plaga del champiñón6. El champiñón se produce en naves cerradas que presentan condiciones similares a un clima tropical húmedo7 y en las que hay moscas infectadas por C. coronatus. Los trabajadores se exponen a ambos factores por periodos prolongados, lo que implica que se encuentran en contacto con las esporas del hongo y, a pesar de ello, no existen reportes de rinoentomoftoromicosis originados en plantas de producción de champiñón.
La relación entre C. coronatus y su amplia gama de hospederos es digna de estudio y se puede considerar un indicio de variación intraespecífica. Basado en lo anterior, el objetivo de este trabajo fue investigar si existe o no variación genética entre aislamientos de C. coronatus provenientes de lesiones en humanos y otras fuentes (suelo, insecto, compost).
Para determinar la variabilidad genética entre aislamientos provenientes de diferentes hospederos se requiere del uso de técnicas moleculares8. El espaciador interno transcrito (ITS por sus siglas en inglés) es el locus más popular en la investigación micológica basada en secuencias; es el más exitoso para la identificación de un gran número de hongos, por lo que se ha designado como un marcador de ADN del código de barras universal para hongos; además, su probada capacidad para el estudio de la variación inter- e intraespecífica lo convierte en una herramienta muy poderosa9-12. De igual modo, la técnica de polimorfismo derivado de la amplificación aleatoria de ADN (RAPD por sus siglas en inglés) ha adquirido diversos usos en el estudio de la diversidad genética; en los hongos ha sido utilizada con éxito principalmente en estudios intraespecíficos13. Se implementaron estas 2 técnicas en 11 aislamientos de C. coronatus y sus resultados se analizaron y discutieron en términos de la distancia genética según la fuente de la que fueron aislados (suelo, insecto y humano).
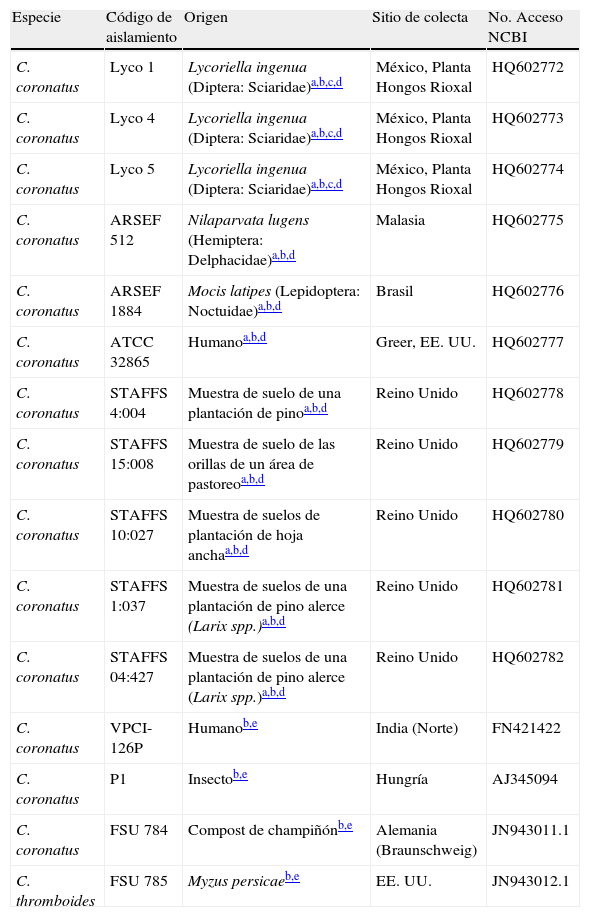
Materiales y métodosAislamientosSe usaron 11 aislamientos de C. coronatus; 8 de ellos se obtuvieron de colecciones públicas y 3 se aislaron por cuenta propia (Lyco 1, 4 y 5). Todos se procesaron para analizarlos con las técnicas RAPD y amplificación de la región ITS1-5.8S rADN-ITS2 (intrones ubicados entre los genes rADN 28S y 18S). La lista de todos los aislamientos y la fuente de que se tomaron se muestra en la tabla 1.
Lista de aislamientos usados
| Especie | Código de aislamiento | Origen | Sitio de colecta | No. Acceso NCBI |
| C. coronatus | Lyco 1 | Lycoriella ingenua (Diptera: Sciaridae)a,b,c,d | México, Planta Hongos Rioxal | HQ602772 |
| C. coronatus | Lyco 4 | Lycoriella ingenua (Diptera: Sciaridae)a,b,c,d | México, Planta Hongos Rioxal | HQ602773 |
| C. coronatus | Lyco 5 | Lycoriella ingenua (Diptera: Sciaridae)a,b,c,d | México, Planta Hongos Rioxal | HQ602774 |
| C. coronatus | ARSEF 512 | Nilaparvata lugens (Hemiptera: Delphacidae)a,b,d | Malasia | HQ602775 |
| C. coronatus | ARSEF 1884 | Mocis latipes (Lepidoptera: Noctuidae)a,b,d | Brasil | HQ602776 |
| C. coronatus | ATCC 32865 | Humanoa,b,d | Greer, EE. UU. | HQ602777 |
| C. coronatus | STAFFS 4:004 | Muestra de suelo de una plantación de pinoa,b,d | Reino Unido | HQ602778 |
| C. coronatus | STAFFS 15:008 | Muestra de suelo de las orillas de un área de pastoreoa,b,d | Reino Unido | HQ602779 |
| C. coronatus | STAFFS 10:027 | Muestra de suelos de plantación de hoja anchaa,b,d | Reino Unido | HQ602780 |
| C. coronatus | STAFFS 1:037 | Muestra de suelos de una plantación de pino alerce (Larix spp.)a,b,d | Reino Unido | HQ602781 |
| C. coronatus | STAFFS 04:427 | Muestra de suelos de una plantación de pino alerce (Larix spp.)a,b,d | Reino Unido | HQ602782 |
| C. coronatus | VPCI-126P | Humanob,e | India (Norte) | FN421422 |
| C. coronatus | P1 | Insectob,e | Hungría | AJ345094 |
| C. coronatus | FSU 784 | Compost de champiñónb,e | Alemania (Braunschweig) | JN943011.1 |
| C. thromboides | FSU 785 | Myzus persicaeb,e | EE. UU. | JN943012.1 |
ARSEF: provenientes de la USDA-ARS Entomopathogenic Fungus Collection (Ithaca, New York EE. UU.); ATCC 32865: proveniente de la American Type Culture Collection (Rockville, MD, EE. UU.); Lyco: estos aislamientos se colectaron de la planta de champiñones Rioxal (México); STAFFS: procedentes de la colección del Dr. Arthur A. Callaghan de la Universidad Staffordshire, College Road, U.K.
Los aislamientos de C. coronatus Lyco 1, 4 y 5 se obtuvieron en la planta de producción de champiñones Hongos Rioxal (según método de Papierok y Hajek14), donde se detectó el brote epizoótico de un hongo entomopatógeno sobre moscas de la especie L. ingenua, que fue identificado por el especialista Arthur A. Callaghan como C. coronatus. Todos los aislamientos presentaron las características morfológicas propias de la especie (por ejemplo, conidios vellosos producidos solo por esta especie). Se realizaron los postulados de Koch y con ellos se comprobó que C. coronatus fue el agente causal de la enfermedad sobre L. ingenua.
Los 11 aislamientos de C. coronatus se mantuvieron a temperatura ambiente y se preservaron en agua destilada estéril. Para su desarrollo, se inocularon en medio agar dextrosa Sabouraud a 25°C.
Extracción de ADNSe obtuvo micelio liofilizado de los 11 aislamientos de C. coronatus siguiendo el método descrito por Guzmán-Franco et al.15. El ADN se extrajo mediante el método CTAB (bromuro de hexacetiltrimetilamonio) modificado de Ahrens y Seemüller16. El macerado se colocó en un tubo Eppendorf con 400μl de solución de lisis (cloruro de sodio 0,4M, Tris-HCl 10mM, EDTA 2mM, PVP 1%) y se mezcló con un agitador vórtex para homogenizar. Enseguida se agregaron 50μl de SDS 20%, 40μl de proteinasa K (10mg/ml) y se incubó a 65°C durante una hora. Después se agregaron 600μl de solución CTAB 3% (Sigma Chemicals, EE. UU.) y se incubó nuevamente durante 45min a 65°C. Posteriormente se adicionó un volumen de cloroformo y alcohol isoamílico (24:1), se mezcló y centrifugó 10min a 14.000rpm, donde se formaron 2 fases; la fase acuosa se colocó en un tubo Eppendorf con un volumen de isopropanol y se incubó a –20°C por 60min; después se centrifugó 10min a 14.000rpm. El ADN fue resuspendido en 50μl de agua destilada estéril; la concentración y calidad del ADN se estimó con un NanoDrop ND-1000 V3.7 (Thermo Scientific®, EE. UU.). Por último, el ADN se visualizó en un gel de agarosa al 1% en 1 X de buffer TBE (Tris0,089M, ácido bórico 0,089M, EDTA 0,002M) teñido con bromuro de etidio (0,5μg ml−1 por 10min) y se fotografió con un fotodocumentador Gel-Doc mod. 2000 (BIO-RAD®, EE. UU.).
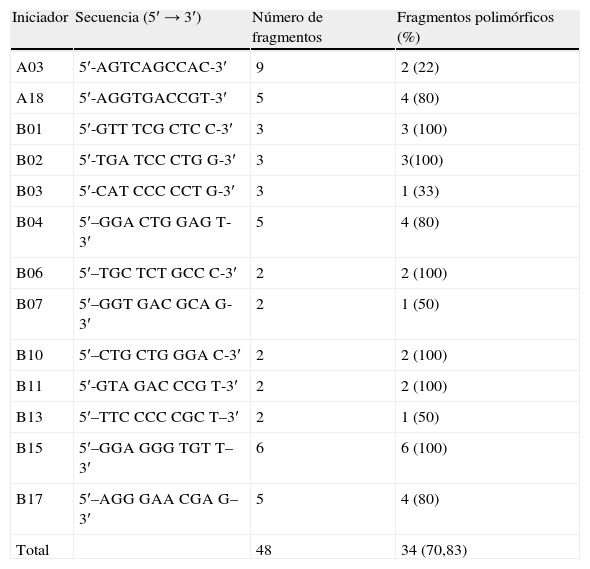
Amplificación RAPDLa reacción en cadena de la polimerasa (PCR por sus siglas en inglés) se realizó en un termociclador My Cycler™ Thermal cycler BIO-RAD modelo 580BR 09275. Se usaron tubos Eppendorf de 0,5ml; el volumen final de la mezcla fue 25μl/PCR y contenía 4μl de ADN (80ng/μl), buffer 10x, cloruro de magnesio 3mM, 2,5mM de cada dNTP (Gene Choice), 1,5 U de Taq polimerasa (Biogenica) y 2μl de cada iniciador (10pmol). Se utilizaron iniciadores aleatorios de la serie A de Operon Technology® y de la serie B de Invitrogen (tabla 2). De 19 iniciadores probados, solo aquellos que dieron resultados reproducibles (3 amplificaciones) se utilizaron en el análisis RAPD, por lo que al final se seleccionaron 13.
Secuencia de los iniciadores RAPD y fragmentos amplificados
| Iniciador | Secuencia (5′ → 3′) | Número de fragmentos | Fragmentos polimórficos (%) |
| A03 | 5′-AGTCAGCCAC-3′ | 9 | 2 (22) |
| A18 | 5′-AGGTGACCGT-3′ | 5 | 4 (80) |
| B01 | 5′-GTT TCG CTC C-3′ | 3 | 3 (100) |
| B02 | 5′-TGA TCC CTG G-3′ | 3 | 3(100) |
| B03 | 5′-CAT CCC CCT G-3′ | 3 | 1 (33) |
| B04 | 5′–GGA CTG GAG T-3′ | 5 | 4 (80) |
| B06 | 5′–TGC TCT GCC C-3′ | 2 | 2 (100) |
| B07 | 5′–GGT GAC GCA G-3′ | 2 | 1 (50) |
| B10 | 5′–CTG CTG GGA C-3′ | 2 | 2 (100) |
| B11 | 5′-GTA GAC CCG T-3′ | 2 | 2 (100) |
| B13 | 5′–TTC CCC CGC T–3′ | 2 | 1 (50) |
| B15 | 5′–GGA GGG TGT T–3′ | 6 | 6 (100) |
| B17 | 5′–AGG GAA CGA G–3′ | 5 | 4 (80) |
| Total | 48 | 34 (70,83) |
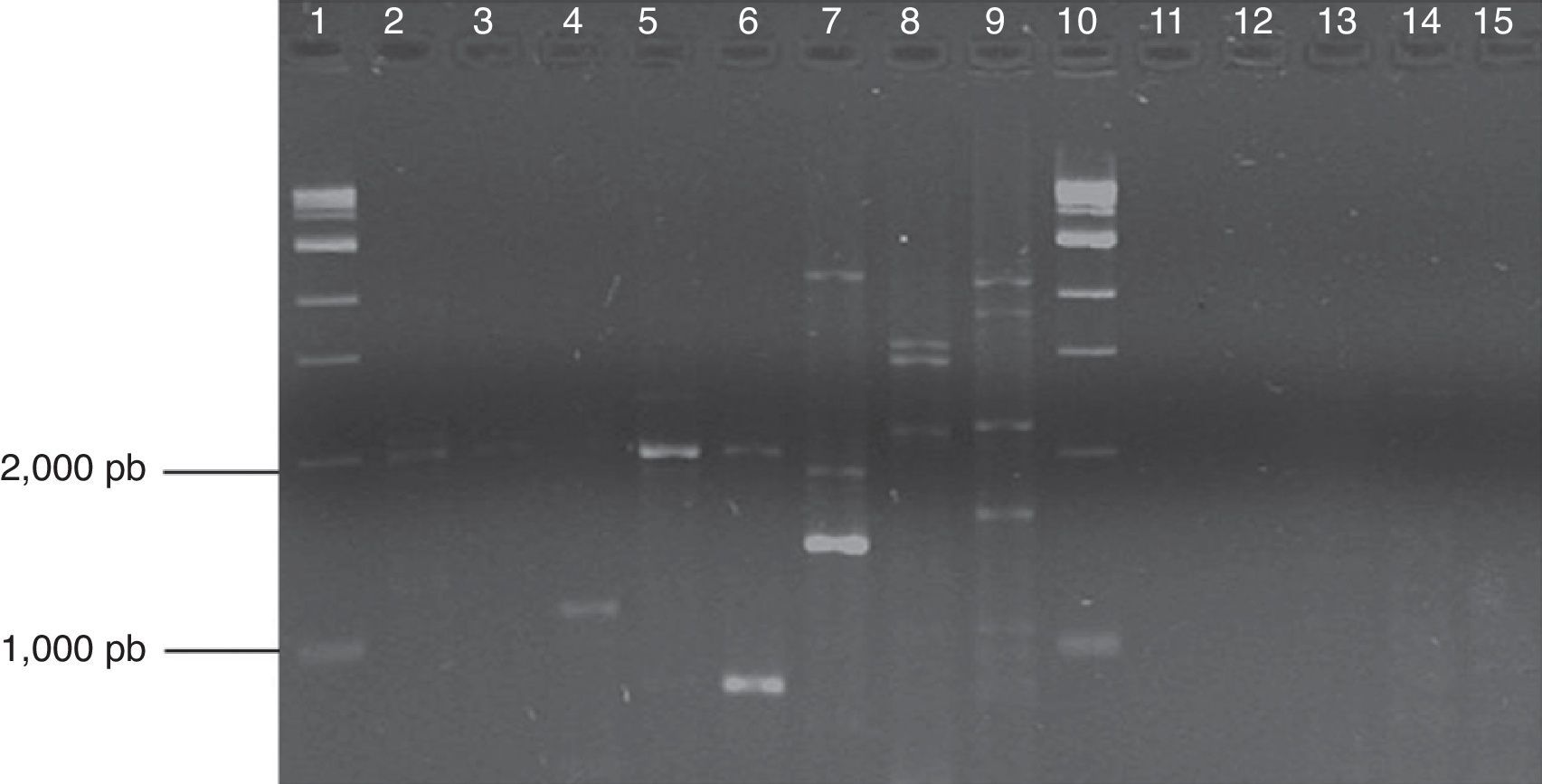
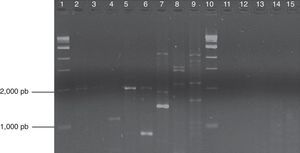
El programa de amplificación fue de un ciclo de desnaturalización inicial a 94°C por unmin, seguido de 38 ciclos de desnaturalización a 94°C por 30 s, hibridación a 35°C por 30 s, y extensión a 72°C por 1,5min, seguido de una extensión final a 72°C durante 5min. El tamaño de los fragmentos amplificados se estimó usando un marcador de peso molecular de 1kb (QIAGEN). En la amplificación de cada iniciador se utilizaron como testigos Zoophthora radicans (aislado de Plutella xylostella) y Cercospora agavicola (aislado de Agave tequilana) (fig. 1), que no se incluyeron en el análisis de datos RAPD. El manejo de estos aislamientos (crecimiento y extracción de ADN) se describe en Guzmán-Franco et al.15 y Ayala-Escobar et al.17.
Productos de PCR con el iniciador RAPD A18 en gel de agarosa al 1%. Carriles 1 y 10: marcadores; carriles 2-4: Lyco 1, 4 y 5; carriles 5 y 6: ARSEF 512 y 1884; carril 7: ATCC 32865; carriles 8 y 9 testigos (Z. radicans y C. agavicola); carril 11-15: STAFFS 4:004, STAFFS 15:008, STAFFS 10:027, STAFFS 1:037, STAFFS 04:427.
La presencia o ausencia de patrones de bandas RAPD se consideró como un carácter independiente y se analizó visualmente a partir de fotografías de los geles de agarosa; la presencia se registraba con 1 y la ausencia con 0. Con estos resultados se construyó una matriz binaria donde se encontraban los polimorfismos de cada uno de los iniciadores. A partir de dicha matriz de similitud y del coeficiente de apareamiento simple, se construyó un dendrograma; para generar y visualizar las relaciones se usó NTSYS 2.0. Para obtener un dendrograma de mayor solidez se realizaron 1.000 réplicas Bootstrap (BS) con Winboot.
Amplificación de la región ITSSe amplificaron las regiones ITS1-5.8 ADNr-ITS2 del ADN ribosomal para los 11 aislamientos de C. coronatus usando los iniciadores universales ITS 5 (5′GGAAGTAAAAGTCGTAACAAGG) e ITS 4 (5′TCCCCGCTTATTGATATGC)18. Las amplificaciones se realizaron en tubos Eppendorf de 0,5ml y el volumen final de la muestra por reacción fue 25μl. Cada reacción contenía 2μl de ADN (80ng/μl), buffer 10x, cloruro de magnesio 2,6mM, 2,5mM de cada dNTP (Gene Choice), 1,5 U/μl de Taq polimerasa, 2μl de cada iniciador (20pmol). Los tubos testigos contenían agua destilada estéril en lugar de ADN. El programa de amplificación fue de un ciclo de desnaturalización inicial a 95°C por 5min, después 35 ciclos de desnaturalización a 95°C por 30 s, hibridación a 50°C por unmin, extensión a 60,3°C por 1,5min y una extensión final a 72°C durante 5min. El tamaño de los fragmentos amplificados se estimó usando marcadores de peso molecular de 1kb y 100 pb (QIAGEN).
Para encontrar diferencias a nivel de nucleótidos entre los aislamientos, los productos de PCR-ITS se enviaron para su limpieza y secuenciación a la empresa Macrogen (Corea). La secuenciación se realizó en ambas direcciones usando los iniciadores universales 4 y 518.
Análisis de datos de la región ITSLas 11 secuencias de ITS se editaron manualmente con Bioedit y FinchTV 1.3. El alineamiento múltiple de secuencias se realizó con Clustal W. Las secuencias se depositaron en el NCBI Genbank (tabla 1). Para construir un árbol filogenético más confiable, adicionales a las 11 secuencias de C. coronatus obtenidas en este estudio, se utilizaron 3 secuencias de C. coronatus y una de Conidiobolus thromboides del NCBI Genbank (tabla 1).
La selección de C. thromboides como grupo externo se hizo a partir de un blast (NCBI Genbank) con las 11 secuencias de C. coronatus y un análisis de parsimonia, cuyos resultados arrojaron que está estrechamente emparentado con C. coronatus pero es diferente en la región ITS.
Con MEGA5 se construyó un árbol filogenético con los métodos de máxima parsimonia, neighbour-joining (NJ) y mínima evolución; en los 2 últimos se usó el modelo de Tamura-Nei. Para evaluar la confianza de la relaciones filogenéticas; la solidez de los nodos se estimó por análisis BS con 2.000 réplicas.
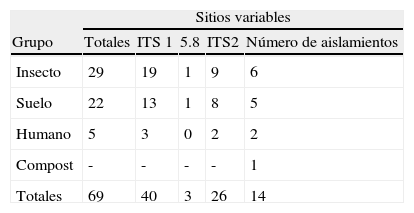
Para analizar más a fondo la diversidad genética de los aislamientos, las secuencias de ITS se agruparon de acuerdo con su sustrato u hospedero: 1) insecto, 2) suelo, 3) humano y 4) compost; con el programa MEGA5 se hizo un recuento para determinar el número de sitios variables en el interior de cada grupo y entre todos los grupos, así como la región en la que se encontraban (ITS1-5.8S rADN-ITS2) (tabla 3). Para estas 2 evaluaciones no se consideró al grupo externo.
ResultadosAnálisis RAPDLos iniciadores seleccionados amplificaron 48 bandas con una variación polimórfica de 22 a 100%; se obtuvieron 34 bandas polimórficas, resultando en 70,83% de polimorfismo; se obtuvieron de 2 a 9 bandas por iniciador (tabla 2). La figura 1 muestra los patrones de bandeo obtenidos por la amplificación con el iniciador A18 y se aprecia la diferencia genética entre los aislamientos evaluados procedentes de diferentes fuentes.
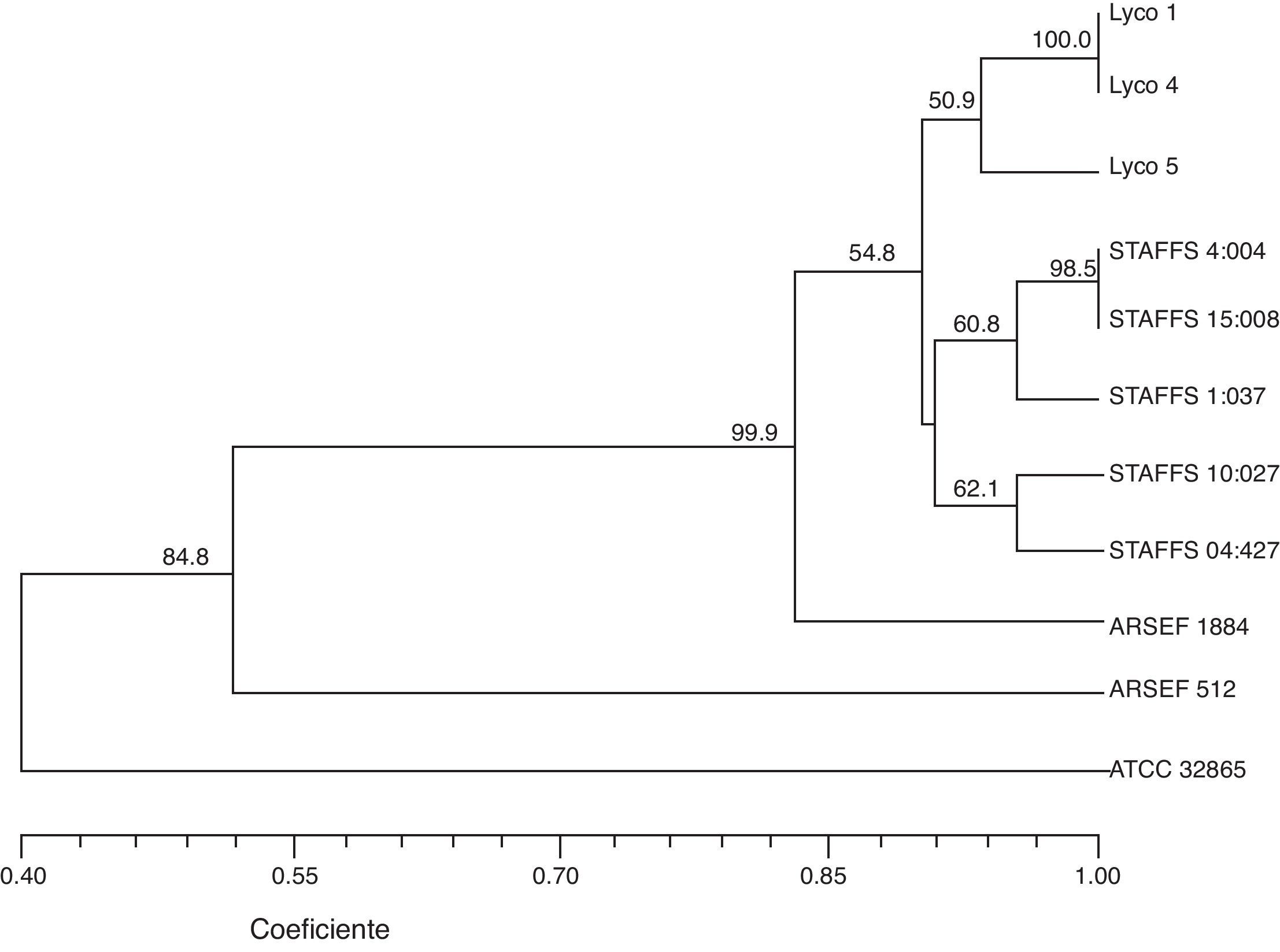
El dendrograma construido a partir del análisis de los patrones de bandeo obtenidos con los 13 iniciadores revela la formación de 4 grupos principales (A, B, C y D) con un 40% de similitud. Los resultados indican un alto nivel de diversidad genética. La estabilidad de los agrupamientos obtenidos se evaluó con un análisis BS con 1.000 réplicas (fig. 1). El grupo A incluye 8 aislamientos divididos en 2 subgrupos, i y ii; el subgrupo i contiene los aislamientos obtenidos de L. ingenua Lyco 1, Lyco 4 y Lyco 5 (Lyco 1 y Lyco 4 tienen un 100% de similitud); el subgrupo ii está formado por los aislamientos de suelo STAFFS 4:004, STAFFS 15:008, STAFFS 1:037, STAFFS 10:027 y STAFFS 04:427 (STAFFS 4:004, STAFFS 15:008 tienen un 100% de similitud). El grupo B solo incluye el aislamiento ARSEF 1884 tomado de Mocis latipes, con 80% de similitud. El grupo C solo incluye el aislamiento ARSEF 512 proveniente de Nilaparvata lugens, con 50% de similitud. En el grupo D solo está el aislamiento ATCC 32865 proveniente de lesiones humanas, con 40% de similitud, y este presenta la mayor distancia genética respecto del resto de los aislamientos (fig. 2).
Análisis de la región ITSLos iniciadores ITS usados18 amplificaron un fragmento de 666 pb (los espacios se consideraron como sitios informativos) correspondiente a las regiones ITS1-5.8S rADN-ITS2 en los 11 aislamientos evaluados. La longitud de cada una de las regiones fue homogénea en todos los aislamientos: ITS1 223 pb; 5.8S rADN, 155 pb; ITS2, 288 pb. El alineamiento y análisis de la secuencias con el programa MEGA5 de la región ITS1-5.8S rADN-ITS2 de aislamientos de C. coronatus (tabla 1) muestra 69 sitios variables entre los 14 aislamientos. De estos, 40 se encuentran en ITS1, convirtiéndola en la región más polimórfica, tanto de todos los grupos como de los aislamientos de cada grupo. La región 5.8S es la más conservada, ya que solo presenta 3 sitios variables entre los 14 aislamientos. En el interior de cada grupo también hubo diferencias en el número de sitios variables totales, siendo el grupo procedente de insectos el de mayor número (29) y el grupo de humanos el más homogéneo (5) (tabla 3).
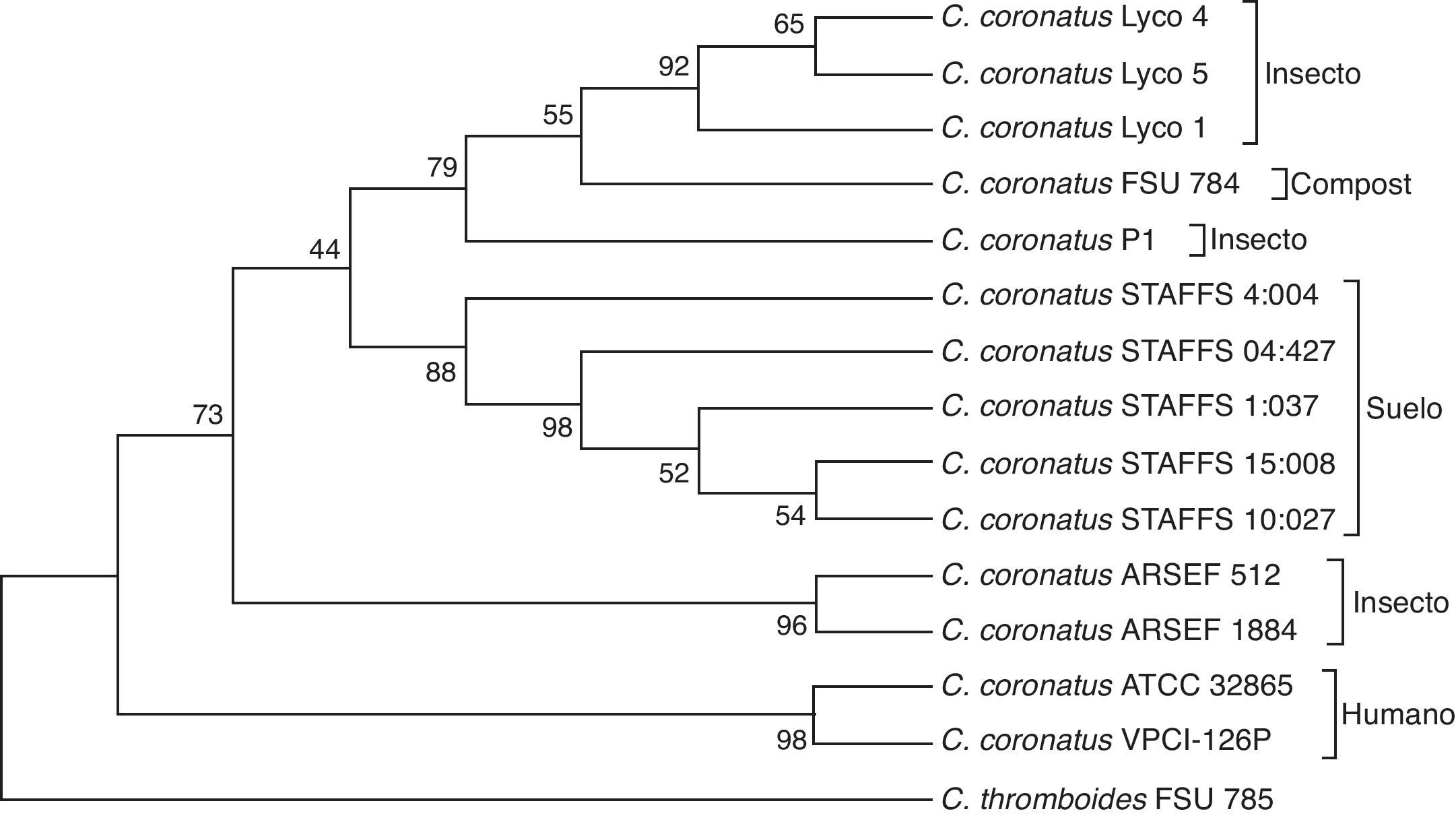
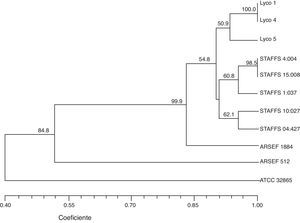
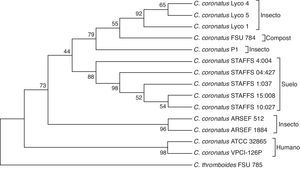
La variabilidad de la región ITS proporcionó información acerca de los patrones de diversificación de C. coronatus. Se construyó un árbol filogenético con los métodos estadísticos de máxima parsimonia, neighbour-joining (NJ) y mínima evolución y usando a C. thromboides como grupo externo. Se obtuvo el mismo resultado con las 3 aproximaciones tanto en la topología como en el soporte de las ramas (valor BS); la región ITS diferenció claramente a C. coronatus de C. tromboides (especie cercanamente relacionada). En la figura 3 se muestra el árbol resultado del análisis de NJ.
Las distancias genéticas entre aislamientos se representan por la longitud de la rama de los grupos que se forman. Se formaron los grupos A, B y C. El grupo A incluye 10 aislamientos divididos en los subgrupos i y ii; el subgrupo i contiene los aislamientos Lyco 1, Lyco 4 y Lyco 5 (obtenidos L. ingenua), FSU 784 (obtenido de compost de champiñón) y P1 (obtenido de insecto). En el subgrupo ii se encuentran los aislamientos STAFFS 4:004, STAFFS 15:008, STAFFS 10:027, STAFFS 1:037 y STAFFS 04:427 (obtenidos de suelo); el valor BS del nodo que une a los subgrupos I y II es bajo (44%), pero el valor BS del nodo que une a los aislamientos del subgrupo i es de 79% y del subgrupo ii es de 88%. El grupo B incluye los aislamientos ARSEF 512 y ARSEF 1884 (aislados de insectos homópteros y coleópteros, respectivamente) y por último, en el grupo C se encuentran los aislamientos ATCC 32865 y VPCI-126P (tomados de lesiones humanas). Este grupo presenta la mayor distancia genética respecto de los demás aislamientos y su valor BS es de 98%.
DiscusiónLos resultados de este trabajo muestran la evidente variación genética entre los aislamientos de C. coronatus evaluados, agrupados según su origen en: 1) insecto, 2) compost, 3) suelo y 4) humano.
A pesar de que los principios de la técnica con secuencias de ITS difieren de los de la técnica RAPD19,20, se cumple con lo que indica Soll21, quien afirma que para que los resultados obtenidos con un método se consideren válidos, deben confirmarse con al menos una técnica diferente. Nuestros resultados son consistentes con ambas técnicas basados en que: 1) existe variación intraespecífica en los aislamientos evaluados; 2) los aislamientos de humanos presentan la mayor divergencia genética respecto del resto de los aislamientos; 3) la mayor distancia genética entre grupos de aislamientos se da entre los provenientes de humanos y los Lycos (aislados de L. ingenua).
Aun cuando resultados como los de Ribes et al.5, Valle et al.3 y Wieloch et al.4 sugieren que no todos los aislamientos son patógenos para el humano y que, a su vez, esto es un indicio de variación intraespecífica del hongo, no hay estudios que lo respalden. Este trabajo es el primero en evaluar y demostrar que existe variación intraespecífica a nivel molecular de C. coronatus relacionada con la fuente de la que fueron tomados.
Estudios similares han evaluado la variación en otros entomoftorales con RAPD y amplificación de ITS22,23. Así como nuestros resultados muestran una clara distancia genética entre aislamientos provenientes de diferentes fuentes, Fargues et al.9 y Morton et al.24 reportaron la relación entre la variación genética y el hospedero.
Existen estudios del complejo Fusarium, que aunque no es entomoftoral, comparte con C. coronatus la característica de infectar una amplia gama de hospederos, incluido el humano. Zhang et al.25 buscaron variación intraespecífica sin éxito en 471 aislamientos tomados de humanos, suelo, animales, aire de hospitales y plantas. La razón de ello es que Fusarium es un hongo multihospedero26, lo que implica que un mismo aislamiento puede infectar un gran número de organismos de diversos phyla.
Cada microorganismo tiene un conjunto de características que funcionan como factores de virulencia en sus hospederos; sin embargo, se desconoce hasta qué punto condicionan los mecanismos de infección en diferentes grupos de hospederos. Una causa de ello es la ausencia de modelos que permitan analizar la virulencia entre grupos infectados27. Tomando en cuenta las diversas fuentes en las que se ha encontrado a C. coronatus y nuestros resultados que muestran claramente la variación intraespecífica entre aislamientos, consideramos que C. coronatus podría servir como modelo para evaluar mecanismos de virulencia que lo hacen específico para cada uno de los grupos de organismos a los que ataca.
Aun cuando nuestros resultados muestran una clara diferencia a nivel molecular entre aislamientos de C. coronatus provenientes de humanos y otras fuentes, en este trabajo no se presentan resultados morfológicos o fisiológicos de los aislamientos para comprobar si las diferencias genéticas se traducen en diferencias fenotípicas; sin embargo, existen observaciones propias (solo con algunos aislamientos) y de otros autores que nos ayudan a inferirlo, además de plantear la posibilidad de que exista algún aislamiento que no sea patógeno de humanos. Hall et al.28 consideran que la capacidad de desarrollarse a 37°C es un criterio para evaluar el potencial de los hongos como patógenos de humanos; Mier et al.29 trabajaron en una cepa proveniente de humano y se desarrolló a 37°C, mientras que Papierok et al.2 estudiaron 10 cepas, de las cuales solo una de origen humano y una aislada del miriápodo Hanseniella unguiculata crecieron a 37°C; el resto provenía de insectos de diversos órdenes. En nuestro caso, se observó el efecto de la temperatura sobre los aislamientos Lyco 1, 4 y 5, encontrando que no hubo desarrollo a 37°C (datos no presentados). Además, observamos que el aislamiento ATCC 32865 (tomado de humano) tardó casi 4 días más en llenar una caja Petri de 85mm de diámetro con respecto a los obtenidos de insectos (datos no presentados) y tenía una apariencia algodonosa, de poca rugosidad y tonalidades grisáceas, características que no se presentaron en los demás aislamientos evaluados. Estas características también sustentan las diferencias entre el aislamiento de humanos y el resto, lo que podría atribuirse a la fuente de la que se obtuvo y sugiere que esta puede ser un factor que genera distancias genéticas. Los resultados de López-Martínez et al.30 también sugieren la variación intraespecífica y que no todos los aislamientos son patógenos de mamíferos; realizaron pruebas en ratones, hámsters y cobayos con una cepa de C. coronatus tomada de Aenolamia postica y aunque hubo una invasión inicial en todos los animales, desapareció 15 días después de la inoculación, lo que indica que el hongo no fue capaz de establecerse e infectarlos. Lo anterior pone de manifiesto la ausencia de patogenicidad de esta cepa en particular en mamíferos.
De esta manera, nuestros resultados sobre la distancia genética con ITS y RAPD entre el aislamiento ATCC 32865 (de humano) y los tomados de insectos, junto con las características fisiológicas y morfológicas de los aislamientos Lycos descritas anteriormente, sugieren que estos no son patógenos para el humano; esta podría ser la razón por la que, pese a que los trabajadores están en contacto con el hongo, no hay reportes de daño en humanos por C. coronatus en plantas productoras de champiñón.
Nuestros resultados no son concluyentes para sugerir el uso de C. coronatus en el control biológico de insectos; hace falta investigación para determinarlo, como ocurrió con la cepa 251 del hongo Purpureocillium lilacinus que es usada actualmente para el control biológico de nematodos, aun cuando el hongo está reportado como patógeno de humanos y animales. Esta cepa es la única que no produce la paecilotoxina, que es el agente causal de daño en humanos31. Cabe mencionar que para C. coronatus ya se identificó el metabolito coronatin-1 en larvas de Galleria mellonella y, aunque hace falta determinar su modo de acción, sería interesante investigar si este metabolito es capaz de causar alteraciones en humanos4.
El tamaño de la muestra y el número de hospederos/sustratos de este trabajo fueron limitados, lo que podría haber afectado el análisis de la diversidad genética y el número de ramas que se formaron en los árboles filogenéticos. Se recomienda realizar pruebas con un mayor número de aislamientos e incluir estudios de morfología y fisiología. A pesar de esas limitaciones, nuestros resultados con las técnicas RAPD e ITS proporcionan información sobre la diversidad intraespecífica de C. coronatus relacionada con el hospedero/sustrato.
Conflicto de interesesLos autores declaran que no existe ningún conflicto de interés.
Agradecemos a la Ing. Iobana Alanís-Martínez, al Dr. Ariel Guzmán-Franco sus valiosas aportaciones. Al Dr. Arthur A. Callaghan, el envío de los aislamientos STAFFS 4:004, STAFFS 15:008, STAFFS 10:027, STAFFS 1:037, STAFFS 04:427, así como al Dr. Richard A. Humber el envío de los aislamientos ARSEF 512 y ARSEF 1884.