La tecnología de BACs-on-Beads utiliza sondas de ADN procedentes de Cromosomas Artificiales Bacterianos o BAC fijados en microesferas Luminex®. La muestra marcada y los ADN de referencia marcados se hibridan respectivamente con las mismas sondas BoBsTM complementarias. Después de la hibridación se leen las intensidades de la señal mediante el sistema instrumental Luminex® 100/200. Se puede aplicar al diagnóstico prenatal rápido con un kit diseñado para descartar aneuploidías para los cromosomas 13, 18, 21 y los cromosomas sexuales así como ganancias y pérdidas de ADN asociadas con 9 síndromes de microdeleción como son: Síndrome de DiGeorge, Síndrome de Williams-Beuren, Síndrome de Prader-Willi, Síndrome de Angelman, Síndrome de Smith-Magenis, Síndrome de Wolf-Hirschhorn, Síndrome de Cri du Chat, Síndrome de Langer-Giedion, y Síndrome de Miller-Dieker. Basados en esta misma tecnología se ha desarrollado un kit llamado KaryoLite-BoBs™, que permite descartar aneuploidías para los 24 cromosomas y que se puede aplicar al estudio de alteraciones cromosómicas numéricas en restos abortivos. En este trabajo se presentan los resultados de nuestro grupo tras la aplicación de la tecnología de BAC-on-Beads™ en 332 muestras de líquido amniótico, 48 muestras de vellosidades coriales y en el estudio de aneuploidías en 71 muestras de restos abortivos.
BACs-on-Beads (BoBs) technology is based on DNA sequences from Bacterial Artificial Chromosomes fixed in Luminex® microspheres. The study sample and the reference DNA are labeled with similar but complementary BoBsTM probes. After the hybridisation, signal intensities were analysed using the Luminex® 100/200 instrumental system. This technology has been applied to rapid prenatal diagnosis with a kit designed to analyse aneuploidy for chromosomes 13, 18, 21 and sex chromosomes, and a panel of 9 microdeletion syndromes: DiGeorge, Williams-Beuren, Prader-Willi, Angelman, Smith-Magenis, Wolf-Hirschhorn, Cri du Chat, Langer-Giedion, and Miller-Dieker syndrome. Based on a similar technology, KaryoLite-BoBs™ kit allows the identification of aneuploidy for all 24 chromosomes, and can be applied to the analysis of products of conception, among other possibilities. In this study we present our current experience in the application of BAC-on-Beads™ technology in 332 amniotic fluid samples; 48 chorionic villus samples and 71 samples from products of conception.
Los defectos congénitos son una de las causas principales de morbimortalidad en las sociedades desarrolladas. De ellos, el 6% es debido a cromosomopatías y se calcula que uno de cada 200 recién nacidos sufre alguna1. Estos defectos congénitos pueden ser causados por una anomalía cromosómica o por una anomalía monogénica. Hay un pequeño grupo de defectos congénitos provocados por causas ambientales, como las infecciones fetales, aunque la mayoría de malformaciones físicas son de origen multifactorial, es decir, en ellas intervienen tanto factores ambientales como genéticos.
En la actualidad, hay un número creciente de estos defectos congénitos que pueden ser detectados precozmente mediante diagnóstico prenatal2. Se dispone de varios métodos de cribado que facilitan un acercamiento al diagnóstico de anomalías congénitas fetales. Un diagnóstico prenatal ecográfico, mediante ecografía de alta resolución, permite poner de manifiesto ciertos rasgos fenotípicos, que se asocian a un riesgo más alto de cromosomopatía. En la población general de bajo riesgo, la efectividad en el cribado mejora si se evalúa conjuntamente marcadores ecográficos y bioquímicos durante el primer trimestre. Concretamente, se trata de determinar la translucencia nucal, la subunidad β libre de la Gonadotropina Coriónica Humana (fß-hCG) y la Proteína Plasmática A asociada al Embarazo (PAPP-A), en lo que se ha denominado test combinado bioquímico-ecográfico del primer trimestre. Con los marcadores tanto bioquímicos como morfológicos obtenidos, se realiza una estimación de riesgo, cuyo valor de corte está situado en aproximadamente 1/250, considerándose un riesgo alto de cromosomopatías aquellos valores mayores a este2.
Si queremos hacer un diagnóstico cromosómico concluyente, la herramienta es el cariotipo en células fetales (amniocitos). El análisis citogenético de las metafases obtenidas tras el cultivo celular es la piedra angular a la hora de realizar un diagnóstico prenatal. El cariotipo convencional es un método altamente fiable para la detección de aneuploidías y reordenamientos estructurales de un tamaño superior a 5-10 millones de pares de bases1. Sin embargo, las técnicas de citogenética clásica presentan entre otros inconvenientes: las limitaciones inherentes a su límite de resolución, ya que no permite la detección de deleciones y duplicaciones de menos de 5 Mb, así como de alteraciones estructurales crípticas; el periodo de espera de resultados es de 1 o 2 semanas (tiempo necesario para llevar a cabo un cultivo celular); y la necesidad de material cultivable vivo. Por este motivo, junto al estudio clásico del cariotipo fetal, se ofrece la posibilidad de un diagnóstico rápido complementario, en el que se estudian solo aquellos cromosomas implicados en las aneuploidías más comunes y compatibles con la vida, que son aquellas que implican a los cromosomas sexuales y a los autosomas 13, 18 y 21. En el plazo de 24-48h tras la recepción de la muestra obtenemos resultados para estas pruebas. Las técnicas de prenatal rápido más usadas actualmente son: hibridación in situ fluorescente (FISH), Quantitative Fluorescence Polymerase Chain Reaction (QF-PCR) y Multiplex Ligation-dependent Probe Amplification (MLPA).
En nuestro laboratorio, en los 2 últimos años, se ha implementado un nuevo test de diagnóstico prenatal rápido basado en la tecnología de BACs-on-Beads (BoBsTM; CE-IVD) (Perkin Elmer, Turku, Finlandia), llamado Prenatal-BoBs™, que utiliza sondas de ADN procedentes de cromosomas artificiales bacterianos o BAC fijados en microesferas Luminex®. La muestra marcada y los ADN de referencia marcados se hibridan respectivamente con las mismas sondas BoBs complementarias. Después de la hibridación se leen las intensidades de la señal mediante el sistema instrumental Luminex® 100/200. El software compara las intensidades de señal de la muestra y las referencias para determinar cambios en el número de copias de ADN en las regiones diana.
Este kit para diagnóstico prenatal rápido se ha diseñado para descartar aneuploidías para los cromosomas 13, 18, 21 y los cromosomas sexuales así como ganancias y pérdidas de ADN asociadas con 9 síndromes de microdeleción como son: Síndrome de DiGeorge, Síndrome de Williams-Beuren, Síndrome de Prader-Willi, Síndrome de Angelman, Síndrome de Smith-Magenis, Síndrome de Wolf-Hirschhorn, Síndrome de Cri du Chat, Síndrome de Langer-Giedion, y Síndrome de Miller-Dieker.
Vialard et al. analizaron retrospectivamente mediante Prenatal-BoBs, 404 muestras prenatales que previamente habían sido estudiadas por métodos convencionales, demostrando la robustez de la tecnología utilizada y estableciendo una tasa de detección conjunta de aneuploidías para los cromosomas 13,18,21,X/Y y de los 9 síndromes de microdeleción de 1/1.7003.
Prenatal-BoBsTM se indica, preferentemente, como herramienta de anticipación para el despistaje de las aneuploidías más comunes, screening bioquímico-ecográfico del primer trimestre positivo, edad materna avanzada, ansiedad materna, embarazo o hijo previo o historia familiar con anomalías cromosómicas, reordenamiento cromosómico en los padres y gestaciones obtenidas mediante técnicas de reproducción asistida.
Basados en esta misma tecnología se ha desarrollado un kit llamado KaryoLite-BoBs™, (Perkin Elmer, Turku, Finlandia), que permite descartar aneuploidías para los 24 cromosomas y que se puede aplicar al estudio de alteraciones cromosómicas numéricas en restos abortivos. El estudio genético de las muestras procedentes de gestaciones no viables se convierte en una herramienta crucial en parejas con gestaciones detenidas (p. ej. tras un tratamiento de reproducción asistida), para establecer la etiología de las pérdidas gestacionales, aportar asesoramiento reproductivo a la pareja, así como plantearse el abordaje de futuros tratamientos.
El objetivo de este trabajo ha sido validar la utilidad de la tecnología de BAC-on-Beads™ en el diagnóstico prenatal rápido y en el estudio de aneuploidías en restos abortivos.
Materiales y métodosPacientes incluidas en los estudiosPacientes con diagnóstico prenatal rápido mediante Prenatal-BoBs™Entre mayo de 2010 y enero de 2012 procesamos en nuestro laboratorio para diagnóstico mediante Prenatal-BoBs™ un total de 380 muestras de las que 332 procedían de líquido amniótico (LA) y 48 de vellosidad corial (VC). Las muestras fueron tomadas y transportadas a temperatura ambiente. Una vez recepcionadas, se llevó a cabo una centrifugación suave en el caso de LA o la toma una pequeña porción de VC. Este material se almacenó a −20°C hasta su procesamiento que se inicia con la extracción de ADN.
En paralelo, se realizó cariotipo convencional a 287 muestras y aCGH (Array-based Comparative Genomic Hybridization) a 9 muestras. Para el cariotipo convencional, el LA se cultivó durante 10-12 días, en medio de cultivo para tal fin, condiciones de esterilidad a 37° C y 5% de CO2. Mediante bandeo GTG, se tiñeron las metafases obtenidas del cultivo previo. Se realizó el análisis de 15-20 metafases de, al menos, 2 cultivos primarios. Los aCGH se utilizaron para la detección de aneuploidias y variaciones patogénicas submicroscópicas asociadas a 97 síndromes (BlueGnome, Cambridge, Reino Unido).
Pacientes con estudio de restos abortivos mediante KaryoLite-BoBs™Entre marzo de 2011 y enero de 2012 se procesaron 71 muestras de restos abortivos procedentes de 67 gestaciones detenidas durante el primer trimestre y obtenidos mediante biopsia dirigida tras histeroembrioscopia. Cuatro de las muestras pertenecían a embarazos gemelares. Las muestras se recogieron y transportaron en suero salino a temperatura ambiente. Una vez recepcionadas se mantuvieron a 4°C hasta su procesamiento. Todas las muestras fueron analizadas exitosamente mediante KaryoLite-BoBs™.
Generalidades de la técnica de BoBs™BoBs™ es una tecnología de detección multiplex que utiliza microesferas en suspensión para revelar la presencia de aneuploidías comunes y ganancias y pérdidas de material genético4. Para ello, usa la tecnología xMAP® (Luminex Corp, Austin, Texas)5 mediante una serie de microesferas marcadas internamente con 2 fluorocromos con espectros en rojo y en rojo lejano. Empleando combinaciones de 10 concentraciones distintas de cada uno de estos 2 flurocromos se consiguen 100 espectros específicos o 100 tipos de microesferas diferentes. Cada microesfera tiene acoplada una secuencia de ADN amplificada en un BAC. Un tercer fluorocromo cuantifica la interacción molecular entre el ADN de la superficie y la secuencia capturada de la muestra problema. Mediante un sistema de citometría de flujo, cada microesfera es detectada por 2 láseres diferentes que determinarán qué microesfera estamos analizando, esto es, qué secuencia lleva unida en su superficie, qué cantidad de ADN problema unido a la superficie.
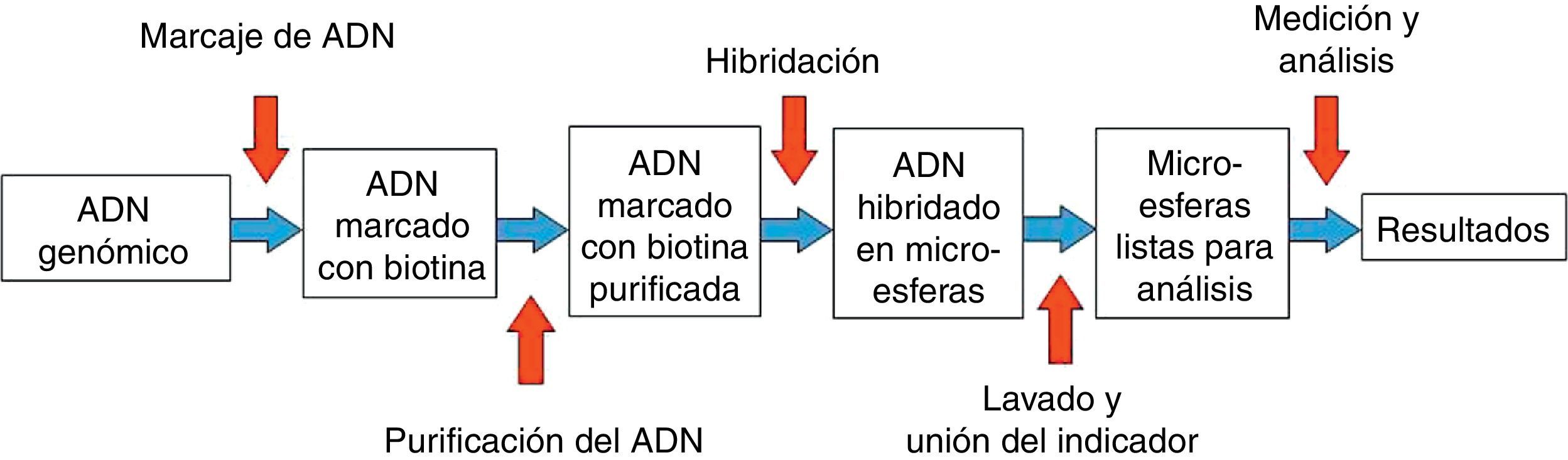
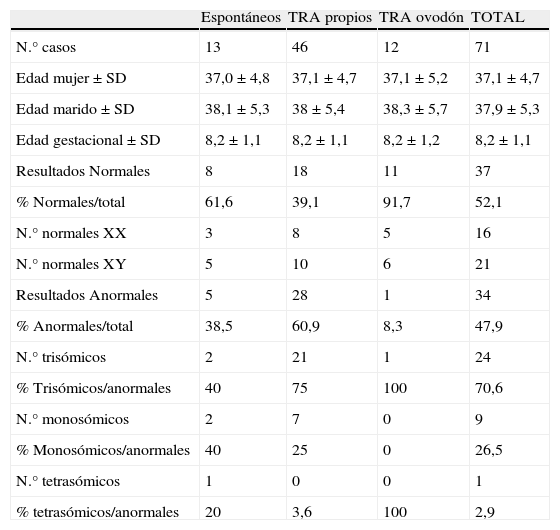
En la figura 1 se resumen las principales etapas del ensayo: 1) obtención del ADN genómico, 2) marcaje del ADN muestra y los ADN de referencia (masculino y femenino) con nucleótidos biotinizados, 3) purificación del ADN marcado, 4) hibridación del ADN marcado y purificado con una mezcla BoBs, 5) tras una serie de lavados las microesferas se incuban con estreptavidina/ficoeritrina que se une al ADN biotinizado, y 6) las microesferas se lavan y se resuspenden de nuevo para realizar la medición mediante un sistema instrumental Luminex®100/200™ con el software xPONENT® 3.1 y el software de análisis BoBsoft 2.0™. El protocolo en el laboratorio requiere unas 24-48 horas (incluyendo extracción de ADN).
El ADN se extrae de forma manual (QIAamp DNA Mini Kit, Qiagen, Inc., Chatsworth, CA, USA), se amplifica y se marca con un conjunto de cebadores cortos y aleatorios, posteriormente se hibrida sobre microesferas durante 16 horas y tras varios lavados se añade un fluoróforo que marca el ADN unido a la superficie de las microesferas. Paralelamente, se hibrida una referencia masculina y otra femenina. Después de varios lavados se separan y analizan las microesferas mediante la tecnología Luminex®. Tras evaluar más de 50 microesferas de cada región del genoma se obtienen unos determinados valores a partir de los cuales el software genera una tabla con los resultados y una representación gráfica de las ganancias y las pérdidas de material genético en las muestras problema. Así, las microesferas que representan regiones del genoma sin cambios en el número de copias deben de tener un valor igual a 1 que indica que el número de copias es igual a las referencias o controles. Las deleciones de regiones genómicas son detectadas por tener valores menores de 1 y las duplicaciones corresponden con valores mayores de 1.
Esta técnica ha sido testada para detectar alteraciones en sangre, tejidos (VC o resto abortivo) o líquido amniótico y ha sido validada con cariotipo convencional. Muestras en las que se conocían las alteraciones, fueron diluidas con muestras de referencia, a distintas concentraciones para simular mosaicismos y definir el límite de detección de mosaicismos o contaminación materna estableciéndose que podría detectar la presencia de otra línea celular en porcentajes superiores al 10-20%3.
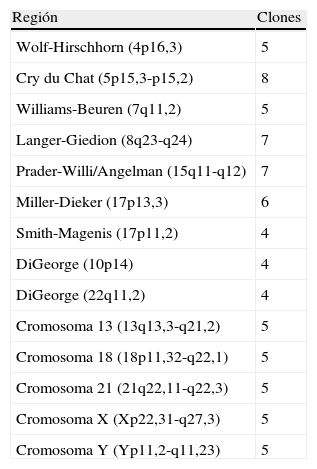
Particularidades del Prenatal-BoBs™Prenatal-BoBs™ evalúa 75 regiones cromosómicas diferentes del genoma. Con este ensayo se pueden detectar aneuploidías comunes de los cromosomas 13, 18, 21, X e Y y los 9 síndromes previamente enumerados (tabla 1). Las microdeleciones estudiadas han sido seleccionadas siguiendo los siguientes criterios: síndromes con prevalencia relativamente alta (1/4.000 hasta 1/200.000), mortalidad y/o morbilidad significativa, signos ecográficos silentes o leves/inespecíficos (p. ej. RCIU), correlación genotipo-fenotipo clara, síndromes no detectables por cariotipo convencional y síndromes en los que la deleción es el mecanismo mayoritario que produce la patología3–5.
descripción de los síndromes de microdeleción y regiones cromosómicas incluidas en Prenatal-BoBs™
| Región | Clones |
| Wolf-Hirschhorn (4p16,3) | 5 |
| Cry du Chat (5p15,3-p15,2) | 8 |
| Williams-Beuren (7q11,2) | 5 |
| Langer-Giedion (8q23-q24) | 7 |
| Prader-Willi/Angelman (15q11-q12) | 7 |
| Miller-Dieker (17p13,3) | 6 |
| Smith-Magenis (17p11,2) | 4 |
| DiGeorge (10p14) | 4 |
| DiGeorge (22q11,2) | 4 |
| Cromosoma 13 (13q13,3-q21,2) | 5 |
| Cromosoma 18 (18p11,32-q22,1) | 5 |
| Cromosoma 21 (21q22,11-q22,3) | 5 |
| Cromosoma X (Xp22,31-q27,3) | 5 |
| Cromosoma Y (Yp11,2-q11,23) | 5 |
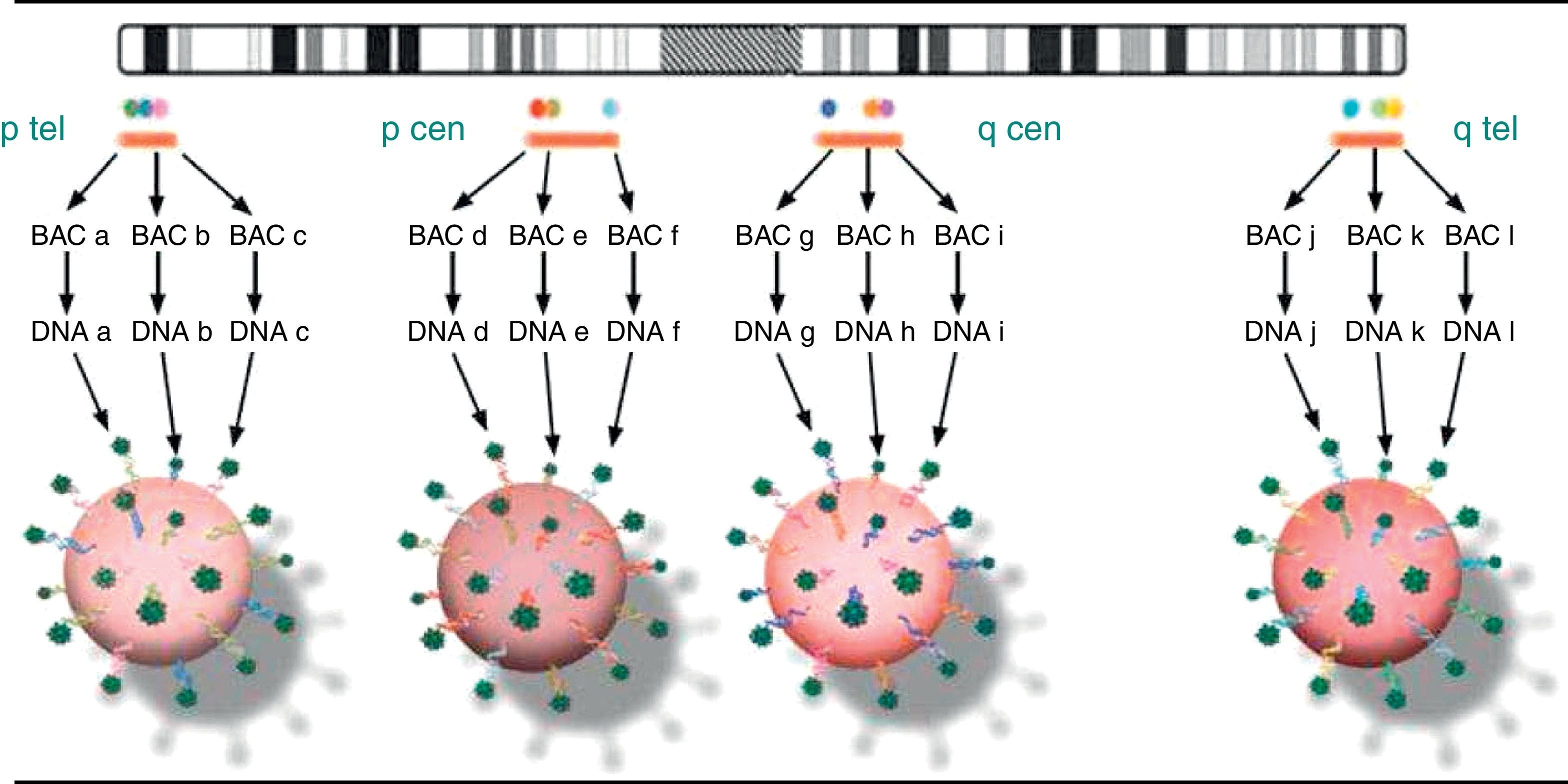
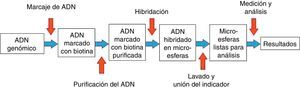
En el caso del KaryoLite-BoBs™, el fundamento técnico es el mismo que el expuesto hasta ahora, con la particularidad de que incluye 91 microesferas cada una de las cuales está compuesta por 3 BACs y mapean en las regiones proximales y terminales de cada brazo de cada uno de los 24 cromosomas (fig. 2). Es importante mencionar que la técnica utilizada para la obtención de las muestras ha sido la histeroembrioscopia dirigida que permite obtener muestras del feto o del trofoblasto, minimizando el riesgo de contaminación materna6.
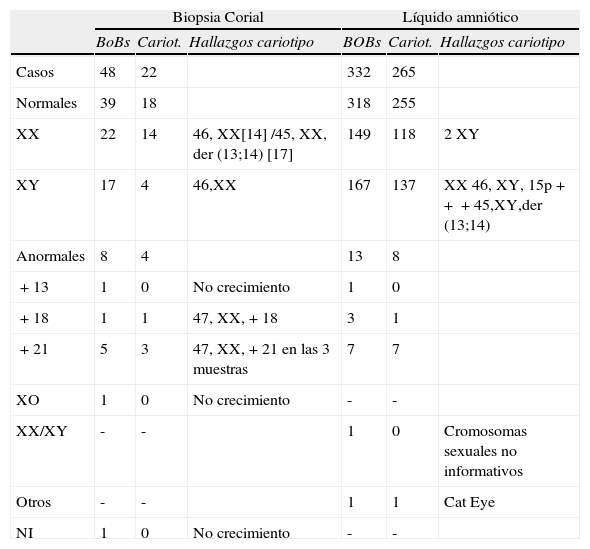
Resultados y conclusionesDiagnóstico prenatal rápido mediante Prenatal-BoBs™Los resultados recopilados tras la utilización de esta técnica se muestran en la tabla 2. Se obtuvieron resultados concluyentes mediante Prenatal-BoBs™ en 379 de las 380 muestras analizadas (99,7%). En 287 muestras se obtuvo un cariotipo convencional (75,5% de los casos). En los líquidos amnióticos, no en todos los casos se pudo obtener crecimiento celular (n=7; 2,1%) o no se solicitó (n=56; 16,9%) y en las biopsias coriales el número de casos sin crecimiento fue mayor (n=18; 37,5%) y los no solicitados algo menor (n=7; 14,6%). Tan solo una vellosidad corial resultó no informativa con la técnica de Prenatal-BoBs™ (2,1%). Y un caso de no informatividad de los cromosomas sexuales (0,3%) en los líquidos amnióticos.
Resultados obtenidos mediante la técnica de Prenatal-BoBs™ en LA y VC
| Biopsia Corial | Líquido amniótico | |||||
| BoBs | Cariot. | Hallazgos cariotipo | BOBs | Cariot. | Hallazgos cariotipo | |
| Casos | 48 | 22 | 332 | 265 | ||
| Normales | 39 | 18 | 318 | 255 | ||
| XX | 22 | 14 | 46, XX[14] /45, XX, der (13;14) [17] | 149 | 118 | 2 XY |
| XY | 17 | 4 | 46,XX | 167 | 137 | XX 46, XY, 15p+++45,XY,der (13;14) |
| Anormales | 8 | 4 | 13 | 8 | ||
| +13 | 1 | 0 | No crecimiento | 1 | 0 | |
| +18 | 1 | 1 | 47, XX,+18 | 3 | 1 | |
| +21 | 5 | 3 | 47, XX,+21 en las 3 muestras | 7 | 7 | |
| XO | 1 | 0 | No crecimiento | - | - | |
| XX/XY | - | - | 1 | 0 | Cromosomas sexuales no informativos | |
| Otros | - | - | 1 | 1 | Cat Eye | |
| NI | 1 | 0 | No crecimiento | - | - | |
NI: no informativos.
Por otro lado el porcentaje de concordancia entre Prenatal-BoBs™ y cariotipo fue 1,4% (n=4), ya que en todos los casos el sexo diagnosticado no coincidía entre ambas técnicas pudiéndose tratar de contaminaciones maternas importantes en aproximadamente la mitad de los casos. El resto de discrepancias fueron anomalías que no abarca el Prenatal-BoBs™, como translocaciones Robertsonianas (n=2; 2,8%) o un caso de un polimorfismo en el cromosoma 15. Una de las limitaciones de esta técnica es su incapacidad para detectar poliploidías. Sin embargo, en nuestra experiencia en los casos en los que también se realizó cariotipo, no encontramos ninguna muestra con poliploidía.
En los casos analizados con aCGH, en todos encontramos concordancia entre el resultado del Prenatal-BoBs™ y el del array. De todos los analizados (n=8), el resultado fue una dotación cromosómica normal excepto en un caso en el que encontramos un síndrome de Cat Eye, duplicación en la región (22q11,2), que fue detectado con ambas técnicas debido a que se localiza en la misma región que el síndrome de DiGeorge, causado por microdeleción que está contemplado dentro del Prenatal-BoBs™.
Estudio de restos abortivos mediante KaryoLite-BoBs™Los resultados obtenidos con esta técnica se muestran en la tabla 3. De un total de 71 muestras, en 58 (81,7%) las pacientes habían concebido mediante el uso de técnicas de reproducción asistida (TRA), 46/58 (79,3%) con ovocitos propios, 12/58 (20,7%) recurrieron a la donación de ovocitos y 13 (18,3%) tuvieron embarazos espontáneos. Estas mujeres con gestaciones espontáneas las consideraremos grupo control. La edad media de los pacientes fue de 37,0 años en el caso de embarazos espontáneos, 37,1 años en TRA con ovocitos propios y 37,1 años en receptoras de ovocitos. No observamos diferencias estadísticamente significativas en cuanto a la edad entre los grupos analizados.
Resultados obtenidos mediante KaryoLite-BoBs™
| Espontáneos | TRA propios | TRA ovodón | TOTAL | |
| N.° casos | 13 | 46 | 12 | 71 |
| Edad mujer±SD | 37,0±4,8 | 37,1±4,7 | 37,1±5,2 | 37,1±4,7 |
| Edad marido±SD | 38,1±5,3 | 38±5,4 | 38,3±5,7 | 37,9±5,3 |
| Edad gestacional±SD | 8,2±1,1 | 8,2±1,1 | 8,2±1,2 | 8,2±1,1 |
| Resultados Normales | 8 | 18 | 11 | 37 |
| % Normales/total | 61,6 | 39,1 | 91,7 | 52,1 |
| N.° normales XX | 3 | 8 | 5 | 16 |
| N.° normales XY | 5 | 10 | 6 | 21 |
| Resultados Anormales | 5 | 28 | 1 | 34 |
| % Anormales/total | 38,5 | 60,9 | 8,3 | 47,9 |
| N.° trisómicos | 2 | 21 | 1 | 24 |
| % Trisómicos/anormales | 40 | 75 | 100 | 70,6 |
| N.° monosómicos | 2 | 7 | 0 | 9 |
| % Monosómicos/anormales | 40 | 25 | 0 | 26,5 |
| N.° tetrasómicos | 1 | 0 | 0 | 1 |
| % tetrasómicos/anormales | 20 | 3,6 | 100 | 2,9 |
La edad gestacional media en el momento de la pérdida fue de 8,2±1,1 semanas en el grupo control, de 8,2±1,1 semanas en mujeres que utilizaron gametos propios y de 8,2±1,2 semanas en el caso de mujeres que usaron una donante de ovocitos. En todos los casos la gestación se detuvo antes de la semana 11. No encontramos diferencias estadísticamente significativas tampoco para la edad gestacional de cada grupo.
De los 13 embarazos espontáneos, 8 (61,5%) de los legrados tuvieron resultado normal mientras que 5 (38,5%) tuvieron resultado anormal. Cuando analizamos los resultados de estudios de legrados de los 58 embarazos de TRA, observamos que 29 (50%) presentaban resultado normal y 29 (50%) resultado anormal, lo que coincidía con los datos bibliográficos de los que disponemos7,8. Si los restos fetales procedían de embarazos tras TRA con ovocitos propios, 18 de los 46 casos (39,1%) tuvieron resultado normal y 28 resultado anormal (60,9%). Por el contrario, en los histerolegrados procedentes de embarazos con ovodón, la mayoría presentaron resultado normal (11/12; 91,7%), mientras que 1 solo presentó resultado anormal (8,3%). Cuando comparamos los resultados de anormalidad en cada uno de los grupos estudiados con el grupo control (38,5%) no observamos diferencias significativas en TRA y ovocitos propios (p=0,2625) y tampoco en donación de ovocitos (p=0,1958). Sin embargo, sí encontramos diferencias estadísticamente significativas (p=0,0035) cuando comparamos resultados de anormalidad entre TRA con ovocitos propios y TRA con donación de ovocitos. Estos resultados citogenéticos en receptoras de ovocitos procedentes de mujeres jóvenes menores de 35 años, con cariotipo normal, y testadas para alteraciones genéticas de alta prevalencia, nos llevan a pensar que son las características del entorno materno y no la calidad ovocitaria, el factor determinante en el desenlace de estas gestaciones.
De los 34 casos estudiados con resultado anormal, 24 fueron trisómicos, 9 monosómicos y uno de ellos (de embarazo espontáneo) tetrasómico. En cuanto a los 24 casos con trisomías detectadas, 2 de 5 (40%) procedían de gestación espontánea, 21 de 28 (75%) de gestación con gameto propio y un solo caso en receptoras de ovocitos. No encontramos diferencias estadísticamente significativas cuando comparamos los 3 grupos estudiados.
Con respecto a los 9 casos monosómicos, 2 de 5 (40%) procedían de gestación espontánea y 7 de 28 (25%) de gestación con gameto propio. No encontramos diferencias estadísticamente significativas cuando comparamos los 2 grupos citados. Ninguna de las muestras procedentes de TRA con donación de ovocitos mostró un resultado monosómico.
ConclusionesPrenatal-BoBs™Prenatal-BoBs™ surge como una tecnología para ofrecer un resultado muy rápido a un coste más reducido que otras técnicas como por ejemplo la FISH. A diferencia de la FISH, este ensayo permite la detección de, además de las aneuploidías de los cromosomas 13, 18 y 21 y de los cromosomas sexuales, la detección de 9 síndromes originados mayoritariamente por microdeleción. Así, se puede considerar como un buen sustituto de FISH y de la QF-PCR porque estas 2 técnicas no proporcionan información sobre ese panel de síndromes.
Entre las ventajas de esta novedosa técnica destacamos que:
- -
Este ensayo no requiere de cultivo celular previo, lo que nos permite obtener resultados en 24-48h
- -
Alto rendimiento
- -
No requiere sangre de los padres
- -
Disponemos de sondas de FISH para confirmar los hallazgos.
- -
Requiere poca cantidad de DNA en comparación con aCGH
- -
Permite el estudio simultáneo de varios pacientes (hasta 94 pacientes)
Entre sus limitaciones encontramos:
- -
Un resultado negativo no excluye la presencia de otros síndromes (desórdenes con otras etiologías no pueden descartarse, como es el caso de las mutaciones puntuales)
- -
No detecta translocaciones equilibradas, ni inversiones
- -
No detecta poliploidías y mosaicismos de bajo grado
- -
No detecta alteraciones segmentales
Prenatal-BoBs™ es un método efectivo y rápido, sin embargo, es apto solo para la detección de alteraciones cromosómicas numéricas grandes y, por tanto, debería complementarse con un cariotipo convencional y/o un aCGH.
KaryoLite-BoBs™Hasta ahora, el estudio citogenético clásico de los restos fetales, se ha llevado a cabo mediante el cultivo celular de los mismos. Con este tipo de técnicas se ha descrito una incidencia de anomalías cromosómicas fetoplacentarias de aproximadamente el 50% de los abortos espontáneos del primer trimestre9. Sin embargo, la tasa de fracaso (no crecimiento) en este tipo de cultivos oscila entre el 5 y el 42%10. No se sabe bien por qué el éxito del crecimiento de los restos fetales es, en ocasiones, tan bajo, aunque se especula con la posibilidad de que sean las propias cromosomopatías existentes en estas células las que no permiten una proliferación celular normal10. Por lo tanto, no siempre es posible el diagnóstico citogenético adecuado de los restos abortivos. En este escenario las técnicas de biología molecular, como la expuesta anteriormente, pueden dar solución a los obstáculos a los que nos enfrentamos en el laboratorio cuando trabajamos con este tipo de muestras.
Dado el fundamento metodológico de KaryoLite-BoBs™, esta técnica nos ha permitido la detección de aneuploidías en el 100% de los tejidos abortivos analizados, mientras que en el cariotipo convencional la obtención de un resultado concluyente está sujeta a la calidad de los cromosomas obtenidos tras el cultivo. Destacaremos, además, que esta técnica no requiere cultivo celular, lo que nos evita problemas de contaminación y nos proporciona resultados en 48h. Puesto que se trata de una técnica basada en la cuantificación del ADN, se pueden infradiagnosticar mosaicismos de bajo grado, poliploidias y anomalias estructurales equilibradas.
Por lo expuesto, consideramos que KaryoLite-BoBs™ se convierte en una interesante herramienta para determinar la etiología cromosómica de las perdidas gestacionales sin necesidad de cultivo celular.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
El presente trabajo no hubiera podido ser realizado sin la contribución de los facultativos de las diferentes Unidades de Obstetricia, Ginecología y Reproducción que realizaron las amniocentesis, biopsia de vellosidades coriales e histeroembroscopias. Queremos agradecer la contribución de las citogenetistas M.a Carmen Martínez y Laura Rodríguez que realizaron los cariotipos de control en todas las muestras y también queremos mencionar el trabajo realizado en el laboratorio por nuestras compañeras Maria Ruíz, Miriam Freixinós, Asunción Martínez y Raquel Gimeno.