El diagnóstico prenatal citogenético durante el primer trimestre de gestación se realiza a partir de biopsias de vellosidad corial. Para la obtención de metafases se utilizan dos métodos: el cultivo corto o semidirecto (STC) y cultivo largo (LTC). La principal ventaja del STC es que no presenta contaminación materna y la del LTC es que no hay descritos en la literatura falsos negativos. Se considera que la combinación de las dos técnicas (STC y LTC) es la estrategia diagnóstica más eficaz para este tipo de estudios.
La técnica de PCR cuantitativa fluorescente (QF-PCR) permite evaluar las aneuploidías más frecuentemente implicadas en el diagnóstico prenatal en 24-48 horas en muestras de vellosidad corial.
El objetivo de este trabajo es evaluar la combinación de QF-PCR y LTC como sustituto de las clásicas STC y LTC para el diagnóstico prenatal en muestras de vellosidad corial. Para ello presentamos nuestra experiencia en 900 muestras de vellosidad corial.
First trimester cytogenetic prenatal diagnosis is performed on chorionic villus biopsies. Two methods are used to obtain metaphases: the short-term or semi-direct culture (STC) and long term culture (LTC). The main advantage of STC is that there is no risk of maternal contamination, and of LTC that no false-negative findings are described in the literature. It is considered that the combination of the two techniques (STC and LTC) is the most effective diagnostic strategy for this type of study.
The technique of quantitative fluorescent PCR (QF-PCR) allows the evaluation of aneuploidy most frequently involved in prenatal diagnosis in 24-48 hours in chorionic villus samples.
The aim of this study is to evaluate the combination of QF-PCR and LTC as a substitute for classical STC and LTC for prenatal diagnosis in chorionic villus samples. We present our experience in 900 chorionic villus samples.
El diagnóstico prenatal citogenético en el 1.er trimestre de gestación se realiza a partir de una muestra de vellosidad corial. Se utilizan de forma rutinaria dos tipos de cultivo: el cultivo corto (STC), en el cual se analizan células procedentes del citotrofoblasto que se dividen espontáneamente, y el cultivo largo (LTC), en el cual se analizan células del mesénquima que solo se dividen tras un cultivo celular que puede durar dos o tres semanas.
El principal problema en el estudio citogenético de este tipo de muestras radica en la detección de mosaicos cromosómicos confinados a la placenta y no presentes en el feto1–6. Este tipo de situaciones son más frecuentes cuando se obtiene el cariotipo a partir del STC que cuando se obtiene a partir del LTC. Esto se debe a que en el LTC las células (provenientes del mesodermo) son embriológicamente mucho más próximas al tejido fetal. Se ha observado un 0,15%7 de falsos positivos con la técnica de STC. Este problema es prácticamente inexistente en el LTC, por lo que diferentes estudios han demostrado que la combinación de ambas o, si no es posible, el realizar únicamente el LTC es la estrategia diagnóstica más indicada para el estudio citogenético en vellosidad corial5,8–10. La técnica STC presenta la menor fiabilidad y puede conducir a la terminación innecesaria del embarazo en algunos casos11,12.
Otro inconveniente derivado del estudio citogenético del STC es que los cromosomas suelen presentar menor resolución de bandas G que el LTC5. A pesar de los inconvenientes del STC es un método ampliamente usado en diferentes laboratorios para el estudio de la vellosidad corial debido a que no hay casos descritos de contaminación materna y puede darse un resultado en 3-4 días. Estas ventajas también las presenta el estudio mediante QF-PCR, una técnica molecular que permite evaluar las aneuploidías más frecuentemente implicadas en el diagnóstico prenatal (los autosomas 13, 18, 21 y los cromosomas sexuales X e Y), detectando un 92% de las anomalías cromosómicas clínicamente relevantes en 24 horas13. La QF-PCR presenta importantes ventajas frente al STC, principalmente que permite el diagnóstico a partir de muy poca cantidad de muestra (gracias a ello se puede destinar gran cantidad de muestra para el cultivo), no necesita tejido vivo y es capaz de determinar la presencia de contaminación materna en una muestra.
El objetivo que nos planteamos es valorar la efectividad de la sustitución del STC por la QF-PCR en la realización del diagnóstico prenatal citogenético en vellosidad corial, combinada con el LTC.
Pacientes y métodosEntre los años 2001 y 2009 se ha realizado el estudio de 900 muestras de vellosidad corial. Las muestras se han obtenido a partir de punciones trans-cervicales o trans-abdominales, en la 10- 12 semana de gestación.
De las 900 muestras, 81 fueron procesadas por los métodos STC y LTC; a 66 se les realizó QF-PCR, STC y LTC; y en 753 QF-PCR y LTC.
El STC se realizó según la técnica clásica de disgregación de la muestra en acético al 60%14. El LTC se realizó mediante la técnica de explantes15 y/o de digestión16. Al llegar la muestra al laboratorio se preparó descartando el material materno y los coágulos, se limpió mediante solución salina y se separaron 2 muestras de aproximadamente 0,1 mg de la vellosidad para realizar la QF-PCR. El resto de la muestra se procesó mediante la técnica de explantes con disgregación mecánica, o mediante la técnica de digestión con disgregación enzimática con colagenasa.
El estudio cromosómico se realizó a partir de extensiones cromosómicas teñidas con colorante Wright para la obtención de bandas G15. La fórmula cromosómica se describió de acuerdo al ISCN (International System Cytogenetics Nomenclature)17. En los cultivos de STC se analizaron 10 metafases y en el LTC 20 metafases pertenecientes a dos cultivos independientes.
La QF-PCR se realizó para detectar aneuploidías de los cromosomas 13, 18, 21, X e Y, siguiendo el protocolo recomendado por el fabricante (Aneufast). El ADN se extrajo a partir de fragmentos de 1-2 mm de vellosidad corial o unas gotas del producto de la digestión con colagenasa16 usando el producto Chelex (Instagene Matrix, Bio-Rad Laboratories, CA, EE. UU.)22. En caso de que el resultado fuera patológico se repetía el estudio a partir de un nuevo fragmento de 1-2 mm diferente de la muestra de vellosidad corial procedente del cultivo citogenético o del material sobrante conservado a 4°C.
ResultadosSe ha obtenido un resultado citogenético en el 94% de los casos. En 59 de los 147 casos analizados por STC (40%) no fue posible estudiar el cariotipo; en 7 de ellos debido a la escasa muestra recibida, en 42 al no obtener metafases y en 10 debido la baja calidad en las metafases.
En 54 de los 900 casos analizados por LTC (6%) no fue posible estudiar el cariotipo; en 22 no se obtuvo crecimiento, en 4 debido al escaso material, en 17 el material era materno y en 11 casos hubo contaminaciones por cándidas, probablemente asociadas al método de extracción de la muestra (extracción trans-cervical en todos los casos). La QF-PCR obtuvo resultados en todos los casos realizados (819 muestras).
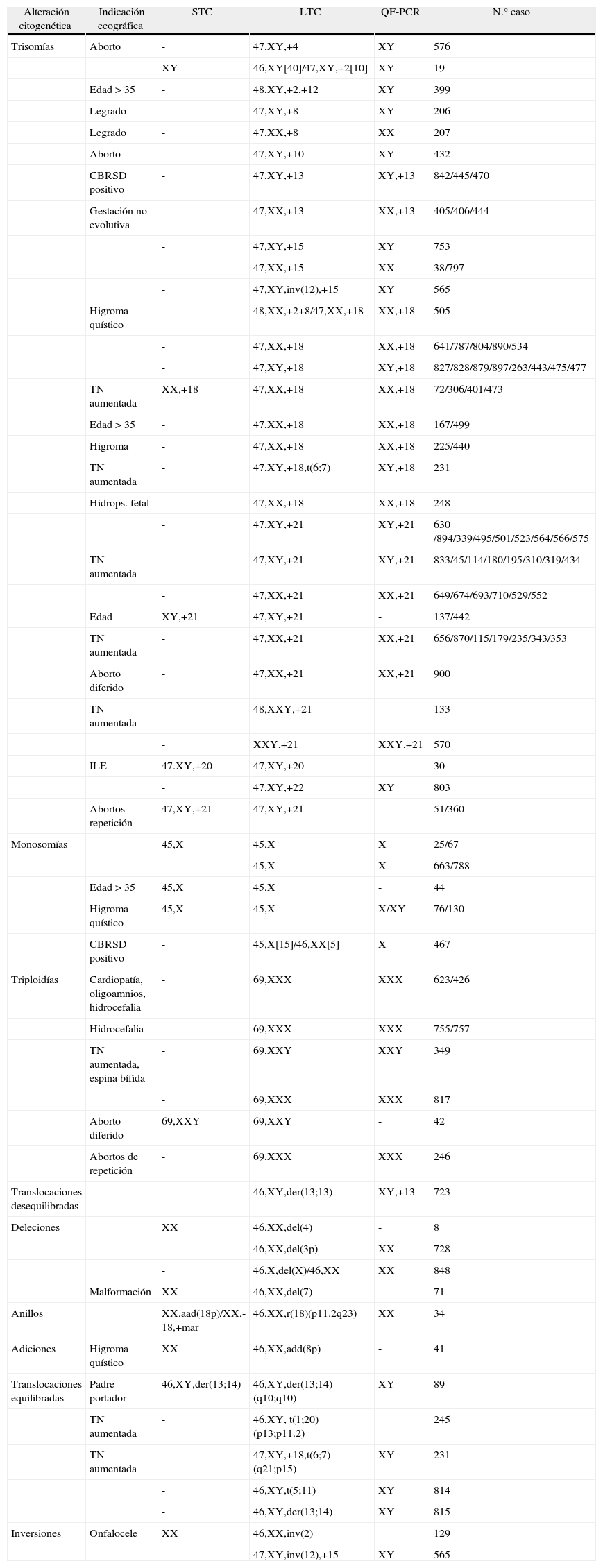
En total, se han detectado 109 alteraciones citogenéticas (tabla 1): 79 trisomías, 8 monosomías X, 8 triploidías, 4 deleciones, 5 translocaciones equilibradas, 2 inversiones, una adición de material cromosómico, un anillo (fig. 1) y una translocación desequilibrada.
Alteraciones citogenéticas detectadas en 109 casos de 900 vellosidades coriales
| Alteración citogenética | Indicación ecográfica | STC | LTC | QF-PCR | N.° caso |
| Trisomías | Aborto | - | 47,XY,+4 | XY | 576 |
| XY | 46,XY[40]/47,XY,+2[10] | XY | 19 | ||
| Edad>35 | - | 48,XY,+2,+12 | XY | 399 | |
| Legrado | - | 47,XY,+8 | XY | 206 | |
| Legrado | - | 47,XX,+8 | XX | 207 | |
| Aborto | - | 47,XY,+10 | XY | 432 | |
| CBRSD positivo | - | 47,XY,+13 | XY,+13 | 842/445/470 | |
| Gestación no evolutiva | - | 47,XX,+13 | XX,+13 | 405/406/444 | |
| - | 47,XY,+15 | XY | 753 | ||
| - | 47,XX,+15 | XX | 38/797 | ||
| - | 47,XY,inv(12),+15 | XY | 565 | ||
| Higroma quístico | - | 48,XX,+2+8/47,XX,+18 | XX,+18 | 505 | |
| - | 47,XX,+18 | XX,+18 | 641/787/804/890/534 | ||
| - | 47,XY,+18 | XY,+18 | 827/828/879/897/263/443/475/477 | ||
| TN aumentada | XX,+18 | 47,XX,+18 | XX,+18 | 72/306/401/473 | |
| Edad>35 | - | 47,XX,+18 | XX,+18 | 167/499 | |
| Higroma | - | 47,XX,+18 | XX,+18 | 225/440 | |
| TN aumentada | - | 47,XY,+18,t(6;7) | XY,+18 | 231 | |
| Hidrops. fetal | - | 47,XX,+18 | XX,+18 | 248 | |
| - | 47,XY,+21 | XY,+21 | 630 /894/339/495/501/523/564/566/575 | ||
| TN aumentada | - | 47,XY,+21 | XY,+21 | 833/45/114/180/195/310/319/434 | |
| - | 47,XX,+21 | XX,+21 | 649/674/693/710/529/552 | ||
| Edad | XY,+21 | 47,XY,+21 | - | 137/442 | |
| TN aumentada | - | 47,XX,+21 | XX,+21 | 656/870/115/179/235/343/353 | |
| Aborto diferido | - | 47,XX,+21 | XX,+21 | 900 | |
| TN aumentada | - | 48,XXY,+21 | 133 | ||
| - | XXY,+21 | XXY,+21 | 570 | ||
| ILE | 47.XY,+20 | 47,XY,+20 | - | 30 | |
| - | 47,XY,+22 | XY | 803 | ||
| Abortos repetición | 47,XY,+21 | 47,XY,+21 | - | 51/360 | |
| Monosomías | 45,X | 45,X | X | 25/67 | |
| - | 45,X | X | 663/788 | ||
| Edad>35 | 45,X | 45,X | - | 44 | |
| Higroma quístico | 45,X | 45,X | X/XY | 76/130 | |
| CBRSD positivo | - | 45,X[15]/46,XX[5] | X | 467 | |
| Triploidías | Cardiopatía, oligoamnios, hidrocefalia | - | 69,XXX | XXX | 623/426 |
| Hidrocefalia | - | 69,XXX | XXX | 755/757 | |
| TN aumentada, espina bífida | - | 69,XXY | XXY | 349 | |
| - | 69,XXX | XXX | 817 | ||
| Aborto diferido | 69,XXY | 69,XXY | - | 42 | |
| Abortos de repetición | - | 69,XXX | XXX | 246 | |
| Translocaciones desequilibradas | - | 46,XY,der(13;13) | XY,+13 | 723 | |
| Deleciones | XX | 46,XX,del(4) | - | 8 | |
| - | 46,XX,del(3p) | XX | 728 | ||
| - | 46,X,del(X)/46,XX | XX | 848 | ||
| Malformación | XX | 46,XX,del(7) | 71 | ||
| Anillos | XX,aad(18p)/XX,-18,+mar | 46,XX,r(18)(p11.2q23) | XX | 34 | |
| Adiciones | Higroma quístico | XX | 46,XX,add(8p) | - | 41 |
| Translocaciones equilibradas | Padre portador | 46,XY,der(13;14) | 46,XY,der(13;14)(q10;q10) | XY | 89 |
| TN aumentada | - | 46,XY, t(1;20)(p13;p11.2) | 245 | ||
| TN aumentada | - | 47,XY,+18,t(6;7)(q21;p15) | XY | 231 | |
| - | 46,XY,t(5;11) | XY | 814 | ||
| - | 46,XY,der(13;14) | XY | 815 | ||
| Inversiones | Onfalocele | XX | 46,XX,inv(2) | 129 | |
| - | 47,XY,inv(12),+15 | XY | 565 |
CBRSD: cribado, selección bioquímica de riesgo de síndrome de Down; ILE: interrupción legal embarazo; LTC: cultivo largo; QF-PCR: técnica de PCR cuantitativa fluorescente; STC: cultivo corto; TN: traslucencia nucal.
En el STC se observaron 3 falsos negativos asociados a baja resolución, 46,XX,del(4)(q31.2), 46,XX,add(8p), 46,XX,del (7)(q32) y un mosaico confinado a placenta, 46,XY/47,XY,+2.
En el LTC se han detectado 2 casos de mosaicismo confinado a placenta, un caso de trisomía 18 en mosaico y otro con cariotipo 48,XY,+2,+12. En ambos casos se observó un cariotipo normal en líquido amniótico. No se conoce ningún caso de falso negativo.
En 9 casos se detectó contaminación materna en el LTC (1%). En dos casos en que la contaminación fue masiva, la QF-PCR permitió detectar el problema y realizar un diagnóstico correcto. En un caso (n.° 428) se diagnosticó un cariotipo 46,XX mediante citogenética convencional. La discrepancia con el resultado obtenido por QF-PCR (XY) llevó a una reevaluación del cultivo que permitió hallar un reducido número de células fetales (4/400). En otro caso (n.° 443) se obtuvo un cariotipo 46,XX en todas las células analizadas procedentes de un cultivo de la digestión de la vellosidad corial. Como la QF-PCR indicaba un sexo masculino y una trisomía 18, se procesaron cultivos adicionales procedentes de explantes, en los que se confirmó el resultado 47,XY,+18. (tabla 2).
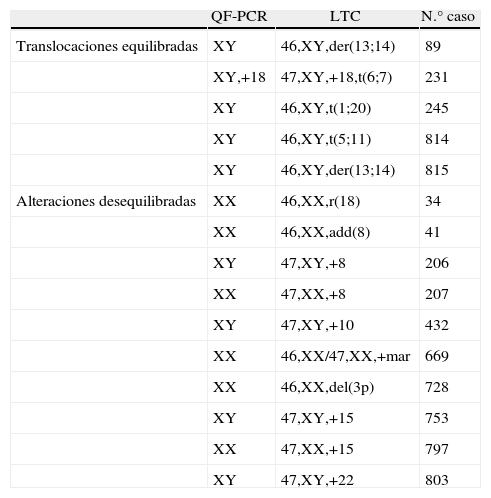
La QF-PCR no pudo detectar 15 anomalías (1,8%) debido a las limitaciones propias de la técnica: 6 aneuploidías (2 casos de trisomía 8, una trisomía 10, dos trisomías 15, una trisomía 22), un cromosoma marcador adicional de muy pequeño tamaño, una deleción de 3p, una adición en el 8, un anillo del cromosoma 18 (fig. 1) y cinco translocaciones equilibradas (tabla 3). En 10 casos (1,2%) se trataba de alteraciones que tenían consecuencias para el feto.
Alteraciones citogenéticas detectadas en LTC y no detectadas en QF-PCR en 819 muestras
| QF-PCR | LTC | N.° caso | |
| Translocaciones equilibradas | XY | 46,XY,der(13;14) | 89 |
| XY,+18 | 47,XY,+18,t(6;7) | 231 | |
| XY | 46,XY,t(1;20) | 245 | |
| XY | 46,XY,t(5;11) | 814 | |
| XY | 46,XY,der(13;14) | 815 | |
| Alteraciones desequilibradas | XX | 46,XX,r(18) | 34 |
| XX | 46,XX,add(8) | 41 | |
| XY | 47,XY,+8 | 206 | |
| XX | 47,XX,+8 | 207 | |
| XY | 47,XY,+10 | 432 | |
| XX | 46,XX/47,XX,+mar | 669 | |
| XX | 46,XX,del(3p) | 728 | |
| XY | 47,XY,+15 | 753 | |
| XX | 47,XX,+15 | 797 | |
| XY | 47,XY,+22 | 803 |
LTC: cultivo largo; QF-PCR: técnica de PCR cuantitativa fluorescente.
En un caso (n.° 848) el resultado de la QF-PCR fue normal y de sexo femenino y el cariotipo en LTC indicó una deleción intersticial del cromosoma X en mosaico, con fórmula cromosómica 46,X,del(X)(q26q28)[24]/46,XX[6]. Los marcadores del cromosoma X analizados mediante la QF-PCR ayudaron a determinar los puntos de rotura con más precisión. Esta alteración detectada en LTC puede tratarse de un mosaicismo confinado a placenta, o bien de una contaminación materna de la línea normal y con afectación fetal. Ambas posibilidades se podrían descartar mediante un análisis en líquido amniótico que no se realizó debido a la decisión de la pareja de continuar la gestación independientemente de la constitución cromosómica fetal.
DiscusiónEl análisis citogenético a partir de muestras de vellosidad corial ha demostrado ser un método fiable para analizar la constitución cromosómica fetal en el primer trimestre, si bien con resultados algo inferiores a los obtenidos en segundo trimestre a partir de líquido amniótico1–5. Existen diferencias entre el análisis a partir de células citotrofoblásticas y mesenquimáticas, que se traducen en una menor fiabilidad del STC. Por otra parte, el LTC presenta el problema de la contaminación materna y la demora del resultado en dos o tres semanas. En consecuencia, es conveniente el análisis en paralelo mediante STC y LTC5,8,18,19. El principal problema en la aplicación en paralelo del STC y LTC es la necesidad de una gran cantidad de muestra, no siempre disponible. En estos casos se recomienda primar el LTC, retrasando el resultado hasta las dos o tres semanas de recepción de la muestra. Adicionalmente, la técnica de STC requiere una gran vitalidad en el tejido analizado, no siempre presente en muestras de difícil extracción o que han sufrido un transporte que a veces puede ser largo. La sustitución del STC por la QF-PCR, una técnica molecular que requiere muy poca muestra y no depende de la presencia de células vivas, nos ha permitido realizar diagnósticos rápidos en la práctica totalidad de las muestras manteniendo la máxima fiabilidad diagnóstica al facilitar el estudio paralelo mediante LTC. En nuestro estudio hemos obtenido un 94% de éxito en la técnica de LTC, algo superior al descrito en la literatura, de aproximadamente un 85%5.
La QF-PCR ha permitido obtener resultados en 22 casos (2,7%) en los que la falta de viabilidad de la muestra impedía cualquier tipo de resultado por técnicas de citogenética convencional. Adicionalmente, al no requerir cultivo celular, la QF-PCR nos ha proporcionado los resultados de 11 casos (1,3%) en los que la contaminación por levaduras de cándidas del cultivo impidió el resultado citogenético clásico.
En la literatura se ha descrito que la presencia de anomalías cromosómicas en mosaico en el estudio prenatal de vellosidades coriales es de un 1-2%, un 80% de las cuales estan confinadas a placenta y no presentes en el feto3,20. La presencia de mosaicos confinados a placenta es superior en STC que en LTC. En nuestra experiencia, sustituir el STC por la QF-PCR nos ha permitido reducir a un 0,22% los mosaicos confinados a placenta, reduciendo la angustia generada en las gestantes y el número de amniocentesis de confirmación. Este descenso se puede deber a dos factores. Primero, la QF-PCR es una técnica muy dirigida a un tipo concreto de anomalías y, por tanto, anomalías numéricas en los cromosomas no analizados y la mayoría de las anomalías estructurales pasarán desapercibidas. Segundo, la QF-PCR no analiza únicamente el citotrofoblasto (tejido en el cual la proporción de mosaicos confinados a placenta es mucho mayor) sino también células del mesénquima.
En nuestra casuística hemos hallado 15 casos (1,8%) en los que la QF-PCR no ha detectado anomalías cromosómicas, 10 de ellas (1,2%) de riesgo potencial para el feto.
Estos datos confirman la utilidad de utilizar ambos ensayos en gestaciones de alto riesgo, ya la QF-PCR ha sido diseñada únicamente para la detección de las aneuploidías más frecuentes21-23.Una posible limitación del LTC es la contaminación materna4. La sustitución del STC (un método sin este problema y que permite el análisis de todos los cromosomas), por la QF-PCR presentaría el riesgo potencial de falsos negativos en el caso de que se produjese una contaminación materna en el LTC en presencia de una aneuploidía no detectable por el ensayo molecular.Sin embargo, en el curso de nuestro estudio, hemos observado 9 casos de contaminación materna en el LTC (1%), una frecuencia más cercana al rango inferior de los valores descritos en la literatura (0,91-6,4%) 1,24. En 7 casos el número de células maternas en el cultivo celular fue minoritario y no existió un riesgo de error diagnóstico. En dos casos la contaminación fue masiva y la QF-PCR permitió detectar el problema evitando un diagnóstico erróneo.
De forma similar a lo descrito en los estudios a partir de STC y LTC, se han publicado varios casos de discordancia entre QF-PCR y LTC25–28. En nuestra experiencia no hemos detectado ningún caso. Este problema, debido a la presencia de mosaicos placentarios, se agudiza debido a la poca representatividad de los pequeños fragmentos de material tradicionalmente utilizados en la QF-PCR. Para minimizar este problema, es recomendable realizar la QF-PCR a partir de material vellositario digerido y no de pequeños fragmentos.
En conclusión, los resultados obtenidos en nuestro estudio demuestran que en el análisis cromosómico prenatal a partir de vellosidades coriales, la combinación de las técnicas de QF-PCR y LTC permite reducir el número de casos sin resultado y el número de falsos positivos observados mediante la combinación clásica de STC y LTC.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.