La proteómica es una nueva tecnología, incluso algunos la llegan a considerar como una nueva ciencia, que permite analizar la expresión de múltiples proteínas a la vez en una única muestra. La proteómica puede facilitarnos identificar nuevos biomarcadores útiles para el pronóstico y el diagnóstico de las enfermedades cardiovasculares. El análisis proteómico está comenzando a dar resultados en el conocimiento de la aterosclerosis, isquemia miocárdica o hipertrofia ventricular, entre otras enfermedades cardiovasculares. Una importante aplicación de la proteómica es la farmacoproteómica, que trata de identificar biomarcadores que permitan predecir la respuesta farmacológica de un paciente. Además, mediante la farmacoproteómica también podemos identificar las variaciones en la expresión proteica asociada a un tratamiento farmacológico específico, lo que facilitará conocer los efectos clase y pleiotrópicos de los fármacos.
Proteomics is a new technology, even being considered as a new science that can analyse the expression of many proteins at the same time in a single sample. Proteomics may help us to identify new biomarkers for use in the diagnosis and prognosis of cardiovascular diseases. Proteomic analysis is starting to produce results in the knowledge of cardiovascular diseases including, atherosclerosis, myocardial ischaemia and ventricular hypertrophy. One important application of proteomics is pharmacoproteomics, which attempts to identify biomarkers that may be able to predict the pharmacological response of a patient. Furthermore, we may be able to use pharmacoproteomics to identify variations in protein expression associated to a specific pharmacological treatment, which will help in understanding the class and pleiotropic action of drugs.
El coste económico, social y humano debido a la alta incidencia de las enfermedades cardiovasculares continúa aumentando. La inversión realizada en investigación en esta área es una de las más altas, junto con la de las enfermedades oncológicas. Sin embargo, a pesar del aparente avance en el conocimiento de las enfermedades cardiovasculares, el número de dianas terapéuticas y de tratamientos realmente efectivos continúa siendo sorprendentemente limitado. Además, el número de biomarcadores cardiovasculares útiles es muy pequeño.
El objetivo último de la medicina moderna es poder alcanzar un tratamiento individualizado del paciente. Conocer la etiología de la enfermedad en cada paciente, la respuesta de cada paciente a los factores de riesgo, los factores que facilitan la progresión de la enfermedad, qué factores genéticos van a hacer al paciente susceptible a esta enfermedad, cuál es el perfil metabólico del paciente y cuáles son las proteínas que expresa el paciente que más nos ayuden a identificar el pronóstico de éste, así como su susceptibilidad al tratamiento, son en el momento actual centro de investigación. Para conseguir todos estos objetivos, debemos utilizar nuevas tecnologías que van desde el conocimiento de los genes contenidos en las células de los individuos (genoma), cuáles de ellos se expresan (transcriptómica), cuál es la funcionalidad de sus proteínas (metabolómica) y cuál es su perfil de expresión proteica (proteómica). Todas estas tecnologías se conocen ahora con el nombre de "nuevas ciencias", las cuales pueden favorecer la transición desde la investigación básica a la práctica clínica, con el fin último de alcanzar el desarrollo de la medicina personalizada.
La secuenciación del genoma humano ha sido sin duda uno de los mayores avances de las ciencias biomédicas de los últimos años. Una de las enseñanzas que nos ha proporcionado la secuenciación del genoma humano es que hay menos genes (alrededor de 30.000) de los que se esperaba. Esto ha llevado a dar una relevancia mayor a la diversidad de proteínas que se forman a partir de estos genes.
Las proteínas son las encargadas directas de las funciones biológicas. Se considera que en el ser humano hay entre 6 y 7 veces más proteínas que genes (entre 200.000 y 250.000 proteínas). Las proteínas, a diferencia de los genes, tienen una complejidad y variabilidad mayores. Mientras que el genoma es relativamente constante (incluso podría considerarse como un componente estático de la célula), el proteoma está continuamente cambiando, muchas veces simplemente por la interacción entre el genoma y el entorno, lo que confiere al proteoma la capacidad de ser dinámico. En este sentido, hay que recordar también que, una vez expresadas las proteínas, éstas están sujetas a modificaciones postranslacionales, entre las que se encuentran la proteólisis, la oxidación de grupos sulfhidrilo, la formación de puentes de disulfuro, las fosforilaciones, las glucosilaciones, las S-nitrosilaciones, la acilación grasa, la oxidación, etc. Estas modificaciones bioquímicas producen diferentes isoformas de una misma proteína, que posiblemente tengan incluso propiedades funcionales diferentes a las de la proteína original.
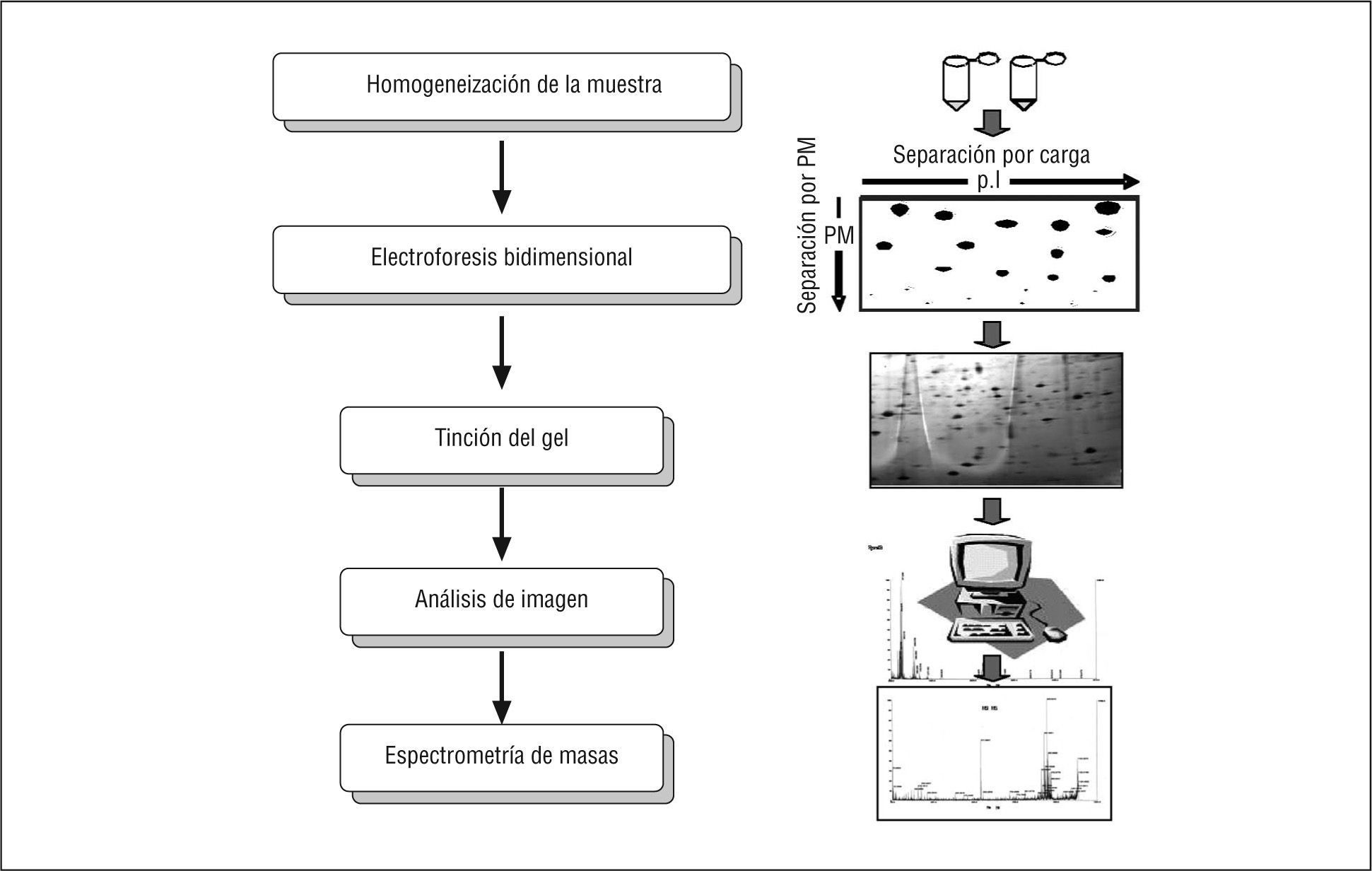
¿En qué consiste un estudio proteómico?Para realizar un estudio proteómico, se necesita integrar una serie de tecnologías. Un estudio proteómico conlleva la realización de electroforesis bidimensional, análisis de imagen, espectrometría de masas y bioinformática, lo que conlleva a contar con expertos en biología, biología molecular, espectrometría de masas y bioinformática (fig. 1).
Además de posibilitar la identificación de múltiples proteínas a la vez, una característica de la proteómica, en comparación con otras técnicas convencionales de identificación de proteínas, es que la proteómica no sólo permite detectar y cuantificar el total en la expresión de una proteína, sino también permite detectar y cuantificar la expresión de distintas isoformas de una misma proteína, algo que hasta el momento no era posible con el resto de técnicas empleadas para el análisis e identificación de proteínas, como el Western blot, el enzimoinmunoanálisis, la citometría de flujo u otras.
La electroforesis bidimensional es la base fundamental utilizada para desarrollar los mapas de expresión proteica o proteomas. La electroforesis bidimensional nos permite separar las proteínas en 2 dimensiones: el peso molecular y el punto isoeléctrico. Esta separación bidimensional de las proteínas dará lugar a un mapa de expresión proteica en el que cada proteína y sus isoformas estarán distribuidas en la coordenada formada por el peso molecular y el punto isoeléctrico. Esta coordenada, en principio, será única para cada proteína; en términos sencillos, es como el documento de identidad de la proteína.
La primera dimensión se realiza mediante el empleo de geles de poliacrilamida con un gradiente de pH inmovilizado, que garantiza la reproducibilidad entre geles. Estos geles están desarrollados sobre unas tiras denominadas IPG (Isoelectric Phocusing Gradient), y en el mercado hay tiras de IPG con distintos rangos de pH. La utilización de un rango amplio de pH (3–10), o uno más estrecho (4–7), dependerá de las características de pH de las proteínas contenidas en la muestra que queremos analizar, o incluso en el interés de amplificar la zona de estudio. Lógicamente, un rango de pH más estrecho resolverá más proteínas contenidas en ese rango, lo que en muchas ocasiones facilita su identificación y cuantificación.
El fundamento de esta primera separación de las proteínas según el punto isoeléctrico es que las proteínas presentes en el extracto a analizar migren a lo largo de la tira IPG al aplicar una corriente eléctrica y se distribuyan por toda su longitud hasta alcanzar cada proteína su punto isoeléctrico, es decir, el punto donde su carga neta es cero, y en ese momento se detienen. Por lo tanto, en la primera dimensión, las proteínas se separan en función de su carga. La segunda dimensión se realiza en geles SDS-PAGE, con lo que se logra la separación de las proteínas según su peso molecular. Una vez realizada la segunda dimensión, las proteínas presentes en el gel deben visualizarse para que podamos cuantificarlas y analizarlas. Hay distintos tipos de tinción del gel que, fundamentalmente, se diferencian según la sensibilidad a la hora de detectar proteínas. Destacan la tinción con azul de Coomassie, tinción radiactiva, tinción fluorescente y la tinción con plata.
Los métodos de tinción más empleados son, sin duda alguna, la tinción con plata y la tinción con azul de Coomassie. La tinción con plata es una de las tinciones más sensibles, perdura en el tiempo y se obtiene una muy buena calidad de la imagen. Además, este tipo de tinción puede ser compatible con el análisis por espectrometría de masas. Sin embargo, el método más recomendado de tinción para realizar la identificación de las proteínas mediante espectrometría de masas es el azul de Coomassie.
Una vez obtenido el gel bidimensional o el mapa de expresión proteico, el siguiente paso es el análisis de la imagen. Hoy día hay diversas herramientas informáticas (PD-QUEST, MELANIE, etc.) que facilitan mucho el estudio de este tipo de imágenes.
El empleo de este tipo de programas (software) va a permitir realizar un análisis completo de los mapas de expresión proteico, que permite incluso la comparación entre distintos grupos de mapas proteicos. Además, estas herramientas normalmente permiten la comparación visual con mapas proteómicos existentes en bases de datos disponibles en la red. La base de datos suiza SWISS-PROT (http://www.expasy.ch) es una de las más conocidas y empleadas.
La aplicación de la espectrometría de masas para identificar proteínas y péptidos se considera el avance principal en la caracterización e identificación de proteínas separadas mediante electroforesis bidimensional. En términos sencillos, la espectrometría de masas consiste en someter a una proteína a una digestión enzimática, normalmente con tripsina, que es capaz de romper la proteína en puntos o localizaciones específicas para obtener una serie de fragmentos peptídicos específicos, cuya masa va a servir para identificar la proteína de interés. En concreto, la tripsina rompe las proteínas en la zona carboxílica de los residuos de lisina y arginina, a menos que estén seguidos por una prolina, lo que resulta en huellas peptídicas de péptidos predecibles.
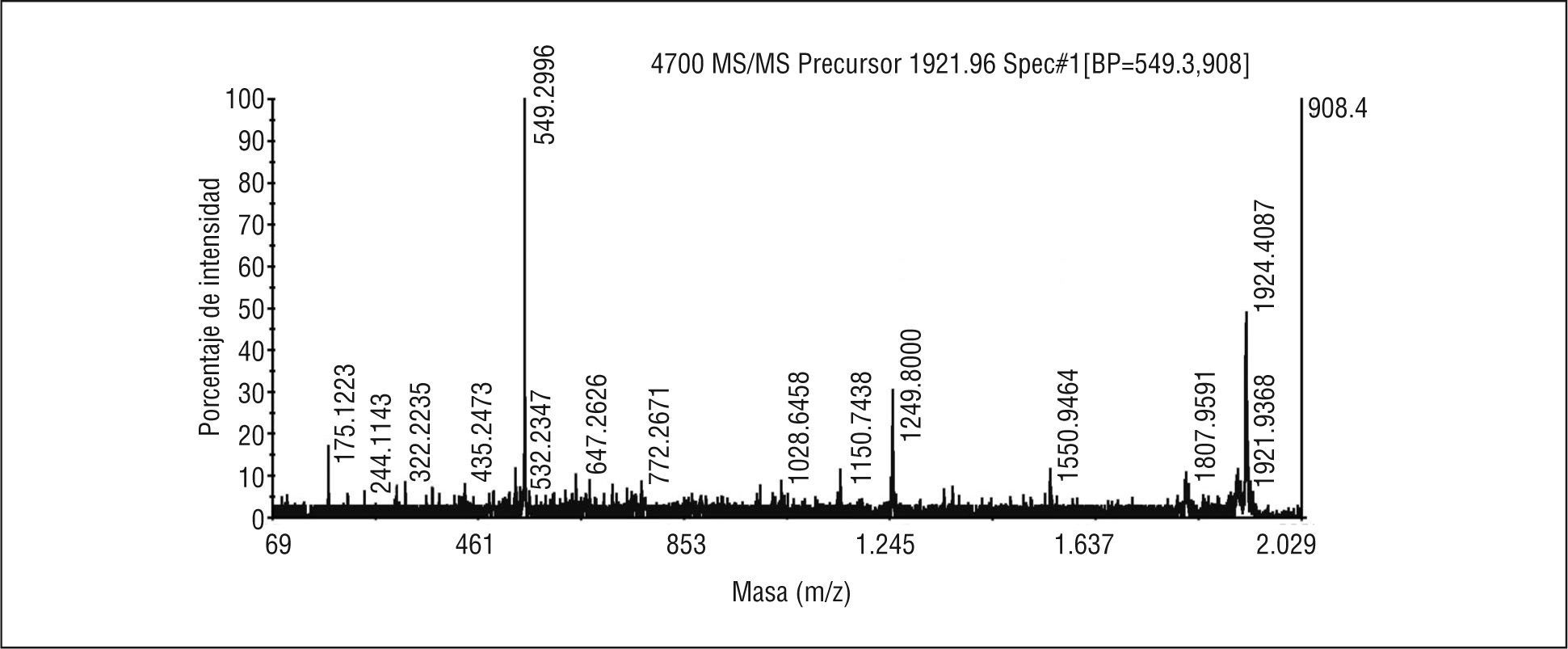
Los fragmentos peptídicos obtenidos son ionizados tras el tratamiento con un láser que les confiere carga. Los fragmentos peptídicos, una vez conferida la carga, se separan en el espectro según su relación masa/carga y, posteriormente, se analizan con un detector, que es capaz de medir la intensidad de cada fragmento (fig. 2).
Ejemplo de un espectro de masas obtenido mediante el análisis por espectrometría de masas en tándem (MS/MS). El eje de las ordenadas viene definido por la relación masa/carga de cada uno de los péptidos y por el porcentaje de intensidad, que indica la cantidad relativa de iones que poseen esta relación carga/masa.
Posteriormente, y mediante otras bases de datos que normalmente son de acceso libre en internet, las huellas peptídicas resultantes del análisis de espectrometría de masas se utilizan para encontrar la homología que nos ayudará a identificar la proteína. La eficiencia de esta técnica es elevada, tanto que incluso la identificación de una secuencia de 5 residuos de aminoácidos puede ser suficiente para identificar la proteína.
Aplicación de la proteómica en la investigación cardiovascularEn líneas generales, la aplicación de la proteómica en el área cardiovascular se ha focalizado en: conocer mecanismos moleculares asociados a la enfermedad, la identificación de nuevos biomarcadores diagnóstico, la identificación de biomarcadores pronósticos y de evolución. El estudio de fármacos a través de la proteómica se ha denominado farmacoproteómica, de la cual hablaremos más adelante.
Identificación de biomarcadores diagnóstico y pronóstico en la enfermedad cardiovascularEn el conocimiento de las enfermedades cardiovasculares, han tenido y tienen un papel fundamental la identificación de factores pronósticos y diagnósticos, como la hipercolesterolemia, la hipertensión o la diabetes. Desde el punto de vista de los biomarcadores, solamente la tropomiosina (para el síndrome coronario agudo [SCA]), o el péptido natriurético cerebral (para el fracaso cardíaco agudo) son actualmente utilizados para el diagnóstico y el pronóstico de los pacientes. Con la utilización de la proteómica se pueden identificar nuevos marcadores de utilidad clínica.
La sangre representa probablemente la fuente más accesible para la búsqueda de biomarcadores diagnósticos y pronósticos. El plasma contiene un rango de proteínas en una concentración desde 55.000.000.000 pg/ml (como la albúmina) a 1 o 5 pg/ml (como las citocinas), con un gran rango de concentraciones entre estos 2 extremos. Como aproximación, el número de proteínas en la sangre humana es superior a 106 moléculas diferentes, que representarían el producto de entre 25.000 a 30.000 genes1. Por todo ello, el potencial de la sangre en contener biomarcadores de utilidad clínica es grande, aunque todavía no se ha analizado en su totalidad, probablemente debido, hasta ahora, a la falta de tecnología para alcanzar este fin.
Proteómica y aterosclerosisEn la investigación de los vasos, la utilización de la proteómica está en sus primeros pasos. Incluso, el proteoma de los vasos normales y patológicos no está aún definido. La dificultad principal para aplicar el análisis proteómico en la enfermedad vascular es la heterogeneidad de la composición celular de la placa aterosclerótica2. El perfil proteico de los vasos normales está dominado por proteínas de las células de músculo liso vascular, pero también contiene proteínas de las células endoteliales o, incluso, de otros tipos celulares, como fibroblastos, mioblastos y células dendríticas. Sin embargo, la aterosclerosis es una enfermedad inflamatoria, lo que implica la interacción de células sanguíneas inflamatorias con la pared vascular, y que complica aún más el componente celular y, por supuesto, proteico de la pared vascular.
El primer intento de analizar la lesión aterosclerótica humana mediante electroforesis bidimensional se realizó hace ahora algo más de 20 años. Stastny et al3 compararon la composición proteica de la lesión fibrograsa humana con la íntima sana. La comparación se realizó de forma visual, ya que en esos años todavía no había programas informáticos para el análisis bioinformático de los geles, por lo que sólo se identificaron los cambios más evidentes. En este trabajo se observó que en la arteria aterosclerótica había un aumento de albúmina, fibrinógeno, inmunoglobulina G, a-1 antitripsina, transferrina, haptoglobina, apolipoproteína AI y apolipoproteína AII3. En un estudio más reciente, You et al4 analizaron arterias coronarias de pacientes con enfermedad de arteria coronaria y los compararon con individuos sanos. El estudio por electroforesis bidimensional reveló un aumento significativo en la expresión de la cadena ligera de ferritina4. Estos resultados estarían relacionados con la hipótesis del "hierro", que propone que el almacén de hierro contribuiría a la formación y la progresión de la placa de ateroma mediante la modulación de la oxidación lipídica. Sin embargo, en este estudio se encontraron muy pocas variaciones entre la arteria sana y enferma, probablemente debido a la gran variabilidad entre los resultados, lo que puede enmascarar las diferencias. También la heterogeneicidad en la composición de la placa aterosclerótica complica la interpretación de los resultados. El aumento en la expresión de la cadena ligera de ferritina podría ser también consecuencia de una infiltración mayor de monocitos en la arteria enferma. En otro trabajo reciente, de Donners et al5, se analizó la lesión avanzada estable con placas conteniendo trombos. Mediante espectrometría de masas identificaron la vinexina-β, una proteína que une vinculina, y α1- antitripsina, cuya expresión estaba limitada en las placas de lesiones temporales comenzando con una mayor expresión en placas con lesiones tempranas comparada con la mayor expresión en placas con lesiones más avanzadas. En las placas avanzadas se identificaron 6 isoformas de aj-antitripsina, una de las cuales sólo se observó en placas que contenían trombo5. Posiblemente, en la placa de ateroma, la α1-antitripsina tenga un efecto protector, e impida la invasión por células inflamatorias. Hay que recordar que la α1-antitripsina tiene actividad antiesclerótica y la pérdida de esta actividad antiesclerótica podría favorecer la adhesión y la acumulación de células inflamatorias, lo cual hace la placa más vulnerable a la rotura.
Se pueden hacer múltiples aproximaciones para el estudio de la placa de ateroma mediante la utilización de la proteómica. Por ejemplo, en medio libre de proteínas se han cultivado muestras de endarterectomía de carótida y se han analizado las proteínas liberadas en el medio de cultivo6. Se encontraron proteínas en el medio solamente en las incubaciones de arterias con placas, pero no en las arterias normales. Entre las proteínas identificadas se encontraron proteínas implicadas en el transporte reverso del colesterol (apolipoproteína B-100; apolipoproteína AI), proteínas asociadas a apoptosis, proteínas relacionadas con la degradación de proteínas y proteínas antioxidantes6. Cuanto más complicada era la lesión aterosclerótica, mayor era el número de proteínas liberadas6.
La proteómica aplicada a la isquemia miocárdicaLa estratificación rápida y eficaz del paciente que llega al servicio de urgencias con un dolor torácico es importante porque ofrece ventajas para el inicio temprano del tratamiento, lo que claramente favorecerá el pronóstico de los pacientes que presentan un SCA. Actualmente, la estratificación de los pacientes con sospecha de tener un SCA comienza con la realización de un electrocardiograma y un examen físico e histórico, en el que se incluyen los antecedentes familiares y los factores de riesgo cardiovascular que presenta el paciente. En el SCA sin elevación del segmento ST, el diagnóstico del infarto de miocardio está principalmente basado en la detección de biomarcadores de necrosis miocárdica. En la actualidad, el biomarcador más utilizado para detectar necrosis miocárdica es la troponina7. La troponina también se utiliza como biomarcador de riesgo de episodio cardíaco, incluidas muerte e isquemia recurrente8. No obstante, tanto la troponina como otros biomarcadores menos específicos se utilizan para detectar necrosis miocárdica, como puede ser la mioglobina o la fracción de mioglobina de la creatincinasa (CKMB). En este sentido, la mioglobina tiene una utilidad limitada, ya que no es poco específica, fundamentalmente cuando coexiste un daño del músculo esquelético y un fallo renal. La CK-MB tiene una sensibilidad similar a la mioglobina, aunque presenta una mayor especificidad, a pesar de que hay algunos autores que describen que no tiene especificidad absoluta. La troponina es muy sensible y más específica para detectar daño miocárdico. No obstante, es importante identificar el SCA antes de que ocurra una necrosis miocárdica. Para intentar identificar mediante la proteómica nuevos biomarcadores útiles para el diagnóstico de SCA, los investigadores han comenzado por conocer cuáles son los cambios en el proteoma del miocardio isquémico. Para ello, se han utilizado fundamentalmente modelos animales.
En un modelo en conejo de isquemia con repercusión miocárdica, el análisis mediante electroforesis bidimensional demostró que múltiples proteínas conocidas y otras no conocidas cambiaban su expresión en el corazón9. Entre ellas se encuentran proteínas del citoesqueleto (troponina C y cadena ligera de la miosina), enzimas del sistema redox, enzimas relacionadas con el metabolismo energético y proteínas de respuesta al estrés. En un modelo porcino, la isquemia miocárdica también demostró alteraciones en diferentes proteínas sistémicas como elevación en las catepsinas B y D y la proteína Hsc7310. Estos cambios se asociaron con una reducción de la apoptosis y se indicó que estas proteínas se activan para proteger el tejido miocárdico contra la isquemia10.
Nuestro grupo estudió recientemente los cambios en la expresión plasmática de proteínas en pacientes durante la fase aguda de un SCA11. El estudio por electroforesis bidimensional del plasma demostró una reducción de algunas isoformas de la α1-antitripsina y de la apolipoproteína AI en los pacientes durante un infarto de miocardio o una angina inestable respecto a los individuos control11. Se encontraron también diferencias en la expresión plasmática de algunas isoformas de proteínas entre los pacientes durante un infarto de miocardio, respecto a pacientes que tenían una angina inestable. En este sentido, las isoformas de la α1-antitripsina 5, 6 y 7 estaban más reducidas en el plasma de los pacientes durante el infarto de miocardio, respecto a los de angina inestable11. Lo mismo ocurría con las 5 isoformas de la apolipoproteína AI identificadas11. Probablemente, la menor expresión de estos 2 biomarcadores, cuya función es la protección miocárdica durante el infarto de miocardio, pueden asociarse a un daño mayor en las células del corazón. En definitiva, a través de la proteómica podemos conocer mejor los mecanismos moleculares que ocurren durante el SCA, lo que además de facilitarnos la identificación de biomarcadores puede desvelarnos más dianas terapéuticas.
Hipertrofia cardíaca y proteómicaLa hipertrofia del ventrículo izquierdo es el mecanismo primario por el que el ventrículo izquierdo responde a la sobrecarga de presión. Una sobrecarga de presión prolongada produce disfunción miocárdica y el desarrollo del fallo cardíaco congestivo. Hay 2 mecanismos principales críticos en el desarrollo de la hipertrofia hipertensiva cardíaca: a) la elevada poscarga del ventrículo izquierdo que induce estrés mecánico, y b) factores humorales/neuronales, como el sistema renina-angiotensina aldosterona y el sistema simpático. No obstante, los mecanismos moleculares mediadores del proceso del remodelado cardíaco implicados en la hipertrofia miocárdica no están completamente definidos12. En un estudio de Jin et al13 en ratas espontáneamente hipertensas, los autores encontraron que la expresión de 20 proteínas estaba cambiada en el miocardio hipertrófico. De ellas, 13 proteínas cambiaron su expresión en las primeras etapas de la hipertensión, antes de que ésta fuera sostenida, y 7 proteínas cambiaron su expresión en el ventrículo izquierdo una vez que el estado hipertensivo era sostenido13. Muchas de las proteínas que cambiaron estaban relacionadas con el metabolismo energético13. Es interesante señalar que en este estudio se identificó una proteína antioxidante, la glutatión transferasa omega-1, que se identificó sólo en el miocardio de las ratas espontáneamente hipertensas, y no en el de las normotensas. Esto podría parecer paradójico, ya que otros investigadores han demostrado valores elevados de esta proteína en el tejido miocárdico, lo que indicaría que en la hipertensión se expresa otra isoforma de esta proteína que no se expresa en la normotensión14. Tomemos este ejemplo para comentar que, al utilizar la proteómica con frecuencia, aparecen proteínas no conocidas o cambios en la expresión de proteínas que hasta la fecha no se habían relacionado con la enfermedad en estudio. Este es uno de los mayores potenciales de los estudios proteómicos y la consecuencia primera es que abre paso al conocimiento de nuevas dianas terapéuticas, que hasta entonces no se habían relacionado con la enfermedad. En este sentido, en un trabajo reciente de Lindsey et al15 se identificaron 123 proteínas en el ventrículo izquierdo hipertrofiado de ratas. De éstas, sólo 32 se habían asociado previamente con la hipertrofia ventricular. El 57% de las proteínas identificadas eran proteínas implicadas en el metabolismo y en la estructura celular, lo cual tiene sentido, ya que en la hipertrofia el miocardiocito aumenta el tamaño, lo que posiblemente implicará un aumento en su demanda metabólica.
Muchos de los estudios sobre los cambios en la expresión de proteínas en el corazón hipertrófico se han centrado en proteínas estructurales y contráctiles. No obstante, en el corazón hipertrófico hay una adaptación compensatoria energética. Probablemente, esta respuesta adaptativa o maladaptativa en el metabolismo puede contribuir de forma negativa a la funcionalidad del miocardio hipertrófico. En el corazón, el adenosintrifosfato (ATP) utilizado en la contracción se sintetiza principalmente en la mitocondria a través de la fosforilación oxidativa. En el corazón normal, la principal fuente de energía es la β-oxidación de los ácidos grasos de cadena larga. Sin embargo, el corazón hipertrófico tiene reducida la utilización de la β-oxidación de los ácidos grasos y estudios indirectos han indicado que su fuente principal de producción de ATP es la glucólisis16,17. De hecho, hay algunas teorías que intentar explicar la reducción en la β-oxidación de los ácidos grasos en el miocardio hipertrofiado. Por ejemplo, se ha indicado una reducción en el contenido de carnitina en el miocardio del corazón hipertrófico18. La carnitina se utiliza para la traslocación de los ácidos grasos, a través de la membrana interna de la mitocondria al sitio de la β-oxidación. No obstante, todo esto no está totalmente establecido.
A través de la proteómica podemos conocer cuáles son los cambios que ocurren en el corazón hipertrofiado en los ensayos que reflejan el metabolismo energético. Algunos resultados preliminares de nuestro grupo parecen indicar que la expresión tanto de proteínas relacionadas con la β-oxidación, como con la glucólisis, está reducida en el ventrículo izquierdo hipertrofiado de ratas espontáneamente hipertensas19.
Una de las estructuras celulares que cambian en el corazón hipertrófico es la mitocondria del cardiomiocito. El papel principal de la mitocondria es la producción de ATP, aunque también está implicada en la homeostasis iónica, la apoptosis, la oxidación de hidratos de carbono y de ácidos grasos, así como en gran diversidad de procesos catabólicos y anabólicos. Como consecuencia de esta diversidad funcional, la disfunción mitocondrial se asocia a diferentes enfermedades, incluidas las enfermedades del corazón. Por lo tanto, el estudio del proteoma mitocondrial comienza también a ser foco de atención.
En forma de predicción, se calcula que el proteoma mitocondrial incluye más de 1.500 proteínas diferentes. En este sentido, hay más proteínas mitocondriales de carácter básico comparadas con las citosólicas. Las proteínas mitocondriales son generalmente pequeñas (< 10 kDa) y están poco descritas en las bases de datos. No obstante, todo ello dificulta su estudio e identificación mediante proteómica. Hay todavía muy pocos estudios realizados sobre el proteoma mitocondrial respecto a la enfermedad cardiovascular. Quizás el más referido sea el realizado por Lin et al20, en el que los autores descubrieron el proteoma de la mitocondria del cardiomiocito en un modelo de estrés crónico en el que se induce disfunción cardíaca. Encontraron que había 11 proteínas mitocondriales que tenían alterada su expresión, respecto a mitocondrias obtenidas de cardiomiocitos de animales control. Estas proteínas estaban implicadas en el ciclo de Krebs y en el metabolismo lipídico, y su expresión estaba disminuida en los animales con disfunción miocárdica. Estas proteínas se identificaron como carnitina palmitoil/transferasa 2, acil CoA-tioesterasa mitocondrial, isocitrato deshidrogenada 3 (NAD+), α-fumarato hidratasa-1 y piruvato deshidrogenasa β.
Farmacoproteómica y enfermedad cardiovascularLa medicina personalizada se basa en las diferencias existentes entre individuos y, por lo tanto, aspira a dar a cada paciente el tratamiento más adecuado de forma personalizada. La farmacoproteómica se puede definir como el estudio de la respuesta farmacológica de un paciente en función de las proteínas que expresa.
En el área cardiovascular, la farmacoproteómica es una rama de reciente utilización y que, entre otras aplicaciones, nos puede ayudar a desvelar el siempre debatido aspecto de la identificación de efectos clase y efectos pleiotrópicos de los fármacos cardiovasculares.
Todavía no hay muchos estudios que analicen los efectos de fármacos en el proteoma de los pacientes cardiovasculares. Para las estatinas, se realizó un estudio en ratas en el que se analizaron las modificaciones en las proteínas del hígado después de administrar lovastatina y fluvastatina21,22. En estos estudios se describieron, a partir de electroforesis bidimensional y espectrometría de masas, modificaciones en el metabolismo de los hidratos de carbono, en proteínas de estrés, en proteínas relacionadas con la homeostasis del calcio y con la actividad de las proteasas. En relación con estos efectos pleiotrópicos de las estatinas, un estudio reciente publicado por nuestro grupo23 analizó las modificaciones en el proteoma plasmático de pacientes moderadamente hipercolesterolémicos tras el tratamiento durante 12 semanas con simvastatina. En este estudio, se observó que los pacientes con hipercolesterolemia moderada tenían aumentado en el plasma la expresión de la isoforma 1 de la cadena gamma del fibrinógeno y 3 isoformas de haptoglobina23. Tras el tratamiento con simvastatina, la isoforma 1 del fibrinógeno disminuyó su expresión en plasma; sin embargo, la isoforma 2 de haptoglobina, proteína reactante de fase aguda, aumentó. La haptoglobina es una proteína que une hemoglobina libre, lo que le confiere propiedades antioxidantes. Por lo tanto, como hipótesis, el aumento de esa isoforma de haptoglobina, observado tras el tratamiento con simvastatina, podría indicar un aumento en la respuesta antioxidante. También se examinaron cambios en la expresión de la apolipoproteína AIV. Como es bien conocido, la apolipoproteína AIV no sólo desempeña un papel importante en el transporte reverso del colesterol, sino que también es una proteína asociada a protección vascular. El tratamiento con simvastatina aumentó los valores plasmáticos de la apolipoproteína AIV23. De todos los cambios descritos anteriormente, los cambios en apolipoproteína AIV y haptoglobina isoforma 2 no se correlacionaron con los valores de colesterol total23. Este tipo de estudios puede ayudar a conocer con más profundidad los mecanismos de acción de las estatinas y, sobre todo, pueden facilitar la identificación de mecanismos pleiotrópicos que nos ayudarán a conocer mejor los beneficios moleculares y celulares de unas estatinas respecto a otras.
Otra de las aplicaciones de la farmacoproteómica es la identificación de biomarcadores de respuesta farmacológica. Es decir, conocer si los cambios en la expresión de una proteína o grupo de proteínas puede asociarse a una respuesta mayor o menor del paciente al fármaco. Un ejemplo reciente de esta posible aplicación de la proteómica es la búsqueda de biomarcadores que nos ayuden a definir a pacientes cuyas plaquetas respondan peor al tratamiento con aspirina.
Se han descrito diferentes mecanismos por los que la aspirina ejerce su efecto antitrombótico; sin embargo, su efecto antitrombótico principal es mediante la inhibición de la actividad de la ciclooxigenasa plaquetaria, lo que bloquea la síntesis de tromboxano A2 (TxA2) por las plaquetas24,25. A pesar de tomar aspirina, algunos pacientes tienen episodios vasculares recurrentes, lo que ha indicado que estos pacientes son aparentemente resistentes a la aspirina, un fenómeno que se ha llamado "resistencia a la aspirina"26,27. En este sentido, en alrededor del 20% de los pacientes que toman de forma habitual aspirina, el fármaco no inhibe sus plaquetas y este porcentaje aumenta hasta un 40% si los pacientes son diabéticos. En el momento actual, hay diferentes hipótesis que intentan explicar el fenómeno de la resistencia a la aspirina, aunque ninguna de ellas por sí misma ha podido justificar la existencia de esta variabilidad en la respuesta plaquetaria28–31. El estudio proteómico del plasma en pacientes resistentes a la aspirina ha demostrado la existencia de un aumento en la expresión de 3 isoformas de la proteína de unión a vitamina D (DBP), comparndo con la expresión de estas isoformas observada en el plasma de pacientes cuyas plaquetas son sensibles a la acción de la aspirina31.
La función principal de la DBP es unir y transportar análogos de la vitamina D. También se ha descrito a esta proteína como activadora de leucocitos32. Ya que los pacientes resistentes a la aspirina tenían elevada la expresión de 3 isoformas de DBP en el plasma, se analizó si había alguna relación entre la DBP y la capacidad de la aspirina para inhibir la ciclooxigenasa 1 plaquetaria. En experimentos in vitro se observó que la presencia de DBP redujo la capacidad de la aspirina de bloquear la producción de TxA2 por las plaquetas31. Estos resultados podrían indicar que, de alguna manera, la DBP impide la acción de la aspirina en la ciclooxigenasa 1 plaquetaria. Por ello, los pacientes con DBP elevada tienen, a pesar del tratamiento con aspirina, una capacidad mayor de producir TxA2 y, por lo tanto, unas plaquetas potencialmente más activas con capacidad de formar trombos en el vaso.
En resumen, la proteómica y la farmacoproteómica pueden ser herramientas muy útiles para desarrollar nuevos fármacos para la búsqueda de biomarcadores pronóstico y de evolución de los pacientes con enfermedades cardiovasculares, e incluso pueden ayudarnos a controlar mejor las interacciones farmacológicas, sobre todo en el ámbito molecular. No obstante, para alcanzar un uso más específico de estas nuevas ciencias, necesitamos primero conocer el proteoma de los órganos implicados: corazón, vasos, células de la sangre, plasma, etc., no ya en condiciones patológicas, sino también en las fisiológicas, lo que aún no se ha realizado en su totalidad.
Los autores agradecen a Begoña Larrea su labor editorial.