En la absorción intestinal de colesterol intervienen distintos transportadores, uno de gran importancia y diana del fármaco ezetimiba, la proteína NPC1L1. Se ha demostrado una asociación entre distintas variantes genéticas de NPC1L1, la eficiencia en la absorción de esteroles y la concentración plasmática de colesterol unido a lipoproteínas de baja densidad. El objetivo de este estudio fue identificar y analizar el efecto funcional de los polimorfismos genéticos potencialmente más relevantes del gen NPC1L1 sobre su actividad transcripcional.
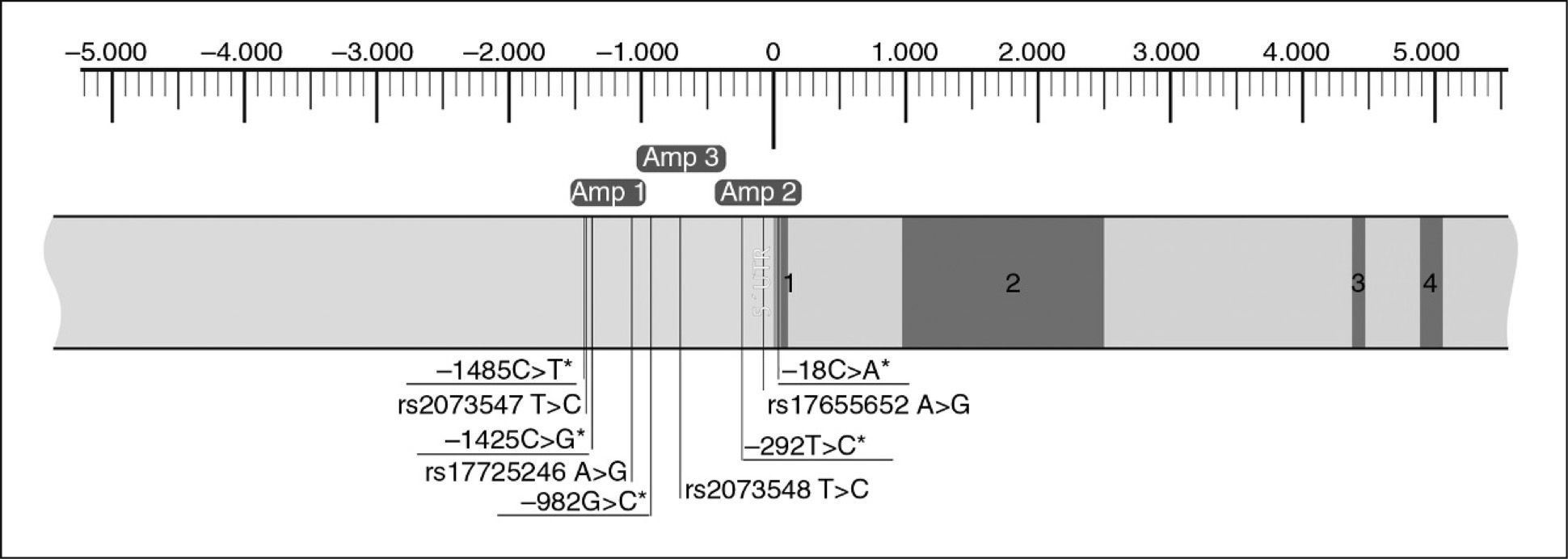
Material y métodosComo zona de estudio se seleccionó 2 kb de la región promotora del gen NPC1L1. Se realizó una búsqueda en bases de datos para localizar variantes descritas en las secuencias seleccionadas. Se diseñaron 3 fragmentos, que se amplificaron mediante PCR y posteriormente se secuenciaron. La funcionalidad del polimorfismo −133A>G se determinó mediante ensayos de retardo en gel y medida de la actividad luciferasa.
ResultadosEl análisis de la zona promotora de 102 sujetos normolipidémicos mostró 5 nuevas variantes polimórficas (−1485C>T, −1425C>G, −982G>C, −292T>C y −18C>A) no identificadas previamente. Los resultados de los ensayos de retardo en gel con el polimorfismo −133A>G revelaron mayor afinidad de unión de las proteínas nucleares con la sonda portadora de la variante −133A. Por otra parte, la actividad del promotor de NPC1L1 con la variante −133G mostró un aumento de 2,5 veces respecto a la variante −133A.
ConclusiónNuestros resultados demuestran que hay diferencias en la afinidad de unión y actividad transcripcional de NPC1L1 en función del polimorfismo −133A>G. Esta variante génica podría contribuir a la variabilidad interindividual de la absorción intestinal de esteroles.
Cholesterol absorption is a process which involves the participation of numerous transporters. One of these transporters is NPC1L1, identified as a critical protein for intestinal cholesterol absorption and the molecular target of ezetimibe. It has been shown that NPC1L1 variants are associated with variations in sterol absorption and plasma levels of LDL-C. The aim of this work was to identify and analyse the potential functional effect on the transcriptional activity of NPC1L1 gene polymorphisms.
Material and methodsThe previously described NPC1L1 promoter variants were located by means of SNP database search. The NPC1L1 promoter was analysed by three PCR reactions followed by automatic capillary sequencing. Functional study of the −133A>G polymorphism was performed by EMSA and Luciferase assays.
ResultsNPC1L1 promoter analysis in 102 normolipemic subjects showed five novel polymorphisms (−1485C>T, −1425C>G, −982G>C, −292T>C y −18C>A). EMSA results revealed that the -133A specific oligonucleotide produced a stronger retarded band than the retarded band corresponding to the −133G allele. Luciferase activity observed with −133G NPC1L1 variant was 2.5-fold higher than that seen with the −133A variant.
ConclusionOur results reveal that the −133A>G polymorphism exerts a significant effect on nuclear extract binding affinity and NPC1L1 transcriptional activity. This gene variant could contribute to interindividual variability of intestinal sterols absorption.
La homeostasis de colesterol está regulada a distintos niveles, de tal forma que se establece un equilibrio entre las fuentes de aporte de colesterol, como son la absorción intestinal y la síntesis endógena, y las vías de eliminación, que incluyen principalmente la excreción y, en menor medida, su utilización metabólica1. La modificación de alguno de estos procesos puede dar lugar a una alteración patológica en la concentración plasmática de colesterol. La absorción intestinal de colesterol y esteroles de origen vegetal es un proceso complejo, formado por múltiples pasos y en el que intervienen distintas enzimas y transportadores2. Dentro de estos transportadores se encuentra la proteína NPC1L1 (Niemann-Pick C1-Like 1), identificada recientemente por el grupo de Altmann como fundamental en la absorción de colesterol y esteroles vegetales3,4 y posteriormente diana molecular del inhibidor de absorción intestinal de colesterol ezetimiba5. La proteína NPC1L1 pertenece a una familia de proteínas transmembrana en la que está incluida la proteína NPC16, defectuosa en la enfermedad de depósito lisosomal Niemann-Pick tipo C7 y con la que comparte el 51% de homología en la secuencia de aminoácidos. En humanos, se encuentra normalmente localizada en compartimientos intracelulares, desplazándose a la membrana plasmática del enterocito en función de la disponibilidad de colesterol celular8,9.
Diversos estudios10,11 han demostrado la influencia de variantes genéticas de NPC1L1 sobre la concentración del colesterol unido a lipoproteínas de baja densidad (cLDL) en respuesta a la inhibición de la absorción por ezetimiba. Por otra parte, recientemente se ha observado que hay una asociación entre los polimorfismos de NPC1L1, la variación en la absorción intestinal de esteroles y la concentración plasmática de cLDL en población normolipémica12. Por todo ello, nos propusimos como objetivo identificar y analizar el efecto funcional sobre la actividad transcripcional de NPC1L1 de las variantes genéticas localizadas en la zona promotora del gen NPC1L1.
Material y métodosSujetosEl grupo de sujetos que se estudió estuvo constituido por 102 sujetos voluntarios sanos normolipidémicos no relacionados, 46 varones y 56 mujeres, con una edad comprendida entre los 25 y los 65 años.
Todos los pacientes aceptaron su inclusión en el estudio previa firma de un consentimiento informado. El estudio fue aprobado por el Comité Ético de Investigación Clínica de Aragón.
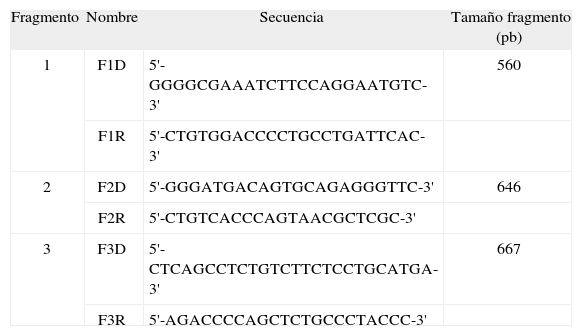
Estudio genéticoComo zona de estudio se seleccionó 2 Kb de la región promotora del gen NPC1L1. Se llevó a cabo una búsqueda en la base de datos dbSNP (http://www.ncbi.nlm.nih.gov/SNP) para localizar variaciones previamente descritas en la secuencia seleccionada del gen NPC1L1 obtenida de la base de datos Ensembl (www.ensembl.org), con código de acceso ENSG00000015520. Se diseñaron 3 fragmentos para la determinación de los polimorfismos descritos en estas zonas y de otras posibles variantes no descritas con anterioridad (fig. 1). Los fragmentos se analizaron mediante 3 reacciones de reacción en cadena de la polimerasa (PCR) amplificadas de forma simultánea en un termociclador GeneAmp®9700 (Applied Biosystems, Foster City, USA). Las condiciones para la amplificación de los 3 fragmentos fueron: 94°C 1min; 35 ciclos (94°C 15s–62°C 15s–72°C 50s); 72°C 5min. Los cebadores empleados para la amplificación de la zona promotora del gen NPC1L1 se diseñaron mediante el programa informático Oligo v.6.0 (MBI). Los tamaños de los fragmentos amplificados y los cebadores utilizados se muestran en la tabla 1. La purificación enzimática de los productos amplificados se llevó a cabo con Exo-SAP-IT™ (United States Biochemicals, Cleveland, EE.UU.), y posteriormente se secuenciaron en un secuenciador automático capilar MegaBACE 500 (GE-Healthcare) utilizando el producto comercial DYEnamic ET Dye Terminator Kit (GE Healthcare).
Secuencias de los cebadores utilizados para la amplificación de la zona promotora del gen NPC1L1 y tamaño de los fragmentos obtenidos
| Fragmento | Nombre | Secuencia | Tamaño fragmento (pb) |
| 1 | F1D | 5'-GGGGCGAAATCTTCCAGGAATGTC-3' | 560 |
| F1R | 5'-CTGTGGACCCCTGCCTGATTCAC-3' | ||
| 2 | F2D | 5'-GGGATGACAGTGCAGAGGGTTC-3' | 646 |
| F2R | 5'-CTGTCACCCAGTAACGCTCGC-3' | ||
| 3 | F3D | 5'-CTCAGCCTCTGTCTTCTCCTGCATGA-3' | 667 |
| F3R | 5'-AGACCCCAGCTCTGCCCTACCC-3' |
Ensayos de retardo en gel o EMSA (Electrophoretic Mobility Shift Assay). Los ensayos de retardo en gel se llevaron a cabo a partir de extractos nucleares obtenidos de cultivos subconfluentes de la línea celular Caco-2 y se utilizaron los siguientes oligonucleótidos (resaltado en negrita la localización del sitio polimórfico –133A>G): 5'-GGCAGGGCTCAGCCTCATT-3' y 5'-GGCAGGGCTCGGCCTCATT-3'. La preparación de los extractos nucleares, el marcaje de los oligonucleótidos y las condiciones de la electroforesis se realizaron tal y como se ha descrito previamente13. La cuantificación de las bandas de retardo obtenidas se realizó mediante el programa de análisis GS-363 (Bio-Rad, Hercules, EE.UU.). Para la realización del ensayo supershift, se empleó 1μg de anticuerpo RXR· (sc-774X) (Santa Cruz Biotechnology, Santa Cruz, EE.UU.).
Mantenimiento de las líneas celulares. Las líneas celulares Hep-G2 (HB-8065™), obtenida a partir de un carcinoma hepatocelular humano, y Caco-2 (HTB-37™), obtenida a partir de un adenocarcinoma colorrectal humano, utilizadas en este trabajo se obtuvieron de la colección ATCC (American Type Culture Collection).
La línea celular Caco-2 se cultivó en medio esencial mínimo de Eagle modificado por Dulbecco (DMEM) suplementado con Glutamax™-I, 4.500mg/l de D-glucosa, 110mg/l de piruvato sódico, un 10% de suero fetal bovino, 100 U/ml de penicilina, 100μg/ml de estreptomicina-sulfato y un 1% de aminoácidos no esenciales. La línea celular Hep-G2 se cultivó en medio esencial mínimo de Eagle modificado por Dulbecco (DMEM) suplementado con Glutamax™-I, 4.500mg/l de D-glucosa, 110mg/l de piruvato sódico, un 5% de suero fetal bovino, 100 U/ml de penicilina y 100μg/ml de estreptomicina-sulfato.
Todas las líneas celulares se cultivaron en frascos de cultivo de 25 y 75cm2 con tapón de filtro, en un incubador termostatizado con regulación electrónica de flujo de CO2, a 37°C, en atmósfera saturada de humedad con un 5% de CO2.
Clonaje en el vector de expresión pGL3-Basic. Para clonar la secuencia de la región promotora del gen NPC1L1, se amplificó una región que contenía el polimorfismo –133A>G situada entre las posiciones −1.796 y +13 (respecto del origen de transcripción del gen) a partir de ADN de sujetos homocigotos para los 2 genotipos. Los cebadores empleados se diseñaron mediante el programa informático Oligo v.6.0 (MBI) tomando como referencia la secuencia ENSG00000015520 (www.ensembl.org) y fueron los siguientes, directo: 5'-AAAAAAAAAAAAGCTAGCGGACTCTATCTCTCTGTGGTGC-3' y reverso: 5'-TTTTTTTTTTTTAAGCTTCCGGCCTCCGCCACCCCAGGTC-3' que contenían los sitios de restricción NheI y HindIII (subrayados), respectivamente.
Los productos de PCR obtenidos fueron digeridos, purificados y posteriormente clonados en los sitios NheI y HindIII del vector de expresión pGL3-Basic (Promega, Madison, EE.UU.). La confirmación de la incorporación de las variantes –133A y –133G en cada uno de las construcciones se llevó a cabo mediante secuenciación capilar en un secuenciador MegaBACE 500 (GE-Healthcare). Para el aislamiento y purificación de ADN plasmídico se utilizó el producto comercial HiSpeed™ Plasmid Midi Kit (Qiagen) siguiendo las instrucciones recomendadas por el fabricante.
Ensayos de transfección transitoria y medida de la actividad luciferasa. Los ensayos de transfección transitoria de las líneas celulares Hep-G2 y Caco-2 se realizaron mediante la técnica de electroporación. Para ello se tomaron 10 × 106 de células de los cultivos rutinarios y se resuspendieron en una mezcla compuesta por 500μl de PBS suplementado con 50μl de MgCl2 10mM, 10μg de ADN plasmídico que contenía el promotor del gen NPC1L1 y 250ng de ADN del plásmido pRL-TK (Promega, Madison, EE.UU.). Este plásmido se utilizó como control interno de transfección para la normalización de la medida de la actividad luciferasa (ratio luciferasa photinus/renilla). Esta mezcla se dispuso en cubetas estériles de electroporación de 4mm y se electroporó a 975μF y 300V en ambas líneas celulares. Tras el pulso eléctrico las células electroporadas se resuspendieron en 5ml de medio de cultivo completo y se incubaron durante 48h a 37°C en atmósfera de O2 y un 5% de CO2 hasta la realización de la determinación de la actividad de luciferasa.
Los ensayos para la determinación de la actividad luciferasa se realizaron mediante luminometría, en un luminómetro LB96V (Berthold, Bad Wildbad, Germany) utilizando el producto comercial Dual Luciferase® Reporter Assay System (Promega, Madison, EE.UU.) siguiendo las instrucciones recomendadas por el fabricante. Las medidas se integraron y analizaron mediante el software WinGlow v.1.25 (Berthold, Bad Wildbad, Germany).
Análisis estadísticoEl análisis estadístico de los datos se realizó mediante el programa informático Statistical Package for the Social Sciences software (SPSS) v. 12.0 (SPSS Inc. Chicago, EE.UU.). En todos los casos se consideró un nivel de significación estadística de p < 0,05. Para cada polimorfismo estudiado se determinaron las frecuencias alélicas y genotípicas. Se comprobó que las frecuencias genotípicas observadas no se desviaban significativamente del equilibrio de Hardy-Weinberg mediante la prueba de χ2. La comparación de los datos normalizados obtenidos en los ensayos de la actividad luciferasa entre 2 grupos se llevó a cabo mediante la prueba de la t de Student.
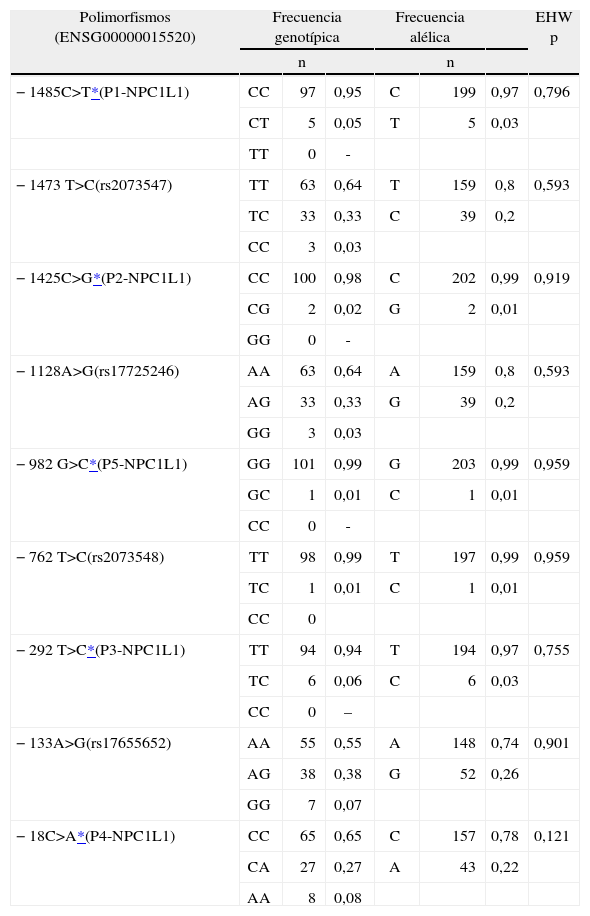
ResultadosEstudio genético de los polimorfismos del promotor del gen NPC1L1. El análisis de la zona promotora llevado a cabo en 102 sujetos normolipémicos mostró la presencia de 5 variantes polimórficas (−1485C>T; –1425C>G; –982G>C; –292T>C, y –18C>A), localizadas a los largo de toda la zona promotora, no identificadas previamente (fig. 1).
Todos los polimorfismos analizados se encontraron en equilibrio de Hardy-Weinberg, ya que no habían diferencias significativas entre estas distribuciones y las frecuencias genotípicas esperadas para una población en equilibrio tras la aplicación de la prueba χ2. En la tabla 2 se muestra la distribución alélica y genotípica de los distintos polimorfismos de NPC1L1 analizados en el grupo de sujetos estudiados.
Distribución genotípica y alélica de los polimorfismos de la zona promotora del gen NPC1L1 analizados
| Polimorfismos (ENSG00000015520) | Frecuencia genotípica | Frecuencia alélica | EHW p | ||||
| n | n | ||||||
| −1485C>T*(P1-NPC1L1) | CC | 97 | 0,95 | C | 199 | 0,97 | 0,796 |
| CT | 5 | 0,05 | T | 5 | 0,03 | ||
| TT | 0 | - | |||||
| −1473T>C(rs2073547) | TT | 63 | 0,64 | T | 159 | 0,8 | 0,593 |
| TC | 33 | 0,33 | C | 39 | 0,2 | ||
| CC | 3 | 0,03 | |||||
| −1425C>G*(P2-NPC1L1) | CC | 100 | 0,98 | C | 202 | 0,99 | 0,919 |
| CG | 2 | 0,02 | G | 2 | 0,01 | ||
| GG | 0 | - | |||||
| −1128A>G(rs17725246) | AA | 63 | 0,64 | A | 159 | 0,8 | 0,593 |
| AG | 33 | 0,33 | G | 39 | 0,2 | ||
| GG | 3 | 0,03 | |||||
| −982G>C*(P5-NPC1L1) | GG | 101 | 0,99 | G | 203 | 0,99 | 0,959 |
| GC | 1 | 0,01 | C | 1 | 0,01 | ||
| CC | 0 | - | |||||
| −762T>C(rs2073548) | TT | 98 | 0,99 | T | 197 | 0,99 | 0,959 |
| TC | 1 | 0,01 | C | 1 | 0,01 | ||
| CC | 0 | ||||||
| −292T>C*(P3-NPC1L1) | TT | 94 | 0,94 | T | 194 | 0,97 | 0,755 |
| TC | 6 | 0,06 | C | 6 | 0,03 | ||
| CC | 0 | – | |||||
| −133A>G(rs17655652) | AA | 55 | 0,55 | A | 148 | 0,74 | 0,901 |
| AG | 38 | 0,38 | G | 52 | 0,26 | ||
| GG | 7 | 0,07 | |||||
| −18C>A*(P4-NPC1L1) | CC | 65 | 0,65 | C | 157 | 0,78 | 0,121 |
| CA | 27 | 0,27 | A | 43 | 0,22 | ||
| AA | 8 | 0,08 | |||||
EHW: equilibrio de Hardy-Weinberg.
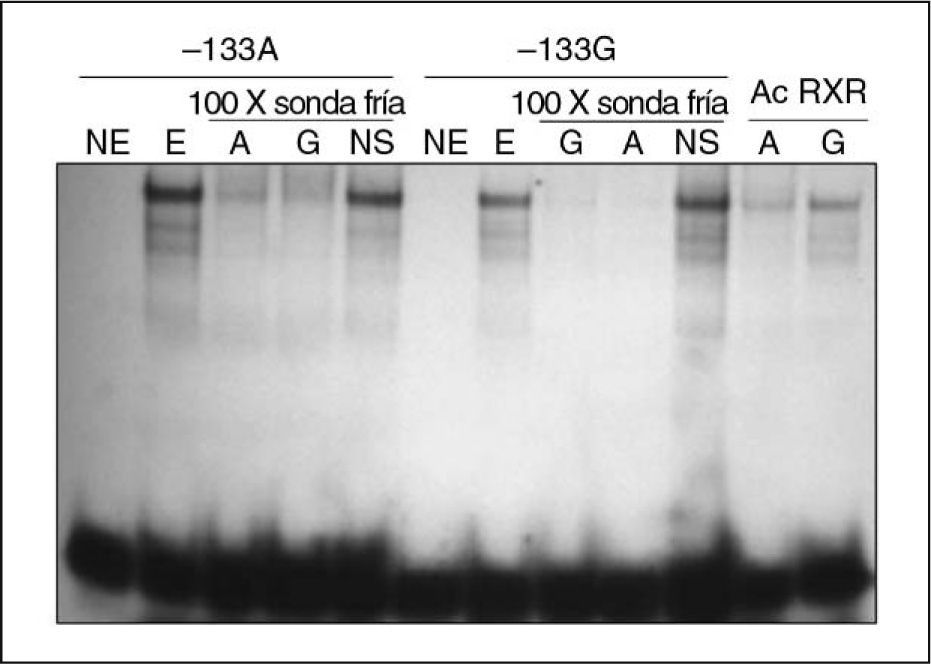
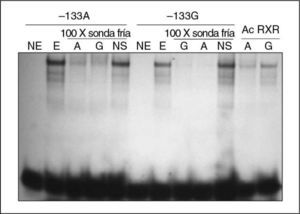
Análisis funcional del polimorfismo –133A>G. Mediante experimentos de retardo en gel o EMSA (Electrophoretic Mobility Shift Assay) se estudió si la presencia del polimorfismo –133A>G podía afectar a la interacción ADN-proteína. Los ensayos se realizaron a partir de extractos nucleares de la línea celular Caco-2 utilizando oligonucleótidos localizados en la posición −143 a −125 (respecto al origen de transcripción) de la secuencia promotora del gen NPC1L1, cada uno de ellos conteniendo la variante –133A o –133G. Los resultados obtenidos mostraron la formación de complejos de las sondas con los extractos nucleares de Caco-2 en presencia de las 2 variantes alélicas del polimorfismo –133A>G. Además, se observó diferente afinidad de unión de las proteínas nucleares en función de la variante alélica presente en la sonda (fig. 2). Los resultados se obtuvieron a partir de 5 experimentos distintos y en todos ellos el oligonucleótido que contenía la variante –133A mostró mayor intensidad (1,76 ± 0,17) en la banda de retardo respecto al oligonucleótido con la variante –133G. Los estudios realizados con herramientas bioinformáticas mostraron que la presencia de esta variante podría alterar la secuencia reconocida por el factor de transcripción RXR. Para comprobar si esto era así, se realizó un ensayo supershift con anticuerpo RXRα Como se puede observar en la figura 2, en presencia del anticuerpo la banda retardada con la sonda portadora del alelo -133A desaparece, lo que sugiere que RXRα es la proteína que se está uniendo a esta secuencia; por el contrario, no sucede lo mismo con la sonda que contiene la variante –133G.
Efecto del polimorfismo –133A>G sobre la afinidad de unión de factores de transcripción. Se muestran los resultados obtenidos de los ensayos de retardo en gel en presencia de sondas con las 2 variantes del polimorfismo –133A>G. En presencia de las 2 sondas se produjo la formación de complejos retardados con extractos nucleares (E) de la línea celular Caco-2, y fue más intenso el retardo mostrado por la sonda que contenía la variante –133A. NE: no extracto nuclear, utilizado como marcador negativo de retardo; NS: no específico; Ac RXR: en presencia de 1μg de anticuerpo RXRα.
Para conocer si el polimorfismo –133A>G afectaba a la actividad transcripcional del promotor del gen NPC1L1, se llevaron a cabo experimentos in vitro de transfección transitoria en las líneas celulares Caco-2 y Hep-G2. Las transfecciones de las construcciones con cada una de las variantes del polimorfismo que se debía estudiar, fueron realizadas mediante electroporación. Los plásmidos utilizados fueron obtenidos a partir de 2 preparaciones diferentes y cotransfectados con el plásmido pRLTK utilizado como control interno de transfección. Además, se realizaron transfecciones en las líneas Caco-2 y Hep-G2 con el plásmido pGL3-Basic, carente de secuencia promotora, que sirvió de control negativo.
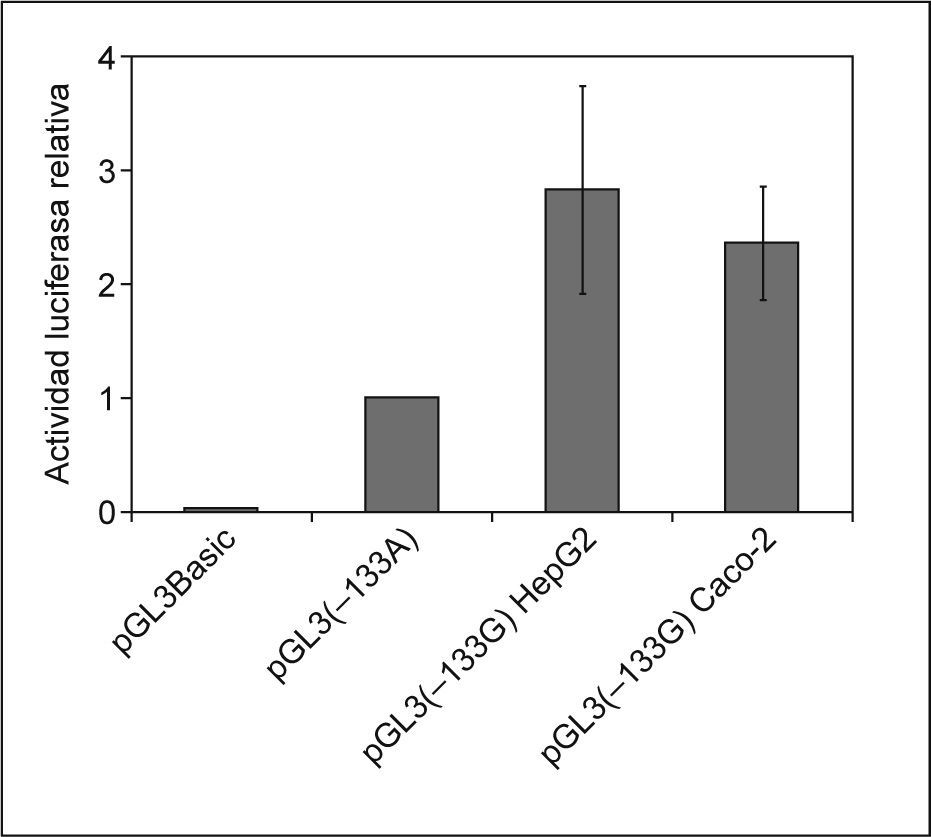
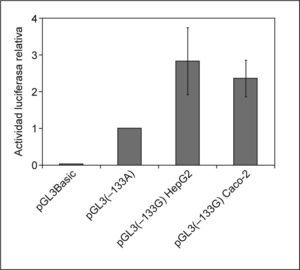
Como era de esperar, la actividad luciferasa del plásmido pGL3-Basic fue prácticamente insignificante en ambas líneas celulares. La actividad luciferasa normalizada de la construcción pGL3 (−133A) que contenía la secuencia promotora con la variante alélica –133A/A se estableció como referencia. Tal y como se muestra en la figura 3, se produjo un incremento de 2,8 y 2,3 veces en la actividad del promotor en las líneas Hep-G2 y Caco-2, respectivamente, en presencia de la variante –133G/G. Estos aumentos resultaron ser estadísticamente significativos (p < 0,05).
Efecto del polimorfismo –133A>G en la actividad transcripcional del promotor del gen NPC1L1 en las líneas celulares Hep-G2 y Caco-2. En la gráfica se representa la media de la actividad luciferasa normalizada. Las barras de error representan la desviación estándar de los 4 experimentos independientes, cada uno de ellos realizado por triplicado en ambas líneas celulares para cada uno de las construcciones.
Las regiones promotoras proximal y distal codifican secuencias diana para la unión de distintos factores de transcripción, reguladores de la transcripción14 y, por tanto, son zonas que pueden contener polimorfismos capaces de modificar de manera relevante la expresión génica15,16. El análisis de las secuencias correspondiente a las variantes estudiadas en la región promotora de NPC1L1 llevado a cabo mediante el software MatInspector17 (Genomatix), determinó posibles sitios de unión para los factores de transcripción NF-1, STAT-1 y STAT-3 alrededor del polimorfismo –1473T>C, el factor c-Myb alrededor del polimorfismo –1128A>G, el factor RXR alrededor del polimorfismo –133A>G y el factor Sp1 alrededor del polimorfismo –18C>A. Este análisis mostró una mayor probabilidad de modificación de la secuencia consenso de unión del factor de transcripción RXR alrededor del polimorfismo –133A>G. El factor de transcripción RXR (RXR α, β, γ) forma parte de los receptores nucleares y forma heterodímeros con otros factores implicados en la regulación del metabolismo lipídico, como FXR, LXR o PPAR18. Teniendo en cuenta el resultado anterior, junto a la elevada frecuencia del alelo minoritario del polimorfismo –133A>G, se seleccionó este último para analizar su posible efecto funcional sobre la transcripción del gen NPC1L1.
Para determinar la posible funcionalidad del polimorfismo –133A>G sobre la actividad transcripcional del gen NPC1L1 se llevaron a cabo en primer lugar ensayos de retardo en gel o EMSA. Los resultados obtenidos en los ensayos de retardo en gel revelaron la formación de complejos de las sondas con los extractos nucleares de Caco-2 en presencia de las 2 variantes alélicas para este polimorfismo y que estas interacciones mostraban diferente afinidad en función de la variante alélica presente en la sonda. Posteriormente, se realizaron ensayos de actividad luciferasa y para ello se transfectaron de manera transitoria construcciones que contenían cada una de las variantes que se tenían que estudiar en las líneas celulares Caco-2 y Hep-G2. Nuestros resultados mostraron un efecto significativo del polimorfismo –133A>G en la actividad transcripcional del promotor del gen NPC1L1. El incremento de la actividad del promotor mostrado en presencia de la variante minoritaria –133G se podría explicar por la modificación de un posible sitio de unión consenso de un factor de transcripción probablemente con efecto represor. Estos datos son coherentes con los resultados obtenidos en los ensayos de retardo en gel donde la sonda que contenía la variante –133A mostró mayor afinidad de unión respecto a la sonda con la variante –133G. La presencia de la variante minoritaria –133G hace que disminuya la afinidad de unión del factor de transcripción con efecto represor por su sitio de reconocimiento y, por lo tanto, disminuya su regulación negativa demostrada con el incremento de la actividad transcripcional en presencia de ésta. El análisis realizado mediante herramientas bioinformáticas determinó que el polimorfismo –133A>G se encontraba en la posible secuencia de unión del factor de transcripción RXR. Para poder determinar la identidad del factor implicado se realizaron ensayos supershift con anticuerpo RXRα. Tal y como se muestra en la figura 2, en presencia de este anticuerpo, la banda retardada con la sonda portadora de la variante –133A desaparece, lo que sugiere que este factor de transcripción es el que se está uniendo a esta secuencia. Además, parece que la presencia del alelo –133G hace que se pierda un sitio de unión para RXR, y que cambie la especificidad de las proteínas por el ADN, puesto que la banda se mantiene en presencia de anticuerpo, lo que sugiere que otra proteína se está uniendo a este sitio.
Analizando nuestros resultados se podría considerar que la disminución de la regulación negativa podría estar asociada a un aumento de la funcionalidad del transportador NPC1L1, dando lugar a un incremento de la absorción intestinal de colesterol. Sin embargo, en nuestro estudio no se determinó la concentración de esteroles vegetales o fitoesteroles, utilizados como marcadores de la absorción intestinal de colesterol19, por lo que sería necesario su determinación en estudios posteriores para verificar que la variación de expresión pudiera dar lugar a un efecto fenotípico real. Es razonable considerar que los efectos mostrados en los ensayos con genes reporteros se puedan correlacionar con variaciones de expresiones in vivo; sin embargo, se debería confirmar la fiabilidad y validez de estos resultados mediante comparación de los datos obtenidos con otras técnicas. A la hora de determinar los posibles efectos sobre la transcripción de variaciones en la secuencia promotora de un gen, la utilización de genes reporteros es uno de los métodos de elección más utilizado y apropiado16. Sin embargo, en este ensayo se utilizan pequeñas cantidades de secuencia de ADN asociada al gen de estudio y, por lo tanto, el posible efecto observado en el análisis de la secuencia se podría suprimir o modular por los efectos de otras secuencias localizadas a lo largo del genoma16.
En conclusión, hay diferencias en la afinidad de unión de proteínas nucleares y en la actividad transcripcional del promotor del gen NPC1L1 en función del polimorfismo –133A>G, posiblemente debidas a la modificación del sitio de unión del factor de transcripción con posible efecto represor RXR. Estas diferencias de expresión observadas podrían estar relacionadas con la variabilidad interindividual en la absorción intestinal de colesterol.
Este trabajo recibió una Mención Especial en el XX Congreso Nacional de la Sociedad Española de Arteriosclerosis celebrado en Valencia en 2007.
Este trabajo ha sido posible gracias a la concesión de una beca predoctoral de la Fundación Cuenca Villoro y al proyecto del FIS PI06/1402.