La fisiología celular es actualmente mejor entendida a partir de sus componentes moleculares interactuantes. Las proteínas son las principales biomoléculas que constituyen las células. Las diferentes proteínas se organizan en asociaciones formando conjuntos, vías, o redes funcionales. Estos conjuntos se encuentran organizados en moléculas receptoras de señales extracelulares, transductoras, sensoras y efectoras de respuestas biológicas. La identificación de las diferentes vías de señalización intracelulares en los diferentes tipos de células ha requerido largas jornadas de trabajo experimental.
Se han identificado más de 300 vías de señalización intracelular en las células humanas, las cuales participan en los procesos celulares básicos y especializados para mantener la homeostasis estructural y funcional. Varias de ellas participan de forma simultánea o cercanamente consecutiva en la generación de un cambio fenotípico celular.
En esta revisión analizamos las principales vías de señalización intracelular que participan en el proceso de proliferación celular y remarcamos la estrategia de utilizar algunos componentes de estas vías como blanco de un potencial intervencionismo terapéutico.
Nowadays, cellular physiology is best understood by analysing their interacting molecular components. Proteins are the major components of the cells. Different proteins are organised in the form of functional clusters, pathways or networks. These molecules are ordered in clusters of receptor molecules of extracellular signals, transducers, sensors and biological response effectors. The identification of these intracellular signaling pathways in different cellular types has required a long journey of experimental work.
More than 300 intracellular signaling pathways have been identified in human cells. They participate in cell homeostasis processes for structural and functional maintenance. Some of them participate simultaneously or in a nearly-consecutive progression to generate a cellular phenotypic change.
In this review, an analysis is performed on the main intracellular signaling pathways that take part in the cellular proliferation process, and the potential use of some components of these pathways as target for therapeutic interventionism are also underlined.
La capacidad de los seres humanos para ver, percibir y responder a señales ambientales depende de la activación de las vías de señalización intracelulares de las diferentes células que los conforman. Estas vías de señalización proporcionan los mecanismos para organizar la información molecular a través de una célula, algo similar a la manera en que el sistema nervioso central transduce la información del ambiente y organiza la respuesta integral de los diferentes órganos del cuerpo. Así, los órganos de los sentidos son sensibles a ciertas formas de estímulos (p.ej., luz, presión u ondas sonoras) dado que los receptores de la superficie celular se unen con ligandos específicos. Una célula puede tener docenas de receptores distintos que simultáneamente reciben señales hacia el interior de la célula al mismo tiempo. Todas las células emplean vías de señalización intracelulares específicas para censar la información de llegada, traducirla, interactuar diferentes moléculas y responder biológicamente generando un patrón fenotípico específico1.
Las vías de señalización consisten, en su mayoría, en una cadena de proteínas que interactúan una con la otra en una secuencia previa establecida evolutivamente. Las plantas y animales comparten ciertos mecanismos básicos de señalización intracelular; sin embargo, algunos componentes organizacionales son predominantes, otros poco frecuentes y algunas vías son únicas en cada reino.
En las últimas décadas se ha identificado una abundante cantidad de vías de señalización intracelular con innumerables componentes moleculares en cada una de ellas; esta caracterización se ha realizado en el contexto de los diferentes procesos celulares de tipos específicos de células tanto en condiciones de salud como de enfermedad. Hemos logrado un entendimiento general de cómo estos grupos de moléculas funcionan a manera de circuitos y sistemas complejos.
El desarrollo y el funcionamiento normales de los organismos procariontes y eucariontes multicelulares son gobernados por un conjunto sucesivo de interacciones celulares cuyo lenguaje son cambios bioquímicos. Los diferentes estados de las células se deben principalmente a patrones de transcripción génica que, al ser traducidos, interactúan con otras moléculas y finalmente regulan diferentes funciones celulares.
La clonación y secuenciación de segmentos de ADN y la purificación y secuenciación de las proteínas han conducido, por ejemplo, a entender como algunos oncogenes codifican factores de crecimiento (como v-Sis) a reconocer sus receptores (como ErbB) y a proteínas de vías de señalización (como v-Src), etc. Igualmente se ha demostrado que la transducción de señales ocurre tanto a través de segundos mensajeros, como de interacciones de proteína-proteína, y proteína-ácido nucleico, o como las condiciones celulares epigenéticas modifican la respuesta efectora debida a las señales extracelulares2.
En este artículo describiremos los principios generales de funcionamiento de las vías de señalización intracelulares y analizaremos la organización modular de las vías que participan en el proceso de proliferación celular, como ejemplo de un modelo molecular complejo que las células emplean para efectuar los diferentes procesos celulares.
Proteínas como biomoléculas participantesLas proteínas son las biomoléculas más abundantes y complejas dentro de las células, y constituyen los elementos estructurales y funcionales de ellas. Las proteínas son las moléculas funcionales del trabajo biológico. Por su codificación directa a partir de genes en la célula humana corresponderían a 22,000 diferentes; sin embargo, este número se incrementa a más de 10 veces debido a las diferentes combinaciones de transcritos de los genes y por los cambios postraduccionales que suceden en el retículo endoplásmico y en el aparato de Golgi. Están compuestas esencialmente de 20 tipos de aminoácidos en diferentes proporciones; las hay pequeñas (40-80 aminoácidos), intermedias y grandes (más de 10,000 aminoácidos), y bioquímicamente se organizan en cadenas de aminoácidos (estructura primaria), en hélices α y hojas β plegadas (estructura secundaria), formando moléculas tridimensionales pequeñas por influencias de atracción y repulsión de sus aminoácidos (estructura terciaria), y constituyendo moléculas tridimensionales mayores por influencia de agrupamientos de otras menores (estructura cuaternaria). Diferentes estudios de afinidad y secuenciación de las proteínas en las últimas décadas han demostrado que segmentos peptídicos específicos o dominios intramoleculares funcionan de manera semiindependiente y a menudo se identifican con funciones específicas, como por ejemplo dominios de unión a cinasas y a fosfolípidos. Las proteínas son biomoléculas que interactúan con pequeñas moléculas de otro tipo o con proteínas adyacentes modificándolas a través de provocarles nuevos cambios intramoleculares, los cuales constituyen indicadores de activación biológica. Dos principales mecanismos de activación o represión del funcionamiento de las proteínas son la fosforilación/desfosforilación y su unión a GTP o GDP1,3,4.
Diversos tipos de proteínas, por su gran versatilidad de tipo, forma, dominios, respuestas e interconectividad con otras proteínas, son frecuentemente componentes principales en la recepción, transducción, regulación y control celular de las señales extracelulares e intracelulares. Las diversas proteínas bioactivadas son la base de la respuesta bioquímica en las vías de señalización intracelular, las cuales conducen finalmente al cambio fenotípico estructural y funcional de las diferentes células. Cada una de las células vivas contiene mecanismos de participación molecular que «prenden» y «apagan» las respuestas de las diferentes señales eléctricas y químicas.
Sistemas de señalización celular. Transducción de señales, sensores y efectores molecularesLa organización funcional de las proteínas intracelulares se realiza en conjuntos, los cuales se ubican generalmente en compartimientos subcelulares específicos. Los componentes básicos de una vía de señalización intracelular incluyen las moléculas de: la señal extracelular, los receptores de la señal, los transductores, los sensores, los efectores y, finalmente, la activación de la respuesta celular para expresar un fenotipo particular1,5. Las señales extracelulares pueden actuar a distancias largas o cortas, son generalmente moléculas grandes e hidrófilas y corresponden a neurotransmisores, hormonas (incluyendo hormonas locales), citocinas, factores de crecimiento, moléculas de superficie celular y de estimulación sensorial. Los caminos de enlace de estas señales con los receptores se realizan por 4 mecanismos: juxtacrino, autocrino, paracrino y endocrino. Las señales extracelulares pueden encontrarse libres en el líquido extracelular o embebidas en la matriz extracelular, y la respuesta depende de que el receptor específico se encuentre presente en la célula. Las proteínas receptoras detectan la señal a nivel de la membrana celular y la transmiten a moléculas transductoras internas, las cuales son de 3 tipos principales: receptores asociados con canales iónicos (actúan de la manera más sencilla y más directa), receptores asociados a proteínasG (monoméricos y heterotriméricos) y los asociados con enzimas (la mayoría con actividad de cinasa) que participan en el control de comportamientos celulares complejos3,4. Muchas moléculas señalizadoras extracelulares interactúan con más de un tipo de receptor. Las señales que llegan a los receptores asociados a proteínasG o a las enzimas se transfieren a sistemas de transmisión complejos formados por cascadas de moléculas de señalización intracelular, y su activación les confiere cambios covalentes y conformacionales específicos que las convierte en los sustratos de la siguiente reacción dentro la vía de señalización. Salvo algunas moléculas pequeñas, como GMP cíclico, AMP cíclico y Ca2+, la mayoría de las moléculas de señalización corresponden a proteínas. En general, las moléculas pequeñas amplifican masivamente la señal y participan como estímulo para la activación simultánea de varias vías de señalización vecinas; las proteínas activadas generalmente transitan por una o pocas vías, y algunas de ellas funcionan como parte secuencial de la vía, otras participan como interruptores moleculares o hubs de una o varias vías, y otras, como moléculas de andamiaje1,6. Las moléculas transductoras llevan la señal a moléculas sensoras y a moléculas efectoras intracelulares; las primeras pueden ser intermediarias o finales en la vía de señalización específica; algunas de las proteínas efectoras (p.ej., factores de transcripción) pueden actuar sobre blancos moleculares únicos o múltiples y conducen procesos como exocitosis, fagocitosis, remodelación de actina, activación de vías metabólicas y expresión génica. La ejecución de programas de interacción molecular a menudo requiere síntesis de novo de proteínas. Las diferentes vías de señalización que emplean las células para regular sus diferentes procesos celulares interrelacionados están interconectadas a través de moléculas hubs, formando redes complejas donde participan varias vías de señalización. Durante su activación, la señal puede viajar en algunas vías de señalización particulares por una ruta preferencial o ruta canónica, o eventualmente por una ruta alterna o ruta no-canónica1. La terminación de la activación de una vía de señalización intracelular implica detener el proceso local en uno o varios de los componentes a través de la acción de enzimas opuestas (p.ej., cinasas vs. fosfatasas) o de inhibidores específicos. Evolutivamente el genoma contiene todos los códigos estructurales de los componentes de las diferentes vías de señalización. Así, por ejemplo, en el proceso de diferenciación cada célula especializada desactiva la codificación de las moléculas que participan en el proceso de proliferación celular y activa la codificación de las moléculas que participan en los procesos de catabolismo especializado o de funcionamiento especializado de los programas del fenotipo estructural y funcional que ejecuta la célula terminalmente diferenciada.
La activación de las vías de señalización intracelular se encuentra estrictamente organizada en espacio y tiempo. El proceso de señalización viaja inicialmente de fuera a dentro, y la respuesta celular de dentro a fuera. Para cada mecanismo de activación molecular on existe un mecanismo de inactivación off. El lenguaje de activación de la vía se establece por la interacción directa de proteína-proteína, o por sus modificaciones covalentes como fosforilación y acetilación, provocadas en la vía por la biomolécula corriente arriba; esta activación requiere la adecuada coordinación temporoespacial de sus componentes. Algunos patrones de la respuesta celular, como aumento del crecimiento y la división celular, implican cambios tanto en la expresión génica y en la síntesis de nuevas proteínas (respuestas lentas), mientras que cambios en el movimiento, en la secreción o en el metabolismo celulares no requieren la participación de la maquinaria nuclear y se producen con mayor rapidez.
El funcionamiento on y off en las vías de señalización intracelular presenta un elevado grado de plasticidad celular, y dichas vías de señalización son constantemente remodeladas por las señales externas e internas requeridas para el mantenimiento de la homeostasis celular; los mecanismos que desactivan una señal son tan importantes como aquellos que la activan. Los comportamientos complejos de las células son frecuentemente controlados por redes de proteincinasas; estas cinasas pueden modular además de los componentes de su propia vía de señalización, componentes de otras vías de señalización cercanas o relacionadas a través de moléculas hubs. La integración adecuada de las múltiples vías de señalamientos intracelulares regula los procesos complejos como desarrollo, proliferación, diferenciación, respuesta al estrés, apoptosis, etc. Las alteraciones en la regulación de las vías de señalización conducen a la inadecuada activación de la respuesta celular y al inicio de muchas enfermedades. La investigación de las vías de señalización intracelulares en los diversos tipos celulares es un área activa, y permanentemente se efectúan nuevos descubrimientos1,6,7.
Principales vías de señalización intracelulares participantes en los diversos procesos celulares estructurales y funcionalesLas vías biomoleculares de señalización intracelular corresponden a conjuntos de proteínas y de otras biomoléculas que se organizan como cascadas de interacciones espaciotemporales y son las responsables de lograr las características fenotípicas celulares. Cada vía de señalización molecular se ubica generalmente en un compartimento subcelular distinto6. La información de las vías de señalamientos o signalosoma es esencial en la construcción de modelos de sistemas biológicos. La Enciclopedia de Kyoto sobre genes y genomas (KEGG) es una de las bases de datos donde se han registrado las principales vías de señalización intracelulares que participan en la regulación de los principales procesos celulares (tabla 1)8; algunas de ellas emplean moléculas mensajeras pequeñas, también conocidas como segundos mensajeros, para la transmisión y amplificación del mensaje, y otras emplean predominantemente proteínas como mensajeros y sensores internos1. El primer grupo está formado, por ejemplo, por las vías del AMP cíclico, del ADP-ribosa cíclico/NAADP, de los canales operados por voltaje (VOC), de los canales operados por receptor (ROC, como K+, Ca2+ o Cl−), la vía de la activación de la fosfolipasaC (PLC) que degrada los fosfatidilinositoles de la membrana celular, la vía de activación de la fosfatidilinositol 3 cinasa (PI3K, que fosforila a PIP2 y forma PIP3), la vía del óxido nítrico/GMP cíclico, la vía de señalización redox, la de fosfolipasa D (PLD), la vía de la esfingomielina, la vía del AMP, la vía del NAD, y otra gran cantidad de vías participantes en el metabolismo. El segundo grupo está formado por las vías de las cinasas activadas por mitógenos (MAPK), la del factor nuclear κB (NF-κB), la de la cinasa Janus/señal transductor y activador de la transcripción (JAK/STAT), SMAD, WNT, Hedgehog, Hippo, Notch, la vía de estrés del retículo endoplásmico, y otras múltiples.
Número de vías de señalización intracelulares participantes en los principales procesos celulares estructurales y funcionales
| Procesos celulares | Número de vías participantes | Ejemplos |
|---|---|---|
| Metabolismo | 130 aprox. | Metabolismo de hidratos de carbono, lípidos, aminoácidos, nucleótidos, factores y vitaminas. Ciclos anabólicos y catabólicos, etc. |
| Procesamientos genéticos/genómicos | 22 aprox. | Transcripción, traducción, cambios postraduccionales, replicación y reparación del ADN |
| Procesamientos de las señales extracelulares (ambientales) | 24 aprox. | Recepción, transducción y respuesta biológica |
| Transportación intracelular de organelos | 4 aprox. | Endocitósis, exocitosis; regulación de autofagia |
| Movilidad celular | 3 aprox. | Remodelación del citoesqueleto, quimiotaxis, etc. |
| Crecimiento celular y muerte | 14 aprox. | Ciclo celular, meiosis, apoptosis, vía de p53, etc. |
| Comunicación intercelular | 4 aprox. | Uniones estrechas, uniones focales, uniones GAP, etc. |
| Vías específicas de las células del sistema inmunológico | 15 aprox. | Vías de señalización de receptores de los linfocitos CD8+ y Cd4+, etc. |
| Vías específicas de las células del sistema endocrino | 12 aprox. | Vías de señalización de la insulina, de los estrógenos, de los andrógenos, etc. |
| Vías específicas de las células del sistema circulatorio | 6 aprox. | Vías de señalización en la contracción de las células cardíacas y de los vasos |
| Vías específicas de las células del sistema digestivo | 10 aprox. | Vías de señalización participantes en la absorción, digestión y secreción, etc. |
| Vías específicas de las células del sistema excretor | 5 aprox. | Vías de señalización participantes en la regulación de la excreción del agua, sodio, bicarbonato, etc. |
| Vías de específicas de las células del sistema nervioso/sensorial | 14 aprox. | Vías de señalización de las sinapsis dopaminérgicas, colinérgicas. Fototransducción, transducción olfatoria, etc. |
| Vías específicas de las células que participan en el desarrollo embrionario | 4 aprox. | Vías de señalización participantes en el desarrollo del eje dorsoventral, etc. |
| Vías específicas de las células que responden a los cambios medioambientales | Múltiples | Ciclos circadianos, interacciones con microorganismos no patógenos |
| Vías específicas de células que responden a agentes infecciosos, tóxicos, drogas etc. | Múltiples | Respuestas antibacterianas, antivirales, etc. |
aprox.: aproximadamente.
La dilucidación de las vías de señalización intracelulares ha implicado la realización de múltiples ensayos con técnicas genómicas y bioquímicas de proteínas en modelos dinámicos de células normales y de células tumorales7. La identificación de una vía en particular requiere muchos años de trabajo experimental. La determinación de la participación y de la importancia de una proteína particular en el proceso de señalización se lleva a cabo a través de diferentes estrategias experimentales frecuentemente combinadas, como la introducción o inactivación de un gen que codifique dicha proteína, la valoración de su expresión y su activación/desactivación postraduccional, todo ello en relación con un efecto celular específico fenotípico. La desactivación de las proteínas que participan en las vías de señalización intracelulares ha sido estudiada por medio del uso de técnicas moleculares genéticas destinadas a bloquear o a disminuir su expresión génica como las de knock-out y de knock-down9. Para explorar las proteínas activadas por fosforilación se emplean anticuerpos específicos o se marcan con ATP radiactivo; para la identificación de las proteínas interactuantes se utilizan ensayos de coinmunoprecipitación, y una vez identificadas las proteínas interactivas, sus dominios activos pueden ser reconocidos por medio de emplear ensayos de ADN recombinante (mutagénesis dirigida) a través de la construcción de proteínas mutantes e investigar su interacción e importancia. Una estrategia que facilita la identificación de las proteínas que participan en alguna vía de señalización intracelular específica es realizando el estudio poblacional de decenas de miles de moscas de la fruta o de nematodos tratados con un mutágeno vs. el grupo testigo, buscando mutantes en los cuales se presenten la alteración de un rasgo fenotípico debido al funcionamiento inadecuado de una o varias vías de señalización; en ellos se identifican los genes alterados que codifican las proteínas que participan en las cascadas de señalización, comparándolos con sus controles1,4. Uno de los eventos que requiere mayor trabajo experimental es determinar la ubicación de una proteína participante corriente abajo o corriente arriba en una vía de señalización, respecto a las otras proteínas participantes.
El estudio experimental acumulativo en esta área ha identificado una gran cantidad de vías de señalamientos moleculares intracelulares relacionadas con funciones celulares globales como metabolismo, proliferación, apoptosis; funciones celulares especializadas como la respuesta inmunológica, interacciones de los factores de transcripción, e interacciones proteína-proteína; y funciones celulares alteradas en diferentes enfermedades10. Esta abundante información ha requerido para su análisis el apoyo bioinformático a través de la construcción de bases de datos de registro e interacciones moleculares (nodos), como la BioPAX, Pathway Interaction Database, Reactome, BioCarta, KEGG, GenMAPP, Genenetwork, Cytoscape, NetPath, entre otras. Muchas de ellas se han construido en colaboración de grupos interdisciplinarios, por ejemplo la base NetPath, que es un proyecto de colaboración del Pandey Lab (John Hopkins University) y del Instituto de Bioinformática (Bangalore, India). Algunas megabases de datos como la del National Center of Biotecnology Information (NCBI), la Pathguide y las WikiPathways11,12 integran múltiples bases de datos de módulos o nodos relacionadas con las vías de señalamientos celulares que participan en diversos tipos celulares normales y en células de diferentes enfermedades. La KEGG13, a partir de la integración de datos genómicos, transcriptómicos, proteómicos y metabolómicos, ha organizado los mapas de más de 300 vías de señalamientos intracelulares humanos relacionados con el metabolismo, con el procesamiento de información genética, con el procesamiento sobre respuesta a cambios ambientales, con los procesos celulares básicos, con diferentes tipos de células diferenciadas de aparatos y sistemas del organismo y con modelos particulares de diferentes enfermedades humanas8, demostrándose que cada proceso celular básico o especializado requiere la participación de varias o múltiples vías de señalización intracelulares que funcionan generalmente de manera simultánea. Estos conjuntos interactuantes de vías de señalización intracelulares que producen un cambio fenotípico se denominan módulos7; ejemplos de módulos de vías de procesos celulares básicos son los del transporte molecular y catabolismo, motilidad celular, crecimiento, muerte y comunicación celulares (todos ellos con sub-vías específicas); ejemplo de módulos identificados de las vías de algunas enfermedades son los de algunos cánceres (colorrectal, pancreático), enfermedades inmunológicas (asma, lupus eritematoso), enfermedades neurodegenerativas (Alzheimer, esclerosis lateral amiotrófica), adicciones (a la cocaína, a la morfina), enfermedades cardiovasculares (miocardiopatía hipertrófica), enfermedades endocrinas y metabólicas (diabetes mellitus tipos1 y 2), enfermedades infecciosas (infección por salmonela, influenzaA)1,6. A través de estos mapas podemos entender cómo la sobreactivación o represión en la expresión de genes ejercida por efectos de mutaciones, epimutaciones o cambios en el número de copias de una región particular del genoma humano modifican la activación o represión de los señalamientos intracelulares específicos y conducen a cambios estructurales y funcionales de las células. Igualmente el estudio de estos mapas moleculares nos facilita el eventual intervencionismo para utilizar o diseñar moléculas con afinidad bioquímica y dirigirlas a la que se encuentra(n) alterada(s), para bloquear o modificar la reacción bioquímica específica, inducir un cambio termodinámico en la vía de señalamiento intracelular y, finalmente, un cambio en la expresión génica y en el fenotipo de la célula. La corrección del funcionamiento en la vía de señalización intracelular alterada podrá limitar o revertir el daño celular, tisular y recuperar la homeostasis14. Como un ejemplo de la participación de múltiples de vías de señalización intracelulares en algunos módulos o programas de procesos fenotípicos celulares, analizaremos brevemente las principales vías de señalización intracelulares que participan en la división y proliferación de células eucariotas.
Módulo de las vías de señalización intracelulares participantes en el ciclo de división celular y proliferación. Blancos potenciales de intervencionismo molecularLas células eucariotas, dependiendo de la activación/desactivación, desviación o modulación en las redes de las vías de señalización del ciclo de división celular y proliferación, pueden transitar por 4 destinos: senescencia, apoptosis, diferenciación y proliferación1,3.
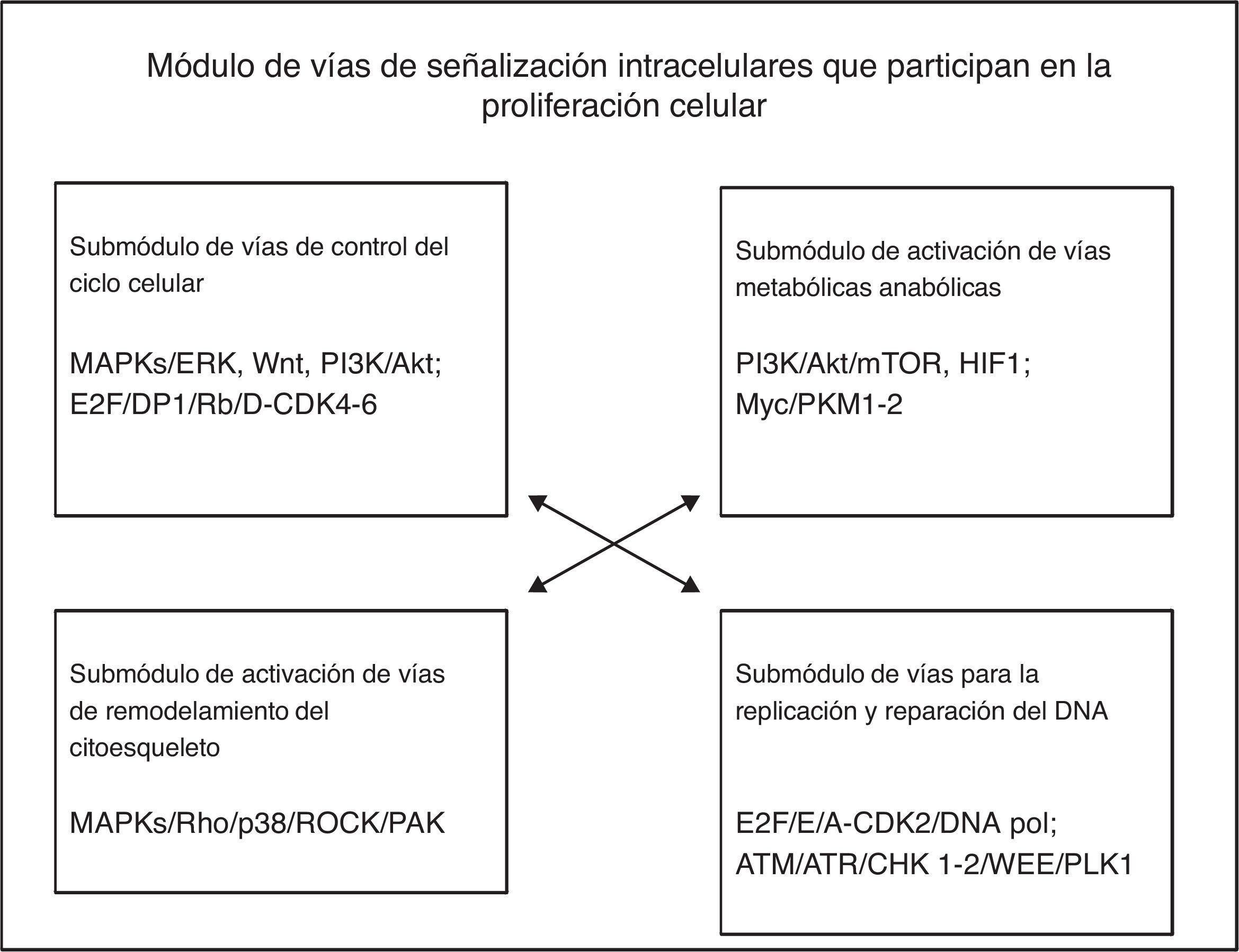
El módulo de división y proliferación celular está constituido por 4 grupos principales de vías de señalización intracelulares: las del control del ciclo celular, las de activación de las vías metabólicas anabólicas, las de activación del remodelación del citoesqueleto y las de la replicación y reparación del ADN. En el proceso de proliferación celular, las células se reproducen a sí mismas por crecimiento intracelular para luego dividirse en 2 copias iguales, lo cual implica que la célula requiere mínimamente incrementar su biomasa y replicar su genoma. Diferentes factores de crecimiento (GF) activan a las células en la interfase para que progresen en el ciclo celular e incrementen su masa celular por medio de aumentar la biosíntesis de macromoléculas mediante la activación de las vías de señalización anabólicas. Los GF activan la vía MAPK/Ras/RAf/ERK para inducir la síntesis de 3 tipos de ciclinas D (D1, D2 y D3) y otras proteínas reguladoras positivas de mitosis (Myc y Jun); la ciclina D1 se une a CDK4/6 para formar un complejo eficaz para activar varios sustratos requeridos para iniciar el ciclo celular, como Myc y AP-1. La ciclina D es regulada por las vías MAPK/Ras, β-catenina-Tcf/LEF, PI3K y Rho/FAK. La disponibilidad de la ciclinaD en la célula es controlada por un balance entre su síntesis dependiente de Ras/Raf/MAPK y la de su estabilidad dependiente de Ras/PI3K/AKT, GSK3 y SCF1.
El ciclo celular se divide en 2 fases principales: la fase M y la interfase. La fase M incluye los fenómenos sucesivos de la mitosis y la citocinesis. La interfase se divide en las fases G1, S y G2, siendo la fase S la equivalente al periodo de síntesis de ADN. En la fase G1, la célula aumenta de tamaño y activa la replicación del ADN; en la fase G2, la célula sigue creciendo y se activa la maquinaria de la mitosis; en la fase M, la célula detiene su crecimiento y se divide en 2 células hijas separándose por citocinesis. Después de la división celular, o fase «posmitosis», cada célula hija entra en la interfase y se prepara para 2 caminos: una próxima división celular o un estado de no-proliferación. En los siguientes párrafos se describen muy someramente las principales moléculas participantes en las vías de señalización del ciclo de división y proliferación/crecimiento celular (para una amplia revisión consultar a Berridge, 2012)1,15.
La fase G1 es llamada fase de crecimiento, y en ella se forman millones de proteínas, enzimas, biolípidos y nucleótidos requeridos para la biosíntesis del citoplasma, membranas celulares, organelos, ADN y ARN. La duración de G1 es muy variable entre las diferentes células, y particularmente se encuentra bajo el control de la proteína p53. Las vías de señalamientos intracelulares que participan a través de los GF son las vías o cascadas proteincinasas activadas por mitógeno (MAPK), la vía WNT que actúa a través de β-catenina y la vía PI3K; estas incrementan la transcripción de los complejos ciclinas/cinasas dependientes de ciclinas (CDK) y particularmente de la ciclinaD, que actúa sobre la proteína retinoblastoma para retirar su efecto inhibitorio a E2F, lo cual activa la formación de las ciclinasE y A, y estas conducen positivamente a la célula a entrar en la faseM. En contraste a estos efectos positivos, las vías del factor de crecimiento de transformaciónβ (TGF-β)/Smads actúan inhibiendo la entrada de la célula al ciclo celular por medio de la expresión de los inhibidores de CDK (familias de INK4 y Cip/Kip) y de p53; particularmente p53 detiene el ciclo cuando el ADN no es correctamente replicado, produciendo las proteínas p21, GADD45 y la 14-3-3, y promoviendo la transcripción de factores apoptóticos15.
Como hemos mencionado, tras a la recepción de los GF por la célula se sintetiza rápidamente la ciclinaD, que persiste a lo largo de todo el ciclo de división celular; en cambio, la ciclinaE aparece como un pico en la parte final de G1, la ciclinaA solo en las fases S y G2, y la ciclinaB se expresa en las fasesG2 y M, mientras que las CDK se producen constitutivamente. En contraste con estos incrementos de las ciclinas, el inhibidor de ciclinas/CDK disminuye desde el inicio de la fase G1, manteniéndose así a lo largo de todas las fases del ciclo celular. Las ciclinas/CDK funcionan como serina-treonina cinasas: la ciclina D/CDK4-6 fosforila la proteína Rb, con lo cual la familia de los factores de transcripción E2F activan la transcripción de los genes de ciclinaE y A, de la fosfatasa cdc25A que remodelan la cromatina. Luego son activados los complejos ciclina E/CDK2 y ciclinaA/CDK2 para iniciar la síntesis del ADN, mientras que la función de la cdc25A permite la activación de la ciclinaB/CDK1 para iniciar el ensamblado del huso mitótico16.
La vías MAPK son clásicamente las vías de señalización más importantes que regulan la proliferación celular, y la cicinaD1 es el principal sensor de las señales extracelulares que conduce la fase de progresión del ciclo de G1 a S17. Analicemos brevemente la complejidad de estas vías y la de la expresión de la ciclinaD1.
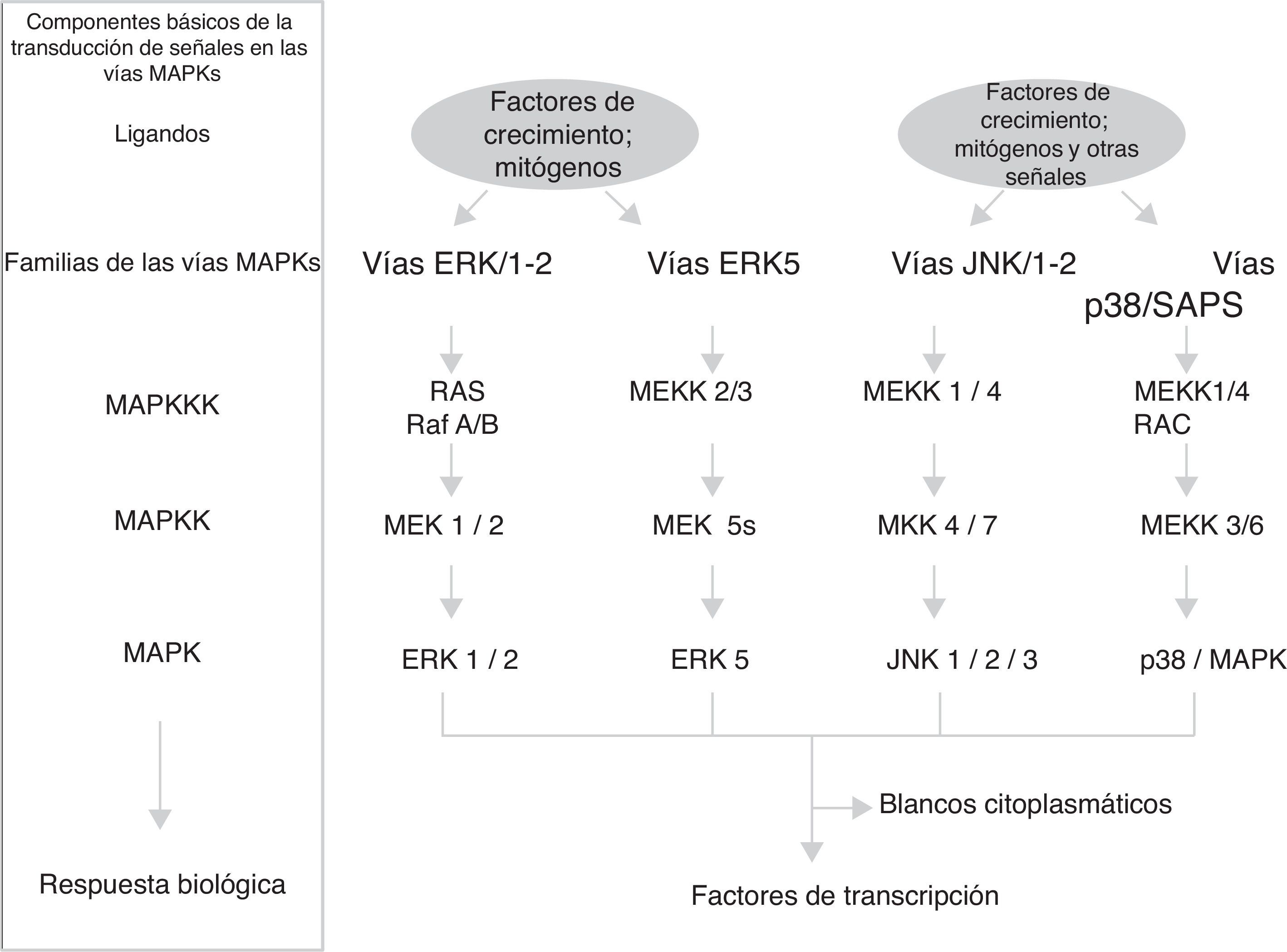
Vías MAPKEstas vías no solo participan en la regulación de la proliferación celular, sino también en la regulación de la diferenciación y de la muerte celular. El módulo de las vías MAPK contiene de 3 a 5 componentes o filas (horizontales) de proteincinasas (fig. 1): una MAP-cinasa-cinasa-cinasa (MAPKKK) que a su vez activa sucesivamente a una MAP-cinasa-cinasa (MAPKK) y a una MAP cinasa (MAPK), esta fosforila a las serinas y treoninas de las proteínas que conducen a cambios en su función y a la expresión de genes. El grupo de las MAP cinasas contiene 3 familias de componentes interrelacionados (distribución vertical) que reciben, transmiten y responden a señales extracelulares diferentes: las ERK, las JNK y las p/38/SAPK; particularmente la familia de las vías de señalización reguladas por señales extracelulares (ERK) se divide en 2 subfamilias (fig. 1): las pequeñas ERK1 y ERK2 y la grande ERK517. Cada cascada o familia/subfamilia responde a distintos factores de crecimiento, de diferenciación y señales extracelulares de estrés y a citocinas inflamatorias; contienen diferentes proteínas de andamiaje y activan a diferentes proteínas o a la expresión de genes (algunos de ellos similares). Esto implica que las vías MAPK corresponden en conjunto a un sistema de cientos de proteínas (muchas de ellas corresponden a isoformas producidas por el procesamiento de la expresión génica de las MAPK) las cuales regulan diferentes funciones fisiológicas celulares al actuar como factores de transcripción o activar enzimas nucleares/citoplasmáticas18,19. Los principales mecanismos temporoespaciales que se han propuesto para que las vías MAPK regulen las funciones celulares son: a)distinta duración e intensidad en la señal extracelular; b)interacciones diferentes de los componentes de las 3 familias con las distintas proteínas de andamiaje; c)diferentes interacciones entre las 3 familias de MAPK; d)compartimentalización subcelular diferente, y e)complejidad intrínseca de los componentes en la distribución vertical de cada cascada17.
Ciclina D1La ciclina D1 es el sensor que integra las señales extracelulares con la maquinaria molecular que, unida con CDK4-6, activa la progresión del ciclo celular. Diferentes factores de transcripción como AP-1, SP-1, E2F, OCT-1, son inducidos por la activación de las vías ERK1/ERK2 y ERK5, o de las vías PI3K/Akt y Wnt/β-catenina (u otras como NF-κB, JAK/STAT) promoviendo la expresión de la ciclina D1 al unirse a su promotor. Algunos conjuntos de factores de transcripción organizados en complejos funcionales como MuvB, B-Myb, FoxM1, DREAM, E2F utilizan diferentes circuitos de feedback para interactuar en tiempos diferentes con la ciclina D1 y conducir la progresión del ciclo celular en sus etapas tempranas y tardía de la fase G120,21.
Como hemos anotado, las células en proliferación incrementan significativamente la producción de ATP y la síntesis de biomoléculas que incluyen lípidos, proteínas y ácidos nucleicos, por lo que reprograman sus vías metabólicas de emplear ciclos catabólicos como el de la fosforilación oxidativa mitocondrial al de transitar por ciclos anabólicos como el de la vía de glucólisis aeróbica y otras rutas anabólicas22,23. La glucólisis produce ATP a mayor velocidad, por sobreexpresión de diversas enzimas que se emplean en esta vía, y proporciona mayor cantidad de moléculas intermediarias para biosíntesis de macromoléculas. El eje regulador del ciclo celular CDK4/pRb/E2F1 es también un importante factor en el control del estatus energético y del proceso anabólico biosintético23.
La célula al activar la vía PI3K/Akt incrementa el gasto de nutrientes, particularmente glucosa y glutamina, y se re-direcciona la utilización de los carbonos por las enzimas glucolíticas como la piruvato cinasa hacia la síntesis de macromoléculas. Durante esta fase, los GF son acoplados a receptores tirosina-cinasa y a otros asociados a proteínasG, los cuales estimulan la formación del inositol 1,4,5trifosfato (IP3) y de diacilglicerol, que liberan Ca2+ y encienden las vías de biosíntesis. La mTOR cinasa, una serina/treoninacinasa que es inhibida por la rapamicina, y el factor de transcripción Myc por medio de la vía PKB/TSC1/2, son las moléculas coordinadoras de la síntesis de proteínas y de ácidos nucleicos, respectivamente. Las relaciones entre las vías de señalización de progresión del ciclo celular y las vías metabólicas son bidireccionales, ambas ejecutan controles de feedback entre ellas24.
Además de estos 2 grupos de vías de señalización, otro grupo de vías de señalización participan en la progresión del ciclo celular: las relacionadas con el remodelado del citoesqueleto intracelular, las cuales conducen modificaciones en la forma de la célula, en el alineamiento de los cromosomas, en el desplazamiento de organelos, en la polarización y en la separación de las 2 células hijas al final de la mitosis; en ellas las proteínas Rho-GTPasa funcionan como moléculas hubs. Las proteínas Rho, miembros de la superfamilia Ras, son reguladores del ensamblaje y organización de las formaciones del citoesqueleto compuestas por actina y microtúbulos, que contribuyen en la formación del huso mitótico y su unión al cinetocoro, coordinan el anillo contráctil y la separación celular durante la citocinesis, etc. Las proteínas Rho incluyen a RhoA, Rac1 y Cdc42 y se interconectan con las vías de señalización MAPK, Wnt/β-catenina y PI3K; particularmente participan en las cascadas JNK y p38/SAPK de las vías MAPK, en las cuales Ras interactúa directamente con cada una de 3 proteínas18, transmitiendo la señal corriente abajo por la filas de las proteincinasas, activando las proteínas de remodelación del citoesqueleto como ROCK, MKLP1, PKN y mDia25,26.
En la fase S se sigue replicando el genoma, cromosomas e histonas, logrando su completa duplicación. El complejo ciclina E/CDK2 contribuye a la progresión del ciclo celular a través de fosforilar a Rb, remover a p21, p27 y p57 e iniciar la síntesis del ADN. Los complejos ciclina/CDK reclutan y afinan la maquinaria molecular de replicación y reparación del ADN. La síntesis del ADN transita por 2 grandes vías de señalización: la síntesis por sí misma y la reparación de un daño previo del ADN por agentes exógenos o endógenos (p.ej., compuestos reactivos de oxígeno)27 o la corrección de errores que suceden en el proceso de replicación (vías de reparación de unión de bases no complementarias). Las vías de respuesta al daño del ADN (DDR) se dividen en aquellas que resarcen lesiones de una cadena (reparación de la escisión de nucleótidos y bases) o de las 2 cadenas (recombinación homóloga y unión de segmentos terminales no homólogos), contando con moléculas que reconocen el daño como Ku70/80, RAD50, PARP, FANC, BRCA, moléculas transductoras como ATM y ATR, y moléculas efectoras como CHK1 y CHK228,29. P53 funciona como una de las principales moléculas coordinadoras entre las vías del ciclo celular y de la síntesis del ADN.
En la fase G2 la célula continúa la fase metabólica biosintética, verifica la fidelidad del ADN replicado y termina la formación del huso mitótico. El complejo ciclina B/CDK1 controla las 2 fases finales del ciclo celular.
En la fase M se realiza la rotura y división nuclear a partir de sus distintas subfases: profase, metafase, anafase, telofase, y termina en la citocinesis. En la fase M se divide el núcleo, citoplasma, organelos y membrana celular en 2 células que contienen cantidades iguales. Los principales eventos de esta fase son la rotura de la envoltura nuclear, el ensamble del huso mitótico, la separación de los cromosomas y la citocinesis. La ciclina B/CDK1 conduce a la formación del huso mitótico organizando la polimerización de redes de microtúbulos para formar su centro de organización (MTOC) y dirigir su orientación; aquí participa la vía de señalización del ensamble del huso mitótico en la cual interviene el complejo de activación de la anafase (APC)1,5. La separación de los cromosomas es modulada por el complejo APC/Plk1/SCF que hidroliza las moléculas de cohesinas. En la citocinesis el complejo MgcRacGAP/RhoA reestructura los filamentos del citoesqueleto para formar un anillo contráctil de actinomiosina26.
De acuerdo con la descripción anterior, el módulo fenotípico por el cual las células progresan en el ciclo de división celular y proliferan, implica la participación de 4 submódulos de vías de señalización intracelulares en forma simultánea o muy cercanamente secuencial, como se muestra en la figura 2. Cada una de las vías puede contener más de 200 componentes organizados en filas horizontales (ordenamientos corriente arriba, corriente abajo) y en filas verticales (distintas cascadas o familias), como sucede particularmente en las vías de las MAPK. A su vez, cada uno de los submódulos de las vías de señalización interconecta directa o indirectamente con los otros submódulos de vías relacionadas directa o indirectamente con el efecto fenotípico de proliferación celular.
A partir del análisis de este modelo, consideramos que en la generación de cada uno de los rasgos o características fenotípicas celulares, participan muy probablemente varias o múltiples vías de señalización intracelulares predominantes y múltiples vías de señalización subpredominantes que se coordinan de forma simultánea o cercanamente sucesiva. En ellas intervienen una gran cantidad de componentes moleculares que pueden ser blancos potenciales para el intervencionismo terapéutico dirigido; estas dianas moleculares pueden corresponder principalmente a las moléculas hubs de las cascadas de señalización de una sola vía, o a aquellas hubs que regulen la interconexión con otras diferentes vías fundamentales para lograr el cambio fenotípico celular específico.
Así, en la proliferación celular diferentes grupos de investigadores han iniciado una gran cantidad de ensayos experimentales preclínicos y clínicos en fasei-ii para tratar de modificar la reprogramación del ciclo celular en las células cancerosas a través de la administración farmacológica de agentes activos dirigidos a alterar las moléculas hubs de sus vías de señalización predominantes y subpredominantes. Una de estas estrategias ha sido la utilización de algunos flavonoides y compuestos derivados de purinas, los cuales modulan a diferentes CDK a través de su efecto inhibitorio sobre los dominios de las serina-treonina cinasas, y con ello su alto índice de proliferación30.
La identificación de las alteraciones en la regulación de las vías de señalización MAPK es un biomarcador fundamental en la oncogénesis de diferentes tumores, por ejemplo, en el carcinoma papilar de tiroides 3 integrantes de las vías MAPK, el receptor y las moléculas de transducción Ras y BRAF, se encuentran implicados en más del 70% de los casos, y se ha observado en algunos pacientes su asociación diferencial en el comportamiento clínico de la enfermedad. Para los pacientes con carcinoma papilar de tiroides en quienes el tratamiento convencional resulta inefectivo, la utilización de inhibidores de receptores tirosincinasas es una alternativa terapéutica31. La determinación de las alteraciones de los componentes de esta vía a través de identificar las mutaciones estructurales y los cambios en los patrones de expresión en este receptor o de RAS/BRAF en las células tumorales son blancos terapéuticos potenciales32,33.
ConclusionesLa identificación de las vías de señalización intracelulares que participan en la generación de los diferentes procesos básicos y especializados que suceden en las células, nos permite entender los mecanismos subyacentes moleculares en la fisiología celular. La dilucidación de las vías de señalización intracelulares, implica la realización de muchos años de trabajo experimental.
Para la generación de los distintos cambios fenotípicos celulares, la célula emplea frecuentemente varias o múltiples vías de señalización intracelular simultáneas o sucesivamente cercanas, como hemos visto en el proceso de la proliferación celular. La identificación de las principales moléculas reguladoras de las vías de señalización en los procesos celulares patológicos permite intentar su modificación y revertir o limitar las alteraciones en los procesos celulares básicos y especializados y con ello y con ello las alteraciones tisulares o de los órganos. Se ha iniciado una era más racional de terapéutica molecular.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.