En paralelo al proyecto de la secuenciación del genoma humano, se han desarrollado varias plataformas tecnológicas que están permitiendo ganar conocimiento sobre la estructura del genoma de las entidades humanas, así como evaluar su utilidad en el abordaje clínico del paciente. En la leucemia linfoblástica aguda (LLA), el cáncer infantil más común, las herramientas genómicas prometen ser útiles para detectar a los pacientes con alto riesgo de recaída, ya sea al diagnóstico o durante el tratamiento (enfermedad mínima residual), además de que permiten identificar los casos en riesgo de presentar reacciones adversas a los tratamientos antineoplásicos y ofrecer una medicina personalizada con esquemas terapéuticos diseñados a la medida del paciente. Un ejemplo claro de esto último es la identificación de polimorfismos de un solo nucleótido (SNPs) en el gen de la tiopurina metil transferasa (TPMT), donde la presencia de dos alelos nulos (homocigotos o heterocigotos compuestos) indica la necesidad de reducir la dosis de la mercaptopurina hasta en un 90% para evitar efectos tóxicos que pueden conducir a la muerte del paciente.
En esta revisión se proporciona una visión global de la genómica de la LLA, describiendo algunas estrategias que contribuyen a la identificación de biomarcadores con potencial utilidad en la práctica clínica.
In parallel to the human genome sequencing project, several technological platforms have been developed that let us gain insight into the genome structure of human entities, as well as evaluate their usefulness in the clinical approach of the patient. Thus, in acute lymphoblastic leukemia (ALL), the most common pediatric malignancy, genomic tools promise to be useful to detect patients at high risk of relapse, either at diagnosis or during treatment (minimal residual disease), and they also increase the possibility to identify cases at risk of adverse reactions to chemotherapy. Therefore, the physician could offer patient-tailored therapeutic schemes. A clear example of the useful genomic tools is the identification of single nucleotide polymorphisms (SNPs) in the thiopurine methyl transferase (TPMT) gene, where the presence of two null alleles (homozygous or compound heterozygous) indicates the need to reduce the dose of mercaptopurine by up to 90% to avoid toxic effects which could lead to the death of the patient.
In this review, we provide an overview of the genomic perspective of ALL, describing some strategies that contribute to the identification of biomarkers with potential clinical application.
En México, el cáncer es un problema prioritario de salud pública por su incidencia y alta tasa de mortalidad1,2. Las estimaciones recientes señalan que anualmente se diagnostican de 2600 a 3120 casos de cáncer en menores de 18 años, siendo el primer lugar como causa de mortalidad en niños de cinco a 14 años de edad. Se ha reportado que el 25% de estos casos de cáncer se deben a leucemia linfoblástica aguda (LLA)1–3. Últimamente se han logrado grandes avances en el tratamiento de la LLA, alcanzándose una tasa de curación de hasta el 90%, a diferencia del 10% que se lograba hace 50 años4–6. Por un lado, este éxito se debe al desarrollo científico y tecnológico que ha permitido el mejoramiento de los antibióticos y el establecimiento del diagnóstico oportuno, la detección temprana de clonas malignas para identificar a los pacientes con alto riesgo de recaída, el descubrimiento de agentes antileucémicos más eficientes y el diseño de esquemas terapéuticos más específicos y con reducidos efectos negativos colaterales. Por otro lado, el mayor conocimiento de la biología de la LLA constituye una base fundamental para que en la actualidad se alcancen altas tasas de remisión de los pacientes5–7. A la fecha, el tratamiento de la LLA se establece con base en el riesgo de recaída que tienen los pacientes, el cual se determina principalmente por la edad y el conteo sanguíneo leucocitario (CSL) al diagnóstico, infiltración a otros órganos, inmunofenotipo y la presencia de alteraciones citogenéticas y moleculares. Así, en comparación con los casos de bajo riesgo o estándar, los pacientes con alto riesgo de recaída son tratados con protocolos más agresivos o terapias innovadoras para alcanzar una mayor tasa de curación4–7.
En paralelo al proyecto de la secuenciación del genoma humano, cuyo primer borrador fue publicado en abril de 20018,9, se han desarrollado varias plataformas tecnológicas que están permitiendo ampliar el conocimiento que se tenía acerca de la estructura del genoma de la LLA, el transcriptoma de los genes codificantes y no codificantes (miRnoma) y del proteoma de un tejido en un tiempo y momento específico, así como sobre los genes que se relacionan con el metabolismo de los fármacos y la respuesta terapéutica (farmacogenómica). Las herramientas genómicas prometen ser útiles para identificar a los pacientes con alto riesgo de recaída al diagnóstico y durante el tratamiento (enfermedad mínima residual) y ofrecer una medicina con esquemas terapéuticos diseñados a la medida del paciente.
En esta revisión se ofrece un panorama global de la genómica de la LLA y se mencionan algunos biomarcadores con potencial aplicación en la práctica clínica.
2Diagnóstico clínico de la LLALa LLA representa un grupo de entidades onco-hematológicas de rápida evolución, clínica y biológicamente heterogéneas, caracterizadas por una proliferación descontrolada de glóbulos blancos inmaduros en la médula ósea (MO) y en la sangre e infiltración de estos a otros tejidos. Los síntomas más comunes incluyen fiebre (causada por la leucemia o por infecciones graves secundarias a la presencia de neutropenia), fatiga, anemia, hemorragias, dolor óseo o articular, petequias y esquimosis10,11. En cuadros clínicos más graves se observa disnea, hepatomegalia, esplenomegalia, linfadenopatías, infiltración al mediastino y al testículo7,10,11.
El diagnóstico diferencial de la LLA se establece con base en las propiedades de tinción citoquímica (negativas a mieloperoxidasas, Sudán negro B, alfa-naftil acetato esterasa) y al inmunofenotipo de las células leucémicas. Hasta hace algunos años, el Grupo Cooperativo Franco-Americano-Británico (FAB) clasificaba a la LLA con base en la morfología de las células leucémicas (subtipo L1, L2 y L3); sin embargo, la clasificación actual se basa en el inmunofenotipo11–13. La LLA pre-B se caracteriza principalmente por la expresión de inmunoglobulinas citoplasmáticas (cIg) y marcadores como CD79a, CD19, HLA-DR y CD10; la LLA cél-B por expresión de inmunoglobulinas de superficie (sIg) y cadenas pesadas μ; finalmente, la LLA cél-T se caracteriza por la expresión de CD3 citoplasmático, CD7, CD5 o CD2. Además, existe un subgrupo de LLA denominada pre-B transicional, que se caracteriza por la expresión citoplasmática de cadenas pesadas μ en las inmunoglobulinas y una débil expresión de estas cadenas en superficie, sin presencia de cadenas ligeras λ o κ 10,11. Un pequeño grupo (<5%) de casos de LLA son de linaje ambiguo, es decir, expresan marcadores linfoides y mieloides (bifenotípica) o presentan dos poblaciones celulares (bilineal)10,13,14.
2.1Citogenética y genética molecularEn cuanto a su genética, la LLA es una entidad compleja y heterogénea. Las aneuploidías (hiperdiploidía alta > 50 cromosomas e hipodiploidía < 44 cromosomas) y las translocaciones cromosómicas son las alteraciones genéticas más comúnmente encontradas en este padecimiento. En leucemias pediátricas de linaje de células B, las translocaciones t(12;21) (TEL-AML / ETV6-RUNX1), t(1;19) (E2A-PBX1 / TCF3-PBX1) y t(9;22) (BCR-ABL) y las fusiones que involucran al gen MLL (principalmente MLL-AF4) son las anomalías genéticas mejor caracterizadas y de mayor frecuencia. En promedio, ETV6-RUNX1 se ha reportado en el 25% (rango de 3-45%) de los casos, seguido por TCF3-PBX1 (13%), BCR-ABL (3-5%) y MLL-AF4 (6.0%)7,10,13–15. En pacientes mexicanos, las primeras tres alteraciones son las más comunes y constituyen alrededor del 17.7 al 28.8% de las anomalías genéticas16–18, aunque se han descrito frecuencias de BCR-ABL de hasta 19% en la población de Veracruz19. Mientras tanto, los rearreglos en el gen MLL se han reportado en el 1.4% de todos los casos y en el 23% de los pacientes menores de 26 meses18,20. Por otro lado, la LLA cél-T se caracteriza por la presencia de mutaciones en NOTCH1 (hasta en el 60% de los casos) y rearreglos en TLX1-HOX11 (5-10%), TLX3-HOX11L2 (20%) y otros (tabla 1)7,15.
Anormalidades genéticas más frecuentemente detectadas por citogenética y biología molecular en LLA
| Linaje | Anormalidades citogenéticas | Genes | Frecuencia % | Pronóstico | |
|---|---|---|---|---|---|
| Otras poblaciones (%) | Niños mexicanos (%) | ||||
| Células B | Hiperdiploidía | 20-307,10,13–15 | 22.916, 8+19 | Favorable | |
| Hipodiploidía | 5-67,10,13–15 | 0+19 | Desfavorable | ||
| t(12;21)(p13;q22) | TEL-AML1 | 257,10,13–15 | 7.4-1416–18 | Favorable | |
| t(1;19)(q23;p13) | E2A-PBX1 | 137,10,13–15 | 7.1-11.517,18 | Desfavorable* | |
| t(9;22)(q34;q11) | BCR-ABL1 | 3-57,10,13–15 | 2-517,18, 19+19 | Desfavorable | |
| t(4;11)(q21;q23) | MLL-AF4 | 67,10,13–15 | 1.418 11++19, 23**20 | Desfavorable | |
| Células T | 9q34 10q24 | NOTCH1 HOX11 | 607,15 207,15 | Se desconoce Se desconoce | Desfavorable |
El conocimiento de las alteraciones genéticas en la LLA ha mejorado favorablemente gracias al constante desarrollo de herramientas tecnológicas que permiten analizar cientos de genes o el genoma completo de cientos de individuos en muy poco tiempo. En esta carrera del conocimiento, las estrategias más útiles han sido los microarreglos de DNA y de expresión, secuenciación masiva (genoma, exoma, transcriptoma, entre otros) y la 5’ exonucleasa21–27.
3.1Secuenciación masiva o en paraleloLas técnicas de secuenciación masiva o de siguiente generación (NGS, del inglés Next Generation Sequencing) son capaces de secuenciar miles de nucleótidos simultáneamente en una sola reacción, cubriendo ya sea todo el genoma (WGS, del inglés Whole Genome Sequencing), solo el exoma, el trascriptoma (genes que se expresan) o un panel de genes seleccionados21. El WGS es costoso y puede ser ineficiente en las regiones ricas en GC (principalmente regiones promotoras), pero a diferencia de otros abordajes, ofrece la posibilidad de identificar alteraciones genéticas estructurales como grandes rearreglos y deleciones/inserciones. Por su parte, el abordaje de secuenciación del exoma es de menor costo que el WGS y es útil para encontrar mutaciones en regiones codificantes. Además, dado que con esta herramienta se puede obtener una amplia cobertura (200X en promedio por genoma haploide), es posible detectar mutaciones en clonas poco representadas, un aspecto de gran relevancia para detectar la enfermedad mínima residual e identificar a pacientes con alto riesgo de recaída21,22.
La secuenciación del transcriptoma involucra el análisis de todos los genes que se expresan (rearreglos génicos, nuevas isoformas), incluyendo los no codificantes como los pequeños (microRNAs) y largos RNAs (LncRNAs del inglés long no coding RNA), entre otros23. Para la detección de todas las formas de RNA, se expresen o no, la estrategia RNA-seq es la herramienta de NGS estándar más popular actualmente21,23.
3.2Microarreglos de DNA y de expresiónLas plataformas de microarreglos han permitido el análisis de alteraciones cromosómicas estructurales (citogenómica) que no podían detectarse con técnicas citogenéticas convencionales o de citogenética molecular, incluyendo translocaciones cromosómicas, inserciones/deleciones, duplicaciones, etcétera28. Además, han contribuido a la identificación de variantes genéticas que confieren riesgo a padecer leucemia mediante el análisis de miles de variantes de polimorfismos de un sólo nucleótido (SNP) y variantes en el número de copias de genes relevantes en la hematopoyesis normal y oncogénica22,28–30. Estos abordajes se plantean sin necesidad de una hipótesis, comparan casos con controles y generan un panorama global de la estructura genética de la LLA. Con los microarreglos de DNA, los cuales incluyen miles de alelos y comparan casos con controles, ha sido posible identificar nuevos loci asociados a la LLA; sin embargo, una gran limitante es que se requiere de un tamaño grande de muestra para detectar alelos de efecto pequeño, lo cual también resulta costoso.
El estudio de expresión de los genes codificantes y no codificantes mediante microarreglos sólo detecta genes representados con sondas en el arreglo; sin embargo, permite identificar nuevos genes involucrados en la etiopatogénesis de la LLA, inferir rutas de señalización y posibles interacciones entre los genes23. Desde el punto de vista clínico, este conocimiento ha permitido el descubrimiento de diversos subtipos moleculares de LLA que correlacionan fuertemente con el pronóstico de la enfermedad25,31–33.
3.35’ exonucleasa y abordaje de gen candidatoLa técnica de abordaje de gen candidato parte de la hipótesis de que ciertos polimorfismos en los genes que participan en los procesos patológicos que caracterizan a la leucemia (proliferación de blastos, linfopenia, leucopenia, plaquetopenia) pueden contribuir al riesgo de padecer la enfermedad, modificar el curso clínico de la misma o influir en la respuesta al tratamiento. Otros genes de interés incluyen a aquellos cuyas variaciones en su secuencia han dado evidencia de asociación con la leucemia en diversas poblaciones, o que se ha demostrado su participación en la oncogénesis hematológica en modelos animales. En el abordaje de gen candidato se compara la frecuencia con la que dichos polimorfismos se presentan en un grupo de individuos afectados con respecto a un grupo de sujetos sanos. Si estas diferencias son estadísticamente significativas, se sugiere que la variante analizada se asocia con la enfermedad en cuestión. Una importante limitante de este abordaje es que se requiere de un grupo grande de pacientes y controles pareados por origen étnico para evitar la obtención de resultados falsos positivos derivados de estratificación poblacional. Sin embargo, es una estrategia que permite identificar genes que tienen un efecto pequeño sobre la enfermedad, además de que es más económica en comparación con otros abordajes. La herramienta genómica más útil para la genotipificación es la técnica de la 5’ exonucleasa, la cual permite el análisis de un SNP en cientos de muestras en menos de 2 horas mediante sondas específicas marcadas con fluorocromos27,34.
4Genómica de la LLAAunque las translocaciones cromosómicas son los marcadores de la LLA, las herramientas genómicas han revelado que otras alteraciones altamente recurrentes están involucradas en su patogénesis, evolución, gravedad y respuesta al tratamiento. La naturaleza de las mutaciones incluye cambios de una sola base, deleciones/inserciones (indel), duplicaciones y variaciones en el número de copias (CNVs). Los genes más afectados son los que participan en la diferenciación de linfocitos (PAX5, IZKF1, EBF1 y LMO2), supresores de tumor y reguladores del ciclo celular (DKN2A/CDKN2B, PTEN y RB1), reguladores de la transcripción y coactivadores (TBL1XR1, ETV6 y ERG), entre otros. Asimismo, se ha reportado que existen diferencias en la frecuencia de estas alteraciones entre casos portadores de translocaciones específicas; por ejemplo, en la LLA BCR-ABL y TEL-AML1 positivas, hay más alteraciones adicionales que en las LLA con rearreglos en MLL. Mientras tanto, en la LLA hipodiploide frecuentemente se detectan mutaciones en IKAROs, IKZF2 y en genes involucrados de la ruta de señalización de RAS. El subtipo de LLA sin anomalías citogenéticas se caracteriza por mutaciones en los factores de transcripción ETS22,32,35–41.
4.1Genes asociadosEn la LLA, pocos casos muestran un patrón de herencia mendeliano, donde los genes TP53, PAX5 y GATA2 son los más reconocidos como responsables de una transmisión autosómica dominante42–45. Más recientemente y con la identificación de las mutaciones germinales p. L349P y N385fs en ETV6, también se observó que la LLA puede presentar un patrón de herencia recesivo. Cabe mencionar que portadores heterocigotos (dos alelos diferentes) para estas mutaciones desarrollaron entidades asociadas a la LLA, como la trombocitopenia y otros tumores sólidos46. Sin embargo, la LLA es principalmente de origen multifactorial, con un patrón de herencia complejo, donde polimorfismos de baja penetrancia en genes que participan en la reparación del DNA, en la respuesta a xenobióticos, en el sistema inmunitario y genes reguladores, predisponen al desarrollo de la enfermedad47,48. Un hallazgo interesante en estos estudios es la identificación de SNPs en el intrón 3 de ARID5B (del inglés AT Rich Interactive Domain 5B) como factores de riesgo para la LLA. De hecho, de las poblaciones analizadas (incluyendo a la mexicana), este es el gen más replicado31,48–55. ARID5B codifica un miembro de la familia de proteínas de unión al DNA con un dominio de interacción rico en AT. La proteína forma un complejo desmetilasa de histona H3K9Me2 con dos dedos de zinc de tipo PHD y está involucrada en la adipogénesis, desarrollo hepático y diferenciación de linfocitos B. Se ha reportado que los portadores homocigotos para alelos de riesgo no solo tienen mayor razón de momios (RM), sino que también responden mejor a tratamientos con metrotexate30,49–54.
Otros genes ampliamente estudiados son IKZF1, CEBPE, PIP4K2A, CDKN2A/CDKN2B, TP53 y GATA329,37,39,40,45,54–58. De este último, las variantes rs3824662 y rs3781093 confieren susceptibilidad a LLA en niños, adolescentes y jóvenes adultos, además de que se asocian con la edad al diagnóstico, la presencia de rearreglos en CRLF2, mutaciones en JAK y deleciones en IKZF1, genes que influencian directamente la trascripción de GATA329,31,38. Asimismo, se ha reportado que alelos de riesgo en IKZF y CEBPE distinguen a la LLA de cél-B de la LLA de cél-T41.
Otros estudios apuntan a que los genes que participan el metabolismo de los hidrocarburos o que tienen un papel potencial en la respuesta inmune innata y adaptativa del hospedero pueden tener relevancia en la etiología de la LLA47,59–63. En el primer grupo se encuentran los citocromos CYP1A1, CYP2D6, CYP2E1 y CYP3A5, así como XRCC1, GSTP1, GSTT1, CYP2E1, NAT2 y NQO1, los cuales también se han asociado con alto riesgo de recaídas tempranas, toxicidad a la quimioterapia, menor resistencia a agentes antineoplásicos y menor sobrevida61,63–65. Algunas asociaciones son dependientes del origen étnico, de interacciones gen-gen, de la edad y del tipo de leucemia. Por ejemplo, el alelo CYP1A1*2A confiere riesgo a LLA en niños caucásicos, y este aumenta si el individuo es homocigoto para CYP1A1*2A y el genotipo nulo GSTM163. En el segundo grupo están IL12A, HLA-DP, HLA-DOA, CD28, FCGR2, GATA3, STAT4 y STAT6, entre otros62. Chang y colaboradores62 observaron que el alelo G del rs583911 de IL12A confiere una RM de 1.52 para LLA en niños sin importar su origen étnico; sin embargo, el haplotipo CCGA-IL12 (rs6602398, rs942201, rs791587 y rs706778) incrementa el RM a 9.15 sólo en los niños de ascendencia hispana. Por su parte, el haplotipo GAC (rs17769459, rs4853546 y rs1031509) de STAT4 tiene un efecto diferencial de riesgo en niños hispanos y no hispanos (RM= 2.65 y 0.30, respectivamente).
5Origen étnico como factor de riesgoLa contribución diferencial de los genes y sus variantes en la etiología de la LLA entre los distintos grupos étnicos es un hallazgo común. Aunque la estratificación poblacional, tamaño de muestra, etc. son factores que podrían influir en este fenómeno, existen cada vez más evidencias de que el fondo genético del paciente, intrínseco a su origen étnico, constituye por sí mismo un factor de riesgo para la enfermedad. Por ejemplo, se ha documentado que las poblaciones nativo-americanas (50.6 casos por millón) tienen mayor prevalencia de LLA en comparación con otras poblaciones (hispana 40.9; caucásica 35.6 y afroamericana 14.8 por millón)66–71. Asimismo, la LLA de células T es hasta 1.7 veces mayor en población afroamericana que en el resto de las poblaciones; la fusión TCF3-PBX1 también está sobre-representada en esta población, mientras que el gen ETV6-RUNX1 es más común en caucásicos que en hispanos71. Utilizando marcadores genéticos de ancestría, se reportó que los niños nativo-americanos tienen un mayor riesgo de recaída independientemente de factores pronósticos como edad al diagnóstico, CSL, linaje y subtipo molecular de leucemia50.
Por otro lado, los genotipos de ARID5B asociados al riesgo de LLA son más frecuentes en poblaciones hispanas en comparación con otros grupos étnicos; adicionalmente, se han identificado variantes específicas para estas poblaciones y para la caucásica (tabla 2)29,31,39,40,45,50,54,55,72–76. El SNP (rs7088318) de PIP4K2A asociado al riesgo de LLA también tiene una distribución diferencial entre los diversos grupos étnicos31,58, por lo tanto, los genotipos de riesgo para LLA en ARID5B y PIP4K2A parecen ser importantes determinantes raciales en la susceptibilidad a la LLA.
Ejemplo de polimorfismos que confieren riesgo a padecer leucemia aguda linfoblástica
| Gen | Polimorfismo | Caucásico | Afroamericano | Asiático | Hispano | Referencias |
|---|---|---|---|---|---|---|
| ARID5B | rs7089424 | 1.7 [1.5-1.9], 0.0001 | – | NS | 2.12 [1.7-2.65], 2.04e-11 | 53 |
| rs7090445 | – | – | – | 2.08 [1.67-2.60], 6.24e-11 | 50,52 | |
| rs4506592 | – | 2.1 [1.3-3.3], 0.015 | 1.4 [1.2-1.7], 2.16e-6 | 0.48 [0.38-0.60], 8.24e-1 | 49 | |
| rs10821936 | 1.8 [1.6 – 2.0], 8.3e-20 | 1.52 [1.1-2.0] | 1.81 [1.4-2.3] | 1.8 [1.4 – 2.3], 1.0e-6 | 50,52,50 | |
| rs10821938 | 1.6 [1.4 – 1.8], 1.4e-14 | – | – | 1.8 [1.4 – 2.3], 2.9e-6 | 50 | |
| rs7087125 | 1.3 [1.7 – 1.5], 9.2e-6 | – | – | NS | 50 | |
| rs2393732 | NS | – | – | 1.4 [1.0-1.8], 0.12 | 50 | |
| IKZF1 | rs7780012 | – | – | – | 0.50 [0.35-0.71] 0.004 | 50 |
| rs11980379 | – | – | NS | 1.47 [1.7-1.85], 0.01 | 50,52,55 | |
| rs4132601 | 1.4 [1.1-1.7], <0.001 | – | – | 1.47 [1.7-1.85], 0.01 | ||
| CEBPE | rs4982731 | – | – | – | 1.69 [1.37-2.08], 1.15e-06 | 50 |
| rs10143875 | – | – | – | 1.67 [1.36-2.07], 1.68e-06 | 50,52 | |
| rs6572981 | – | – | NS | 0.53 [0.39-0.70], 1.6e-05 | 56 | |
| rs2239629 | 1.19 [1.11-1.28], 0.01 | – | – | 0.57 [0.40-0.81], 0.001 | ||
| PIP4K2A | rs7088318 | 1.28 [1.14-1.35], 0.001 | 1.48 [1.21-1.8], 0.001 | 1.24 [1.05-1.46], 0.001 | 1.29 [1.12-1.49], 0.001 | 58 |
| rs4748793 | 1.29 [1.24-1.55], 0.001 | NS | NS | 1.22 [1.06-1.43], 0.001 | ||
| GATA3 | rs3824662 | 1.7 [1.48-2.12], 2.8e-10 | – | – | – | 49,57 |
| rs3781093 | 1.7 [1.44-2.08], 3.2e-9 | – | – | – | ||
| CYP1A1 | *2A | NS | NS | – | 2.70 [1.27–5.74], 0.010 | 72 |
| *2B | NS | NS | – | 3.28 [1.40–7.69], 0.006 | 72 | |
| *2C# | NS | NS | – | 2.47 [1.13–5.38], 0.023 | 72 | |
| MLL | rs525549 | NS | – | – | 2.84 [1.37-8.18], 0.008a | 73 |
| rs6589664# | 0.40 [0.02-0.95], 0.04 | – | – | 2.32 [1.07-5.03], 0.034b | 73 | |
| EP300 | rs20551 | NS | – | – | 2.46[1.04-5.83], 0.040c | 73 |
| CDKN2B | rs662463 | – | 1.55 [1.16–2.06], 0.002 | – | NS | 73 |
| XRCC1 | rs Ars399Gln | NS | – | 1.72 [1.33, 2.23], 0.0001 | NS+ | 75,76 |
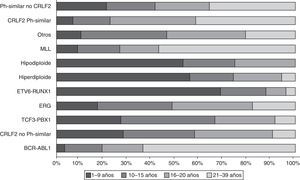
Los estudios de expresión génica de la LLA, además de los análisis de alteraciones en el número de copias en el DNA, han enfatizado la gran heterogeneidad de la enfermedad y han permitido establecer el diagnóstico diferencial e identificar nuevos subtipos de leucemia28,33,38,77,78. Existe un grupo de pacientes BCR-ABL negativos (15%) que tienen un perfil de expresión génica similar al que se observa en los portadores de este transcrito quimérico. Este subgrupo tiene un rango de alteraciones genéticas y estructurales de genes que activan el desarrollo linfoide, receptores de citocinas y rutas de señalización de cinasas. Más del 50% de los casos llevan rearreglos en CRLF1, de los cuales cerca de la mitad tienen mutaciones en JAK38. Datos recientes revelan la presencia de más de 10 subtipos de LLA pre-B clasificados como hiperdiploides, hipodiploides, TEL-AML1 positivos, TCF3-PBX1 positivos, BCR-ABL positivos, similar a BCR-ABL, rearreglos en MLL, con rearreglos en MYC, con y sin rearreglos en CRLF2, desregulación de ERG y con alteraciones en PAX5, mismos que difieren en su distribución entre grupos de edad (fig. 1)26,32. De la misma forma, en la LLA de cél-T se han identificado 9 subtipos moleculares de la enfermedad, incluyendo pre-T temprana (12%), con desregulación en TLX3 (20%), TAL1 (15-18%), LMO2 (10%), TLX1 (7%), positivos a la translocaciones t(10;11) (10%), MLL-ENL (2-3%), NUP214-ABL1 (6%) y t(7;9) (<1%)26,32,77. Cabe recalcar que en un análisis de perfiles de expresión en niños con recaída temprana y tardía, se reportó que la sobreexpresión de FOXM1, exonucleasa NEF-sp, BIRC5, NCAPH, GTSE1, CENPM, KIAA0101, C10orf56, BUB1B, UBE2V1, POLQ y TMEM97 son indicadores de recaída, mientras que PAICS, TYMS, IMPA2, CAD, ATIC y GART, correlacionan con recaída tardía26,78.
Los genes que definen los distintos perfiles de expresión en la LLA infantil son útiles en la clasificación de la enfermedad en adultos, particularmente para los casos positivos a las translocaciones t(1;19), t(12;21) y 11q2312,78.
7Epigenoma de la LLAEn la leucemogénesis, la desregulación epigenética tiene un papel fundamental. Se han descrito distintas señales epigenéticas para los diferentes subtipos de la LLA, donde es muy común encontrar mutaciones en genes que codifican modificadores del epigenoma, los cuales a su vez se asocian con falla terapéutica79–87. La hipometilación global e hipermetilación de islas ricas en CpG de las regiones promotoras de algunos genes son características de las células leucémicas. La hipometilación y la hipermetilación pueden influir en la expresión anormal de genes, promoviendo o regulando la proliferación celular, respectivamente86. Los genes comúnmente metilados en LLA incluyen a los que participan en rutas de señalización (TIE1, MOS, CAMLG y GPRC5C), regulación del ciclo celular y proliferación (MCTS1 y DGKG), factores y reguladores de la transcripción (PROP1, TAF3, H2AFY2, ELF5, ZBTB16, CNOT1 y TADA2A) y genes homeóticos (HOXA5 y HOXA6)82.
Las modificaciones epigenéticas también explican en cierto grado la resistencia al tratamiento antineoplásico69. De hecho, se ha sugerido que la resistencia reversible al tratamiento con inhibidores de NOTCH1 en la LLA de cél-T es un fenómeno mediado por mecanismos epigenéticos79. El uso de fármacos dirigidos a la reversión de patrones de metilación en genes específicos es una alternativa potencial de tratamiento anti-leucémico.
Otro mecanismo epigenético es la acción de los RNAs no codificantes, que juegan un papel fundamental en la biología del cáncer actuando como reguladores epigenéticos, potenciadores y reguladores de la interacción intra e intercromosómica. En diversos estudios se han detectado los miRNAs que frecuentemente se encuentran desregulados en su expresión: miR-99, -100, -125, -126, -128a/b, -146a, -155, -181a/c, -195, -198, -210, -221, -223, -425-5p, -663 y -708, entre otros81,84–87. Algunos de ellos se han reportado como indicadores de pronóstico y de respuesta al tratamiento79,80; otros se han asociado con el linaje de las células leucémicas, por ejemplo, miR-155, -128b y -210, los cuales permiten discriminar ente LLA y leucemia mieloide aguda (LMA)79,84,85. La sobreexpresión de miR-128b también correlaciona con la LLA MLL-AF4 positivo85. Los perfiles de expresión de los miRNAs permiten discriminar entre los linajes81 y pueden ser útiles para predecir la respuesta al tratamiento (miR-10a, -33, -27a, etc.) o monitorear la enfermedad después del mismo (tabla 3)80–87.
Ejemplos de miRNAs con relevancia clínica potencial y su correlación con el subtipo genómico de la LLA
| Biomarcador | Tipo de LLA | MicroRNAs | Subtipo molecular |
|---|---|---|---|
| Diagnóstico | Células B | miR-425-5p, miR-191 y miR-128 | E2A-PBX1 |
| miR-146b y miR-126 | BCR-ABL1 | ||
| miR-383, miR-125b, miR-99a y miR-100 | Hiperdiploidía y TEL-AML1 | ||
| miR-629 miR-196b, miR-17-92 | MLL-AF4 Rearreglos en MLL | ||
| Células T Linaje mezclado | miR-148a, miR-424 y miR-151* miR-196 | ||
| Remisión | miR-223 y miR27a | ||
| Recaída | miR-210, miR-708 | ||
| Mal pronóstico | miR-33, miR-215, miR-369-5p, miR-496, miR-518d y miR-599 | ||
| Buen pronóstico | miR-10a, miR-134, miR-214, miR-484, miR-572, miR-580, miR-624 y miR-627 |
Hasta el momento, existen pocas investigaciones cuyo objetivo sea conocer la interacción entre el genoma y el ambiente como una variable en el desarrollo de la LLA. Los factores ambientales más explorados son la exposición a rayos X, tabaco, pesticidas, insecticidas, pintura, agua clorada y consumo de alcohol47,59–61,88–91. En este aspecto, los genes mejor estudiados incluyen a los miembros de la familia de los citocromos, GSTM y MDR. Se ha reportado que los alelos *2A/*2B de CYP1A1 incrementan hasta 5 veces más el riesgo a LLA en niños expuestos a pesticidas durante el desarrollo intrauterino y la niñez. Mientras tanto, niños expuestos a altos niveles de trialometanos en el agua potable tienen hasta nueve veces más de riesgo a LLA si son portadores del alelo CYP2E1*5 o GSTT146. También se ha documentado que el genotipo nulo GSTM1 y el alelo CYP2E1*5 confieren alto riesgo a LLA en niños de madres que consumen alcohol durante el tercer trimestre del embarazo y durante la lactancia59. De la misma forma, la ausencia del haplotipo CGACC (-T1761C, -G9893A, Ex7+A131G, C1188T, C11599G) de CYP1A1 confiere riesgo para LLA en niños que tienen al menos un padre fumador88. Haplotipos en CYP2C8 y MDR1 (ABCB1) fueron identificados como factores de riesgo a LLA en niños hispanos de Estados Unidos expuestos a pintura e insecticidas; el haplotipo CGC (C1236T, G2677T/A, C3435T) de MDR1 se asoció a protección a LLA ante la exposición a insecticidas intradomiciliarios89,90. Recientemente, Lupo y colaboradores91 sugirieron que el rs1804742 de la metilen-tetrahidofolato reductasa podría conferir riesgo para tener hijos afectados con LLA en madres con bajo consumo de ácido fólico.
9La genómica en la clasificación y pronóstico de la LLADurante varios años, las características clínicas, citomorfológicas, inmunofenotípicas y moleculares de las células leucémicas han permitido establecer el diagnóstico de la LLA y han contribuido a al diseño de esquemas terapéuticos para mantener el control de la enfermedad. De acuerdo con estas características se valora el riesgo de recaída; los pacientes con bajo riesgo son tratados con terapias menos agresivas, mientras que los tratamientos más agresivos y tóxicos o terapias innovadoras están reservados para los pacientes con alto riesgo de recaída9,32,91,92. La masa inicial de células leucémicas, reflejada por el CSL, edad y presencia de enfermedad extra-medular son factores pronósticos que correlacionan con las probabilidades de sobrevida libre de enfermedad (SLE)92. Los niños < 1 año de edad, > 10 años de edad y los que tienen CSL > 50x109/l tienen peor pronóstico que los de menor edad (1-9.99 años) y que presentan CSL <50x109/l. Factores como infiltración al sistema nervioso, involucramiento testicular en varones, trisomía 21 u origen étnico, pueden influir en el pronóstico13,93. Con respecto al inmunofenotipo, la LLA pre-B se asocia con un pronóstico más favorable, mientras que la co-expresión de antígenos mieloides en blastos linfoides (My+ ALL) y el inmunofenotipo cél-T son factores asociados a una menor SLE10,39,41.
La hiperdiploidía, hipodiploidía, presencia de BCR-ABL, TEL-AML1 y alteraciones que involucran al gen MLL son las anomalías citogenéticas y moleculares de pronóstico mejor caracterizadas. Se ha observado in vitro que la hipodiploidía correlaciona con un SLE menor a 40%, mientras que la hiperdiploidía se asocia con mayor apoptosis y sensibilidad a una gran variedad de agentes quimioterapéuticos, y por ende mejor respuesta al tratamiento. La expresión de TEL-AML1 en algunas poblaciones es un marcador de buen pronóstico; los casos portadores tienen una SLE de más del 90% a 5 años con protocolos que contienen asparaginasa. Los transcritos quiméricos BCR-ABL y MLL-AF4 están asociados a un mal pronóstico; los pacientes portadores son candidatos para terapias más innovadoras, incluyendo trasplante de MO durante la primera remisión. De hecho, los pacientes portadores de BCR-ABL son los que presentan mayores dificultades en el tratamiento porque, a pesar de que un subgrupo de ellos puede ser tratado con un inhibidor de la cinasa (imatinib), el efecto es transitorio y el riesgo de recaída es muy elevado10,94–96.
Se ha realizado un gran esfuerzo para conocer el componente genético de la LLA e identificar todas las lesiones genéticas que contribuyen a la leucemogénesis y a la falla terapéutica. Los estudios de asociación y de secuenciación del genoma y transcriptoma han mostrado que pacientes con LLA pre-B, BCR-ABL1 positivos y deleciones en IKZF1, tienen una SLE más corta y alto riesgo a recaída95; los casos con deleciones en CDKN2A/B sin rearreglos cromosómicos corresponden a subgrupos de pacientes con alto riesgo de falla al tratamiento96. La pérdida del gen supresor de tumor CDKN2A/B ocurre en más del 40% de los casos de LLA pre-B; por otro lado, el SNP rs3824662 de GATA3ha sido asociado con respuesta temprana al tratamiento95. Otros genes asociados a recaída son VPREB1, EBF y ARID5B50,78. En este sentido, los SNPs rs7923074 (RM= 1.2), rs10821938 (RM= 1.2), rs4948488 (RM= 1.41), rs2893881 (RM= 1.45) y rs6479778 (RM= 1.48) en ARID5B se asocian con el riesgo de recaída en pacientes de origen hispano y caucásico, siendo la población hispana la que presenta una mayor frecuencia de estas variantes50. Por otro lado, se ha reportado que la presencia de deleciones y expresión reducida de MSH6 correlacionan con la resistencia al tratamiento con mercatopurina y prednisona e indirectamente con recaída97.
Dentro del grupo de la LLA cél-T considerado de mal pronóstico, los pacientes portadores de alteraciones que desregulan la expresión de TLX1 (7%) presentan bajo riesgo de recaída97.
Mei y colaboradores84 sugieren que los miRNAs también pueden ser útiles como biomarcadores de pronóstico y predictores de sensibilidad al tratamiento. Por ejemplo, la baja expresión de miR-210 al diagnóstico correlaciona con mayor tasa de recaída o falla a la inducción; cuando es sobreexpresado en las líneas celulares REH y RS4;11, estas son más sensibles al tratamiento con daunorubicina, L-asparaginasa y vincristina.
10Blancos terapéuticosLa identificación de anormalidades cromosómicas en las neoplasias hematológicas ha sido fundamental en el descubrimiento de blancos terapéuticos para el abordaje de la enfermedad. Uno de los primeros éxitos en el tratamiento de la leucemia fue el uso del ácido retinoico en pacientes con leucemia promielocítica aguda portadores de la t(15,17)(q22;q12) (PML-RARA)98. Posteriormente, la identificación de un inhibidor de tirocina cinasas (imatinib) permitió tratar de manera eficiente a los pacientes positivos a t(9; 22) (q34, q11) (BCR-ABL) con leucemia mieloide crónica y LLA99.
Algunos tratamientos se encuentran en fase clínica I, por ejemplo, los inhibidores de JAK (ruxolitinib) en pacientes con rearreglos en CRLF2 y mutaciones en JAK1 y JAK2.
La identificación de alteraciones de expresión de genes también ofrece oportunidades para el desarrollo de nuevos blancos terapéuticos. Por ejemplo, el subtipo de LLA cél-T, que tiene un perfil de expresión parecido al de timocitos normales en estadios tempranos de diferenciación, tiene un pronóstico muy pobre, por lo que se sugiere que estos casos sean tratados con altas dosis de dexametasona99. Recientemente se reportó que los genes TPMT (mercaptopurina, tioguanina, azatioprina), CYP2D6 (codeína, tramadol, oxicodeína, amitriptilina, ondansetrón, fluoxetina, paroxetina), CYP2C19 (clopidrogrel) y SLCO1B1 (simvastatina, potencialmente metotrexato) han sido incluidos en el registro médico electrónico para su implementación clínica, siendo el genotipo de TPMT el de mayor relevancia para el tratamiento de los niños con LLA100–104.
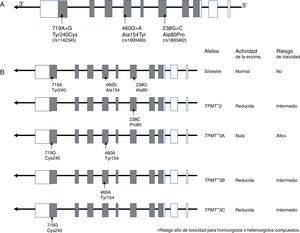
10.1Gen TPMTLa TPMT es una enzima de localización citoplasmática que participa en el metabolismo de los análogos de purinas utilizados en el tratamiento anti-leucémico e inmunosupresor, como la 6-mercaptopurina (6-MP), tioguanina (6-TG) y azatioprina. TPMT inactiva a las tiopurinas, por lo que en su ausencia se incrementan los metabolitos activos (nucleótidos de tioguaninas: TGNs) de estos fármacos anti-neoplásicos en las células hematopoyéticas, y con ello se presenta un alto riesgo de hematoxicidad que pone en peligro la vida del paciente. Actualmente se sabe que las variantes TPMT rs180462 (c.238G>A), rs1800460 (c.460G>A) y rs1142345 (c.719A>G) correlacionan con la actividad de la enzima y constituyen factores predictores de toxicidad y efectividad de tratamientos basados en tiopurinas, por lo que tienen un significado clínico importante en el abordaje de la LLA y de otras entidades autoinmunes. Los alelos TMPT*2, (c.238G>A, Arg80Pro) provocan una reducción de la actividad de la enzima; los pacientes que son heterocigotos para un alelo mutante tienen riesgo intermedio de toxicidad, mientras que los homocigotos y los heterocigotos compuestos muestran una actividad enzimática muy baja o indetectable, y el riesgo de un efecto tóxico es muy alto (fig. 2). Estos pacientes deben ser tratados con dosis más bajas de mercaptopurina para disminuir la toxicidad y no comprometer el tratamiento; se sugiere una reducción del 90% en la dosis en pacientes homocigotos y del 30 hasta el 70% en casos portadores de un solo alelo deficiente101–103.
11Tratamientos en fase preclínicaA pesar de los grandes avances en el tratamiento de la LLA, un porcentaje de pacientes aún muere como consecuencia de la enfermedad o de efectos secundarios asociados al tratamiento. La identificación de nuevos blancos terapéuticos permite el desarrollo de nuevas alternativas de tratamiento: inhibición/inducción de los genes alterados, inmunoterapia, etc105–108.
En pacientes con LLA que muestran un perfil de expresión parecido a BCR-ABL y que son portadores de las fusiones génicas EBF1-PDGFB o NUP214-ABL1, el tratamiento con imatinib, un inhibidor de ABL y de PDGFB, se encuentra en fase preclínica de evaluación105.
La LLA cél-T es una entidad de baja frecuencia y altamente agresiva; los portadores de mutaciones en el gen NOTCH1 no responden a los tratamientos convencionales y tienen una alta tasa de recaída y mortalidad. Para su tratamiento, se encuentran en desarrollo inhibidores de NOTCH1 (GSIs), los cuales no han sido llevados a la fase clínica porque existen evidencias experimentales de que las células leucémicas son refractarias al tratamiento con GSIs, un mecanismo probablemente mediado por fenómenos epigenéticos106.
Otros genes blanco en el tratamiento de la LLA cél-T son BDR4 y BCL2. BDR4 es una proteína que se encuentra sobre-expresada en esta neoplasia y se ha descrito como un potencial promotor de la expresión de MYC y la molécula antiapotótica BcL-2. De hecho, como alternativa del tratamiento en la LLA cél-T, se propone el uso de inhibidores de NOTCH1, BDR4 y BCL2 al mismo tiempo107.
La inmunoterapia como alternativa de tratamiento es de reciente interés; los blancos terapéuticos son los antígenos de superficie de blastos leucémicos. Para ello, se están desarrollando anticuerpos desnudos y no conjugados, inmunoconjugados e inmunotoxinas, receptores de antígenos quiméricos, anticuerpos de cadena única bi-específicos que se acoplan a linfocitos T, entre otros108.
12Genómica y LLA en MéxicoLa aplicación de las tecnologías genómicas en la investigación de la LLA infantil están sumando una plétora de datos que confirman su complejidad genómica. Además, contribuyen a la detección de biomarcadores que son potencialmente útiles para detectar a los pacientes en riesgo alto a recaída o toxicidad a tratamientos antes de que existan manifestaciones clínicas de la enfermedad. Actualmente, las plataformas de NGS permiten el estudio de grupos de genes que comúnmente están afectados en cáncer y que son portadores de mutaciones “accionables”, para las cuales existen tratamientos farmacológicos en fase preclínica (inhibidores de JAK en pacientes con rearreglos en CRLF2 y mutaciones en JAK1 y JAK2). Los genes afectados pueden ser utilizados como biomarcadores de la enfermedad mínima residual (EMR)106,107.
En México, las investigaciones genómicas en la LLA sugieren a FLT3, DEFA1 y hsa-miRNA-511 como potenciales marcadores de riesgo a recaída o de respuesta a tratamiento33; no obstante, son datos que deben ser replicados en poblaciones independientes antes de su implementación en el área clínica. Lo que está ampliamente demostrado es que la detección de BCR-ABL y la genotipificación de TPMT al diagnóstico deben realizarse en todos los niños con LLA, y que la primera opción de tratamiento para portadores de BCR-ABL es el mesilato de imatinib en lugar de trasplante de médula ósea4,6,102. En el caso de portadores de uno (heterocigotos) o dos (homocigotos o heterocigotos compuestos) alelos nulos de TPMT, que en conjunto son alrededor del 8% de los niños mexicanos con LLA109,110, se debe reducir la dosis de tiopurinas del 30 al 90% para disminuir el riesgo de mielosupresión y potencial muerte de los pacientes99–104. Sin embargo, aunque se están haciendo grandes esfuerzos, el uso de ambos biomarcadores en el área clínica es todavía limitado en nuestro país. No estamos lejos de constituirlas como herramientas diagnósticas en el abordaje clínico de los pacientes atendidos en instituciones hospitalarias públicas, pero por el momento, el estudio molecular, en particular la detección de BCR-ABL al diagnóstico, está sólo disponible en algunos centros hospitalarios de tercer nivel16–18. El reto es que médicos e investigadores sumemos esfuerzos para encontrar rutas que faciliten la optimización de los recursos técnicos y de infraestructura para potenciar la traslación de la investigación genómica a una medicina personalizada en los niños mexicanos con LLA.
Las herramientas ómicas han revelado que, además de las alteraciones somáticas adquiridas reconocidas como marcadores de la LLA (aneuploidias, translocaciones como BCR-ABL, TEL-AML1), las variaciones genéticas germinales o heredadas tienen importantes implicaciones en el riesgo a LLA, en la respuesta a medicamentos o en la toxicidad a la terapia antineoplásica. Existen ejemplos claros como el uso del genotipo TPMT en el tratamiento de la LLA infantil, pero se requieren más estudios que integren la información genética somática y germinal para predecir el riesgo de recaída de los pacientes y detectar la EMR, con el objetivo final de aplicar este conocimiento y ofrecer un tratamiento oportuno y una medicina personalizada. Así, los estudios genómicos subsecuentes deberán incorporar información clínica, patológica y molecular para identificar los alelos que contribuyen a la malignidad de las entidades humanas.
FinanciamientoNinguno.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.