Un microarray es básicamente un soporte sólido de pequeño tamaño (micro) que contiene cientos de miles de pequeños depósitos (spots) de sondas (moléculas de ácido desoxirribonucleico [ADN]) ordenadas en filas y columnas formando una matriz (array). Cada una de las sondas utilizadas se deposita en un único spot y es capaz de identificar, de manera específica, un determinado ácido ribonucleico (ARN) mensajero, una determinada región de ADN o una variación genética, dependiendo del tipo de microarray (tabla 1).
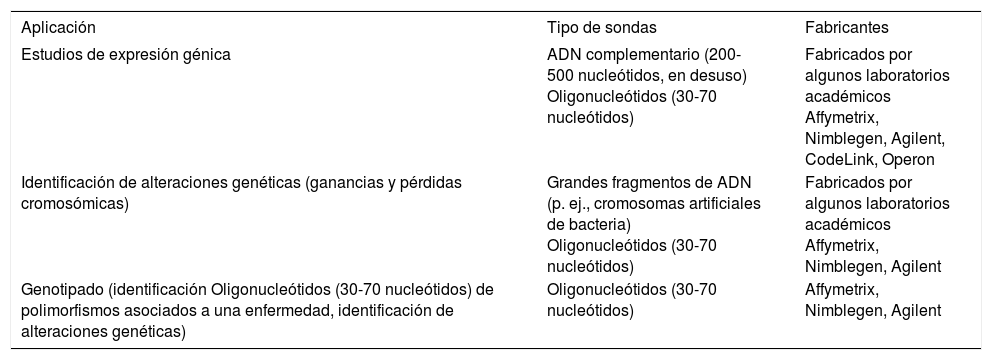
Características y aplicaciones de diferentes tipos de microarrays
| Aplicación | Tipo de sondas | Fabricantes |
|---|---|---|
| Estudios de expresión génica | ADN complementario (200-500 nucleótidos, en desuso) Oligonucleótidos (30-70 nucleótidos) | Fabricados por algunos laboratorios académicos Affymetrix, Nimblegen, Agilent, CodeLink, Operon |
| Identificación de alteraciones genéticas (ganancias y pérdidas cromosómicas) | Grandes fragmentos de ADN (p. ej., cromosomas artificiales de bacteria) Oligonucleótidos (30-70 nucleótidos) | Fabricados por algunos laboratorios académicos Affymetrix, Nimblegen, Agilent |
| Genotipado (identificación Oligonucleótidos (30-70 nucleótidos) de polimorfismos asociados a una enfermedad, identificación de alteraciones genéticas) | Oligonucleótidos (30-70 nucleótidos) | Affymetrix, Nimblegen, Agilent |
ADN: ácido desoxirribonucleico.
Un experimento de microarrays comprende básicamente 3 pasos: a) diseño y fabricación del microarray; b) preparación de la muestra, marcaje e hibridación, y c) escaneado y análisis de los resultados.
Puntos clave
Los primeros microarrays se desarrollaron a mediados de la década de los años noventa para estudiar la expresión de miles de genes de forma simultánea. Estos primeros microarrays constaban de un cristal similar a los utilizados en los estudios histológicos sobre el que se habían depositado, con la ayuda de un robot, pequeñas gotitas que contenían sondas de ADN complementario de varios centenares de nucleótidos, específicas para cada uno de los genes cuyos valores de expresión se pretendía estudiar1–3. De estos primeros arrays fabricados en entornos académicos, se pasó rápidamente a los microarrays comerciales que se utilizan en la actualidad (tabla 1). Desde un punto de vista tecnológico, hay básicamente 2 formatos disponibles comercialmente: a) microarrays en los que la sonda de ADN se deposita mediante un robot en el soporte sólido (en la actualidad se utilizan sondas de 30-70 nucleótidos denominadas oligonucleótidos que presentan una especificidad mayor y un coste menor que las sondas de ADN complementario utilizadas inicialmente), y b) microarrays en los que los oligonucleótidos se sintetizan directamente en el soporte sólido.
El sistema comercial más utilizado es el GeneChip de la compañía Affymetrix (www.affymetrix.com). Este sistema usa una técnica de síntesis de oligonucleótidos in situ, similar a la utilizada en la fabricación de los microchips de los ordenadores. El sistema se completa con el equipamiento necesario para los pasos de hibridación, lavado y escaneado del microarray, específicamente diseñado para su uso con esta plataforma. La compañía Nimblegen (www.nimblegen.com) también comercializa microarrays de oligonucleótidos sintetizados in situ mediante un sistema diferente al utilizado por la compañía Affymetrix. En cuanto a los microarrays fabricados mediante la deposición e inmovilización de oligonucleótidos presintetizados, la compañía Agilent (www.chem.agilemt.com) es la que ofrece una variedad mayor de microarrays. otras compañías que utilizan este sistema de fabricación son CodeLink (www.appliedmicroarrays.com) y Operon (www.operon.com). Hoy día, la mayoría de los fabricantes de microarrays ofrecen un catálogo extenso de productos para diferentes aplicaciones, entre las que se encuentran los estudios de expresión génica, la determinación de alteraciones genéticas (también conocidos como CGH-arrays) y los estudios de variabilidad genética (tabla 1).
Preparación de la muestra, marcaje e hibridaciónEl siguiente paso comprende la preparación del ARN o ADN de la muestra problema para su hibridación al microarray. Una vez aislado el ARN (estudios de expresión) o ADN (determinación de alteraciones genéticas o estudios de variabilidad genética) con técnicas estándares, se marca con una molécula fluorescente mediante una serie de reacciones enzimáticas. A continuación, las moléculas de ARN o ADN marcadas con fluorescencia se incuban con el microarray durante un tiempo prolongado para permitir la hibridación de la muestra con las sondas correspondientes. Tras la hibridación, el microarray se lava con determinadas soluciones que eliminan el exceso de ARN o ADN marcado no unido a las sondas.
Escaneado y análisis de resultadosPor último, los microarrays se digitalizan con un escáner láser, y se obtiene una imagen que refleja con exactitud la disposición espacial de las diferentes sondas del microarray y en las que la intensidad de cada spot es proporcional al número de moléculas de ARN o ADN marcado que han hibridado con la sonda correspondiente (fig. 1). La cantidad de información que se genera con este tipo de experimentos es enorme: por ejemplo, un experimento en el que se ha analizado el perfil de expresión en 20 muestras diferentes mediante un microarray capaz de detectar la expresión de 30.000 genes de forma simultánea, generará una tabla de 600.000 valores de fluorescencia (fig. 2). Para manejar esta cantidad enorme de información, se han desarrollado múltiples aplicaciones informáticas que utilizan diferentes aproximaciones estadísticas y que permiten identificar, por ejemplo, qué perfiles de expresión se asocian con un determinado estadio patológico o qué funciones o rutas biológicas están implicadas en una enfermedad determinada.
En la figura se muestra el digitalizado de un microarray de la marca CodeLink después de la hibridación con una muestra de ácido ribonucleico (ARN) marcada con un compuesto fluorescente. Este microarray contiene más de 20.000 sondas específicas para otros tantos genes, dispuestas de manera ordenada en una superficie de tan sólo × 76mm. Cada punto contiene una única sonda y, por tanto, reconoce a un único gen. Las diferencias en intensidad que se observan entre los puntos son proporcionales a la cantidad de ARN marcado hibridado a cada sonda y, por tanto, reflejan la concentración del ARN correspondiente en la muestra de partida.
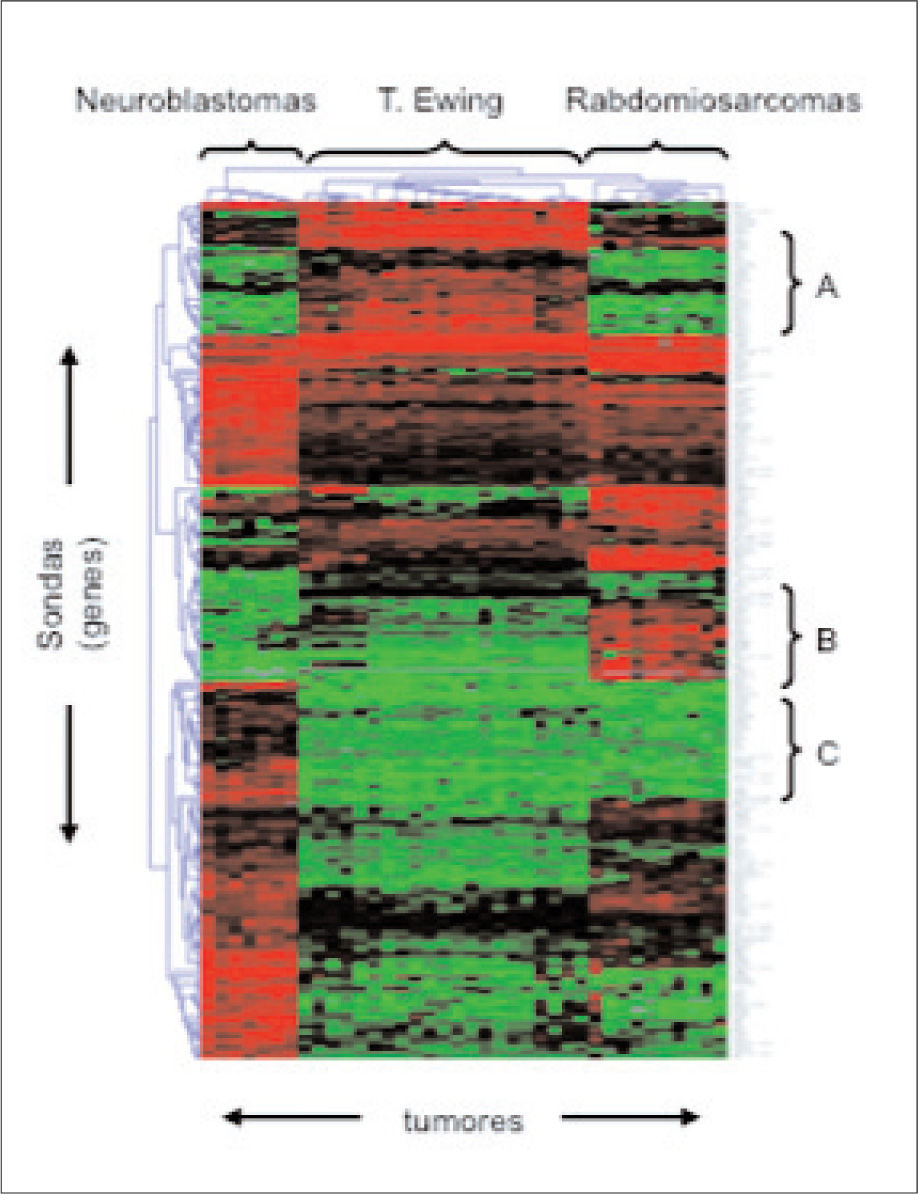
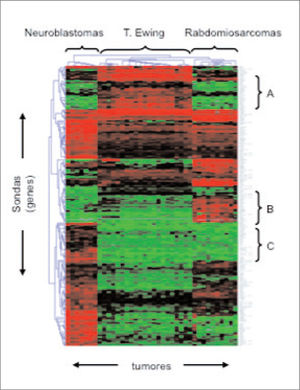
En la figura se muestra un experimento de microarrays realizado con una serie de neuroblastomas, tumores de la familia Ewing y rabdomiosarcomas. Los datos de fluorescencia de cada microarray se procesaron mediante una aplicación informática, lo que da como resultado la imagen que se muestra en la figura. Cada línea vertical corresponde a un tumor y cada línea horizontal, a un gen. Los genes que se expresan a valores elevados en un determinado tumor se muestran en rojo, mientras que los genes que se expresan a valores bajos se muestran en verde. Un análisis estadístico de correlación segrega cada tipo tumoral por separado e identifica grupos de genes que sólo se expresan a valores elevados en tumores de Ewing (grupo A), rabdomiosarcomas (grupo B) o neuroblastomas (grupo C), y que pueden, por tanto, servir para definir mejor a estas entidades desde un punto de vista molecular.
A continuación, se describirán algunos ejemplos que servirán para ilustrar el impacto que la tecnología de los micro-arrays aplicada al análisis de la expresión génica está teniendo en nuestro conocimiento de la enfermedad pediátrica.
Utilidad de los microarrays en oncología pediátricaLa adecuada clasificación de un determinado tipo tumoral es un aspecto crítico, tanto para el diagnóstico, como para la asignación del protocolo terapéutico más adecuado. La posibilidad de analizar la expresión de miles de genes de forma simultánea mediante la técnica de microarrays permite, al menos en teoría, identificar una huella molecular propia y exclusiva de cada tipo tumoral, y esta característica puede, por tanto, utilizarse a la hora de clasificar el tumor. Uno de los primeros trabajos que demostró la aplicabilidad de la técnica de microarrays para la clasificación de tumores se llevó a cabo precisamente en el campo de la oncología pediátrica. Golub et al4 demostraron que el perfil de expresión de un grupo de 50 genes era suficiente para diferenciar la leucemia linfoblástica aguda de la leucemia mieloide aguda, y que esta huella molecular podía utilizarse para clasificar adecuadamente estos 2 tipos de cáncer. Este estudio pionero representó la prueba de que los microarrays pueden utilizarse como herramienta diagnóstica a la hora de clasificar diferentes tipos tumorales. Estudios posteriores han identificado, por ejemplo, huellas moleculares que definen diferentes tipos de leucemia linfoblástica aguda5–8 y subtipos que responden diferencialmente a diferentes tratamientos9–11.
El grupo de tumores denominados de células redondas, pequeñas y azules, está formado por entidades tan diversas como neuroblastoma, rabdomiosarcoma, linfoma no hodgkiniano y tumores de la familia Ewing. Debido a su apariencia morfológica similar, el diagnóstico diferencial de estos tumores presenta con bastante frecuencia cierta dificultad en la práctica clínica. Con el objeto de identificar una huella molecular que pudiera definir a cada uno de estos tumores, Khan et al12 analizaron mediante microarrays una serie de tumores y líneas celulares de cada tipo tumoral. De esta manera, identificaron, utilizando redes neuronales artificiales, una huella molecular capaz de clasificar con total precisión cada uno de los diferentes tipos tumorales.
El uso de los microarrays también ha resultado ser de extraordinaria utilidad a la hora de identificar nuevos marcadores de pronóstico. Por ejemplo, el análisis del perfil de expresión génica de una serie de meduloblastomas mediante micro-arrays permitió la identificación de STK15, una cinasa de serina/treonina, como un marcador independiente de pronóstico13, y en un estudio realizado en 136 neuroblastomas se pudo identificar un grupo de 70 genes capaz de definir el pronóstico en tumores de riesgo intermedio14.
Los estudios de microarrays también son extraordinariamente útiles para identificar nuevas dianas terapéuticas en tumores pediátricos. Los tumores de la familia Ewing se caracterizan por la presencia de translocaciones cromosómicas que dan lugar a proteínas quiméricas que fusionan los genes EWS y FLI1. Esta proteína quimérica actúa como un factor de transcripción aberrante, por lo que se cree que los genes que se alteran por EWS/FLI1 en células de tumores de Ewing desempeñan un papel importante en el desarrollo del tumor. Recientemente, después de utilizar una serie de sistemas celulares modelo y la técnica de microarrays, hemos demostrado que el neuropéptido colecistocinina es uno de los genes inducidos por la oncoproteína EWS/FLI1 en células de tumor de Ewing15. La colecistocinina actúa además como un factor de crecimiento autocrino en estos tumores, por lo que estrategias terapéuticas encaminadas a bloquear la unión de la colecistocinina a sus receptores pudieran representar un nuevo tratamiento terapéutico para esta familia de tumores tan agresivos.
Utilidad de los microarrays en enfermedades metabólicas y autoinmunitarias de la infanciaLas enfermedades que afectan a la generación de energía por la mitocondria son genéticamente heterogéneas, y afectan a varios genes mitocondriales y nucleares, que codifican para las diferentes subunidades del complejo I multiproteico de la cadena respiratoria. Kirby et al16 emplearon la técnica de microarrays para estudiar el perfil de expresión génica en 2 pacientes con una deficiencia letal en el complejo I mitocondrial. Estos investigadores encontraron que la expresión del gen NDUFS6, que codifica para una subunidad del mencionado complejo, se encontraba reducida en las líneas celulares derivadas de estos pacientes. Análisis posteriores demostraron que este gen se encontraba mutado en estos pacientes. Este estudio demuestra que los análisis de expresión génica realizados con la técnica de microarrays pueden ser útiles a la hora de identificar nuevos genes relacionados con enfermedades complejas.
La artritis juvenil idiopática de aparición sistémica representa aproximadamente el 10% de los casos de artritis que se inician en la infancia. El tratamiento de estos pacientes es todo un reto para los reumatólogos pediatras, tanto por su cronicidad, como por las consecuencias que esta enfermedad tiene para la actividad diaria normal del niño. Los mecanismos patogénicos de esta enfermedad no se conocen bien, aunque se han correlacionado valores elevados de interleucina (iL) 6 con la actividad sistémica de la enfermedad y con el desarrollo de la artritis17,18. Recientemente, Pascual et al19, 20 utilizaron la técnica de microarrays para profundizar en los mecanismos moleculares de esta enfermedad. Estos autores incubaron células mononucleares de sangre periférica de donantes sanos con suero autólogo o suero proveniente de pacientes con artritis, y analizaron el perfil de expresión génica inducido en las células mononucleares tras la exposición al suero. observaron que el suero proveniente de pacientes con artritis indujo la transcripción de numerosos genes relacionados con la inmunidad innata, incluidos varios miembros de la familia de la IL-1 y varias quimiocinas implicadas en la quimiotaxis de neutrófilos. Estos genes también se encontraron sobreexpresados en las células mononucleares de pacientes con artritis en comparación a las de individuos sanos. Los autores concluyeron que los pacientes con artritis juvenil sistémica tienen alterada la producción de IL-1. Asimismo observaron que el tratamiento con antagonistas de la IL-1 resultó beneficioso para pacientes que fueron resistentes a los tratamientos agresivos convencionales. En este caso, el empleo de microarrays permitió identificar la IL-1 como uno de los principales mediadores de la cascada inflamatoria en esta enfermedad, proporcionando además una base racional para el uso de antagonistas de la IL-1 en su tratamiento.
Otra enfermedad autoinmunitaria grave que afecta a múltiples órganos, como la piel, los riñones y el sistema nervioso central, es el lupus eritematoso sistémico (LES). Aproximadamente, el 20% de todos los pacientes que presentan LES son niños y, como con tantas otras enfermedades autoinmunitarias, los mecanismos patogénicos son en gran parte desconocidos. Bennett et al21 utilizaron la técnica de los microarrays para analizar el perfil de expresión génica de células mononucleares de sangre periférica aisladas de niños con LES en fase activa. Cuando compararon a pacientes con LES frente a controles sanos, encontraron que un elevado número de los genes diferencialmente expresados correspondían a genes inducidos en respuesta con el interferón. Lo curioso es que esta huella molecular no se observó en pacientes con artritis juvenil crónica, lo cual indica que el patrón de expresión génica distingue el LES de otros desórdenes autoinmunitarios.
Conclusiones y perspectivas futurasLa técnica de los microarrays ha cambiado la forma de estudiar y abordar los problemas biológicos. Hemos pasado de abordajes en los que se analizaba la posible función de un único gen en una determinada enfermedad, partiendo de una hipótesis preconcebida, a abordajes en los que se estudia el sistema biológico en su conjunto y que permiten identificar genes y alteraciones que no hubieran sido posibles identificar utilizando los abordajes convencionales. Todo ello está, por tanto, generando una gran cantidad de nuevos conocimientos sobre las bases moleculares de la enfermedad. El próximo paso será trasladar estos resultados a la práctica clínica mediante el desarrollo de metodologías que se puedan implementar de forma sencilla y económica, y de esta manera contribuir a un mejor diagnóstico, pronóstico y terapéutica de las enfermedades pediátricas.