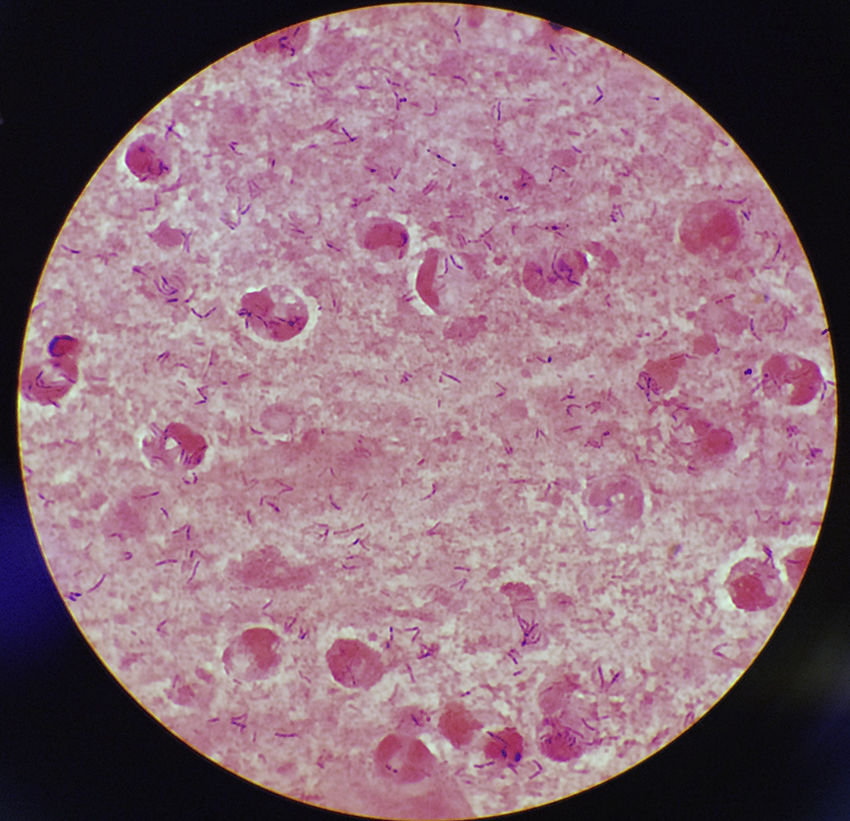
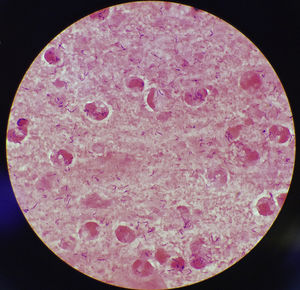
Se trata de una mujer de 77 años que acudió a su médico de atención primaria por dolor de garganta, tos, somnolencia, expectoración verdosa y afebril (36,7°C). Presentaba como antecedentes de interés diabetes mellitus secundaria a tratamiento esteroideo y bronquiectasias. Entre su historial de vacunación reciente, se hallaban la vacuna de la gripe y la vacuna del tétanos-difteria, esta última administrada en 2003. No tenía antecedentes de contacto con animales o viajes recientes. Se recogió una muestra de esputo para cultivo y en la tinción de Gram se observaron abundantes leucocitos y bacilos grampositivos (fig. 1). Después de 24 h de incubación, en agar Columbia CNA con 5% sangre de carnero y agar chocolate se aislaron en cultivo puro colonias muy pequeñas (∼1mm diámetro), catalasa positiva, grises y brillantes con una pequeña zona de beta-hemólisis. El microorganismo se identificó como Corynebacterium diphtheriae (C. diphtheriae) biotipo mitis/belfanti mediante API Coryne (bioMérieux; Bionumber 0000324; 90.2%) y MALDI-TOF (MALDI Biotyper® Microflex LT, Bruker Daltonik GmbH; puntuación: 1.786). Se secuenció el gen codificante de la subunidad ß de la ARN polimerasa (rpoB) confirmando dicha identificación1. No se detectó mediante PCR2 la presencia del gen de la toxina diftérica (tox) y tampoco su expresión mediante la prueba de Elek3. La sensibilidad antimicrobiana fue determinada mediante el método ɛ-test (bioMérieux) e interpretada según las recomendaciones establecidas por CLSI (www.clsi.org). Las concentraciones mínimas inhibitorias (μg/ml) fueron: penicilina (0,25, sensible [S]), cefotaxima (1,5, intermedio [I]), cefepime (1,5, I), imipenem (0,125, S), meropenem (0,064, S), eritromicina (0,016, S), ciprofloxacino (>32, resistente), tetraciclina (0,75, S), clindamicina (0,25, S), cotrimoxazol (0,064, S), rifampicina (0,002, S), y linezolid (0,25, S). Se instauró tratamiento antibiótico con eritromicina 500mg/día durante 14 días con evolución favorable.
Desde 1986 se han publicado 3 artículos en España de difteria respiratoria y cutánea por cepas toxigénicas y/o no toxigénicas (NT)4–6. Este es el primer caso documentado de difteria respiratoria por biotipo belfanti NT en nuestro país. El principal reservorio de C. diphtheriae es el hombre, sin embargo, se han aislado ambos tipos de cepas en animales domésticos e incluso salvajes sin evidencia de transmisión zoonótica7.
C. diphtheriae está compuesto de 4 biotipos: gravis, mitis, intermedius, y belfanti. El biotipo belfanti presenta tropismo respiratorio teniendo ventaja a la hora de colonizar o infectar el tracto respiratorio superior frente a otros biotipos, describiéndose fundamentalmente en casos de rinitis atrófica crónica primaria (ozena)8. Se ha aislado más frecuentemente en áreas geográficas con alta cobertura vacunal como Norteamérica y Europa (36-100%). Durante la época postepidémica ha ido aumentando su presencia en Europa reemplazando a las cepas toxigénicas, que en su mayoría pertenecen a los biotipos gravis y mitis9,10. Las cepas NT pueden causar infecciones graves como miocarditis, endocarditis, bacteriemia, artritis séptica, osteomielitis, neuritis y epiglotitis8–10. Su mecanismo de patogenicidad aún no es comprendido y se conoce poco sobre los genes responsables de la colonización, invasión y supervivencia en el hospedador, así como de otros factores de virulencia fuera de la producción de la toxina.
MALDI-TOF ha sido descrito como una herramienta útil, coste-efectiva y rápida para la identificación de este microorganismo junto con otras especies toxigénicas como Corynebacterium ulcerans y Corynebacterium pseudotuberculosis, aunque no es capaz de diferenciar las cepas toxigénicas de las NT11. Además, la secuenciación del gen rpoB ha mostrado mejor poder discriminatorio (91%) que la secuenciación del ARNr16S (81%)12. Estos métodos deben de ir siempre acompañados de la detección y expresión del gen tox, siendo la ausencia de toxina diftérica algo habitual en biotipo belfanti, el cual ha mostrado gran diversidad clonal con respecto al resto de biotipos9,10.
A lo largo de estos años, se ha observado un aumento de la resistencia antimicrobiana en cepas NT. C. diphtheriae biotipo belfanti suele presentar mayor sensibilidad que el resto de biotipos. Sin embargo, se ha descrito sensibilidad reducida a ciprofloxacino, cefotaxima y cefepime, no considerándose buenas opciones de tratamiento. La eritromicina o penicilina son el tratamiento de elección, pudiendo ser ambas no efectivas en algunos casos por la descripción de cepas resistentes8–10.
Este caso muestra la necesidad de monitorizar la expansión de las cepas de C. diphtheriae que circulan en España, no solo aquellas cepas que son toxigénicas si no también las NT que deben considerarse patógenos emergentes.
Los autores quieren agradecer a Silvia Herrera León del Centro Nacional de Microbiología de Majadahonda (ISCIII) llevar a cabo la caracterización del aislamiento.