El cáncer de pulmón es la principal causa de muerte por cáncer en nuestro país. El cáncer de pulmón de células no pequeñas (CPCNP) representa el paradigma de la medicina personalizada. El objetivo principal de este trabajo es estudiar la frecuencia en nuestro medio de las variantes clínicamente significativas más frecuentemente descritas en CPCNP.
Material y métodosSe estudia la expresión inmunohistoquímica de TTF1, p40 y PD-L1 y la frecuencia de variantes genéticas mediante secuenciación masiva (NGS) con un panel de 52 genes, en 174 muestras incluidas en parafina de CPNCP en 169 pacientes (111 hombres y 52 mujeres) de la provincia de Cádiz.
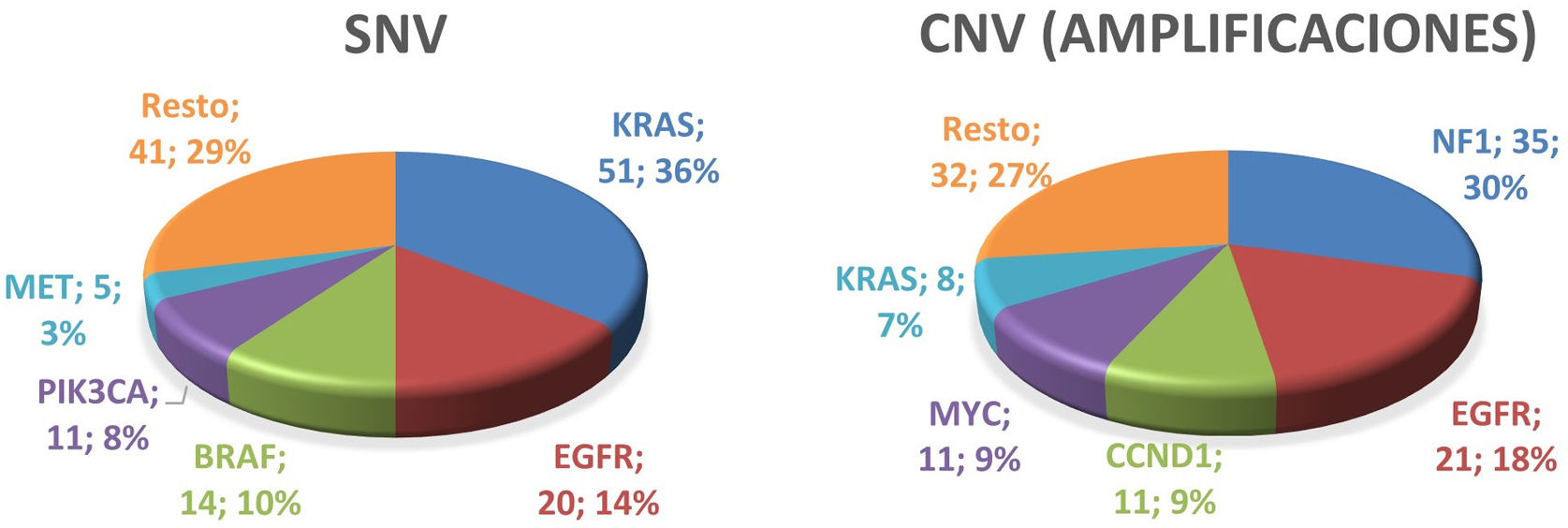
ResultadosLa expresión inmunohistoquímica de TTF1, p40 y PD-L1 fue positiva en el 87%, el 0% y el 46% de los adenocarcinomas y en el 0%, el 100% y el 41% de los carcinomas escamosos. En NGS, las variantes de un solo nucleótido (SNV) más frecuentes fueron KRAS (36%), EGFR (14%), BRAF (10%), PIK3CA (8%) y MET (3%). Las variantes en el número de copias (CNV) más frecuentes fueron las amplificaciones en NF1 (30%), EGFR (18%), CCND1 (9%), MYC (9%) y KRAS (7%). En mujeres, las SNV en EGFR fueron más frecuentes que en hombres (p<0,0001). El adenocarcinoma es el tipo histológico más frecuente con SNV en KRAS (p=0,007361) o en EGFR (p<0,0001). En 16 pacientes (9,47%) se detectaron fusiones génicas, 9 casos en el gen MET.
ConclusionesDetectamos nuevas asociaciones entre expresión inmunohistoquímica y algunas variantes génicas, que podrían tener impacto en el tratamiento de pacientes de CPNCP.
Lung cancer is the leading cause of cancer death in our country. Non-small cell lung cancer (NSCLC) represents the paradigm of personalized medicine. The main objective of this study is analysing the distribution of the most frequently described clinically significant variants in NSCLC, in our environment.
Material and methodsWe studied the immunohistochemical expression of TTF1, p40 and PD-L1 and the genetic variants frequency using Next-Generation Sequencing (NGS) with a panel of 52 genes, in 174 NSCLC paraffin-embedded samples in 169 patients (111 men and 52 women) from the province of Cádiz.
ResultsThe immunohistochemical expression of TTF1, p40 and PD-L1 was positive in 87%, 0% and 46% in adenocarcinoma, and 0%, 100% and 41% in squamous cell carcinoma. In NGS, the most common single nucleotide variants (SNVs) were KRAS (36%), EGFR (14%), BRAF (10%), PIK3CA (8%), and MET (3%). The most frequent copy number variants (CNVs) were amplifications in NF1 (30%), EGFR (18%), CCND1 (9%), MYC (9%) and KRAS (7%). In women, SNV in EGFR are more frequent than in men (P<.0001). Adenocarcinoma is the most frequent histological type with SNV in KRAS (P=.007361) or in EGFR (P<.0001). Gene fusions were detected in 16 patients (9.47%), in 9 cases in the MET gene.
ConclusionsWe detected associations, not described so far, between immunohistochemical expression and specific gene variants, which could have an impact on the treatment of NSCLC patients.
El cáncer de pulmón es la principal causa de muerte por cáncer, con una estimación de 1,79 millones de muertes por año en todo el mundo1. La incidencia del cáncer de pulmón está influida por factores ambientales y de estilo de vida, como puede ser el consumo de tabaco y de alcohol, la polución del aire y la dieta. Se ha visto que también existe una susceptibilidad genética, de manera que se describe un riesgo aumentado de desarrollar cáncer de pulmón en fumadores que son portadores de ciertas variantes de genes2. El cáncer de pulmón de células no pequeñas (CPNCP) y, dentro de este, el adenocarcinoma, es el tipo histológico de cáncer de pulmón más frecuente hoy en día, ya que engloba más del 50% de todos los casos de cáncer de pulmón3,4,5.
En España, la incidencia es de 29.188 casos nuevos, con 22.930 muertes al año, siendo la principal causa de muerte por cáncer1.
Históricamente, el tratamiento se basaba en distintas combinaciones de fármacos quimioterápicos. Sin embargo, el estudio a nivel molecular del CPNCP ha permitido desarrollar fármacos dirigidos contra dianas terapéuticas concretas, como erlotinib para EGFR y crizotinib para ALK, que han permitido aumentar la supervivencia de estos pacientes6. También la inmunoterapia tiene su papel dentro del abanico terapéutico, pues fármacos como el pembrolizumab permiten el tratamiento en pacientes con alta expresión inmunohistoquímica de PD-L1. Por tanto, a día de hoy, el CPNCP representa el paradigma de la medicina personalizada, y el estudio inmunohistoquímico y molecular se ha vuelto hoy en día un procedimiento de vital importancia antes de decidir cualquier tipo de tratamiento7.
En The Cancer Genome Atlas (TCGA) se publicaron los resultados del análisis molecular de 1.144 casos de carcinoma de pulmón (660 adenocarcinomas y 484 carcinomas escamosos), realizando técnicas de secuenciación de ácido desoxirribonucleico (ADN), incluyendo el análisis del número de copias de genes y el análisis proteómico. Se observó que las variantes de un solo nucleótido (SNV) más frecuentes que aparecen en estos tumores están en los genes TP53 (67,7%) y TTN (59,6%). En este estudio se encontraron alteraciones moleculares comunes en el adenocarcinoma y en el carcinoma escamoso de pulmón en solo 6 genes (TP53, RB1, ARID1A, CDKN2A, PIK3CA y NF1), destacando una frecuencia mucho más alta de alteraciones en TP53 (86%), en CDKN2A (16%) y en PIK3CA (11%) en el carcinoma escamoso. En el adenocarcinoma se describieron frecuencias altas de mutaciones en KRAS (32%), EGFR (16%) y MET (4%)8.
El receptor del factor de crecimiento epidérmico, o EGFR, es un receptor tirosín-quinasa que pertenece a la familia de receptores de claseI9. Su activación provoca la proliferación, la diferenciación, la adhesión celular y la protección frente a la apoptosis10, y también está relacionado con la capacidad invasora tumoral11. Según los datos recogidos en el TCGA, las variantes en el gen EGFR se dan con mayor frecuencia en mujeres (67%) que en hombres (33%)8.
Se han identificado otros genes que son relevantes para el tratamiento en el CPNCP. En algunos casos, con eficacia contrastada pero con una baja frecuencia en las variantes (ALK, ROS1), y en otros casos, aunque son variantes que se presentan con mayor frecuencia (KRAS, BRAF, PICK3CA, MET), su impacto terapéutico aún está en estudio o se desconoce12-15.
El objetivo principal del presente estudio es analizar la frecuencia observada de las variantes detectadas en el tumor primario en CPNCP en pacientes de la provincia de Cádiz, utilizando un panel de 52 genes, mediante secuenciación masiva o Next Generation Sequencing (NGS), incluyendo su potencial clasificación terapéutica.
Material y métodosSe seleccionan pacientes con el diagnóstico de carcinoma de pulmón en el Hospital Universitario Puerta del Mar y en el Hospital Universitario de Jerez de la Frontera mediante una búsqueda, en el sistema de información Vitropath®, de biopsias, piezas quirúrgicas o citologías, entre octubre de 2020 y abril de 2022, de pacientes con diagnóstico de carcinoma de pulmón, excluyendo el carcinoma de células pequeñas, en las que se hubieran realizado estudio de NGS. Se excluyeron dos casos con diagnósticos de sarcoma indiferenciado de célula pequeña de pulmón y linfoma de Hodgkin de afectación pulmonar.
Se obtuvieron un total de 174 muestras de CPNCP en las que se había realizado NGS. De ellas, se excluyeron 3 muestras en las que el estudio molecular fue no valorable. De esta forma, se obtuvieron resultados técnicamente satisfactorios en la NGS de 171 muestras de 169 pacientes distintos.
El estudio fue aprobado por el comité de ética de investigación clínica (CEIC) de la provincia de Cádiz, con fecha de 25 de septiembre de 2020, número de protocolo 96.20/129-N-20.
Estudio inmunohistoquímicoSe realizó un estudio inmunohistoquímico (IHC) en muestras fijadas en formol e incluidas en parafina (FFIP). De cada bloque de parafina se obtuvieron secciones de 3μm de espesor que se colocaron en portaobjetos Super Frost® para tinción IHC estándar.
Se utilizaron los siguientes anticuerpos primarios, siguiendo las recomendaciones de la OMS16,17:
- •
TTF1 (Clona SPT24, Master Diagnóstica®, Granada). Se realizó en 159 muestras. Tiempo de incubación: 24min. La expresión de este marcador se evaluó como positiva o negativa según su expresión nuclear en las células tumorales.
- •
P40 (Clona ZR8, Master Diagnóstica®, Granada). Se realizó en 120 muestras. Tiempo de incubación: 28min. Su expresión también se evaluó como positiva o negativa, valorando los núcleos de las células tumorales.
- •
PD-L1 (Clona SP263, Roche Diagnostics, Barcelona). Se estudió la expresión en 157 muestras. Tiempo de incubación: 36min. Su expresión se evaluó siguiendo la guía de interpretación de «VENTANA PD-L1 (SP263) Assay®»18, indicando el porcentaje de células tumorales con una clara tinción de membrana de cualquier intensidad en la señal del cromógeno. Se agruparon como casos positivos para PD-L1 todos los que tenían expresión igual o mayor del 1% en las células tumorales, y negativos, aquellos con una expresión menor del 1%.
Las técnicas inmunohistoquímicas se automatizaron completamente en un instrumento Roche Ventana BenchMark ULTRA IHC/ISH® (Ventana Medical Systems. Inc., Tucson, AZ, EE.UU.), utilizando el kit de detección DAB Optiview® (Roche Diagnostics, Barcelona).
Extracción de ADNLos estudios moleculares se realizaron en la Unidad de Gestión Clínica (UGC) de Anatomía Patológica del Hospital Universitario Puerta del Mar.
Las secciones de tejido se desparafinaron con xileno y alcohol, se hidrolizaron con 4μl de proteasa por espécimen y se centrifugaron 30segundos a 10.000g. El ADN y el ácido ribonucleico (ARN) se extrajeron utilizando el MagMAX™ FFPE DNA/RNA Ultra Kit® (Thermo Fisher Scientific, Texas, EE.UU.), siguiendo el protocolo determinado por el fabricante. La cuantificación fluorométrica se realizó utilizando el fluorómetro Qubit 2.0® con los kits Qubit ds DNA/RNA HS Assay kits® y el kit Genejet RNA Cleanup and Concentration Micro kit® (Thermo Fisher Scientific, Inc.), siguiendo los protocolos proporcionados por el fabricante y considerándose una concentración apropiada de ácido nucleico cuando fuera mayor de 30ng/μl.
Preparación de librerías y templadoLas librerías de ADN y ARN se construyeron con los kits Oncomine® Focus Assay, Chef-Ready Library y con Ion 530™ kit Chef (Thermo Fisher Scientific, Texas, EE.UU.), siguiendo las instrucciones proporcionadas por el fabricante. Se utilizaron como mínimo 10ng de ADN genómico y de ARN por microplaca. Se utilizaron los Ion Code Barcode® (1-8) (DNA) y Ion Code Barcode® 9-16 (RNA) para ligarlos y purificarlos para obtener aproximadamente 100pM. Todos los pasos para la preparación de las librerías y el templado fueron automatizados utilizando Ion Chef Robot™ (Thermo Fisher Scientific, Texas, EE.UU.).
Secuenciación NGSSe prepararon 8 librerías de ADN y 8 de ARN por carrera en un chip Ion 530™ (Thermo Fisher Scientific, Texas, EE.UU.). Después de cargar el chip, se realizó el análisis de secuencias utilizando el Ion GeneStudio™ S5 System (Thermo Fisher Scientific, Texas, EE.UU.). El panel utilizado fue el Oncomine® Focus Assay (OFA) (Thermo Fisher Scientific, Texas, EE.UU.), diseñado para cubrir 52 genes, especialmente relevantes en cáncer, incluyendo: 35 genes para SNV (Hotspot), e incluyendo en este grupo, además de las SNV, las variantes de múltiples nucleótidos (MNV) y las inserciones o deleciones (INDEL); 19 genes para el estudio de variantes en el número de copias (CNV), y 23 genes para el estudio de fusiones19.
Análisis bioinformáticoLos archivos de secuenciación Binary Alignment Map (BAM) se analizaron con el software Ion Reporter™ (Versión 5.16, Thermo Fisher Scientific, Texas, EE.UU.) y se subieron anonimizadas al sistema en la nube del servidor de Ion Reporter™ bajo una licencia institucional (https://ionreporter.thermofisher.com). La determinación de las variantes y el análisis de datos se realizaron como describieron Park et al.20. Utilizamos Fusions-Single Sample workflow, y el análisis inicial se realizó utilizando Oncomine Variant Annotator en Ion Reporter™.
Para identificar y anotar las variantes de tipo SNV, INDEL y CNV, se aplicaron los siguientes filtros en Ion Reporter™: límite de frecuencia alélica para decidir si es una variante: SNV 0,05, INDEL 0,15, MNV 0,15; cobertura total requerida de lecturas de 500 en SNV, 1.500 lecturas en INDEL y en MNV; proporción de alelos variantes provenientes de una hebra, SNV 0,96, INDEL 0,9; media de los valores absolutos de todas las diferencias pareadas <0,4; 5% para el intervalo de confianza en ploidías de CNV; 4,5 o más copias más de lo normal para la identificación de CNV para las amplificaciones, y número de copias <1 para las deleciones. Para la evaluación de CNV solo se incluyeron los casos que mostraban un Phred Quality Score de 100.
Para la identificación detallada y la revisión manual de las variantes se seleccionó la opción de No filter y se descargaron los archivos tab-separated values (TSV) de todas las variantes del Ion ReporterSystem y se reanalizaron utilizando Microsoft Excel® (v16.0). Se aplicaron los siguientes filtros adicionales a los aplicados previamente en Ion Reporter™, para la exclusión de las siguientes variantes en SNV: intrónicas o en regiones no traducidas (UTR), las que tienen un valor «sinónimo» en el campo «efecto de la variante», las clasificadas como polimorfismos en la base de datos «UCSC Common SNPs», aquellas que sean clasificadas como «benignas» en ClinVar, y las que tienen una probabilidad de ser variantes reales («p-value») menor de 0,05. En las variantes en CNV no se aplicaron filtros adicionales a los descritos en Ion Reporter™.
Para la clasificación de variantes según su patogenicidad (patogénicas, probablemente patogénicas y variantes de significado incierto o VUS), se utilizó la plataforma OncoKDM® (https://api.oncokdm.com/) de la empresa OncoDNA® (Gosselies, Bélgica). Además, también se realizó un estudio bioinformático interno de confirmación utilizando las siguientes bases de datos para estudiar las variantes: dbNSP (https://www.ncbi.nlm.nih.gov/snp/), COSMIC (https://cancer.sanger.ac.uk/cosmic), DECIPHER (https://decipher.sanger.ac.uk/) y ClinVar (https://www.ncbi.nlm.nih.gov/clinvar/).
Análisis estadísticoSe estudiaron las variables clinicopatológicas por su posible asociación con la presencia de las variantes estudiadas. Las variables categóricas se analizaron mediante el test de Fisher en tablas de contingencia. Las variables numéricas se analizaron utilizando el test de Spearman. Para analizar las asociaciones entre las variables categóricas y numéricas se utilizó el test no paramétrico de Wilcoxon, que compara las diferencias entre las medias. Se consideraron como significativos los valores para un p-valor menor a 0,05. Los datos fueron analizados utilizando el paquete R v.4.1.1 (R Development Core Team).
ResultadosPoblación estudiadaEl estudio incluye 171 muestras de 169 pacientes con CPNCP, incluyendo 52 mujeres (30,77%) y 117 hombres (69,23%). La edad media de los pacientes al diagnóstico de cáncer de pulmón fue de 65,29años (rango: 38 a 86años), con una edad media de 63,94años en mujeres (rango: 43 a 85años) y de 65,89años en varones (rango: 38 a 86años). No se aprecian diferencias estadísticamente significativas en la edad, al diagnóstico de los pacientes, entre hombres y mujeres (p=0,2368).
La tabla 1 resume las características de la población estudiada.
Características de la población de estudio
| Característica estudiada | Población estudiada |
|---|---|
| Edad media (rango) | 65,39 años (38 a 86 años) |
| Hombres: número de casos (frecuencia) | Hombres: 117 (69,23%) |
| Mujeres: número de casos (frecuencia) | Mujeres: 52 (30,77%) |
| Adenocarcinoma: número de casos (frecuencia) | 134 (79,29%) |
| Carcinoma escamoso: número de casos (frecuencia) | 24 (12,24%) |
| Carcinoma adenoescamoso: número de casos (frecuencia) | 8 (4,70%) |
| Carcinoma neuroendocrino: número de casos (frecuencia) | 2 (1,18%) |
| Carcinoma PDCG: número de casos (frecuencia) | 1 (0,6%) |
PDCG: pobremente diferenciado de célula grande.
El tipo histológico de la neoplasia de pulmón más frecuente fue el adenocarcinoma (79,29%), estando presente en 134 pacientes con una edad media de 64,39años (rango: 38 a 86años), seguido de lejos por el carcinoma escamoso en 24 pacientes (14,2%), con una edad media de 68,54años (rango: 53 a 82años), el carcinoma adenoescamoso en 8 pacientes (4,70%), con una edad media de 71,62años (rango: 56 a 84años) y el carcinoma neuroendocrino de células grandes en 2 pacientes (1,18%) varones de 55 y 65años. Finalmente, a una paciente (mujer de 68años) se le diagnosticó carcinoma pobremente diferenciado de célula grande (PDCG) (0,6%). A pesar de que el adenocarcinoma tiene tendencia a presentarse a una edad más joven, mientras que el carcinoma adenoescamoso parece presentarse en pacientes de mayor edad, la diferencia no fue estadísticamente significativa (p=0,05037). Tampoco se apreciaron diferencias estadísticamente significativas en el sexo entre los distintos tipos histológicos (p=0,2836).
De los 134 casos de adenocarcinoma, se realizó un estudio inmunohistoquímico en 125 (93,28%), de los cuales 109 (87,2%) fueron positivos para TTF1. De los 24 casos de carcinoma escamoso, se realizó un estudio inmunohistoquímico en 20 (83,33%), donde la totalidad de estos casos eran positivos para p40 y negativos para TTF1. Los 8 casos de carcinoma adenoescamoso eran positivos tanto para TTF1 como para p40. En el carcinoma neuroendocrino de célula grande, uno de los casos era positivo para TTF1 y negativo para p40, mientras que el otro caso era negativo para ambos marcadores. El único caso de carcinoma pobremente diferenciado de célula grande también fue negativo para los dos marcadores.
Se estudió la expresión de PD-L1 en 131 (97,76%) de los casos de adenocarcinoma, de los cuales 71 (54,20%) casos fueron negativos, 29 (22,13%) casos tuvieron del 1% al 50% de expresión de PD-L1, 14 (10,69%) casos expresaron del 51% al 80% y 17 (12,98%) casos expresaron más del 80% de PD-L1. En los casos de carcinoma escamoso, se estudió la expresión de PD-L1 en 22 (91,67%) pacientes, de los cuales 13 fueron negativos (59,09%), 4 (18,18%) lo expresaban del 1% al 50%, 4 (18,18%) del 51% al 80% y 1 (4,54%) caso expresaba más del 80% de PD-L1. En el carcinoma adenoescamoso, la expresión de PD-L1 se estudió en 7 (87,50%) casos, de los cuales 5 (71,42%) fueron negativos, 1 (14,29%) caso expresó un 50% y el otro caso (14,29%) expresó un 95%. En ambos casos de carcinoma neuroendocrino de célula grande se estudió la expresión de PD-L1, siendo uno negativo y el otro positivo, con una expresión del 95%. En el caso de carcinoma PDCG no pudo estudiarse la expresión de PD-L1. La expresión de PD-L1 no mostró relación estadísticamente significativa con la edad (p=0,6384), con el sexo (p=0,4173), ni con el tipo histológico (p=0,8943).
Estudio molecular mediante Next Generation SequencingLas variantes más frecuentes del panel de 52 genes estudiados mediante Next Generation Sequencing (NGS), encontradas en CPNCP se muestran en la figura 1. De estas variantes, describimos con mayor detalle las encontradas en los genes EGFR, KRAS, BRAF, PIK3CA y MET, ya que son las que aparecen en mayor proporción en nuestra población de estudio.
SNVEl estudio de las SNV, realizado en los 169 pacientes, mostró variantes relevantes de este tipo en 108 de ellos (63,91%), siendo 68 hombres (62,96%) y 40 mujeres (37,04%). La edad media de esta población fue de 65,68años (rango: 43 a 86años). Se han identificado 142 mutaciones relevantes, incluyendo en este grupo SNV, INDEL y MNV, siendo en su mayoría SNV (130 variantes, 91,55%), seguido de INDEL (9 variantes, 6,34%) y de MNV (3 variantes, 2,11%).
Los genes más frecuentemente afectados por SNV fueron KRAS, EGFR, BRAF, PIK3CA y MET (fig. 1). Estos cinco genes se vieron afectados por SNV en un total de 89 pacientes (52,66%).
El adenocarcinoma fue el tipo histológico más frecuente en las neoplasias con variantes SNV de KRAS (48 casos, 94,12%), con una diferencia estadísticamente significativa respecto a los tumores sin SNV de KRAS (p=0,007361).
En EGFR, se identificaron 20 pacientes con variantes, y en 17 casos (85%) se detectaron en mujeres. La mayor frecuencia de mujeres en los pacientes con variantes SNV en EGFR es estadísticamente significativa (p<0,0001) respecto a los casos que no tienen variantes en este gen. Además, el único tipo histológico presente en tumores con SNV de EGFR fue el adenocarcinoma. El resto de los resultados de los genes quedan recogidos en la tabla 2. No se aprecian diferencias estadísticamente significativas en el sexo, en la edad ni en el tipo histológico entre los pacientes con variantes de tipo SNV en los genes BRAF, PIK3CA y MET y los pacientes que no presentan variantes en estos genes.
Características de la población en las variantes más frecuentes detectadas
| SNV (SNV +INDEL +MNV) | CNV (deleciones y amplificaciones) | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| Característica / Gen | KRAS | EGFR | BRAF | PIK3CA | MET | Deleciones (todas) | NF1AMP | EGFRAMP | CCND1AMP | MYCAMP | KRASAMP |
| Edad media (años) | 62,21 | 67 | 68,14 | 67,82 | 68,2 | 64,72 | 65,71 | 65,43 | 65 | 61,45 | 61.38 |
| Hombres, n (%) | 35 (68,6) | 3 (15) | 8 (87,14) | 7 (63,64) | 4 (80) | 21 (13,21) | 28 (17,72) | 13 (61,9) | 8 (72,7) | 8 (72,7) | 5 (62,5) |
| Mujeres, n (%) | 16 (31,4) | 17a (85) | 6 (42,86) | 4 (36,66) | 1 (20) | 7 (9,21) | 7 (9,21) | 7 (38,1) | 3 (27,3) | 3 (27,3) | 3 (37,5) |
| Adenocarcinoma, n (%) | 48a (94,12) | 20a (100) | 13 (92,86) | 9 (81,82) | 4 (80) | 23 (82,14) | 31 (88,6) | 19 (90,5) | 7 (63,6) | 9 (81,8) | 7 (87,5) |
| Ca escamoso, n (%) | 1 (1,96) | 0 (0) | 1 (7,14) | 2 (18,18) | 0 (0) | 3 (10,71) | 2 (5,7) | 1 (4,8) | 4 (36,4) | 2 (18,2) | 1 (12,5) |
| Ca adenoescamoso, n (%) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 1 (20) | 2 (7,14) | 2 (5,7) | 1 (4,8) | 0 (0) | 0 (0) | 0 (0) |
| Ca neuroendocrino, n (%) | 1 (1,96) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) |
| Ca PDCG, n (%) | 1 (1,96) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) |
| Patogénico. Tier I, n (%) | 9 (17,65) | 13 (65) | 4 (28,57) | 0 (0) | 0 (0) | 2 (7,14) | 0 (0) | 1 (5) | 1 (9,09) | 1 (9,09) | 0 (0) |
| Patogénico. Tier II, n (%) | 38 (74,51) | 0 (0) | 2 (14,29) | 7 (63,64) | 0 (0) | 2 (7,14) | 2 (5,71) | 2 (10) | 0 (0) | 1 (9,09) | 0 (0) |
| Patogénico. Tier III, n (%) | 0 (0) | 0 (0) | 4 (28,57) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) |
| Prob. Patogén. Tier II, n (%) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 3 (10,71) | 0 (0) | 18 (90) | 9 (81,81) | 8 (72,73) | 0 (0) |
| Prob. Patogén. Tier III, n (%) | 0 (0) | 1 (5) | 1 (7,14) | 1 (9,09) | 0 (0) | 1 (3,57) | 0 (0) | 0 (0) | 0 (0) | 0 (0) | 0 (0) |
| VUS. Tier III, n (%) | 1 (1,96) | 5 (25) | 2 (14,29) | 3 (27,27) | 1 (20) | 20 (71,43) | 33 (94,29) | 0 (0) | 0 (0) | 0 (0) | 8 (100) |
La expresión inmunohistoquímica de PD-L1 fue significativamente mayor (p=0,02797) en las neoplasias que no presentaban variantes en EGFR respecto a las que sí presentaban variantes en ese gen. En el resto de los genes estudiados no se encontró una asociación significativa con la expresión de PD-L1 (KRAS: p=0,8122; BRAF: p=0,7973; PIK3CA: p=0,3574; MET: p=0,3598).
CNVEl estudio molecular de CNV se realizó con éxito en 169 pacientes, donde se identificaron 192 variantes relevantes en 95 (56,21%) de estos pacientes, siendo 69 (58,97%) hombres y 26 (50%) mujeres. La edad media de esta población fue de 64,94años (rango: 46 a 86años). De las 192 variantes detectadas, 28 (14,58%) eran deleciones y 164 (85,42%), amplificaciones.
La tabla 2 recoge las características de los pacientes con CNV.
No se apreciaron diferencias estadísticamente significativas en el sexo (p=0,7387), en la edad al diagnóstico (p=0,7599) ni en el tipo histológico (p=0,5974), entre los pacientes que presentaban deleciones y los que no. Los genes que presentaron deleciones con mayor frecuencia fueron JAK2 (4 casos), AR, FGFR1 y KIT (3casos, cada uno). Solo se detectaron dos deleciones patogénicas de impacto terapéutico «TierI», en los genes ALK y KIT.
En cuanto a las amplificaciones, se observaron en el 54,7% de los hombres (con una media de 1,84 amplificaciones/paciente) y en el 50% de las mujeres (con una media de 1,77 amplificaciones/paciente). La edad media de estos pacientes fue de 64,72años (rango: 46 a 84años). En las neoplasias donde se identificaron amplificaciones, también el adenocarcinoma fue el tipo histológico predominante, con 132 amplificaciones (80,49%), seguido por el carcinoma escamoso (24 amplificaciones, 14,63%), el carcinoma adenoescamoso (5 casos, 3,05%) y el carcinoma neuroendocrino de célula grande (3 amplificaciones, 1,83%).
El gen que presentó amplificaciones con mayor frecuencia fue NF1. La edad media de este grupo de pacientes fue de 65,71años (rango: 48 a 84años) (tabla 1). No se aprecian diferencias estadísticamente significativas en el sexo (p=0,232), en la edad (p=0,3386) ni en el tipo histológico (p=0,2009) entre los pacientes con amplificaciones en NF1 y los que no presentan amplificaciones en este gen. En NF1 se detectaron dos amplificaciones patogénicas (5,71%), ambas clasificadas como «TierII» en su impacto terapéutico.
El resto de los genes que presentaron amplificaciones quedan recogidos en la tabla 2. No se aprecian diferencias estadísticamente significativas en el sexo, en la edad ni en el tipo histológico entre los pacientes con amplificaciones en los genes EGFR, CCND1, MYC y KRAS y los que no presentan amplificaciones en estos genes.
Fusiones (translocaciones)En 16 pacientes (9,47%) se detectaron fusiones génicas. La edad media de estos pacientes fue de 63,94años (rango: 51 a 75años), siendo 10 (62,5%) hombres y 6 (37,5%) mujeres. Se detectaron un total de 18 fusiones, en EGFR, MET, KT3, ALK, ROS1, NTRK, RET y TMPRSS2:ERG (tabla 3).
Clasificación de fusiones
| Genes | N° pacientes / N° muestras | Sexo | Edad media | Tipo histológico | Clasificación |
|---|---|---|---|---|---|
| MET | 9 / 9 | 5 hombres / 4 mujeres | 63,94 años | Adenocarcinoma | PatogénicaTier I |
| RET | 1 / 1 | 1 hombre | 56 años | Carcinoma adenoescamoso | PatogénicaTier I |
| AKT3 | 1 / 1 | 1 hombre | 66 años | Carcinoma escamoso | PatogénicaTier III |
| ALK | 1 / 1 | 1 hombre | 66 años | Adenocarcinoma | PatogénicaTier II |
| EGFR | 1 / 1 | 1 mujer | 67 años | Carcinoma escamoso | PatogénicaTier II |
| ROS1 | 1 / 2a | 1 mujer | 72 años | Adenocarcinoma | PatogénicaTier II |
| NTRK | 1 / 1 | 1 hombre | 72 años | Carcinoma adenoescamoso | PatogénicaTier III |
| ERG | 1 / 2a | 1 hombre | 68 años | Adenocarcinoma | PatogénicaTier III |
Se han estudiado las asociaciones entre los distintos tipos de mutaciones. Las asociaciones entre SNV en distintos genes, detectadas en 28 pacientes, quedan reflejadas en la tabla 4.
Asociaciones entre genes con variantes dentro de las SNV
| Gen | Segundo gen afectado |
|---|---|
| BRAF | IDH1IDH2METPIK3CA |
| CTNNB1 | BRCA1RETKRASFGFR3 |
| EGFR | EGFR (x3)KRAS (x2)MTORERBB2 |
| ERBB2 | EGFRKRAS |
| HRAS | APCKRASMET |
| IDH2 | CDK4BRAF |
| KRAS | APCCDK4CTNNB1EGFR (x2)ERBB2 (x2)FBXW7FGFR3FGFR4HRASJAK2KITKRASMAP2K1METPIK3CA |
| MET | APCBRAFHRASKRAS |
| NRAS | JAK1 |
| PIK3CA | BRAFIDH1KRASROS1 |
Las asociaciones entre variantes de tipo SNV y CNV fueron las más frecuentes (57 pacientes), destacando la asociación de SNV en KRAS con CNV en los genes AR, BIRC2, CCND1, CDK4, CDK6, DCUN1D1, EGFR, ERBB2, FGFR1, FGFR2, JAK3 y KRAS; la asociación de SNV en EGFR con CNV en los genes ALK, AR, BIRC2, CCND1, CDK4, EGFR, KIT, MYC y NF1, y la asociación de SNV en PIK3CA con CNV en los genes AR, CDK4, EGFR, ERBB2, FGFR1, FGFR3, JAK1, KRAS, MYC y NF1.
Otras asociaciones encontradas fueron entre SNV y fusiones (6 pacientes) y entre CNV y fusiones (10 pacientes). Además, en cinco pacientes se encontraron simultáneamente variantes de tipo SNV, CNV y fusiones.
DiscusiónEs importante estudiar las variantes que intervienen en el desarrollo del cáncer de pulmón para poder trabajar en tratamientos efectivos. Los estudios de caracterización molecular indican que las mutaciones en los genes EGFR, PIK3CA, BRAF y KRAS se identifican en el 40% de los casos de CPNCP21. En nuestra serie, la frecuencia de alteraciones en esos cuatro genes fue ligeramente mayor, observándose SNV en el 50,89% de los pacientes.
Además de los datos disponibles en TCGA (1.144 casos de CPNCP)22-24, en 2018 se realizó un estudio, llamado Thoracic Tumors Registry (TTR), que incluyó 6.600 pacientes con CPNCP provenientes de 56 hospitales españoles25. En ambas series, la edad media de los pacientes (66,37 y 64años, respectivamente) fue muy similar a la de nuestra serie (65,29años). El porcentaje de hombres en TCGA (58,8%) fue algo menor que en TTR y en nuestra serie (73,4% y 69,23%, respectivamente). La mayor proporción de adenocarcinoma fue aún más marcada en nuestra serie (79,29%) que en TCGA o en TTR (57,69% y 63,8%, respectivamente).
El panel de NGS utilizado en nuestra serie se centra en alteraciones moleculares con posible impacto terapéutico y no incluye las SNV más frecuentes detectadas en CPNCP en TCGA (TP53 y TTN)24. La frecuencia de alteraciones de tipo SNV fue algo superior en nuestra serie y en TTR, respecto a TCGA, en los genes KRAS (35,9% y 28,4%, respectivamente, frente al 19,4%) y EGFR (14,1% y 18,1%, respectivamente, frente al 10,2%), debido a la mayor proporción de adenocarcinoma en ambas series. En otros genes, como BRAF, la frecuencia era similar en nuestra serie (9,86%) y en TCGA (6,1%), pero menor en TTR (3,3%). No se describen las alteraciones de PIK3CA ni MET en TTR, pero en nuestra serie (7,75%, 3,52%) y en TCGA (8,2%, 3%) presentaron proporciones muy parecidas.
Las mutaciones SNV en KRAS y en EGFR fueron mutaciones muy raras en el carcinoma escamoso, en TCGA (3,60% y 11,1%, respectivamente) y aún más infrecuentes en nuestra serie (1,96% y 0%, respectivamente). En TTR no se realizó el análisis molecular por tipo histológico.
En las CNV, la frecuencia de deleciones en nuestra serie fue similar a las encontradas en TCGA, predominando la afectación de los genes JAK2 (en torno al 2,5% en ambas series), AR (entre el 0,8% y el 1,8%, respectivamente), FGFR1 (entre el 1% y el 1,8%, respectivamente) y KIT (>0,1% en TCGA y un solo caso «TierI» en nuestra serie). TCGA no recoge ninguna CNV de tipo deleción en ALK, pero en nuestra serie detectamos un caso de esta variante con impacto terapéutico significativo, que hasta la fecha es la primera descrita en la literatura. En TTR, no se detectaron deleciones.
La frecuencia global de las amplificaciones de nuestra serie fue superior a las observadas en TCGA, sobre todo en el gen NF1, con un 21,34% en nuestra serie frente a un 0,5% en TCGA. Por ello, es importante filtrar solo las variantes que tienen impacto terapéutico, que en nuestra serie solo fueron 2 casos clasificados como «patogénico, TierII» en el gen NF1 (1,18%) y un caso clasificado como «patogénico, TierI» en EGFR (0,59% en nuestra serie, frente al 6% en TCGA). La frecuencia global de otros genes (incluyendo su clasificación desde VUS hasta patogénicas) sí fue similar a la descrita en TCGA, como en CCND1 (7,80% TCGA, 6,71% nuestra serie) y en KRAS (5,10% TCGA, 4,88% nuestra serie). En otros genes, como FGFR1, la frecuencia de amplificaciones en nuestra serie fue muy inferior (3,66%) a las de TCGA y TTR (8,9% y 7,7%, respectivamente), mientras que en MET las frecuencias de amplificaciones fueron algo superiores en TTR (7,10%) respecto a TCGA (1,70%) y a nuestra serie (3,66%).
En cuanto a las fusiones, en TCGA y en TTR se han detectado en ALK (2%-11,4%), ROS1 (3%-4,3%) y RET (1%-2,3%). En nuestra serie, las fusiones en los genes ALK (0,59%) y ROS1 (1,18%) se detectaron en una menor proporción. Hay fusiones en otros genes en nuestra serie que no vienen recogidas en TTR, como MET (5,33%) o EGFR (0,59%)22-24.
Las fusiones descritas en la literatura de MET suelen ser con los genes CD47, KIF5B y STARD3NL2,3. Se han descrito fusiones del gen EGFR con RAD51 y SHC14.
En este estudio hemos encontrado la fusión de TMPRSS2:ERG, que es característica del carcinoma de próstata. En la literatura se ha descrito dicha mutación en un paciente con carcinoma de próstata y de pulmón concomitante, aunque finalmente el carcinoma de pulmón se consideró metástasis del de próstata debido a esta mutación26. Sin embargo, en nuestro estudio el paciente no tenía carcinoma de próstata.
No se ha establecido ninguna asociación estadísticamente significativa entre la edad de los pacientes y la alteración genética detectada, pero sí se ha observado relación entre el sexo de los pacientes y las alteraciones en EGFR, donde, como describe la bibliografía8, las mujeres tienen mayor predisposición a presentar variantes en este gen. También se han detectado asociaciones entre el tipo histológico y las variantes SNV en KRAS y EGFR, pues las alteraciones en KRAS se encuentran en mayor proporción en el adenocarcinoma, y en EGFR, en nuestra serie, solo se han encontrado alteraciones moleculares en el adenocarcinoma8.
Aunque existe bibliografía contradictoria al respecto, se ha descrito una relación estadísticamente significativa entre los niveles de expresión de PD-L1 y los tumores con mutación en el gen EGFR, al igual que en los resultados de nuestro estudio. Aunque en la literatura se ha encontrado relación entre la expresión de PD-L1 y el sexo o el subtipo histológico27,28, en nuestro trabajo no hemos podido confirmar estos hallazgos.
La existencia de más de una mutación en el mismo tumor es especialmente relevante hoy día en cáncer de pulmón de célula no pequeña, pues estas asociaciones tienen implicaciones terapéuticas y pronósticas. En la serie de Martorell et al.29 de 326 pacientes de CPNCP, el 12% de los casos con estadioIV tenían múltiples mutaciones, destacando la coexistencia de SNV de EGFR con reordenamientos en ALK y con otras SNV en KRAS, BRAF y PIK3CA. Un ejemplo de la importancia de estas asociaciones es la coexistencia de SNV en EGFR con mutaciones en el exón 14 de MET y con amplificaciones de MET (tres casos en nuestra serie), que se asocian con una resistencia a los inhibidores EGFR30.
Una de las limitaciones de este estudio es el sesgo de selección de los pacientes a los que se realizan estudios de secuenciación masiva, por lo que la distribución de algunas variables clínicas y anatomopatológicas, como el tipo histológico, no corresponden a su frecuencia real en los pacientes de cáncer de pulmón.
El panel de NGS utilizado en este estudio solo estudia las áreas de interés o hotspots en las SNV. En general, los paneles utilizados en la práctica clínica se centran, sobre todo, en aquellos genes que se asocian con posibles dianas terapéuticas conocidas hasta la fecha, para lo cual es necesario saber qué genes se examinan solo en las áreas más significativas (hotspots) y qué genes son secuenciados completamente por el panel31.
La utilización de las técnicas de NGS en los departamentos de anatomía patológica ha demostrado ser más coste-eficiente que las estrategias basadas en la determinación secuencial de estudios moleculares mediante reacción en cadena de la polimerasa (PCR)32,33.
ConclusionesEn este estudio hemos realizado un examen exhaustivo de las variantes génicas encontradas en los pacientes de carcinoma de pulmón de células no pequeñas de la provincia de Cádiz. Creemos que estos datos son muy relevantes para ampliar el estudio de las posibles variables génicas que pueden encontrarse en pacientes con CPNCP, pues es el primero que se realiza de estas características en la provincia. De hecho, hemos obtenido datos no descritos con anterioridad en la literatura, como la deleción en ALK, o la fusión de TMPRSS2:ERG. La descripción de nuevas variantes génicas ayudará a desarrollar nuevos tratamientos para el cáncer de pulmón de células no pequeñas.
Responsabilidades éticasEste trabajo es un estudio retrospectivo que no implica modificación en el manejo clínico de los pacientes seleccionados. Se ha seguido la legislación española y europea de protección de datos y de investigación biomédica, así como los principios éticos de la Declaración de Helsinki, en su versión más reciente.
FinanciaciónEl presente trabajo ha sido financiado parcialmente por la Convocatoria de Ayudas del Sistema Andaluz del Conocimiento para la Contratación de Jóvenes Investigadores y Personal Técnico de Apoyo de I+D+I, número de expediente: ACOJIN21-06, y la Convocatoria ITI Cádiz con Fondos FEDER, número de expediente PI-0032-2017.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.