El objetivo de este estudio fue analizar las características genotípicas de los aislados de Enterococcus con resistencia adquirida a vancomicina (ERV) obtenidos durante un período de 3 años y 2 meses en el Hospital Clínic de Barcelona.
MétodosSe incluyeron en el estudio todos los aislados de ERV obtenidos en el período de enero de 2004 a marzo de 2007. Se analizó el mecanismo de resistencia a vancomicina y las resistencias asociadas a otros antibióticos. Los aislados ERV fueron tipificados por electroforesis en campo pulsante (PFGE) y multi-locus-sequence-typing (MLST).
ResultadosSe obtuvieron 39 ERV que fueron identificados como Enterococcus faecium y representaron el 2% del total de enterococos aislados durante el período de tiempo estudiado. El genotipo vanA fue detectado en 38 de los aislados y el genotipo vanB2 en uno adicional. Los 39 ERV fueron clasificados en 13 pulsotipos diferentes (A-M) por PFGE, incluyendo un pulsotipo principal, A, que agrupaba 13 aislados. La secuencia tipo fue identificada por MLST en 24 de las cepas (con patrones diferentes o estrechamente relacionados) y todas ellas fueron adscritas al complejo clonal CC17, excepto 2 que fueron adscritas al complejo CC9. Todas las cepas mostraron un fenotipo de multirresistencia, incluyendo en muchos casos ampicilina, ciprofoxacina, eritromicina, estreptomicina, gentamicina, kanamicina y cloranfenicol, albergando múltiples genes de resistencia asociados. Los genes esp y/o hyl fueron detectados en 37 de los ERV.
ConclusiónTodas las cepas, excepto una, presentaron el genotipo vanA y formaban parte mayoritariamente del complejo clonal CC17.
The objective of this study was to analyse the genotypic characteristics of all Enterococcus isolates with acquired vancomycin resistance (VRE) recovered in the Hospital Clinic (Barcelona, Spain) in a period of three years and two months.
MethodsAll VRE isolated in the referred Hospital in the period January 2004-March 2007 were included in the study. The vancomycin resistance mechanism was investigated, as well as other antibiotic resistance mechanisms. Isolates were also typed by pulsed-field-gel-electrophoresis (PFGE) and multi-locus-sequence-typing (MLST).
ResultsThirty-nine VRE were recovered, all being identified as E. faecium, representing 2% of total enterococci obtained in that period. Thirty-eight of them carried the vanA gene, and one isolate the vanB2 gene. The 39 VRE were classified into 13 different pulsotypes (A-M), with one main pulsotype, A, which included 13 isolates. The sequence type was identified by MLST in 24 VRE (with unrelated or closely-related PFGE patterns), and they were ascribed to the clonal complex CC17, but two classified as CC9. All VRE showed a multiresistance phenotype, including, in most cases ampicillin, ciprofloxacin, erythromycin, streptomycin, gentamicin, kanamycin and chloramphenicol, harbouring multiple antibiotic resistance genes. The presence of esp and/or hyl genes was identified in 37 VRE.
ConclusionAll VRE, but one, showed the vanA genotype and they were mostly ascribed to the high-risk clonal complex CC17.
Los enterococos, que forman parte de la microbiota intestinal de personas y animales, son importantes patógenos nosocomiales. El género Enterococcus se caracteriza por presentar resistencia intrínseca a un gran número de antibióticos, incluyendo betalactámicos, clindamicina o bajas concentraciones de aminoglucósidos y por su capacidad para adquirir nuevas resistencias, disminuyendo las posibilidades terapéuticas para el tratamiento de estas infecciones. Por ello, el interés hacia los enterococos aumentó notablemente cuando se detectaron las primeras cepas con resistencia adquirida a vancomicina (ERV) en 1988, en Francia y Reino Unido1,2.
La resistencia a los glucopéptidos ha aumentado en diversos países, representando un importante problema para el tratamiento de las infecciones enterocócicas, aunque la prevalencia de ERV en España es aún baja, según los datos del European Antimicrobial Resistance Surveillance System (EARSS) (< 5%)3. Se han descrito 8 genotipos de resistencia adquirida a glucopéptidos (vanA, vanB, vanD, vanE, vanG, vanL, vanN y vanM)4, pero son los genotipos vanA y vanB los más comúnmente detectados. La mayoría de las infecciones enterocócicas están causadas por la especie Enterococcus faecalis, sin embargo, la resistencia a vancomicina es más frecuente en la especie E. faecium5.
El aumento en la detección de E. faecium en el ambiente hospitalario está relacionado con la expansión, en todo el mundo, de un complejo clonal específico (CC17). En este complejo clonal se agrupan la mayoría de los aislados que causan infecciones humanas y brotes hospitalarios en todo el mundo6,7. Las cepas CC17 frecuentemente presentan resistencia a ampicilina y fluoroquinolonas y esta línea genética está asociada en la mayoría de los casos a la presencia del gen de virulencia esp y en algunos también a hyl8.Estas características podrían haber favorecido la expansión global de este complejo clonal.
El objetivo de este trabajo ha sido analizar las características genotípicas de las cepas clínicas ERV obtenidas durante un período de 3 años y 2 meses en el Hospital Clínic de Barcelona. También se han evaluado sus genes de virulencia y las líneas genéticas circulantes de ERV en dicho hospital.
MétodosCepas estudiadasSe incluyeron en el estudio todas las cepas clínicas de ERV aisladas en el Hospital Clínic de Barcelona en el período comprendido entre enero de 2004 y marzo de 2007, y que fueron obtenidas en el contexto de un brote epidémico. No se incluyeron los aislados correspondientes a estudios de colonización.
Tanto la identificación de especie como la determinación de la sensibilidad a diferentes antimicrobianos se realizó con el sistema Phoenix (Becton Dickinson). La caracterización de especie se confirmó mediante la técnica de reacción en cadena de la polimerasa (PCR) utilizando cebadores específicos de especie9. En las cepas resistentes a glucopéptidos el fenotipo de resistencia fue corroborado mediante el método de E-test en placas de agar Müller-Hinton.
Caracterización del genotipo de resistencia a antibióticos y de virulenciaLa caracterización del genotipo de resistencia a vancomicina se realizó determinando por PCR la presencia de los genes vanA, vanB, vanC1, y vanC2/C3 asociados con la resistencia a glucopéptidos10. Los amplicones obtenidos fueron secuenciados para confirmar el mecanismo y para determinar el alelo en caso del genotipo vanB (vanB1, vanB2 o vanB3). Asimismo, se estudió la presencia de otros genes de resistencia a eritromicina (erm[A] y erm[B]), tetraciclina (tet[M] y tet[L]), cloranfenicol [catA] y resistencia de alto nivel a aminoglucósidos (RAN) (ant[6]-Ia, aac[6’]-Ie-aph[2”]-Ia y aph[3’]-IIIa) por PCR con cebadores y condiciones específicas10. La presencia de los genes de virulencia esp y hyl fue también determinada por PCR11.
Estudio de la diversidad clonalEl estudio de la clonalidad de los aislados se realizó mediante electroforesis de campo pulsante (PFGE): tras la lisis del DNA cromosómico, embebido en bloques de agarosa, se llevó a cabo su restricción con la enzima SmaI durante 4h a 30°C y posteriormente se cargaron en un gel de agarosa al 1,2% y se realizó la electroforesis en un aparato CHEF DRII (Bio-Rad) con las siguientes condiciones: 6V/cm2 con pulsos de 5s y 35s durante 24h. Los patrones de macrorrestricción obtenidos se interpretaron según los criterios de Tenover et al., 199512.
Del mismo modo, se estudió la secuencia tipo (ST) por MLST en cepas seleccionadas (24 cepas, correspondientes a todas las cepas con pulsotipos diferentes o estrechamente relacionados). Para este propósito se amplificaron 7 genes constitutivos (atpA, ddl, gdh, purK, gyd, pstS y adk) que fueron secuenciados. Las secuencias obtenidas fueron comparadas con las incluidas en la base de datos http://efaecium.mlst.net/, para asignar un alelo a cada uno de los genes. La combinación de los 7 alelos correspondió con una secuencia tipo (ST), englobada en un determinado complejo clonal (CC). Las secuencias tipo de algunas de las cepas habían sido obtenidas en un estudio previo11.
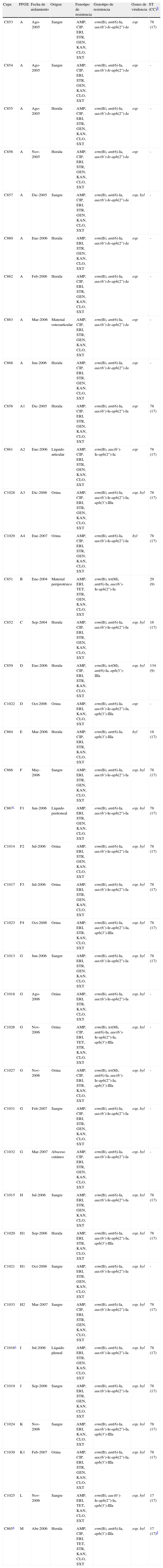
ResultadosEpidemiologíaEntre enero de 2004 y marzo del 2007 se obtuvieron un total de 1.940 aislados clínicos de enterococos, de los cuales se detectaron 39 VRE, todos ellos de la especie E. faecium, que representa un 2% del total de enterococos y un 9,1% del total de E. faecium en dicho período. No se aisló ninguna cepa de E. faecalis ni de otras especies de enterococo, resistentes a la vancomicina, durante el período de estudio. Estas 39 cepas VRE fueron aisladas de diferentes localizaciones, entre las cuales cabe destacar sangre (31%), heridas (31%) y orina (26%), entre otras (tabla 1). Todas las cepas fueron aisladas de diferentes pacientes, excepto dos de ellas (C867 y C1016) que fueron obtenidas del mismo paciente (de líquido peritoneal y de líquido pleural), y ambas se incluyeron en el estudio ya que presentaron patrones de PFGE diferentes. Las infecciones causadas por dichas cepas eran de origen nosocomial y se recuperaron en el contexto de un brote epidémico detectado en diversas áreas del hospital durante el período de tiempo comentado.
Características fenotípicas y genotípicas de las cepas Enterococcus faecium vanA y vanB2 aisladas en el Hospital Clínic de Barcelonaa
| Cepa | PFGE | Fecha de aislamiento | Origen | Fenotipo de resistencia | Genotipo de resistencia | Genes de virulencia | ST (CC)b |
| C853 | A | Ago-2005 | Sangre | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp | 78 (17) |
| C854 | A | Ago-2005 | Sangre | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp | - |
| C855 | A | Ago-2005 | Herida | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp | - |
| C856 | A | Nov-2005 | Herida | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp | - |
| C857 | A | Dic-2005 | Sangre | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | - |
| C860 | A | Ene-2006 | Herida | AMP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp | - |
| C862 | A | Feb-2006 | Herida | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp | - |
| C863 | A | Mar-2006 | Material osteoarticular | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp | - |
| C868 | A | Jun-2006 | Herida | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp | - |
| C858 | A1 | Dic-2005 | Herida | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp | 78 (17) |
| C861 | A2 | Ene-2006 | Líquido articular | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), aac(6’)-Ie-aph(2”)-Ia | esp | 78 (17) |
| C1028 | A3 | Dic-2006 | Orina | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia, aph(3’)-IIIa | esp, hyl | 78 (17) |
| C1029 | A4 | Ene-2007 | Orina | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | hyl | 78 (17) |
| C851 | B | Ene-2004 | Material periprotésico | AMP, ERI, TET, STR, GEN, KAN, CLO, SXT | erm(B), tet(M), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | 29 (9) | |
| C852 | C | Sep-2004 | Herida | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | 16 (17) |
| C859 | D | Ene-2006 | Herida | AMP, CIP, ERI, STR, KAN, CLO, SXT | erm(B), tet(M), ant(6)-Ia, aph(3’)-IIIa | esp, hyl | 134 (9) |
| C1022 | D | Oct-2006 | Orina | AMP, ERI, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia, aph(3’)-IIIa | esp | - |
| C864 | E | Mar-2006 | Herida | AMP, CIP, ERI, STR, KAN, CLO, SXT | erm(B), ant(6)-Ia, aph(3’)-IIIa | hyl | 18 (17) |
| C866 | F | May-2006 | Sangre | AMP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | 78 (17) |
| C867c | F1 | Jun-2006 | Líquido peritoneal | AMP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | 78 (17) |
| C1014 | F2 | Jul-2006 | Orina | AMP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | 78 (17) |
| C1017 | F3 | Jul-2006 | Orina | AMP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | 78 (17) |
| C1023 | F4 | Oct-2006 | Orina | AMP, ERI, STR, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia, aph(3’)-IIIa | esp, hyl | 78 (17) |
| C1013 | G | Jun-2006 | Sangre | AMP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | 78 (17) |
| C1018 | G | Ago-2006 | Orina | AMP, ERI, STR, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | - |
| C1026 | G | Nov-2006 | Orina | AMP, CIP, ERI, TET, STR, KAN, CLO, SXT | erm(B), tet(M), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia, aph(3’)-IIIa | esp, hyl | - |
| C1027 | G | Nov-2006 | Orina | AMP, CIP, ERI, STR, KAN, CLO, SXT | erm(B), tet(M), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia, aph(3’)-IIIa | esp, hyl | - |
| C1031 | G | Feb-2007 | Sangre | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | - |
| C1032 | G | Mar-2007 | Absceso cutáneo | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | - |
| C1015 | H | Jul-2006 | Sangre | AMP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | 78 (17) |
| C1020 | H1 | Sep-2006 | Herida | AMP, ERI, STR, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia, aph(3’)-IIIa | esp, hyl | 78 (17) |
| C1021 | H1 | Oct-2006 | Sangre | AMP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | - |
| C1033 | H2 | Mar-2007 | Sangre | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | 78 (17) |
| C1016c | I | Jul-2006 | Líquido pleural | AMP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | 78 (17) |
| C1019 | J | Sep-2006 | Sangre | AMP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia | esp, hyl | 78 (17) |
| C1024 | K | Nov-2006 | Sangre | AMP, ERI, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia, aph(3’)-IIIa | esp, hyl | 78 (17) |
| C1030 | K1 | Feb-2007 | Orina | AMP, CIP, ERI, STR, GEN, KAN, CLO, SXT | erm(B), ant(6)-Ia, aac(6’)-Ie-aph(2”)-Ia, aph(3’)-IIIa | esp, hyl | 78 (17) |
| C1025 | L | Nov-2006 | Sangre | AMP, ERI, TET, KAN, CLO, SXT | erm(B), aac(6’)-Ie-aph(2”)-Ia, aph(3’)-IIIa | esp, hyl | 17 (17) |
| C865a | M | Abr-2006 | Herida | AMP, CIP, ERI, TET, STR, KAN, CLO, SXT | erm(B), ant(6)-Ia, aph(3’)-IIIa | esp, hyl | 17 (17)d |
AMP: ampicilina; CIP: ciprofloxacina; CLO: cloranfenicol; ERI: eritromicina; GEN: gentamicina; KAN: kanamicina; STR: estreptomicina; SXT: trimetoprima-sulfametoxazol; TET: tetraciclina.
Todas las cepas mostraron resistencia a vancomicina con valores de concentración mínima inhibitoria (CMI) de entre 16-> 256mg/l, y todas, excepto una (CMI = 3mg/l), presentaron también resistencia a teicoplanina con CMI de 16-> 64mg/l. Se estudió el mecanismo de resistencia a vancomicina, resultando 38 cepas portadoras del gen vanA. En una cepa se detectó el gen vanB (con el alelo vanB2). Todas las cepas mostraron corresistencia a ampicilina, eritromicina, cloranfenicol, kanamicina (RAN) y trimetoprima-sulfametoxazol, 36 cepas presentaron además resistencia a estreptomicina (RAN), 28 a gentamicina (RAN), 22 a ciprofloxacina y 4 cepas a tetraciclina. Se encontraron asimismo los genes de resistencia asociados (número de cepas): erm(B) (39), ant(6)-Ia (38), aac[6’]-Ie-aph[2”]-Ia (36), aph(3’)-IIIa (12), y tet(M) (4); ninguna de las cepas resultó positiva para el gen catA (tabla 1). Todas las cepas fueron sensibles a linezolid (CMI ≤ 2mg/l), tigeciclina (CMI ≤ 0,125mg/l) y daptomicina (CMI ≤ 4mg/l).
La presencia de los genes esp y hyl fue también estudiada y se encontró que 25 cepas poseían ambos genes, 11 solo el gen esp, 2 hyl y una de ellas no presentó ninguno de los genes de virulencia indicados (tabla 1).
Caracterización clonalLa tipificación de las cepas por PFGE reveló la presencia de 13 patrones electroforéticos diferentes (A-M). Entre ellos se detectó un clon mayoritario en el que se agrupaban 13 cepas con pulsotipos iguales o estrechamente relacionados (A-A4) que fueron obtenidas en el período de agosto de 2005 a enero de 2007. Otros clones agrupaban también varias cepas: 6 cepas con el mismo patrón (G), 5 cepas con patrones relacionados (F-F4) y 4 cepas con patrones relacionados (H-H2) (tabla 1). Las 2 cepas aisladas del mismo paciente presentaron patrones diferentes, F1 e I.
Se analizó la secuencia tipo de 24 cepas del estudio mediante MLST, correspondientes a 13 pulsotipos diferentes y a 11 pulsotipos estrechamente relacionados; hay que señalar que las secuencias tipo de 18 de estas cepas fueron caracterizadas en un estudio previo10. Dos cepas, C851 y C859, mostraron las secuencias tipo ST9 (correspondiente al pulsotipo B, sensible a ciprofloxacino y carente de esp y hyl) y ST134 (pulsotipo D), respectivamente, englobadas en el complejo clonal CC9. El resto de secuencias tipo encontradas estaban englobadas dentro del complejo CC17, siendo la secuencia ST78 la más frecuente, encontrada en 18 cepas agrupadas en 7 pulsotipos (A, F, G, H, I, J y K), además de ST17, detectada en 2 cepas (pulsotipos L y M), y ST16 y ST18, encontradas en una cepa cada una de los pulsotipos C y E, respectivamente. Es destacable que la única cepa con genotipo vanB2, C865, presentó la secuencia ST17, pero con una deleción de 9 nucleótidos (3 aminoácidos) en el alelo ddl.
DiscusiónEn general, la prevalencia de ERV en los hospitales españoles es baja3, acorde con los resultados obtenidos también en nuestro hospital, en el que el 2% de los enterococos obtenidos en el período analizado presentaron resistencia adquirida a vancomicina, y todos ellos pertenecían a la especie E. faecium (representando un 9% del total de E. faecium en este período). Es interesante destacar que en los años siguientes (2008-2011) solamente se obtuvieron 2 cepas ERV en dicho hospital (de la especie E. faecium) representado < 0,5% del total de los enterococos aislados.
En relación con el período de estudio (2004-2007), se ha detectado la presencia de una serie de clones, indicando posibles situaciones epidémicas, que se aíslan en presencia de otras cepas ERV no relacionadas clonalmente. Estos resultados concuerdan con lo referido por otros autores, que señalan la presencia de clones mayoritarios junto a la presencia de clones minoritarios en otros hospitales europeos4,13. Algunos autores postulan que se observa un cambio en la epidemiología de ERV en Europa, aumentando la aparición de brotes epidémicos, cuando anteriormente se trataba de un problema mayormente de origen comunitario4. En España se han descrito varios brotes hospitalarios, causados tanto por ERV con genotipo vanA14–18, como con genotipo vanB27,19–21. Analizando las especies de enterococo implicadas en estos brotes, en general, aquellos causados por cepas vanA están más asociados a la especie E. faecalis, mientras que los causados por cepas vanB2 están asociados mayoritariamente a la especie E. faecium. En nuestro estudio, sin embargo, la mayoría de las cepas fueron E. faecium con genotipo vanA. Las cepas de E. faecium analizadas eran mayoritariamente del complejo clonal CC17, similar a las cepas implicadas en brotes descritos tanto en España como en otros países7,15,19,20. Únicamente 2 cepas resultaron adscritas al complejo CC9, más vinculado al origen aviar22, destacando que una de ellas fue la primera cepa ERV aislada en el estudio en 2004. Posteriormente se observó una expansión de la línea genética ST78, incluida en el CC17, relacionada con distintos perfiles de PFGE. Las cepas pertenecientes al complejo clonal CC17 poseen una especial capacidad de adaptación al ambiente hospitalario. Se ha reportado la capacidad de esta línea genética para colonizar diferentes ambientes hospitalarios y es posible que cepas de este linaje previamente sensibles a vancomicina se hayan hecho dominantes y posteriormente hayan adquirido la resistencia a vancomicina por transferencia horizontal de los determinantes de resistencia, bien de otros microorganismos anaerobios portadores de estos determinantes de resistencia23, convirtiéndose en la especie predominante en este ambiente, como puede observarse en este estudio. El hecho de que estas cepas CC17 estén adaptadas al ambiente hospitalario hace más difícil su erradicación4 poniendo de manifiesto la importancia del control rápido y efectivo de este tipo de bacterias resistentes para evitar la transmisión de la resistencia a glucopéptidos a otros microorganismos de gran relevancia clínica, como Staphylococcus aureus multirresistentes.
La mayoría de las cepas presentaron un fenotipo de multirresistencia. Todas las cepas mostraron resistencia a ampicilina y ciprofloxacina (excepto 2), resistencias comúnmente detectadas en el linaje CC17. Estas resistencias podrían estar relacionadas con mutaciones en las proteínas reparadoras del DNA, MutS y MutL, especialmente detectadas en cepas del complejo CC17, que han sido relacionadas con fenotipos hipermutadores, pudiendo facilitar la adquisición de dichas resistencias24. Todas las cepas eran, asimismo, resistentes a eritromicina y cloranfenicol, evidenciando la posible asociación entre los genes de resistencia a vancomicina, principalmente vanA, y los de resistencia a eritromicina y cloranfenicol. Este asociación ha sido descrita entre los genes vanA y erm(B)25, aunque no puede descartarse la presencia de otras relaciones teniendo en cuenta que son genes comúnmente localizados en plásmidos. De este modo, la selección de uno de estos tipos de resistencia puede conllevar la selección de cepas multirresistentes.
La detección de los genes de virulencia esp e hyl en casi todas nuestras cepas (con la excepción de una que era CC9) pone de nuevo de manifiesto el enriquecimiento en estos genes de la línea genética CC17, especialmente en esp, como ya ha sido comunicado previamente8. La mayoría de las cepas poseía ambos genes, sin embargo aquellas pertenecientes al clon A albergaban únicamente esp, siendo este clon además cronológicamente es el más antiguo entre los clones que se agrupan en ST78. Este hecho podría justificarse con la adquisición posterior de hyl que pondría de relevancia la capacidad de estas cepas para acumular determinantes que facilitan su persistencia.
Sería interesante continuar con los estudios de vigilancia de cepas ERV en el hospital analizado para valorar su evolución futura tanto en lo relativo a la prevalencia como a los genotipos o clones circulantes, con el objetivo de detectar posibles cambios epidemiológicos que puedan ocurrir y tomar las medidas de control pertinentes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
M. López disfruta de una beca (Formación de Personal Investigador) de la Comunidad Autónoma de La Rioja. La investigación de C. Torres está financiada por el proyecto SAF2009-08570 del Ministerio de Ciencia e Innovación.