El diagnóstico de las micosis invasoras es un reto difícil por la baja sensibilidad de los métodos tradicionales y conlleva retrasos diagnósticos y terapéuticos. Los objetivos de este trabajo de revisión han sido resumir el estado actual de las técnicas de diagnóstico molecular de las enfermedades fúngicas invasoras y aclarar el papel real que desempeñan en la práctica clínica. Los métodos microbiológicos convencionales pueden ser complementados con técnicas moleculares que permitan una identificación más rápida y certera de los aislamientos clínicos. La detección de biomarcadores (β-glucano, galactomanano) es útil en pacientes inmunodeficientes y son criterios diagnósticos para la EORTC/MSG. La detección de ácidos nucleicos es todavía una herramienta diagnóstica complementaria, útil en el diagnóstico de las micosis por mohos. Finalmente, la detección combinada de biomarcadores puede mejorar el diagnóstico pero su aplicabilidad no es tan sencilla y requiere de estudios adicionales que permitan valorar adecuadamente su validez.
Diagnosis of invasive mycoses is a difficult challenge due to the limitations and low sensitivity of traditional microbiology methods which lead to diagnostic and therapeutic delays. The aim of this review is to summarise the state of the art of the molecular diagnosis of invasive fungal disease and to clarify its current role in the clinical practice. Conventional microbiological methods could be complemented with molecular methods in the rapid and definitive identification of fungal isolates. Biomarkers (β-glucan, galactomannan) are very useful in immunocompromised patients and have been included as probable invasive mycoses by the EORTC/MSG. Nucleic acid detection is currently used as a complementary tool for diagnosis. However, PCR can be very useful in mould invasive mycoses. Finally, the combined detection using biomarkers can improve the diagnosis. However, their applicability in the microbiology laboratory is not so easy and further studies are required for the appropriate evaluation of its clinical usefulness.
El diagnóstico de las micosis invasoras continúa siendo un difícil reto, sobre todo por la escasa especificidad de las manifestaciones clínicas, la inexistencia de hallazgos radiológicos que sean patognomónicos y la baja sensibilidad y relativa lentitud de los métodos microbiológicos1–4. Las limitaciones inherentes y la escasa sensibilidad de los métodos de diagnóstico microbiológico tradicionales como la observación al microscopio de las muestras clínicas, su cultivo en medios artificiales y la posterior identificación de los hongos aislados en dichos medios mediante diversas técnicas, implican un retraso diagnóstico que muchas veces posterga la instauración del tratamiento antifúngico más apropiado para la enfermedad fúngica que padece el enfermo y además favorecen la aparición de daños orgánicos irreversibles. De hecho, diversos estudios asocian la alta morbimortalidad de las micosis invasoras con estos retrasos tanto diagnósticos como terapéuticos5–7.
Una de las posibles vías de solución de este problema diagnóstico es el desarrollo de técnicas microbiológicas que no necesiten basarse en el cultivo de aquellas muestras clínicas que se consideren significativas o que, si se basan en el cultivo y aislamiento del agente patógeno, reduzcan de una forma significativa el tiempo necesario para alcanzar la identificación de certeza de los aislamientos fúngicos obtenidos. Además, para que estos métodos diagnósticos sean eficientes desde el punto de vista clínico, sería importante que aportaran una información adicional valiosa sobre las características de estos aislamientos, como son aquellas propiedades relacionadas con la resistencia potencial a los fármacos antifúngicos, los factores de virulencia o patogenicidad o datos epidemiológicos que mejoren la comprensión de la patogenia y de la epidemiología de las micosis invasoras.
Los métodos diagnósticos independientes del cultivo, disponibles en la actualidad, tienen distintas bases científicas, dianas moleculares y técnicas de detección para alcanzar un diagnóstico fiable de la infección fúngica en el menor tiempo posible. Muchos de estos métodos detectan moléculas específicas e importantes de los hongos más frecuentemente implicados en patología humana y, sensu lato, podríamos definir como un método de diagnóstico molecular a aquel que permite detectar la presencia de estas moléculas fúngicas en las muestras clínicas. También incluiríamos en esta definición a aquellos métodos que detectan estas moléculas una vez que el hongo ha sido aislado en un medio de cultivo y que pueden facilitar una correcta identificación del género, especie o subespecie (genotipo, biotipo, variedad, etc.). En el primero de los casos, la detección molecular va a permitir un diagnóstico probablemente más rápido de la micosis invasora porque la detección de los componentes del hongo en muestras como la sangre, el suero, el lavado broncoalveolar o el líquido cefalorraquídeo se anticipará en la mayoría de los casos al binomio convencional cultivo-identificación. En la segunda de las posibilidades, los métodos moleculares facilitarían la identificación del agente etiológico aislado cuando los métodos morfológicos, bioquímicos o fisiológicos convencionales supongan un trabajo oneroso o no permitan obtener una identificación de la especie con un mínimo de certeza y rapidez.
Si consideramos este sentido amplio, podemos incluir dentro del diagnóstico molecular de las micosis invasoras a todos aquellos métodos que permiten detectar componentes fúngicos como el 1-3-β-D-glucano (BG), antígenos como el manano (MN), el galactomanano (GM) o el glucuroxilomanano (GXM), proteínas o ácidos nucleicos (AN). Algunos de estos biomarcadores, como el GXM o el GM, han sido validados en diferentes estudios clínicos para los diagnósticos de la criptococosis en pacientes inmunodeficientes y de la aspergilosis invasora en pacientes oncohematológicos, respectivamente2,8–12. Debemos tener en cuenta que tanto el instrumental como la metodología necesarios para realizar la detección de GXM y GM están al alcance de la mayoría de los laboratorios clínicos. La detección de BG o proteínas y la amplificación y secuenciación de AN implica la utilización de equipos y métodos más complejos, pero que ya están disponibles en muchos laboratorios de hospitales terciarios y en centros de referencia. Sin embargo, debemos precisar que la detección de AN o proteínas se encuentra en el terreno etéreo de la investigación clínica y que aunque los resultados son muy prometedores, todavía se debe recorrer un importante camino para alcanzar una correcta estandarización de la metodología empleada13–16.
Se han publicado recientemente varios documentos y guías de consenso con recomendaciones sobre el diagnóstico y tratamiento de las micosis invasoras2,17–20 y, en concreto, Ayats et al8 han publicado una actualizada revisión sobre el diagnóstico de la enfermedad fúngica invasora y propuesto una serie de recomendaciones de gran utilidad. Debido a esto, en esta revisión vamos a intentar contestar a la pregunta planteada en el título sobre qué diagnóstico de laboratorio es el más adecuado en el paciente con sospecha de padecer una infección fúngica invasora, si el diagnóstico convencional o el molecular, o si en el momento actual ambos son necesarios y complementarios. Para alcanzar este objetivo, realizaremos una somera revisión de los métodos existentes resaltando sus puntos fuertes y débiles, cuáles son las intersecciones y cruces de caminos entre diagnóstico convencional y molecular, y qué hechos pueden permitir un refuerzo mutuo que redunde en una mejora del diagnóstico de las micosis invasoras.
Diagnóstico micológico convencionalEntre los métodos de diagnóstico micológico considerados como convencionales podemos destacar el estudio macro y microscópico de las muestras clínicas, su cultivo en medios artificiales para poder aislar al agente causal de la micosis o la evaluación de la respuesta inmune del enfermo frente a este, principalmente mediante la detección de anticuerpos frente a diferentes antígenos fúngicos específicos3,21.
El estudio microscópico de los tejidos o de las muestras de citología puede permitir observar estructuras características de los hongos o la respuesta inflamatoria del enfermo frente a estos patógenos. Este estudio anatomopatológico realizado por un experto sigue siendo uno de los pilares diagnósticos básicos para poder confirmar una micosis invasora. Sin embargo, tiene muchas limitaciones tanto por la necesidad de que lo realicen personas con gran experiencia en la anatomía patológica de las infecciones fúngicas, lo que no siempre es posible, como porque la observación de estas estructuras características de los hongos solo es posible cuando son abundantes y esto ocurre de forma más habitual en los estadios avanzados de la infección, cuando los daños orgánicos son importantes y, muchas veces, irreversibles. La realización del estudio anatomopatológico no debe impedir que se realice un cultivo de la muestra porque el aislamiento es indispensable por ahora para poder identificar correctamente al hongo patógeno. Estas técnicas microscópicas también son útiles en los estudios necrópsicos para confirmar la etiología fúngica de la enfermedad que ha causado el fallecimiento del paciente. Debemos precisar que la labor del microbiólogo es muy importante en este diagnóstico basado en la microscopía. En concreto, la observación de muestras cutáneas, respiratorias u obtenidas por aspiración de diferentes lesiones, tratadas con KOH o teñidas con coloraciones microbiológicas, como la de azul de metileno, azul de toluidina, gram o metenamina de plata, o con calcoflúor o anticuerpos marcados con fluoresceína, pueden permitir un diagnóstico presuntivo temprano de algunas micosis invasoras, como aspergilosis, candidiasis, fusariosis, mucormicosis o neumocistosis 3,8,22–26.
En la sensibilidad del diagnóstico mediante estudio microscópico influyen diferentes aspectos técnicos, como los aumentos empleados en la observación, las tinciones aplicadas a la muestra o la necesaria evaluación de un número elevado de campos para que no pasen desapercibidos los hongos cuando su concentración es baja. Es importante tener en cuenta que no siempre las estructuras fúngicas van a tener unas características tan específicas, que permitan la identificación del hongo patógeno, y que esta identificación será con frecuencia solo la del género fúngico porque es difícil, muchas veces imposible, establecer la identificación de la especie fúngica concreta que está provocando las lesiones en los tejidos24,25. Esta detección se ve facilitada con el empleo de tinciones como la tinción con hematoxilina-eosina y PAS que permiten evaluar la respuesta inflamatoria o la tinción de metenamina de plata (Gomori-Grocott) que, como las anteriores, resalta las formas celulares fúngicas y permiten determinar si el hongo implicado es unicelular o levaduriforme (como Candida) o filamentoso (como Fusarium), si la hifa es regular y tabicada (como en Scedosporium) o deforme y sifonada (como en los mucorales), si el micelio es hialino (como en Aspergillus) o dematiáceo (como en Alternaria). Como no es posible obtener con certeza la identidad del hongo, se están desarrollando métodos de inmuno-histoquímica, hibridación in situ o amplificación de ácido nucleicos en las muestras de tejidos que serían una combinación eficaz de métodos convencionales y moleculares 27–30. Hay una serie de trabajos recientes que pueden permitir al lector interesado profundizar en estos métodos pero debemos tener en cuenta que no son técnicas estandarizadas y la experiencia se reduce a pequeñas series de casos o a casos anecdóticos27,29,31. Sin embargo, el interés por desarrollar un diagnóstico anatomopatológico molecular rápido y específico ha estimulado la creación de grupos internacionales, como el Fungal Infection Study Group de la European Society of Clinical Microbiology and Infectious Diseases, con el objetivo de consensuar técnicas y criterios que permitan una mejora en su fiabilidad28.
El cultivo de las muestras clínicas en medios microbiológicos apropiados es necesario para el diagnóstico etiológico porque permite el aislamiento del agente causante de la micosis, su identificación y la realización de estudios posteriores que permitan determinar su sensibilidad in vitro a los antifúngicos, la presencia de determinados factores de virulencia o su tipificación epidemiológica32–40. La mayoría de los hongos crecen bien en medios de cultivos estándares, como el agar sangre o el chocolate, pero los medios de cultivo específicos para hongos como el agar glucosado de Sabouraud, el agar patata o los medios cromógenos diferenciales, facilitan el crecimiento fúngico8,22,41. En muchas ocasiones, es necesaria la adición al medio de cultivo de fármacos antibacterianos, como cloranfenicol o gentamicina, que inhiban el crecimiento bacteriano, y permitir así un crecimiento fúngico más abundante. Los medios cromógenos han supuesto un importante avance en el diagnóstico de las candidiasis ya que muchos de ellos permiten la identificación presuntiva, con un elevado índice de certeza, de Candida albicans, Candida dubliniensis, Candida glabrata, Candida krusei y Candida tropicalis, en base al color de las colonias que se han desarrollado en el medio, lo que facilita la elección del fármaco antifúngico más adecuado para el tratamiento42,43.
La automatización de los hemocultivos ha permitido acortar el tiempo de diagnóstico en aquellas micosis invasoras, como la candidiasis, en las que la fungemia es relativamente frecuente. Sin embargo, debemos precisar que los hemocultivos con crecimiento de Candida siguen estando cercanos al 50% de los casos de candidiasis invasora, cifra claramente mejorable44–46. Otras micosis invasoras, como fusariosis y escedosporiasis, pueden cursar con episodios de fungemia y los hemocultivos permitir el aislamiento del hongo, pero en la mayoría de las micosis invasoras causadas por mohos el rendimiento de estos cultivos es muy pobre22,45,47,48. El aislamiento de hongos en medios de cultivo en los que se han sembrado muestras clínicas que no son habitualmente estériles, como las muestras respiratorias, debe valorarse con cautela y, en estos casos, todavía tiene una mayor relevancia realizar una correcta evaluación de la situación clínica del paciente. El valor de un aislamiento clínico, aunque pueda ser el de un hongo contaminante habitual, es muchísimo mayor si el paciente reúne las condiciones clínicas necesarias para ser considerado de alto riesgo de padecer una infección fúngica invasora.
La identificación del hongo aislado se va a basar en la morfología macroscópica de las colonias y en la microscópica del hongo y sus propiedades bioquímicas, fisiológicas e inmunológicas8. La identificación en pocos minutos de cuatro especies importantes de Candida, como C. albicans, C. dubliniensis, C. glabrata y C. krusei, se puede realizar con pruebas de aglutinación de partículas látex49–51 o de asimilación de trehalosa52. La amplificación y secuenciación de ácidos nucleicos fúngicos de los aislamientos clínicos o la identificación por espectrometría de masas (MALDI-TOF o Matrix-Assisted Laser Desorption/Ionization-Time-Of-Flight) es cada vez más asequible, tanto por su rapidez como por sus costes13,38,53–60. Ferroni et al14 han estudiado la utilidad del MALDI-TOF para identificar a los agentes patógenos en los hemocultivos con resultados interesantes y rápidos (2h). Estas técnicas están desplazando en muchos laboratorios a los métodos tradicionales de identificación y esta identificación molecular es todavía más importante para aquellos hongos cuya identificación por métodos tradicionales es poco fiable, demasiado laboriosa o que únicamente puede ser realizada en laboratorios de referencia por un número reducido de micólogos expertos36.
Existen varias técnicas aún no completamente estandarizadas que permiten las identificaciones mediante amplificación de ADN de patógenos comunes de los géneros Aspergillus y Candida61. Sin embargo, son más útiles las desarrolladas para la identificación de aquellas especies menos comunes de hongos filamentosos o de las especies, denominadas crípticas, hasta ahora clasificadas dentro de especies-grupo como Aspergillus fumigatus, C. albicans, C. glabrata, Candida parapsilosis, Scedosporium apiospermum o Sporothrix schenkii33,40,59,62–66, que pueden presentar grandes diferencias en sus perfiles de sensibilidad-resistencia a los fármacos antifúngicos o de evolución clínica de las micosis que causan35,38,55,60,67–69. En algunos trabajos se ha detectado ADN de Candida en botellas de hemocultivo con una PCR multiplex en tándem con posterior identificación de la especie70–72. Otros métodos basados en técnicas de hibridación in situ con sondas fluorescentes PNA-FISH detectan la presencia de C. albicans o C. parapsilosis, C. glabrata o C. krusei y C. tropicalis en los frascos de hemocultivos, en los tejidos de biopsia o permiten su identificación a partir de los medios de subcultivo30,56,58,73,74. Esta identificación rápida y certera es importante porque un error en la identificación puede llevar al empleo de un tratamiento inadecuado puesto que, en ocasiones, varias especies de un mismo género pueden tener muy diferente sensibilidad a los fármacos antifúngicos.
La detección de anticuerpos tiene una utilidad limitada en el diagnóstico de las micosis invasoras. Se ha considerado que la producción de anticuerpos está disminuida en pacientes inmunodeficientes, lo que reduce la utilidad diagnóstica de su detección. Sin embargo, varios estudios han mostrado que este no es un hecho generalizado y que es posible detectar mediante enzimoinmunoensayo e inmunofluorescencia anticuerpos antimicelio de C. albicans (CAGTA) en pacientes oncohematológicos con un importante potencial diagnóstico y pronóstico en la candidiasis invasora75–78. La segunda fase de un estudio multicéntrico está evaluando la utilidad diagnóstica y el valor pronóstico de la detección de CAGTA mediante la prueba Candida albicans IFA IgG (Laboratorios Vircell, España), en pacientes críticos ingresados en UCI españolas79–81. Sin embargo, los mejores resultados se han obtenido combinando la detección de anticuerpos con otras técnicas de detección de MN o BG82,83.
Diagnóstico molecularEl diagnóstico molecular tiene la gran ventaja de que no necesita basarse en el cultivo del microorganismo y la detección de biomarcadores fúngicos ofrece grandes esperanzas a corto plazo 8,83. Este hecho a priori permite un diagnóstico más rápido por no ser necesaria la espera de 24-48h (en ocasiones semanas) antes de obtener colonias del agente patógeno en los medios de cultivo. Sin embargo, este diagnóstico debe aspirar a informar sobre las características de los hongos aislados en la misma proporción, detalle e importancia que lo hace el diagnóstico convencional, lo que no es tan sencillo.
Uno de los primeros métodos moleculares que demostró una gran utilidad y eficacia desde su comercialización en los años 1970 ha sido la detección de GXM, un antígeno capsular de Cryptococcus neoformans. Diferentes estudios y la práctica clínica en miles de laboratorios de microbiología clínica han mostrado la gran utilidad diagnóstica y pronóstica de detectar GXM mediante una prueba de aglutinación de látex o por enzimoinmunoensayo (ELISA), en líquido cefalorraquídeo o en suero de pacientes infectados por el VIH (evidencia científica AI)8 o pacientes oncohematológicos con criptococosis meníngea y diseminada (evidencia científica AII)12.
Otros métodos desarrollados posteriormente para la detección de MN de Candida o de GM de Aspergillus no han permitido alcanzar unos logros diagnósticos similares y deben utilizarse con más cautela en los laboratorios de microbiología clínica. Dos trabajos recientes revisan en profundidad la contribución de los biomarcadores serológicos, como la detección de MN o de anticuerpos anti-Candida, en el diagnóstico de la candidiasis invasora 83,84.
El GM es un componente de la pared celular de Aspergillus, que se puede detectar mediante un ELISA con el anticuerpo monoclonal EBA-2 (Platelia Aspergillus, Bio-Rad, Francia) en el suero, lavado broncoalveolar, biopsias, orina o líquidos cefalorraquídeo, pericárdico y pleural de los enfermos con aspergilosis invasora3,8. En las tablas 1 y 2 se muestra un resumen de la metodología, indicaciones y limitaciones diagnósticas de la detección de GM. Dos metaanálisis han mostrado que la sensibilidad media de la prueba está alrededor del 70% y la especificidad media está cercana al 90%85,86. Su mayor utilidad diagnóstica se ha descrito en los pacientes oncohematológicos con alto riesgo de sufrir una aspergilosis invasora. En los pacientes con neutropenia prolongada, después de la quimioterapia o de la recepción de un trasplante alogénico de precursores hematopoyéticos, la sensibilidad de la prueba es mayor del 85% y la especificidad se sitúa por encima del 95%, con valores predictivos de la prueba positiva (VPP) y negativa (VPN) superiores al 85 y 95%, respectivamente9,82,87. En este grupo de pacientes, el GM puede ser detectado antes de que las manifestaciones clínicas y radiológicas aparezcan, potenciando su valor como biomarcador que facilitaría el comienzo más temprano del tratamiento antifúngico dirigido contra Aspergillus. La concentración de GM en suero es un reflejo de la carga fúngica en los tejidos y su disminución, mantenimiento o incremento, podrían orientar sobre la evolución de la infección y la respuesta al tratamiento empleado87,88. La detección de GM en el lavado broncoalveolar de pacientes oncohematológicos con neutropenia presenta también un valor diagnóstico alto (sensibilidad alrededor del 90%) y se han descrito valores aceptables en pacientes críticos inmunodeprimidos y en receptores de trasplante de pulmón89,90. Por el contrario, los valores diagnósticos de la detección de GM en suero, lavado broncoalveolar u otras muestras clínicas representativas han sido peores en pacientes sin neutropenia, críticos o receptores de trasplante de hígado u otros órganos sólidos (sensibilidad≤50%), en los que se observa una menor angioinvasión por Aspergillus91. En pacientes pediátricos, la detección de GM en suero ha mostrado valores diagnósticos similares a los encontrados en adultos26, con valores VPP y VPN del 54 y 83%, respectivamente, en pacientes oncológicos y del 70 y 92%, respectivamente, en receptores de trasplante de progenitores hematopoyéticos. La detección de GM se ha incluido como criterio micológico de aspergilosis invasora probable en las definiciones de consenso de la EORTC/MSG (European Organization for Research and Treatment of Cancer/Mycoses Study Group del National Institute of Allergy and Infectious Diseases)2,12.
Evidencia científica de las recomendaciones diagnósticas8
| Categoría de la evidencia | |
| A | Técnica diagnóstica útil o fiable, que permite recomendar su uso en todos los casos. |
| B | Técnica diagnóstica con una utilidad moderada que puede ser recomendable emplear en determinadas ocasiones. |
| C | Técnica diagnóstica cuya utilidad no ha sido demostrada. |
| Calidad de la evidencia | |
| I | Evidencia obtenida en por lo menos un estudio multicéntrico, con un diseño correcto, de casos y controles o de cohortes. |
| II | Evidencia obtenida en por lo menos un estudio de casos y controles o de cohortes realizado en un solo centro, de múltiples series o de resultados relevantes de series no controladas. |
| III | Evidencia obtenida de opiniones de expertos, basadas en la experiencia clínica, casos anecdóticos o estudios descriptivos |
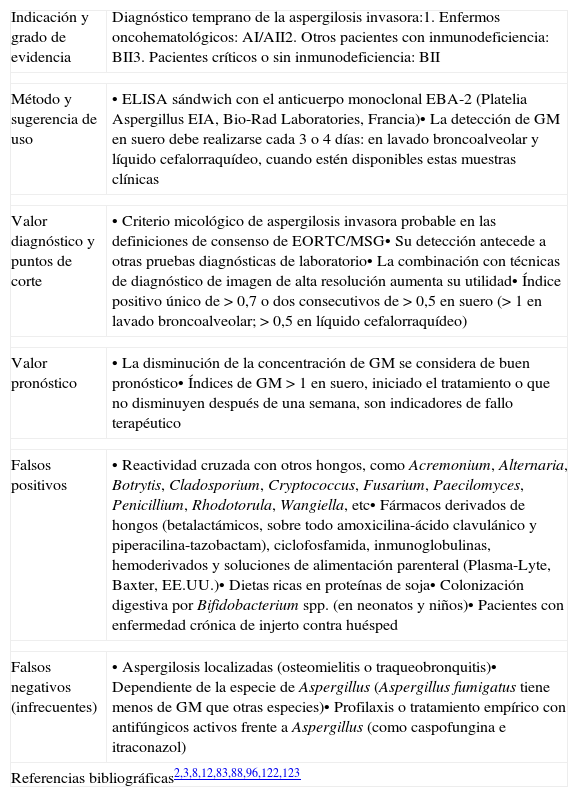
Indicaciones y limitaciones diagnósticas de la detección de galactomanano
| Indicación y grado de evidencia | Diagnóstico temprano de la aspergilosis invasora:1. Enfermos oncohematológicos: AI/AII2. Otros pacientes con inmunodeficiencia: BII3. Pacientes críticos o sin inmunodeficiencia: BII |
| Método y sugerencia de uso | • ELISA sándwich con el anticuerpo monoclonal EBA-2 (Platelia Aspergillus EIA, Bio-Rad Laboratories, Francia)• La detección de GM en suero debe realizarse cada 3 o 4 días: en lavado broncoalveolar y líquido cefalorraquídeo, cuando estén disponibles estas muestras clínicas |
| Valor diagnóstico y puntos de corte | • Criterio micológico de aspergilosis invasora probable en las definiciones de consenso de EORTC/MSG• Su detección antecede a otras pruebas diagnósticas de laboratorio• La combinación con técnicas de diagnóstico de imagen de alta resolución aumenta su utilidad• Índice positivo único de>0,7 o dos consecutivos de>0,5 en suero (>1 en lavado broncoalveolar;>0,5 en líquido cefalorraquídeo) |
| Valor pronóstico | • La disminución de la concentración de GM se considera de buen pronóstico• Índices de GM>1 en suero, iniciado el tratamiento o que no disminuyen después de una semana, son indicadores de fallo terapéutico |
| Falsos positivos | • Reactividad cruzada con otros hongos, como Acremonium, Alternaria, Botrytis, Cladosporium, Cryptococcus, Fusarium, Paecilomyces, Penicillium, Rhodotorula, Wangiella, etc• Fármacos derivados de hongos (betalactámicos, sobre todo amoxicilina-ácido clavulánico y piperacilina-tazobactam), ciclofosfamida, inmunoglobulinas, hemoderivados y soluciones de alimentación parenteral (Plasma-Lyte, Baxter, EE.UU.)• Dietas ricas en proteínas de soja• Colonización digestiva por Bifidobacterium spp. (en neonatos y niños)• Pacientes con enfermedad crónica de injerto contra huésped |
| Falsos negativos (infrecuentes) | • Aspergilosis localizadas (osteomielitis o traqueobronquitis)• Dependiente de la especie de Aspergillus (Aspergillus fumigatus tiene menos de GM que otras especies)• Profilaxis o tratamiento empírico con antifúngicos activos frente a Aspergillus (como caspofungina e itraconazol) |
| Referencias bibliográficas2,3,8,12,83,88,96,122,123 | |
EORTC/MSG: European Organization for Research and Treatment of Cancer/Mycoses Study Group del National Institute of Allergy and Infectious Diseases; GM: galactomanano.
El BG es un componente de la pared celular de la mayoría de los hongos. Se libera durante el desarrollo de la infección y puede detectarse en el suero de los pacientes, comportándose como un biomarcador panfúngico que no es específico de ninguna micosis invasora concreta. Su utilidad es mayor en aspergilosis, candidiasis y neumocistosis, y mínima en el diagnóstico de criptococosis y mucormicosis: Cryptococcus y mucorales liberan in vitro cantidades muy bajas de BG (<200pg/ml), mientras que Candida y Aspergillus liberan una media de 2.119 y 1.915pg/ml, respectivamente8. En la tabla 3 se muestra un resumen de la metodología, indicaciones y limitaciones diagnósticas.
Indicaciones y limitaciones diagnósticas de la detección de 1-3-β-D-glucano
| Indicación y grado de evidencia | Diagnóstico temprano de micosis invasora (biomarcador panfúngico):1. Aspergilosis y candidiasis invasoras: AI-BII2. Neumocistosis: BII3. Criptococosis y mucormicosis: BIII |
| Métodos y sugerencia de uso | • Fungitell (Associates of Cape Cod Inc., EE.UU.)• Wako WB003 (Wako Pure Chemical Industries, Japón)• Fungitec G (Seikagaku Kogyo Corporation, Japón)• B-G Star (Maruha Corporation, Japón)• La detección de BG en suero debería realizarse cada 3 o 4 días |
| Valor diagnóstico y puntos de corte (micosis invasora probable) | • Criterio micológico de micosis invasora probable en las definiciones de consenso de EORTC/MSG• Su detección antecede a otras pruebas diagnósticas de laboratorio• La combinación con la detección de GM o CAGTA aumenta su valor diagnóstico en aspergilosis y candidiasis invasoras, respectivamente• >60-80pg/ml (Fungitell) o 7pg/ml (Wako) en suero |
| Valor pronóstico | • La disminución de la concentración de BG se considera de buen pronóstico |
| Falsos positivos | • Contaminación de los materiales de laboratorio• Bacteriemia por cocos grampositivos (Streptococcus) y bacilos gramnegativos (Alcaligenes y Pseudomonas aeruginosa)• Contacto con gasas y esponjas durante procesos quirúrgicos• Hemodiálisis con membranas o filtros de acetato de celulosa• Tratamiento intravenoso con albúmina, inmunoglobulinas, factores de coagulación y/o proteínas plasmáticas• Algunos fármacos antineoplásicos (lentinano y polisacárido K) y antibacterianos (amoxicilina-ácido clavulánico y piperacilina-tazobactam) |
| Falsos negativos (infrecuentes) | • Sueros hiperpigmentados (bilirrubina y triglicéridos elevados)• Profilaxis y tratamiento empírico con antifúngicos• Azitromicina y pentamidina intravenosas |
| Referencias bibliográficas2,3,8,12,82,83,88,92,93,96 | |
BG: 1-3-β-D-glucano; CAGTA: anticuerpos antimicelio de Candida albicans; EORTC/MSG: European Organization for Research and Treatment of Cancer/Mycoses Study Group del National Institute of Allergy and Infectious Diseases; GM: galactomanano.
La detección de BG se realiza mediante una técnica muy sensible basada en la activación de la cascada de coagulación del cangrejo de herradura (Limulus polyphemus y otras especies). La interpretación de los resultados deben realizarla profesionales con experiencia en la prueba debido a su complejidad. Existen varias pruebas comercializadas que han demostrado una sensibilidad superior al 60% y una especificidad entre el 85 y el 100% en enfermos con neutropenia y micosis invasoras12,82,88,92,93. La prueba denominada Fungitell (Associates of Cape Cod, Inc., EE.UU.) es la más utilizada en Europa y EE.UU.82,88. Koo et al92 han realizado un estudio retrospectivo que incluía 871 pacientes con riesgo de sufrir una micosis invasora a los que se les realizaron 1.308 pruebas de detección de BG con el método Fungitell. Se diagnosticaron 228 casos de micosis invasora probada o probable y la sensibilidad y especificidad de la detección de BG (≥80pg/ml) fueron 64 y 84%, respectivamente. La sensibilidad de la prueba aumentaba cuando se realizaba de forma seriada. La sensibilidad de la detección de BG era mayor en la aspergilosis invasora que en la candidiasis invasora y permitía excluir la presencia de neumocistosis. En el 71,4% de los pacientes con neumonía por Pneumocystis jiroveci se encontraba una concentración alta de BG (>500pg/ml), que con frecuencia precedía en varios días al diagnóstico microbiológico convencional. El empleo de albúmina, inmunoglobulinas intravenosas y/o la realización de hemodiálisis con membranas de celulosa se han asociado con concentraciones más elevadas de BG. Sin embargo, el tratamiento antifúngico empírico no reducía la sensibilidad de la prueba ni siquiera en aquellos pacientes que habían recibido antifúngicos durante más de siete días3,8.
Un metaanálisis reciente de Karageorgopoulos et al94 ha comprobado la utilidad diagnóstica de la detección de BG en pacientes con micosis invasoras (exceptuando la neumocistosis) basándose en las definiciones de EORTC/MSG. Los 16 estudios seleccionados incluían 2.979 pacientes de los cuales 595 sufrían micosis invasoras probadas o probables. La sensibilidad y la especificidad diagnósticas de la detección de BG fueron 76,8 y 85,3%, respectivamente. La detección del BG también se ha incluido como criterio micológico de micosis invasora probable en las definiciones de consenso de la EORTC/MSG2. Sin embargo, el grado de evidencia para su recomendación en pacientes oncohematológicos por la tercera European Conference on Infections in Leukemia (ECIL3)12 ha sido menor, de utilidad moderada (BII), que para el GM (AII).
A pesar del avance que han supuesto las pruebas existentes para la detección de GM y BG en la práctica clínica, ambas tienen importantes limitaciones diagnósticas que han estimulado la búsqueda y desarrollo de pruebas alternativas, entre las que destacan las de amplificación y detección de AN. Entre las distintas técnicas existentes, la reacción en cadena de la polimerasa (PCR) genera grandes esperanzas por su capacidad de amplificar pequeñas cantidades de ADN fúngico (entre 1 y 10 fg) en las muestras clínicas hasta concentraciones detectables. Por otro lado, son técnica versátiles ya que permiten la detección e identificación de una especie fúngica en concreto, empleando para ello como objetivo secuencias de ADN específicas de dicha especie (PCR específica) o bien la detección de múltiples especies, por ejemplo en un cribado diagnóstico empleando como objetivo secuencias de regiones comunes del genoma presentes en hongos de diferentes géneros (PCR panfúngica). Aunque han sido descritas diversas regiones utilizadas como diana de amplificación, las regiones variables del ADN ribosómico, como los espaciadores transcriptores internos (internal transcribed spacers) ITS1 e ITS2, son las más utilizadas3,8,95,96.
El mayor problema de todas estas técnicas radica en la falta de estandarización de los métodos empleados. La mayoría de los estudios se han realizado con técnicas desarrolladas en el propio laboratorio que realizaba el estudio, pero cuya validación era escasa lo que condicionaba la reproducibilidad de las técnicas descritas cuando se realizaban en otros laboratorios. El principal objetivo que se persigue en la actualidad es la obtención de una prueba validada y estandarizada que permita el diagnóstico de la aspergilosis invasora, sin olvidar el de las candidiasis, criptococosis, neumocistosis y de las micosis emergentes causadas por hongos filamentosos, cuyo diagnóstico es aún más difícil3,96,97. Se están realizando importantes esfuerzos para estandarizar las condiciones diagnósticas de la detección de AN y, tal vez, uno de los proyectos de mayor alcance es la European Aspergillus PCR initiative bajo los auspicios de la International Society of Human and Animal Mycology (ISHAM) que intenta conseguir un amplio consenso metodológico98–101. Sin embargo, todavía no se han realizado estudios suficientes como para respaldar la utilidad clínica de estas técnicas, por lo que aún la detección de AN no ha sido incluida como biomarcador de micosis invasora en las definiciones de EORTC/MSG2 o de la ECIL312.
La estandarización de las técnicas de detección de AN tiene que alcanzar puntos muy diversos y críticos, como la definición de aquellas muestras clínicas más adecuadas (sangre, suero, lavado broncoalveolar, etc.), el volumen de las mismas, su conservación y la periodicidad de su obtención. Una vez definidos los parámetros estándar en la fase preanalítica, deben normalizarse y validarse los distintos métodos que se emplearán para las distintas muestras clínicas. Así, se ha de estandarizar y validar el método de extracción de ADN (o ARN) para cada una de las muestras previamente definidas como de utilidad clínica, empleando en la medida de lo posible métodos comerciales y siguiendo las especificaciones del fabricante; o si se realizan modificaciones de las mismas, ofrecer una descripción detallada de estos cambios. Por último, se deben definir todos aquellos parámetros que nos permitan cuantificar la cantidad de ADN o ARN obtenido en esta fase, así como su pureza para disponer de una medida cuantitativa y fiable que permita la comparación entre las diferentes técnicas de extracción disponibles102–104. Este paso es clave y condiciona los resultados obtenidos en las fases posteriores, ya que la mayor o menor cantidad de AN, así como su pureza, puede ser la causa de la obtención de resultados dispares aunque sea utilizado el mismo protocolo de amplificación de AN. Este hecho condicionará, en última estancia, los resultados falsos positivos y falsos negativos de la técnica y, en consecuencia, su sensibilidad, especificidad, VPP y VPN.
Para la validación de la fase de amplificación y detección de AN es necesaria la estandarización de múltiples pasos, cuya descripción detallada está por encima del objetivo de esta revisión. De forma somera se puede resaltar que la validación y estandarización de esta fase depende en primer lugar de si la técnica que vamos a realizar es cualitativa o cuantitativa. Los requisitos necesarios para la estandarización y validación son menores para las técnicas cualitativas. No obstante, hay puntos comunes en ambos procedimientos de validación que sí convienen comentar. En primer lugar, para conseguir una mayor eficiencia de la técnica de amplificación de AN se debe optimizar la concentración de magnesio, de polimerasa, de los cebadores o primers empleados en la reacción y las condiciones de amplificación104,105. En segundo lugar, los pasos iniciales de la validación y estandarización de la especificidad de la técnica obligan a la secuenciación del producto de PCR obtenido para verificar la detección del microorganismo o microorganismos, así como la obtención de resultados sistemáticamente negativos en al menos diez muestras control negativas y en otras tantas muestras control potencialmente reactivas con una cantidad elevada de ADN en las mismas. Habitualmente, estas muestras de control potencialmente reactivas incluyen microorganismos genéticamente relacionados con el microorganismo que es objetivo de estudio y en una cantidad superior a las 100.000 copias/ml.
Por último, los pasos iniciales de la validación y estandarización de la sensibilidad de la técnica incluyen la existencia de un material adecuado de referencia tanto en su pureza como en su cuantificación, a fin de determinar el límite de detección de la técnica mediante dilución sucesiva de una cantidad determinada de la muestra control en las diferentes matrices de las distintas muestras consideradas en la fase preanalítica. La verificación de la sensibilidad analítica se realizará en al menos diez muestras positivas y otras diez muestras positivas cuya carga fúngica esté alrededor del límite de detección de la técnica. En el caso de las técnicas de amplificación y detección de AN cuantitativas, es necesario verificar la linealidad de la técnica siguiendo los procedimientos estándar descritos. Además y si es posible, en todas estas pruebas de validación es deseable que haya la mayor representatividad de variaciones genómicas del microorganismo que se desea detectar102,104–106.
La última fase de validación y estandarización en las técnicas de amplificación y detección de AN es la fase de análisis de resultados. En ella se debe de definir en caso de emplear una técnica de PCR en tiempo real (PCRtr) el cálculo de valor del punto de corte (Ct o Cp -en inglés crossing point o crossing threshold-), que es aquel valor del tiempo en el que la fluorescencia registrada por el termociclador supera un umbral preestablecido por encima del cual se considera positivo el resultado de la PCR. Otro aspecto que debe incluirse en esta fase es el significado clínico exacto de un resultado positivo (infección, colonización o contaminación) ya que es importante para la valoración y la toma de decisión que el médico realice sobre el estado clínico del paciente, así como de la necesidad o no de instaurar un tratamiento antifúngico3,8,84,97.
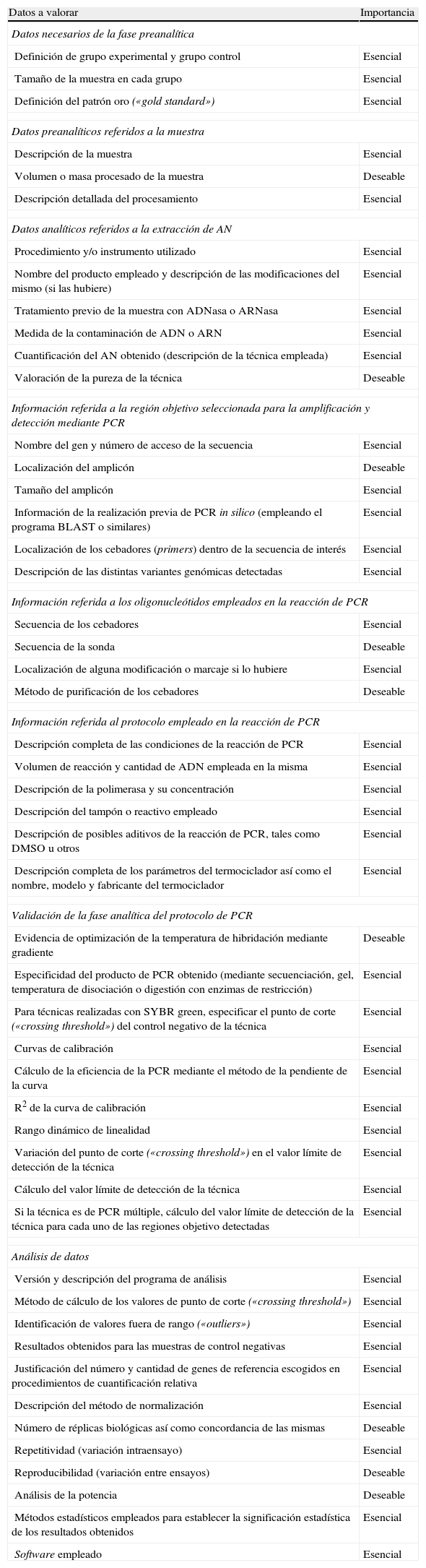
Una mejor comprensión del origen del ADN fúngico y su cinética en el transcurso de la invasión fúngica permitirá seleccionar las mejores muestras clínicas para el diagnóstico, mejores técnicas preanalíticas de procesamiento y el mejor protocolo de extracción de ADN con el objeto de obtener los mejores resultados de sensibilidad y especificidad posibles. Las recientes revisiones de Bretagne95,107,108 son de lectura recomendable para una adecuada comprensión de los múltiples factores que influyen sobre este proceso de estandarización y que deben tenerse en cuenta para evitar o limitar los resultados falsos positivos y falsos negativos y así mejorar la validez diagnóstica de la detección de ADN fúngico. De entre los factores considerados por Bretagne, merece la pena citar dos por sus implicaciones en la reducción de falsos positivos y negativos. Evitar la contaminación con productos amplificados previamente o con hongos ambientales (presencia de conidiosporas de hongos aerovagantes como Aspergillus) puede reducir los resultados falsos positivos; mientras que el uso de controles internos de amplificación puede limitar los falsos negativos. No obstante, el empleo de controles internos puede conllevar problemas adicionales, como la elevación del límite de detección de la técnica debido a un efecto competitivo entre el ADN de la región del microorganismo elegida como diana para la detección y el ADN del control interno. Por tanto, su elección es importante y debe hacerse cuidadosamente a fin de no interferir con la técnica y, sin embargo, beneficiarnos de su uso para la detección de resultados falsos negativos por inhibición de la reacción de PCR por distintas causas104,106. Las técnicas cuantitativas o PCRtr son probablemente las más adecuadas para lograr esta estandarización, ya que en los últimos años se han definido los requisitos mínimos de información para la publicación de experimentos empleando PCRtr que facilitan la comparación y la estandarización entre distintas técnicas de PCR (MIQE checklist–tabla 4-)15,108,109.
Requisitos mínimos de información para la publicación de experimentos empleando PCRtr (MIQE checklist)
| Datos a valorar | Importancia |
| Datos necesarios de la fase preanalítica | |
| Definición de grupo experimental y grupo control | Esencial |
| Tamaño de la muestra en cada grupo | Esencial |
| Definición del patrón oro («gold standard») | Esencial |
| Datos preanalíticos referidos a la muestra | |
| Descripción de la muestra | Esencial |
| Volumen o masa procesado de la muestra | Deseable |
| Descripción detallada del procesamiento | Esencial |
| Datos analíticos referidos a la extracción de AN | |
| Procedimiento y/o instrumento utilizado | Esencial |
| Nombre del producto empleado y descripción de las modificaciones del mismo (si las hubiere) | Esencial |
| Tratamiento previo de la muestra con ADNasa o ARNasa | Esencial |
| Medida de la contaminación de ADN o ARN | Esencial |
| Cuantificación del AN obtenido (descripción de la técnica empleada) | Esencial |
| Valoración de la pureza de la técnica | Deseable |
| Información referida a la región objetivo seleccionada para la amplificación y detección mediante PCR | |
| Nombre del gen y número de acceso de la secuencia | Esencial |
| Localización del amplicón | Deseable |
| Tamaño del amplicón | Esencial |
| Información de la realización previa de PCR in silico (empleando el programa BLAST o similares) | Esencial |
| Localización de los cebadores (primers) dentro de la secuencia de interés | Esencial |
| Descripción de las distintas variantes genómicas detectadas | Esencial |
| Información referida a los oligonucleótidos empleados en la reacción de PCR | |
| Secuencia de los cebadores | Esencial |
| Secuencia de la sonda | Deseable |
| Localización de alguna modificación o marcaje si lo hubiere | Esencial |
| Método de purificación de los cebadores | Deseable |
| Información referida al protocolo empleado en la reacción de PCR | |
| Descripción completa de las condiciones de la reacción de PCR | Esencial |
| Volumen de reacción y cantidad de ADN empleada en la misma | Esencial |
| Descripción de la polimerasa y su concentración | Esencial |
| Descripción del tampón o reactivo empleado | Esencial |
| Descripción de posibles aditivos de la reacción de PCR, tales como DMSO u otros | Esencial |
| Descripción completa de los parámetros del termociclador así como el nombre, modelo y fabricante del termociclador | Esencial |
| Validación de la fase analítica del protocolo de PCR | |
| Evidencia de optimización de la temperatura de hibridación mediante gradiente | Deseable |
| Especificidad del producto de PCR obtenido (mediante secuenciación, gel, temperatura de disociación o digestión con enzimas de restricción) | Esencial |
| Para técnicas realizadas con SYBR green, especificar el punto de corte («crossing threshold») del control negativo de la técnica | Esencial |
| Curvas de calibración | Esencial |
| Cálculo de la eficiencia de la PCR mediante el método de la pendiente de la curva | Esencial |
| R2 de la curva de calibración | Esencial |
| Rango dinámico de linealidad | Esencial |
| Variación del punto de corte («crossing threshold») en el valor límite de detección de la técnica | Esencial |
| Cálculo del valor límite de detección de la técnica | Esencial |
| Si la técnica es de PCR múltiple, cálculo del valor límite de detección de la técnica para cada uno de las regiones objetivo detectadas | Esencial |
| Análisis de datos | |
| Versión y descripción del programa de análisis | Esencial |
| Método de cálculo de los valores de punto de corte («crossing threshold») | Esencial |
| Identificación de valores fuera de rango («outliers») | Esencial |
| Resultados obtenidos para las muestras de control negativas | Esencial |
| Justificación del número y cantidad de genes de referencia escogidos en procedimientos de cuantificación relativa | Esencial |
| Descripción del método de normalización | Esencial |
| Número de réplicas biológicas así como concordancia de las mismas | Deseable |
| Repetitividad (variación intraensayo) | Esencial |
| Reproducibilidad (variación entre ensayos) | Deseable |
| Análisis de la potencia | Deseable |
| Métodos estadísticos empleados para establecer la significación estadística de los resultados obtenidos | Esencial |
| Software empleado | Esencial |
Modificado de15,107,109.
La mayoría de los estudios de detección de AN se han realizado con el fin de diagnosticar de manera temprana la aspergilosis invasora en pacientes inmunodeficientes. La falta de un método estandarizado causa resultados dispares en la literatura y en muchos estudios los casos de aspergilosis invasora probada o probable son pocos, lo que influye de forma importante en la sensibilidad y especificidad de la prueba y dificultan tanto la comparación de los estudios como la inferencia de conclusiones sólidas. Mengoli et al16 han realizado un metaanálisis que incluye 16 estudios realizados empleando diferentes técnicas y condiciones de PCR para detectar ADN de Aspergillus en sangre completa, suero y/o plasma de 1.618 pacientes. A pesar de la dificultad de realizar la comparación, los datos mostraban que un resultado positivo tenía una sensibilidad del 88% y una especificidad del 75% para el diagnóstico de la aspergilosis invasora. La obtención de dos o más resultados positivos incrementaba de forma significativa la utilidad diagnóstica; apoyando la realización de determinaciones seriadas como en los casos de la detección de BG y GM. Cuenca-Estrella et al96 también describen una sensibilidad para la aspergilosis invasora superior al 90% cuando se utiliza como criterio diagnóstico la obtención de dos pruebas de PCR consecutivas positivas.
Las muestras de sangre no han permitido obtener una sensibilidad adecuada probablemente por la escasa presencia de ADN de Aspergillus en la sangre o la presencia de inhibidores de la PCR, bien en la sangre del enfermo o en las sustancias empleadas para que esta no coagule o en la obtención del plasma y suero (heparina, hemoglobina, etc.)96,101,108. El aumento del volumen de muestra empleado hasta 1-3ml puede facilitar la detección de ADN fúngico; sin embargo, podría interferir en el resultado obtenido con las muestras de aquellos pacientes con leucocitosis elevada, inhibiendo la PCR por exceso de ADN en la reacción. Por otro lado, el uso de técnicas de extracción automatizada de AN reduciría la manipulación de la muestra al mínimo y, por tanto, las contaminaciones con ADN ambiental, decreciendo el porcentaje de falsos positivos. La utilización de PCRtr, que requiere menos manipulaciones y tiene un límite de detección menor que la técnica de PCR convencional, permitiría desechar los viejos protocolos de PCR anidada, que son una fuente frecuente de contaminación y de resultados falsos positivos y, por tanto, contribuiría a una mejora técnica substancial y a la disminución del número de falsos positivos. La utilización de otro tipo de muestras clínicas también ofrece resultados esperanzadores y hay varios estudios que muestran la eficacia diagnóstica de la utilización de lavados broncoalveolares o biopsias de parénquima pulmonar28–31,110,111. El problema principal que está asociado a la obtención de estas muestras es el estado clínico del paciente que en muchas ocasiones no permite realizar técnicas invasoras.
Hay varios estudios de detección de AN valorando su utilidad diagnóstica en candidiasis y neumocistosis y, en menor número, en mucormicosis y otras infecciones por hongos filamentosos. La mayoría de los estudios respaldan la utilidad diagnóstica de la detección de ADN por PCR, pero existe una importante variabilidad técnica que hace difícil la comparación de resultados. Avni et al11 han realizado una extensa revisión y un metaanálisis de la utilidad diagnóstica de la PCR en la candidiasis invasora. Han incluido 54 estudios en los que se empleaba la sangre como muestra clínica adecuada para detectar ADN de Candida y han evaluado la utilidad de esta detección en 4.694 pacientes (963 con candidiasis invasora probada o probable). La sensibilidad y la especificidad eran máximas (100%) cuando se comparaban personas sanas y personas con candidiasis invasora. Estos valores se mantenían elevados (95 y 92%, respectivamente) en pacientes con sospecha de candidiasis invasora y la especificidad se mantenía por encima del 90% cuando se analizaba con diferentes grupos control. Los mejores datos se obtenían cuando la muestra era sangre completa, la diana de amplificación los genes del ARNr o del P450, siendo el límite de detección de ADN≤10UFC/ml. Además, en los pacientes con candidiasis invasora probada o probable, la detección de ADN era positiva entre el 78 y el 91% (media, 85%) mientras que el crecimiento de Candida en hemocultivo variaba entre el 29 y el 46% (media, 38%).
La comercialización reciente con marcaje CE (Comunidad Europea) de varias pruebas basadas en la técnica de PCRtr, como LightCycler SeptiFast Test MGRADE (Roche Molecular Diagnostics, EE.UU.) y MycoAssay Aspergillus (Myconostica, Gran Bretaña), va a permitir un estudio en condiciones estandarizadas de su utilidad112–114. La prueba LightCycler SeptiFast detecta ADN de varios patógenos bacterianos y fúngicos, entre ellos A. fumigatus, C. albicans, C. glabrata, C. krusei C. tropicalis y C. parapsilosis mediante una PCRtr45,70,115. Se han publicado varios estudios sobre su utilidad en el diagnóstico de infecciones bacterianas y fúngicas en pacientes febriles con neutropenia y enfermos críticos con sospecha de padecer sepsis grave. Aunque los datos son todavía escasos y preliminares, orientan a que esta técnica puede ser complementaria del hemocultivo y de otros biomarcadores fúngicos116–119.
Una propuesta para mejorar el diagnóstico fúngico es la detección combinada de varios biomarcadores, como AN, BG, CAGTA, GM y MN. Esta combinación disminuiría el número de falsos positivos y mejoraría la especificidad. Varios estudios han mostrado una mejoría clara de la utilidad diagnóstica al disminuir tanto los resultados falsos positivos como negativos12,82,83,88,111,120,121. Sin embargo, son necesarios estudios multicéntricos que permitan contrastar las fortalezas y debilidades de la combinación de técnicas, algunas de ellas laboriosas y costosas.
ConclusionesEl diagnóstico de las micosis invasoras se debe basar en una visión global de la situación del enfermo valorando los datos clínicos, radiológicos y las diferentes pruebas de diagnóstico microbiológico y anatomopatológico. Los métodos micológicos convencionales tienen una capacidad diagnóstica limitada pero pueden ser complementados con técnicas moleculares (DiversiLab, MALDI-TOF, PCR, PNA-FISH, etc.), que permitan una identificación más rápida y certera de los hongos en los hemocultivos, aislados en cultivos o presentes en las muestras de tejidos. La detección de biomarcadores, como BG, GM y GXM, es útil en las micosis invasoras en pacientes inmunodeficientes y son considerados criterios diagnósticos por la EORTC/MSG. Los estudios muestran que la detección de GM en suero y de GXM en suero y líquido cefalorraquídeo tiene gran utilidad (AI-AII) y la de BG una utilidad más moderada (BII) en pacientes oncohematológicos. El diagnóstico de las micosis invasoras por PCR puede ser una alternativa útil en un futuro próximo tanto para el diagnóstico como para la detección de resistencias potenciales y la monitorización del tratamiento antifúngico. Sin embargo, su falta actual de estandarización y validación en las distintas fases que la componen es la razón por la que la EORTC/MSG sigue sin incluirla como criterio diagnóstico de micosis invasora. De entre las distintas técnicas disponibles, la PCRtr parece ser la técnica más ventajosa y conveniente. En la actualidad existen al menos dos sistemas comerciales (SeptiFast y MycoAssay), cuyos resultados preliminares son esperanzadores por estar comercializados con marcaje CE y por ende ser susceptibles de estandarización. Los principales inconvenientes de ambos métodos son su elevado coste por determinación y la escasa experiencia clínica existente, lo que exige tener mucha prudencia en su recomendación. Por lo tanto, la PCR debe ser todavía considerada una herramienta complementaria de los métodos de diagnóstico convencional y no su sustituto. Sin embargo, la detección de AN puede ser muy útil en el diagnóstico de todas aquellas micosis causadas por hongos filamentosos, donde el hemocultivo tiene una rentabilidad muy pobre y en la identificación mediante PCR y posterior secuenciación de aislamientos obtenidos mediante técnicas convencionales. Finalmente, la detección combinada de varios biomarcadores, como GM, BG y AN, pueden mejorar el diagnóstico debido a que desciende el número de resultados falsos positivos, mejorando la especificidad global y, por ende, el VPP. Sin embargo, aunque debería considerase su empleo sobre todo en hospitales con una población importante de pacientes con importantes factores de riesgo para padecer una micosis invasiva, su aplicabilidad en el laboratorio no es tan sencilla y requiere de estudios adicionales que permitan valorar adecuadamente su validez.
Conflicto de interesesEn los últimos cinco años, GQ y EE han recibido fondos de investigación de la Consejería de Educación, Universidades e Investigación y del Departamento de Industria, Comercio y Turismo del Gobierno Vasco-Eusko Jaurlaritza, del Fondo de Investigación Sanitaria (FIS), de la Universidad del País Vasco-Euskal Herriko Unibertsitatea, de Astellas Pharma y de Pfizer. GQ ha recibido honorarios por ponencias de Astellas Pharma, Esteve y Pfizer.
GQ y EE pertenecen al Grupo de Investigación Consolidado del Sistema Universitario Vasco GIC07 123-IT-222–07 (Departamento de Educación, Universidades e Investigación, Gobierno Vasco) y su trabajo de investigación está financiado parcialmente por los proyectos S-PR09UN01 y S-PR10UN03 (Saiotek 2009 y 2010, Departamento de Industria, Comercio y Turismo, Gobierno Vasco) y PI11/00203 (Fondo de Investigación Sanitaria, Ministerio de Sanidad y Consumo).