Estratificación de la población general con base en las variantes genotípicas para seleccionar a aquellas mujeres de alto riesgo a desarrollar un cáncer de mama que puedan ser candidatas a un seguimiento individualizado.
Material y métodosSe ha realizado un estudio caso-control en 856 mujeres con cáncer de mama y 839 mujeres controles de la población general pareadas por edad, analizando la asociación entre el riesgo a desarrollar cáncer de mama y un grupo de variantes basado en 76 polimorfismos de un cambio de base (SNP) de susceptibilidad.
ResultadosSe han establecido 2curvas de casos y controles con base en las odds ratio (OR) genotípicas que diferencian las 2poblaciones con significación estadística (p = 2,293×10-15). Asimismo, se ha estratificado la población de casos y controles e identificado un 14% de la población que se encontraría en el grupo de alto riesgo con una OR > 2 (> 25% probabilidades de desarrollar un cáncer de mama). Este grupo sería candidato a un seguimiento individualizado.
ConclusionesEl Polygenic Risk Score es un predictor del riesgo del cáncer de mama independiente que puede ayudar a seleccionar mujeres con alto riesgo para establecer medidas de seguimiento y tratamiento individualizado en función del riesgo genético.
To stratify the general population based on genotypic variants in order to select women at high risk of breast cancer who could be candidates for individualized follow-up.
Material and methodsWe performed a case-control study in 856 women with breast cancer and 839 aged-matched control women from the general population. We analysed the association between the risk of developing breast cancer and a group of variants based on 76 susceptibility single nucleotide polymorphisms.
ResultsTwo case-control curves were established based on genotypic odds ratios (OR) that differentiated the 2populations with statistical significance (P=2.293×10-15). Stratification of the case-control population showed that 14% of the population would be at high risk, with an OR>2 (> 25% probability of developing breast cancer). Persons in this group would be candidates for individualized follow-up.
ConclusionsThe Polygenic Risk Score is an independent predictor of breast cancer risk that may help to select women at high risk, with a view to establishing individualised follow-up and treatment according to genetic risk.
La predicción del riesgo individualizado de desarrollar una enfermedad es uno de los principales objetivos de la genética. En el caso del cáncer de mama se conocen diferentes parámetros que pueden modificar el riesgo de una mujer. La historia familiar es uno de ellos. Una mujer cuya madre desarrolló un cáncer de mama tiene un riesgo doble que el de la población general, mientras que si han sido su hermana y su madre este riesgo es 3veces superior1. Las bases genéticas que están detrás de estas probabilidades se empezaron a conocer a finales del siglo pasado con el descubrimiento de los genes BRCA1 y 2 como responsables del síndrome de cáncer de mama y ovario hereditario. Estos genes de alto riesgo (6 o 7 veces superior al de la población general) explican un 20% de las familias con cáncer de mama y ovario. Posteriormente, se han identificado otros genes de moderado riesgo (entre 2 y 4 veces un riesgo mayor), como son PALB2, CHECK2, ATM o BRIP1, que explicarían alrededor de un 5% del riesgo de cáncer familiar2. Finalmente, los estudios de asociación del genoma (GWAS) han llevado al descubrimiento de muchas variantes comunes (polimorfismos de un cambio de base, SNP) que están asociadas a riesgo de cáncer. En este sentido, 2grandes estudios internacionales, el proyecto europeo Collaborative Oncological Gene Environment Study (COGS) y el proyecto Oncoarray3,4 analizando cientos de miles de SNP en alrededor de 200.000 mujeres han ampliado el número de SNP a 135, que ayudarían a explicar hasta un 18% del riesgo de cáncer familiar y a identificar a mujeres de la población general con un riesgo mayor de desarrollar cáncer de mama. A nivel individual, estos SNP no son lo suficientemente potentes para incrementar el riesgo (odds ratio [OR] < 1,5), pero la combinación de ellos (conocida como Polygenic Risk Score [PRS]) podría dar un nivel de riesgo que permitiera diferenciar a las mujeres con un mayor riesgo de desarrollar un cáncer de mama, permitiendo estratificar a la población y aplicar medidas individualizadas de seguimiento y tratamiento5.
Estudios previos han demostrado la utilidad a la hora de estratificar a la población con base en el PRS y el valor predictivo del mismo es tanto mayor y más acertado cuanto mayor es el número de SNP6, como se identificó al comparar los datos obtenidos inicialmente para 10 SNP y observar que eran menores que para 70 SNP. Estos últimos, obtenidos del proyecto COGS y de otros GWAS, muestran una OR de 2 para las mujeres estratificadas en el quintil más alto al compararlo con la mediana (OR = 1) de la población y un riesgo a lo largo de la vida de un 29% frente a un 3% para el grupo con el riesgo más alto y bajo, respectivamente3,7. Estos y otros datos muestran el potencial que puede tener la estratificación basada en los factores genéticos y las implicaciones preventivas que podría suponer.
En este estudio presentamos los datos obtenidos analizando un conjunto de 76 SNP asociados previamente a cáncer de mama en el proyecto COGS y otros trabajos, sobre un total de 856 casos de cáncer de mama y 839 mujeres sanas de nuestra población, con el objetivo de replicar estas asociaciones en población española y analizar el poder del PRS en la identificación y selección de mujeres de alto riesgo para desarrollar un cáncer de mama.
Material y métodosReclutamiento de mujeresSe analizaron 2grupos de mujeres emparejadas por rangos de edad:
- –
Un grupo de 992 casos de cáncer de mama consecutivos y no seleccionados procedentes de 4 hospitales: Hospital Universitario La Paz, Fundación Jiménez Díaz y Hospital de Fuenlabrada (Madrid), y del Hospital Monte Naranco (Oviedo). Estas mujeres fueron reclutadas entre los años 2005 y 2009.
- –
Un grupo control procedente del Colegio de Abogados de Madrid y del Instituto Palacios, formado por 927 mujeres sanas, también reclutadas entre 2005 y 2009.
Ambos grupos han sido descritos previamente8.
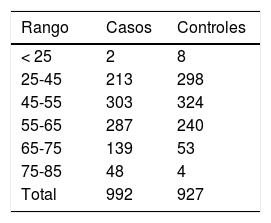
La distribución de los casos y los controles por rango de edad se encuentra recogida en la tabla 1.
Recogida de datosTodas las mujeres rellenaron un cuestionario incluyendo los posibles antecedentes familiares. Los casos y los controles que habían sido estudiadas previamente para BRCA1 y 2 fueron descartadas del estudio. La edad media al diagnóstico de los casos fue de 55,3 años y la del grupo control de 51,5. Un consentimiento informado fue obtenido de todas las mujeres, y el estudio fue aprobado por el comité de ética del Hospital Universitario La Paz y llevado a cabo en consonancia con el código ético de la OMS (Declaración de Helsinki).
Procesamiento de muestras y genotipificadoSe obtuvo una muestra de sangre de todas las mujeres, y su ADN se extrajo de forma automática utilizando un extractor de ADN (MagNA Pure, Roche, Mannheim, Alemania), posteriormente se cuantificó el ADN mediante técnica fluorimétrica Picogreen y finalmente fueron genotipificadas en el Centro Nacional de Genotipado (CEGEN) del CNIO, en Madrid, usando un array de genotipificación custom de Openarray que contiene los 76 SNP asociados a cáncer de mama procedentes del estudio COGS3. Los análisis se basaron en el estudio de los SNP que se habían encontrado asociados en dichos estudios con un valor de p < 5×10–8, que presentaban una frecuencia alélica mínima (MAF) > 1% y unas OR > 1,05 o < 0,85. Todas las variantes, con sus OR y MAF, se encuentran en la tabla S1 (Material suplementario). Como control de calidad de los datos, se estableció que aquellas muestras que presentaran una tasa de éxito o «call rate» inferior al 95%, es decir, que no tuvieran dato genotípico de 7 o más SNP de los 76 seleccionados, serían excluidas del análisis. En los análisis, el genotipo de cada variante se representó por el número de copias del alelo de riesgo (0, 1, 2).
Polygenic Risk ScoreSe calculó la OR genotípica de cada mujer (ORG) recopilando el riesgo asociado a cada uno de los 76 SNP de susceptibilidad para cada individuo usando la fórmula:
donde β es el logaritmo de la OR del alelo minoritario del SNPk asociado al cáncer de mama yxes el número de alelos de dicho SNPk (0, 1, 2). Las OR para cada variante fueron tomadas del proyecto COGS. El PRS fue normalizado con base en la mediana de los valores de PRS obtenidos en la población control.
Estudios estadísticosTodos los estudios estadísticos fueron realizados en la plataforma R. Las gráficas de distribuciones de los valores de PRS para todas las categorías fueron realizadas usando el paquete estadístico ggplot de R9. La diferencia entre las distribuciones de PRS de las muestras control y de los casos fueron estadísticamente evaluadas mediante la prueba estadística de Wilcoxon. La asociación entre el riesgo de cáncer y el PRS fue modelada usando una regresión logística y ajustando por edad.
Para la evaluación estadística de la proporción entre casos y controles asociados a las categorías extremas (bajo riesgo y alto riesgo) se realizó un test binomial. Los cálculos fueron realizados usando el paquete estadístico stats de R10 (R Foundation for Statistical Computing, Viena, Austria).
ResultadosDistribución de casos y controlesHemos genotipificado los 76 SNP en cada uno de los casos y controles, y asignado la OR individual para cada SNP del que son portadores con base en las OR descritas en el proyecto COGS3 para obtener el riesgo genotípico global (ORG) de cada una de las muestras. Este riesgo está basado en un modelo multiplicativo simple donde se considera el valor en homocigosis como el doble de la odds obtenida en heterocigosis. Después se ha normalizado con respecto a la mediana de la población control. Los SNP y la OR se encuentran en la tabla S1 (material suplementario). Este análisis nos permite ver si la población de casos y controles se puede separar con base en su riesgo genotípico.
De este estudio se han descartado todas aquellas muestras que tuvieron un «call rate» menor del 95% o, lo que es igual, que alrededor de 7 o más SNP fallaran en la genotipificación. Un total de 856 casos y 839 controles pasaron el filtro y constituyen la base de este estudio.
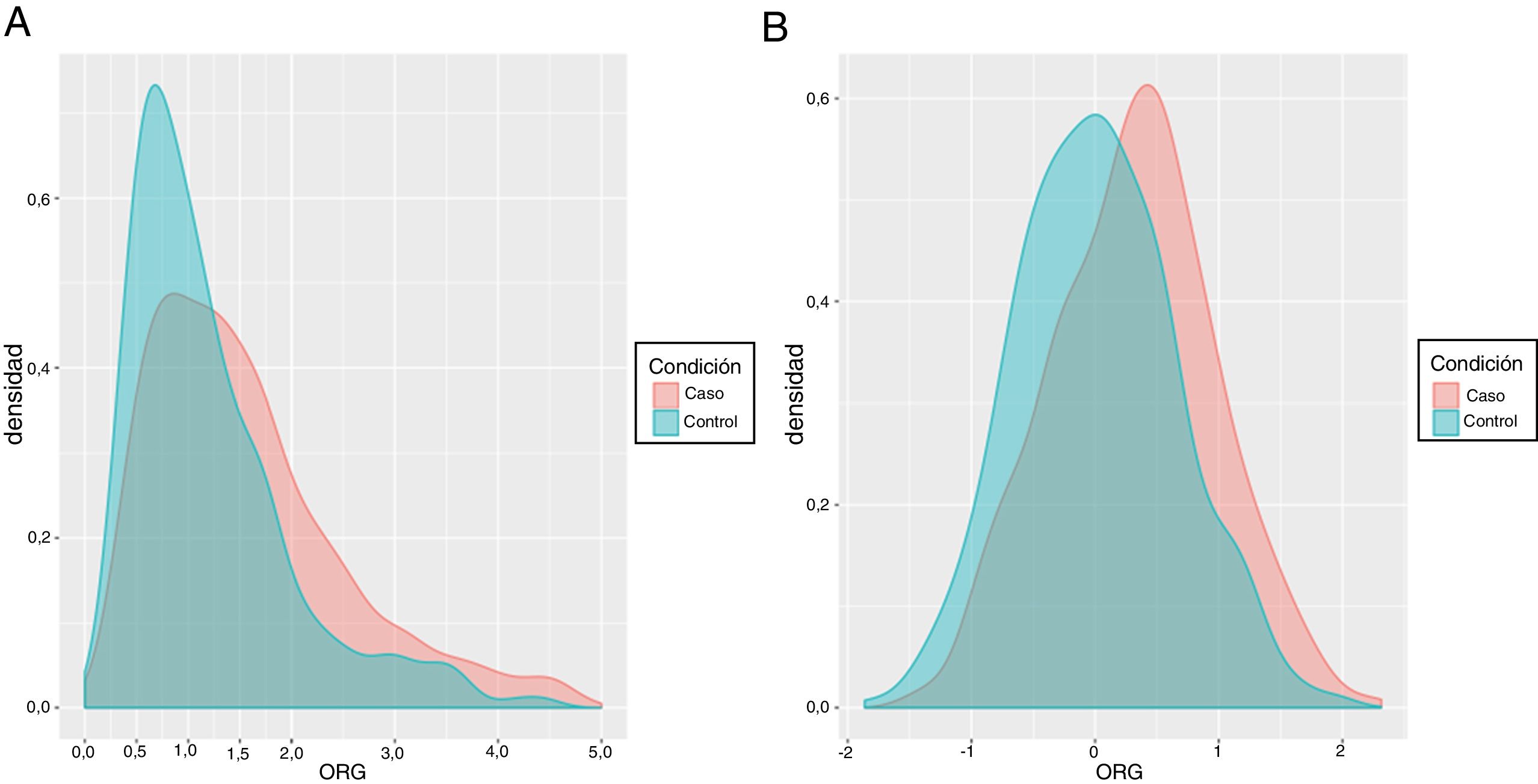
La distribución de las 2poblaciones con base en la ORG obtenida después de normalizar por la mediana de la población control se muestra en la figura 1A.
A) Distribución del riesgo genético de casos y controles después de normalizar por la mediana de la población control. En horizontal el ORG con la mediana de 1 y los rangos de riesgo de 0,5 a 2. B). Distribución del riesgo genético de casos y controles después de hacer una transformación logarítmica de la figura anterior. Las ORG tienen unos valores diferentes (mediana = 0) de la figura 1 A dado que se trabaja con logaritmos en base 2 (log2 [1] = 0).
Las 2curvas de casos y controles se separan de forma estadísticamente significativa (p = 2,29×10–5), aunque no presenten una distribución normal, debido a que las colas tienen una elevada dispersión. Una de las transformaciones más sencillas para evitar esto y equilibrar las 2curvas es usar la transformación logarítmica. La nueva distribución en base logarítmica normalizada por la mediana de la población control se observa en la figura 1B.
Aquí se aprecia claramente cómo las 2curvas se separan. Cuantos más alelos de riesgo acumula una persona, más riesgo tiene de desarrollar cáncer, de ahí que la curva de pacientes con cáncer se encuentra más desplazada hacia la derecha y que los controles que se encuentran a la derecha de la mediana irán incrementando su riesgo cuanto más desplazamiento presenten. Aunque hay casos con un PRS pequeño (< 0,5) y controles con un PRS elevado (> 2), la distribución de las curvas presenta una diferencia estadísticamente significativa (p = 2,29×10–15). Una vez distribuida la población control y normalizada en función de la mediana (OR = 1) se puede establecer el threshold para controles de bajo riesgo en < 0,5; para riesgo poblacional entre 0,5 y 1,5; riesgo moderado en 1,5-2 y, finalmente, alto riesgo para OR > 2 (fig. 1).
Al establecer la asociación entre PRS y cáncer de mama en las 2curvas comparativas de casos y controles, obtenemos una OR de 0,57 (IC del 95% de 0,38869-0,8427, p = 0,003907) para la categoría de bajo riesgo (OR < 0,5), y en el otro extremo, para la categoría de alto riesgo (> 2), la OR es 2,57 (IC del 95% de 1,8071-3,3409, p = 9,01×10–10), es decir, una probabilidad que es la mitad y más del doble, respectivamente, de ser un caso las muestras que se agrupan en uno u otro grupo.
Estratificación de la población con base en el riesgo genéticoLa ORG de cada uno de los casos y controles nos permite clasificar a la población objeto de estudio en cada una de las 4 categorías establecidas. Para ello se multiplica la OR de cada uno de los SNP por el número de alelos (0, 1, 2) que presente la persona. El conjunto de todos los SNP presentes en la mujer en estudio nos dará la ORG global. En la tabla 2 se observa cómo para cada SNP del que es portadora una mujer se obtiene una OR final. Esta OR habrá que normalizarlo con respecto a la mediana de la población control, como se ha indicado previamente.
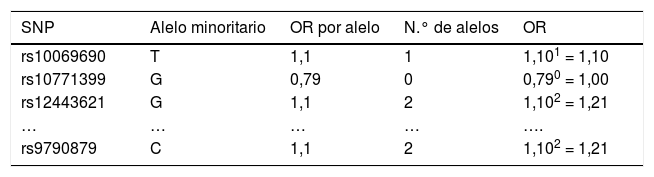
Ejemplo del cálculo de ORG de cada mujer con base en la ORG de cada uno de los SNP de los que es portadora
| SNP | Alelo minoritario | OR por alelo | N.° de alelos | OR |
|---|---|---|---|---|
| rs10069690 | T | 1,1 | 1 | 1,101 = 1,10 |
| rs10771399 | G | 0,79 | 0 | 0,790 = 1,00 |
| rs12443621 | G | 1,1 | 2 | 1,102 = 1,21 |
| … | … | … | … | …. |
| rs9790879 | C | 1,1 | 2 | 1,102 = 1,21 |
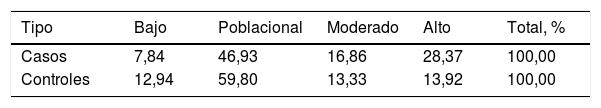
Una vez calculadas y normalizadas las ORG individuales de cada persona, podemos clasificarlas en cada una de las 4 categorías establecidas: riesgo bajo (OR < 0,5); poblacional (OR > 0,5 y < 1,5); moderado (OR > 1,5 y < 2) y riesgo alto (OR > 2). En la tabla 3 se recoge la distribución porcentual de la población española de casos y controles que hemos analizado.
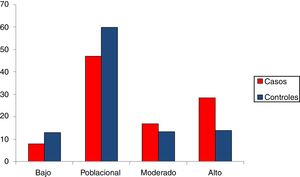
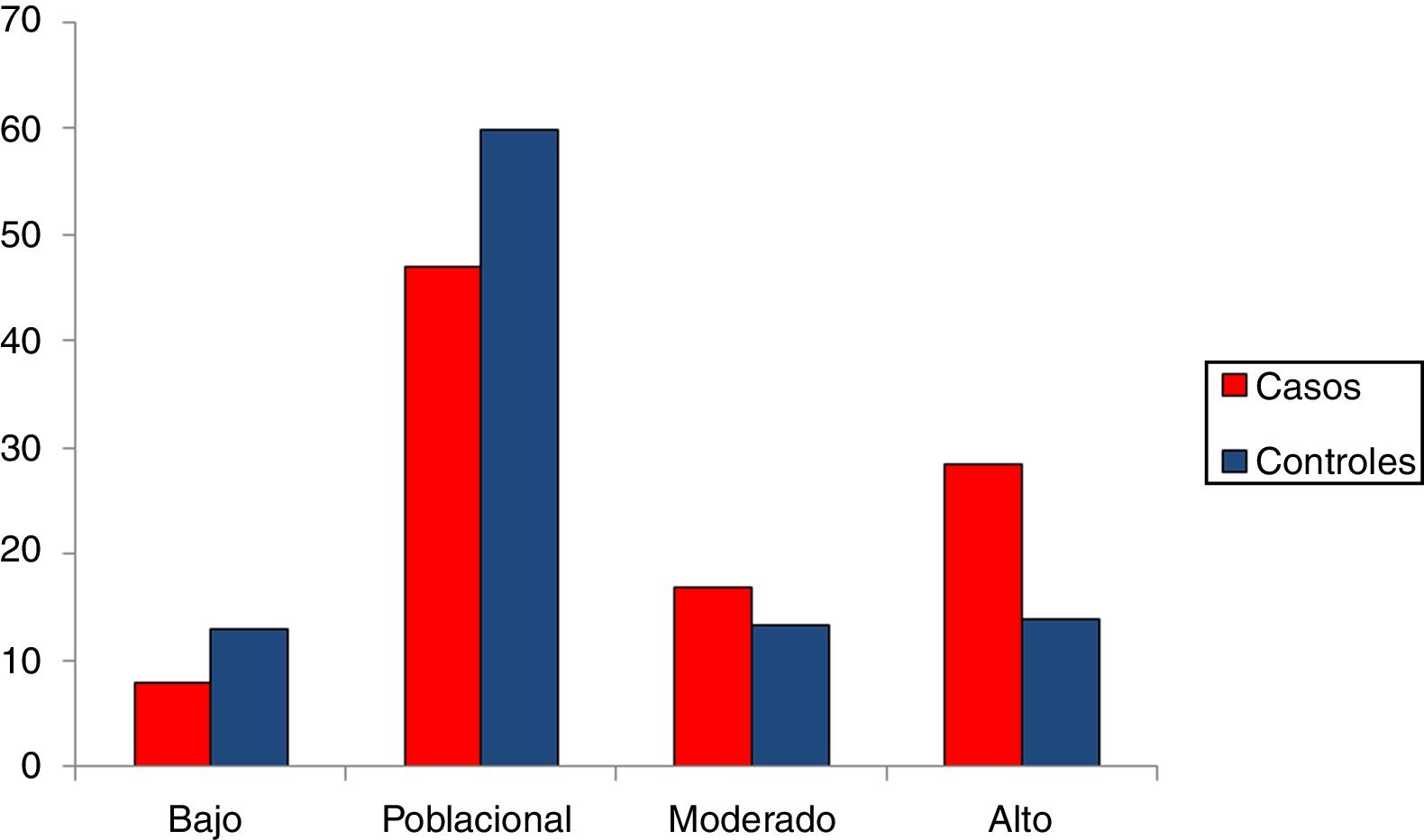
Alrededor de un 8% de los casos de cáncer de mama presentan una ORG baja, mientras que en el grupo control el porcentaje es de un 13%. Por el contrario, un 28% de los casos tienen una ORG alta, mientras que en el grupo control el porcentaje es de un 14%.
En la figura 2 se encuentran representados los 4grupos de riesgo. Desde un punto de vista de seguimiento podemos considerar 2grandes grupos en la población control: un 73% de la población se agrupa en un riesgo bajo poblacional con base en su genotipo (OR de 1,5 como máximo), mientras que el 27% restante se clasificaría como moderado-alto riesgo (OR > 1,5). Más concretamente, el 13,92% sería la población de más alto riesgo de desarrollar cáncer de mama, con una OR superior a 2, equivalente a un 20-25% de probabilidades de desarrollar un cáncer. Esta sería la población candidata a un seguimiento más intenso e individualizado.
Para la valoración del enriquecimiento en el número de controles, en el caso de la categoría de «bajo riesgo» y en el número de casos en la categoría de «alto riesgo», se realizó el test binomial.
En el caso de la categoría «bajo riesgo», el valor de p obtenido es estadísticamente significativo, obteniendo un valor de 0,00614. Este valor indica que la proporción de los controles en la categoría de bajo riesgo es claramente superior a la que cabría esperar por aleatoriedad.
En el caso de la categoría «alto riesgo», la proporción de casos frente a los controles presenta una frecuencia estadísticamente significativa, con un valor de p de 4,9×10–8, claramente alejado de la aleatoriedad.
DiscusiónEn el presente trabajo hemos estratificado a las mujeres españolas de la población general con base en el riesgo que presentan de desarrollar un cáncer de mama debido a su componente genético usando datos de 76 variantes o SNP que se resumen en el concepto de PRS. Nuestros datos indican que las curvas de distribución de riesgo genético de las mujeres control y mujeres con cáncer se diferencian perfectamente y estas diferencias son estadísticamente significativas (fig. 1). En general, y aunque existen SNP protectores, podemos decir que cuantos más SNP de susceptibilidad presenta una persona, mayor riesgo tiene y esto se observa porque existe un mayor desplazamiento de la distribución hacia la derecha (fig. 2). Además, el algoritmo genético permite estratificar a la población en 4 grupos de riesgo y seleccionar un grupo de mujeres de alto riesgo que serían candidatas a un seguimiento más individualizado.
El PRS es el conjunto de SNP de susceptibilidad que presenta una persona y el análisis se realiza asumiendo que los SNP son independientes entre sí y que no presentan interacciones, tal como se indica en estudios previos7,11. Este PRS va incrementando a medida que se trabaja con un mayor número de SNP, como se ha comprobado en trabajos previos con 10, 18 y 70 SNP, y esto se asocia con un mayor riesgo de cáncer de mama6,7,12. Actualmente, hay más de 300 SNP de susceptibilidad y el conjunto de todos ellos en próximos estudios pueden dar un riesgo mayor y más acertado.
La ORG nos permite estratificar a la población en 4subgrupos con base en las ORG. Una OR > 2 se asocia con el grupo de alto riesgo y una OR< 0,5 con el grupo de bajo riesgo. La OR poblacional se situaría entre un 0,5 y 1,5, mientras que el 1,5-2 correspondería a la zona de moderado riesgo. Esta estratificación nos permite seleccionar a la población de alto riesgo que en nuestro caso representa un 14% de las mujeres de la población general que debería tener medidas especiales de seguimiento. Por ejemplo, podrían beneficiarse de la quimioprevención, como se está realizando actualmente en un ensayo (STAR) con mujeres en riesgo bien por ser BRCAX, o con riesgo de supervivencia total mayor de 20%, o bien odds a partir de 1,69 solo por la edad, a las que se les suministra tamoxifeno13. De la misma manera, el PRS podría identificar a mujeres candidatas a un cribado intensivo combinando mamografía y resonancia magnética cada 6meses, dado que la Sociedad Americana del Cáncer recomienda una resonancia magnética para aquellas mujeres con un riesgo de supervivencia total > 25%14.
El poder del PRS se podría ver incrementado si se incorpora a su algoritmo otros parámetros de riesgo, como la densidad mamográfica, la historia familiar o factores hormonales. La incorporación de otros parámetros de riesgo a nivel individual modificables, como el índice de masa corporal o la ingesta de alcohol, actuarían multiplicativamente incrementando el PRS final7,15,16. Y en este último caso, al ser parámetros modificables podrían ser la base para una intervención que reduciría el riesgo final. En esta línea se han realizado estudios que muestran cómo aquellas mujeres en los deciles más bajos y más altos de riesgo se mueven entre un 4 y un 23,5% de desarrollar un cáncer, pero si desaparecen los factores modificables el riesgo se reduciría alrededor de un 30%17. Por ello, además de las medidas preventivas basadas en los cribados y la quimioprevención, la estratificación de mujeres permitiría reducir el riesgo de aquellas que presentan hábitos modificables como los mencionados anteriormente.
En conclusión, el PRS es un predictor del riesgo del cáncer de mama independiente que puede ayudar a seleccionar mujeres con alto riesgo para establecer medidas de seguimiento y tratamiento individualizado en función del riesgo genético. La incorporación de parámetros no genéticos al algoritmo de riesgo podría incrementar notablemente el poder de selección de este grupo de riesgo.
FinanciaciónEl presente trabajo ha sido parcialmente financiado por el Instituto de Salud Carlos III (Madrid) (Fondo Investigaciones Sanitarias PI16/00440) y con Fondos FEDER; Proyecto Europeo FP7-COGS (Grant 223175 Health); IVACE (Valencia) (IMIDA/2016/75, IMIDTA/2018/76). Los financiadores no han tenido ningún papel en el diseño del estudio, recogida de datos y análisis, decisión de publicación o preparación del manuscrito.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Agradecemos a Alicia Barroso, Victoria Fernandez, Belén Herranz de Sistemas Genómicos por su ayuda técnica; a Sonia Santillán y Christian Moya por el asesoramiento genético desde la Unidad de Genética Médica de Sistemas Genómicos; al Banco de muestras de Oviedo por las muestras suministradas.






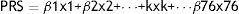
![A) Distribución del riesgo genético de casos y controles después de normalizar por la mediana de la población control. En horizontal el ORG con la mediana de 1 y los rangos de riesgo de 0,5 a 2. B). Distribución del riesgo genético de casos y controles después de hacer una transformación logarítmica de la figura anterior. Las ORG tienen unos valores diferentes (mediana = 0) de la figura 1 A dado que se trabaja con logaritmos en base 2 (log2 [1] = 0). A) Distribución del riesgo genético de casos y controles después de normalizar por la mediana de la población control. En horizontal el ORG con la mediana de 1 y los rangos de riesgo de 0,5 a 2. B). Distribución del riesgo genético de casos y controles después de hacer una transformación logarítmica de la figura anterior. Las ORG tienen unos valores diferentes (mediana = 0) de la figura 1 A dado que se trabaja con logaritmos en base 2 (log2 [1] = 0).](https://static.elsevier.es/multimedia/02141582/0000003200000003/v1_201910101101/S0214158219300489/v1_201910101101/es/main.assets/thumbnail/gr1.jpeg?xkr=ue/ImdikoIMrsJoerZ+w96p5LBcBpyJTqfwgorxm+Ow=)