A pesar de utilizar criterios histológicos e inmunohistoquímicos, no somos capaces de reflejar la heterogeneidad del cáncer de mama. En 2012 se realiza el estudio Molecular Taxonomy of Breast Cancer International Consortium (METABRIC), el cual analiza la arquitectura genómica y de transcripción en 2000 cánceres de mama. Aparecieron subtipos moleculares con gran implicación. Tal es la importancia de la biología molecular que, en el AJCC-TNM8 (2017), se incorporaron grupos pronósticos con base en la expresión de biomarcadores (RE, RP, HER2, Ki67). Estos grupos complementan a la clasificación tradicional y añade un enfoque biológico al puramente anatómico existente. Hemos analizado el estudio METABRIC, haciendo hincapié en la nueva línea de investigación que aportó. Realizamos una exhaustiva búsqueda bibliográfica en las principales bases de datos, obteniendo los artículos que exponen los resultados del METABRIC. Desglosamos los 10 grupos integradores descubiertos recientemente, sus variaciones genéticas y su implicación para nuestra práctica clínica. Comprobamos que la clasificación actual del cáncer de mama no es lo suficientemente precisa, cuyas incongruencias se explican por los grupos integradores. Sientan los cimientos para una nueva clasificación o para refinar los subtipos existentes.
Despite using histological and immunohistochemical criteria, we are unable to reflect the heterogeneity of breast cancer. In 2012 METABRIC analyzed the genomic and transcriptional architecture of 2000 breast cancers. Molecular subtypes were found to be highly implicated. Such is the importance of molecular biology that, in AJCC-TNM8 (2017), prognostic groups based on biomarker expression (ER, PR, HER2, and Ki67) were incorporated. These groups complement the traditional classification and add a biological approach to the existing purely anatomical one. We have analyzed the METABRIC study, emphasizing the new line of research it contributed. We did an exhaustive literature search in the main databases, obtaining the articles presenting the METABRIC results. We broke down the 10 recently discovered integrative clusters, their genetic variations and their implication for our clinical practice. We found that the current classification of breast cancer is not enough accurate, the inconsistencies of which are explained by the integrative clusters. They lay the foundation for a new classification or for refining existing subtypes.
El cáncer de mama es el más frecuente en mujeres. Supone la tercera neoplasia maligna en España, por detrás del cáncer colorrectal y de próstata, superando en prevalencia al cáncer de pulmón. La Sociedad Española de Oncología Médica (SEOM) estima que en el año 2021 los casos de cánceres diagnosticados en España ascenderán a 276.239. El cáncer de mama supondrá el 12% de ellos, 33.375 casos1.
La mayor parte del diagnóstico se realiza dentro de los programas de detección precoz que existen en nuestro país. Engloban a las mujeres de entre 45-50 hasta los 69 años, con variaciones entre cada Comunidad Autónoma. La Sociedad Europea de Imagen Mamaria (SEDIM), en conjunto con la Sociedad Europea de Imagen Mamaria (EUSOBI), recomienda realizar un cribado poblacional a todas las mujeres de entre 40 a 70 años y a las que tienen antecedentes familiares directos (madre, padre, hijos, hermana…) de neoplasias relacionadas2.
El 80% de los tumores malignos de la mama son clasificados como infiltrantes de tipo no especial, seguido de un 10% de lobulillares invasivos. Dado que la clasificación histológica no refleja la heterogeneidad de esta enfermedad, el estudio inmunofenotípico se ha estandarizado. Este análisis clasifica a las lesiones según su índice mitótico (ki67) y la sobreexpresión de receptores para estrógenos, progesterona o HER2/neu3.
Los receptores de esteroides sexuales (estrógenos y progesterona) se expresan en el 75% de los cánceres y estimulan el crecimiento del epitelio normal y neoplásico. El factor de crecimiento epidérmico humano HER2 lo expresan el 15% de los casos y es un indicador de mal pronóstico. Sin embargo, disponemos de terapia específica frente a estos receptores. Las lesiones malignas que no expresan receptores de estrógenos, progesterona ni HER2 se denominan tumores triple negativo. Tienen un mal pronóstico por su alto riesgo de recidiva a cinco años4.
En 2012, el estudio Molecular Taxonomy of Breast Cancer International Consortium (METABRIC) realizó un análisis de la arquitectura genómica y de transcripción de 2.000 cánceres de mama5. Aparecieron así nuevas plataformas genómicas y se descubrieron aberraciones compartidas entre los subtipos moleculares, aportando significación pronóstica6.
El valor de la inmunohistoquímica se puso de manifiesto en el año 2013, en St. Gallen. Se redactó un consenso que clasificaba el cáncer de mama en cuatro grandes grupos (Luminal A, Luminal B, Triple negativo y subtipo HER2) con base en la biología molecular e inmunohistoquímica (tabla 1).
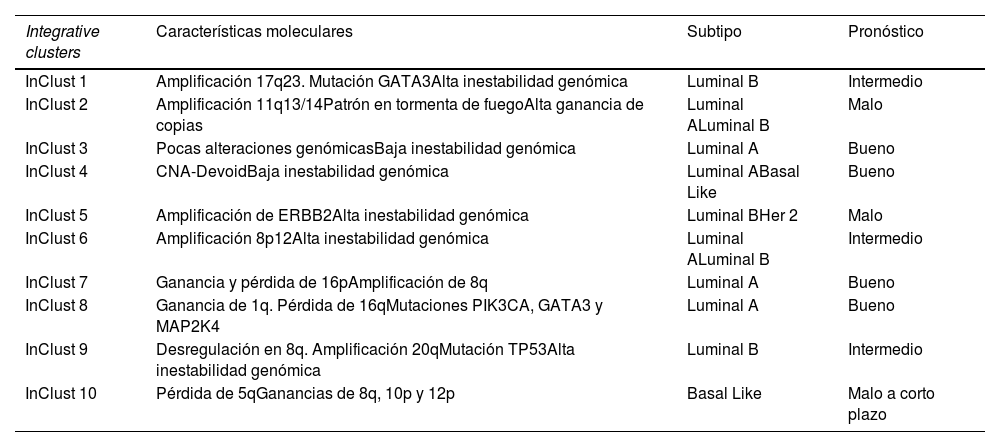
Grupos integradores. Características moleculares, subtipo portador más frecuente y pronóstico otorgado
| Integrative clusters | Características moleculares | Subtipo | Pronóstico |
|---|---|---|---|
| InClust 1 | Amplificación 17q23. Mutación GATA3Alta inestabilidad genómica | Luminal B | Intermedio |
| InClust 2 | Amplificación 11q13/14Patrón en tormenta de fuegoAlta ganancia de copias | Luminal ALuminal B | Malo |
| InClust 3 | Pocas alteraciones genómicasBaja inestabilidad genómica | Luminal A | Bueno |
| InClust 4 | CNA-DevoidBaja inestabilidad genómica | Luminal ABasal Like | Bueno |
| InClust 5 | Amplificación de ERBB2Alta inestabilidad genómica | Luminal BHer 2 | Malo |
| InClust 6 | Amplificación 8p12Alta inestabilidad genómica | Luminal ALuminal B | Intermedio |
| InClust 7 | Ganancia y pérdida de 16pAmplificación de 8q | Luminal A | Bueno |
| InClust 8 | Ganancia de 1q. Pérdida de 16qMutaciones PIK3CA, GATA3 y MAP2K4 | Luminal A | Bueno |
| InClust 9 | Desregulación en 8q. Amplificación 20qMutación TP53Alta inestabilidad genómica | Luminal B | Intermedio |
| InClust 10 | Pérdida de 5qGanancias de 8q, 10p y 12p | Basal Like | Malo a corto plazo |
La escala TNM del Comité Estadounidense Conjunto sobre el Cáncer (AJCC) se basaba en la extensión anatómica, sin reflejar la heterogénea biología del cáncer de mama. Por este motivo, en su última edición del 2017 (AJCC-TNM8) se incorporaron grupos pronósticos teniendo en cuenta la expresión de biomarcadores, la inmunohistoquímica y genética. Estos datos ayudan a evaluar el comportamiento molecular de las células neoplásicas, complementando de esta manera la clasificación tradicional basada en la extensión de la enfermedad7.
Material y métodoHemos realizado una revisión de la literatura existente, profundizando en la línea de trabajo del estudio METABRIC. De los 2.000 cánceres de mama recogidos, aproximadamente la mitad de ellos se utilizaron para desgranar su biología molecular y generar nuevas líneas de investigación. En el resto de los casos se comprobó la reproducibilidad de los grupos integradores y su posibilidad de extrapolarlos a la población general. METABRIC aporta una nueva línea de investigación en el cáncer de mama. Explicando el comportamiento anómalo de determinados tumores.
Hemos realizado una exhaustiva búsqueda bibliográfica usando los operadores lógicos (booleanos) en las principales bases de datos médicos; PubMed, EMBASE y Cochrane Library. Las palabras clave utilizadas fueron: «breast cancer», «molecular classification» e «Integrative clusters». La estrategia de búsqueda empleó tanto los términos del Medical Subject Headings (MeSH) como los Descriptores de Ciencias de la Salud (DeCS). Restringimos las referencias bibliográficas a los artículos posteriores a 2012, año en el que METABRIC publica su estudio. Priorizamos los de mayor calidad metodológica e impacto. Obtuvimos cinco artículos cuyo tema central fue la actualización molecular del cáncer de mama.
La referencia al METABRIC como marco de trabajo es común en los estudios analizados. Por este motivo se utiliza el estudio METABRIC y los artículos de nuestra búsqueda como base de investigación.
ResultadosEn la última actualización de los subtipos intrínsecos se añade el Luminal B enriquecido con HER2. Esta combinación de receptores hormonales y HER2 se explica gracias a los grupos integradores. Éstos son variaciones genéticas adquiridas y heredadas que contribuyen a una expresión genética anormal6.
El METABRIC describe un grupo de alteraciones genéticas que se comportan como oncogenes y permiten clasificar el cáncer de mama en grupos integradores con características propias8.
Se descubren 10 grupos integradores (integrative clusters) asociados a un determinado número de copias aberrantes5. Cada uno de ellos confiere unas características clínicas determinadas que refina aún más los subgrupos existentes9. Hasta la fecha representan la taxonomía molecular más extensa del cáncer de mama8. Recogen características de los subgrupos, crea otros nuevos e incluso algunos son compartidos por subtipos moleculares.
A continuación, se desglosan cada uno de estos 10 grupos integradores (abreviado internacionalmente como «InClust»).
InClust 1:
Fundamentalmente se encuentra en el cáncer tipo Luminal B6. Confiere un pronóstico intermedio. Se caracteriza por amplificación 17q23 e intensa inestabilidad genómica. La mutación representativa de este grupo es la GATA3, diferenciándola del resto de Luminal B. la amplificación de 17q23 afecta a genes adyacentes como RPS6KB1, PPM1D. El primero está implicado en la ruta de los mTOR, actuando sobre la proliferación celular. El segundo está en relación con la p53, implicada en la ruta de la apoptosis10. Esto nos podría proporcionar dianas terapéuticas.
InClust 2:
Se encuentra tanto en el cáncer tipo Luminal A como Luminal B. Es el subtipo de peor pronóstico dentro de todos los Luminal5,6. Está caracterizado por amplificación 11q13/14, patrón en tormenta de fuego y una alta ganancia de número de copias. En esta región encontramos varios genes conductores, relacionados con cáncer de mama y ovario, como CCND1 (11q13.3), EMSY (11q13.5), PAK1 (11q14.1) y RSF1 (11q14.1)10. Se caracteriza por un bajo infiltrado inflamatorio en comparación con el InClust 111.
InClust 3:
Relacionado con el grupo Luminal A. Clínicamente estos tumores presentan bajo grado y poca incidencia de afectación ganglionar10. Tiene muy pocas alteraciones genómicas y se caracteriza por su baja inestabilidad genómica8. Es uno de los subgrupos más prevalentes. Confiere el mejor pronóstico de todos los grupos integradores, con una supervivencia alrededor del 90% a los 10 años10.
InClust 4:
Es compartido por tumores de tipo Luminal A y triple negativo6,9. Confiere buen pronóstico, supervivencia del 80% a los 10 años del diagnóstico, explicado por la infiltración de células no tumorales8. Molecularmente se caracteriza por una baja inestabilidad genómica. Tiene un número mínimo de copias, desprovisto de aberraciones, el llamado «CNA-Devoid» o alteración de número de copias sin aberración. Alrededor del 20% presentan deleciones en el receptor de Células-T (TCR), localizado en los cromosomas 7 y 1410. Histológicamente tienen un gran infiltrado linfocitario9. Dentro de este InClust distinguimos dos entidades, una con mejor pronóstico y comportamiento luminal y otra con comportamiento basal, conservando el infiltrado linfocitario8.
InClust 5:
Representado tanto en cáncer tipo luminal como no luminal. Está caracterizado por un alto nivel de amplificación del 17q12, lo que implica específicamente la amplificación del ERBB26,9. Encontramos los tumores HER2 con RE negativo (HER2 puro), representando el 58%, y los Luminales B HER2 enriquecido (42%)10. Consta de alta inestabilidad genómica. Este subgrupo es el de peor supervivencia. Alrededor del 45% a los 10 años5,10.
InClust 6:
Se encuentra en tumores con receptor de estrógeno positivo, comprendiendo tanto Luminal A como Luminal B9. Se caracteriza por una amplificación específica del 8p12, común en los en los canceres de mama con RE positivo10. Tiene una alta inestabilidad genómica y un pronóstico intermedio6. Junto con el InClust 2, es el de peor pronóstico en el cáncer hormonal puro (HER2-), debiendo estudiarse como factor pronóstico del mismo10.
InClust 7:
Fundamentalmente en tumores tipo Luminal A9. Tienen buen pronóstico, con una supervivencia a los 10 años alrededor del 80%. Molecularmente caracterizado por la ganancia de 16p y pérdida de 16q, aparte de amplificación de 8q. Consta también de la mayor frecuencia de mutaciones en MAP3K1 y CTCF dentro de todos los grupos integradores10.
InClust 8:
Muy similar al InClust 7, también aparece en los Luminal A y tiene una supervivencia alrededor del 80% a los 10 años10. Las diferencias aparecen a nivel molecular, con ganancia de 1q y pérdida de 16q, este evento es bastante común6. Aparecen mutaciones en PIK3CA, GATA3 y MAP2K410.
InClust 9:
Aparece en varios subtipos intrínsecos, pero fundamentalmente aparece en Luminal B HER2 negativo6,10. Tienen un pronóstico intermedio, alrededor del 60% a los 10 años5. Se caracteriza por una alta inestabilidad genómica, dominada por la mutación de TP53. Molecularmente encontramos una desregulación en 8q y una amplificación de 20q9.
InClust 10:
Representan fundamentalmente tumores Triple Negativo. Tienen una alta mortalidad dentro de los primeros cinco años del diagnóstico, pero superados estos, la mortalidad se estabiliza. Clínicamente son mujeres jóvenes con un cáncer de alto grado y pobre diferenciación10. Molecularmente se caracterizan por una pérdida de 5q y ganancias de 8q, 10p y 12p6, genéticamente no suelen tener amplificaciones, pero sí alteración en el número de copias, daño en la reparación del ADN y alteración de genes implicados en la apoptosis (CDC20, AURKB, BCL2…)10.
DiscusiónLa clasificación tradicional del cáncer de mama según las características patológicas y la IHQ (RE, RP, HER2) está afianzada por su validez, rentabilidad y aplicabilidad clínica. Con el avance tecnológico se revelaron alteraciones transcriptómicas, epigenéticas, genómicas y proteómicas no conocidas5,6. Proporcionaron una descripción más completa de la biología del cáncer de mama, aportando información sobre su estratificación e identificando nuevos genes impulsores y biomarcadores6.
METABRIC genera un subgrupo molecular robusto. El tamaño del estudio permitió analizar los loci de rasgo cuantitativo. Identificando cada locus que contribuye al fenotipo tumoral5. Los genes que contienen la mayoría de las mutaciones implicadas son conocidos, pero las alteraciones en el número de copias (CNA) de regiones genómicas suponen un avance8. La expresión génica junto con las alteraciones en el número de copias nos aporta una mejor apreciación de la heterogeneidad en los subgrupos moleculares existentes10.
Los grupos integradores proporcionan información sobre los posibles impulsores moleculares y comportamiento biológico de determinados tumores10. La correlación entre los cambios en el número de copias y la expresión génica hace que los grupos integradores difieran de la clasificación clásica, proporcionando subtipos más refinados8.
El uso de los grupos integradores debe ser complementario con los subtipos tradicionales, ya que ambos juegan un papel importante en el manejo del paciente9. Los grupos integradores, aparte de tener implicaciones en el desarrollo de objetivos terapéuticos, ayudan a comprender cómo las alteraciones en el número de copias afectan a la expresión del cáncer de mama5,10. Estos nuevos subgrupos aportan un nuevo marco objetivo para la investigación.
En el futuro se deberá abordar con más detalle la capacidad del tumor de evolucionar, cambiar el fenotipo y adquirir cambios genómicos. Los 10 grupos integradores se superponen cuando los enmarcamos en la clasificación inmunohistoquímica clásica. Esto se debe a que miden aspectos de las principales propiedades biológicas de los tumores8.
La secuenciación de próxima generación (NGS) permitirá conocer al completo las mutaciones somáticas, reordenamientos estructurales, alteraciones en el número de copias y epigenética. La lista de los genes conductores está aumentando, agrega una complejidad creciente y a la vez permite dilucidar los mecanismos que impulsan cada subtipo tumoral. A pesar de todo, se necesitan más datos empíricos para apoyar los nuevos descubrimientos6,10.
El futuro de la clasificación del cáncer de mama integrará información clínica del paciente, información histopatológica del tumor e información molecular revelada por perfiles genómicos, transcriptómicos y proteómicos. Proporcionando pruebas diagnósticas, pronósticas y predictivas más específicas para cada subtipo.10
La información derivada de la clasificación molecular debe ser complementaria a la clasificación histopatológica. Es esperable que, en el futuro, haya más información sobre la genética del cáncer de mama. Servirá como base para una nueva clasificación o para una división más fina entre los subtipos existentes6. Los grupos integradores cambian nuestro enfoque respecto al cáncer de mama, aportan una nueva clasificación, nuevas características biológicas e implicación en el manejo6,10. El análisis genético a distintos niveles del tumor nos aporta importante información complementaria diagnóstica, pronóstica y predictiva, fundamental para una patología avanzada de precisión. Sin embargo, actualmente la mayoría de los hospitales no constan de la infraestructura necesaria para realizar este análisis de manera rutinaria, reservándose solamente para los pacientes que no responden al tratamiento convencional o con un tumor no clasificable mediante inmunohistoquímica. Esperamos que en un futuro no muy lejano tengamos disponible la genética del cáncer en la práctica diaria para poder ofrecer el mejor tratamiento dirigido posible.
FinanciaciónEste trabajo no ha recibido ningún tipo de financiación.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.