Las mutaciones del gen forkhead-box-O1 (FoxO1) en el locus 13q14.1 provocan alteraciones en los parámetros bioquímicos conduciendo al envejecimiento prematuro. La proteína FoxO1 participa en la regulación de procesos bioquímicos, que influyen en la regulación del perfil lipídico y glucémico. Estos parámetros son un factor de riesgo de mortalidad en la población anciana. El objeto del estudio fue investigar la relación entre el locus FoxO1 y los marcadores metabólico-nutricionales.
Material y métodosSe investigaron los polimorfismos de nucleótido único (SNP, del inglés single-nucleotide polymorphisms) rs2721069, rs4943794 y rs7981045 en 594 ancianos hospitalizados (65-99 años) en una sección geriátrica, probando la asociación con los marcadores biológicos mediante el análisis de covarianza (ANCOVA) y del modelo estadístico Genotype Score.
ResultadosEl análisis de ANCOVA bajo distintos modelos genéticos reveló una asociación significativa. Asumiendo un modelo genético dominante se observó una asociación significativa con los niveles de la glucosa para rs2721069 (p=0,034) y rs4943794 (p=0,012). Para rs4943794 se observó también una asociación significativa si considerado libre de modelos genéticos (p=0,039) confirmada en el modelo aditivo (p=0,012). El modelo estadístico Genotype Score confirmó una asociación significativa entre FoxO1 SNP y la glucosa, teniendo en cuenta los SNP rs2721069 y rs4943794 en conjunto (p=0,048; β=3,198).
ConclusionesEl envejecimiento es un proceso complejo, resultante de la interacción entre varios factores, como los ambientales y los genéticos. Nuestros hallazgos sugieren que el locus FoxO1 puede influir en los niveles séricos de glucemia en pacientes hospitalizados mayores, siendo entonces uno de los factores genéticos que contribuyen a un envejecimiento saludable.
Mutations of forkhead-box-O1 (FOXO1) gene at locus 13q14.1 cause changes in biochemical parameters leading to premature aging. Protein FoxO1 participates in the regulation of biochemical pathways, including those influencing the regulation of lipid profile and glucose metabolism. These parameters are a risk factor for all-cause mortality in the elderly population. The aim of this study was to investigate the relationship between FOXO1 locus and metabolic-nutritional markers.
Material and methodsSingle-nucleotide polymorphisms (SNP) rs2721069, rs4943794 and rs7981045 were determined in 594 hospitalized elderly (65-99 years), patients consecutively admitted to a geriatric ward, and tested the association of FOXO1 variants with biological markers by the analyses of co-variance (ANCOVA) and by Genotype Score Model statistic.
ResultsThe ANCOVA analysis, under different genetic models, revealed significant associations. In particular, assuming a dominant genetic model, a significant association with serum levels of fasting glucose was observed for rs2721069 (P=.034) and rs4943794 (P=.012). For rs4943794 a significant association assuming a free genetic model (P=.039) and an additive one (P=.012) was also observed. No significant relationship was observed between rs7981045 and the analyzed markers. The Genotype Score Model analysis confirmed a significant association between FOXO1 SNP and fasting glucose, taking the SNP rs2721069 and rs4943794 together (P=.048; β=3.198).
ConclusionsAging is a complex process, resulting from the interaction between several factors, including environmental and genetic ones. Our findings suggest that FOXO1 locus may influence blood glucose levels in hospitalized older patients, thus being one of the genetic factors contributing to healthy aging.
Los factores genéticos asociados con la longevidad humana y el envejecimiento saludable siguen siendo desconocidos. Las estimaciones de la longevidad relacionada con la herencia deriva de los registros individuales y de grandes poblaciones sugieren un componente genético significativo, pero modesto a la vida humana (la herencia ∼15 a 30%)1. Una serie de posibles genes candidatos en una variedad de procesos biológicos se han asociado con la longevidad en organismos modelo. Genes implicados en la regulación de la reparación del ADN y los genes en la vía de señalización evolutivamente conservada del factor de crecimiento insulina-similar2,3 se están convirtiendo en una gran promesa para el esclarecimiento futuro de la fisiología subyacente el control de duración de vida. Muchos de estos genes tienen homólogos humanos y, por lo tanto, tienen el potencial de proporcionar ideas sobre la longevidad humana4,5.
Mutación en los genes que afectan la señalización endocrina, las respuestas al estrés, el metabolismo y los telómeros pueden aumentar la vida útil de organismos modelos. Estas mutaciones han revelado vías conservadas evolutivamente para el envejecimiento, algunos de los cuales parecen extender la vida en respuesta a estímulos sensoriales o a la restricción calórica. Muchas mutaciones que afectan a las vías de la longevidad retrasan las enfermedades relacionadas con la edad, y el análisis molecular de estas vías conduce a una comprensión de la forma en que estos 2 procesos (el envejecimiento y la susceptibilidad a la enfermedad) están vinculados.
Las proteínas FOXO son un subgrupo de la familia Forkhead de factores de transcripción. Esta familia se caracteriza por un dominio conservado de unión al ADN (la «Forkhead box», o FOX) y cuenta con más de 100 miembros en los seres humanos que se clasifican de FOXA a FOXR sobre la base de similitudes en la secuencia y participan en funciones muy diversas. Miembros de la clase «O» muestran la característica de ser regulados por la vía de señalización insulina/PI3K/Akt.
Los genes FOXO fueron identificados por primera vez en seres humanos cuando 3 miembros de la familia fueron encontrados en las translocaciones cromosómicas en los rabdomiosarcomas y las leucemias mieloides agudas. Poco después que los factores FOXO fueron identificados en las células tumorales humanas, el papel crucial de la DAF-16 en la longevidad fue descubierto en los gusanos. La actividad de DAF-16 ha demostrado ser regulada negativamente por la vía de señalización insulina/PI3K/Akt. Experimentos posteriores en células de mamíferos mostraron que las proteínas de mamíferos FOXO fueron fosforiladas directamente e inhibidas por Akt en respuesta a la estimulación del factor de crecimiento insulina-similar. Así, los factores FOXO son mediadores evolutivamente conservados de señalización de la insulina y del factor de crecimiento.
Los factores FOXO han demostrado prolongar la vida de los invertebrados. El vermis, DAF-16, activa un programa de genes que extiende la longevidad mediante la promoción de la resistencia al estrés oxidativo, a los agentes patógenos y a los daños a la estructura de las proteínas. Las mutaciones en el receptor de la insulina o PI3K prolonga la longevidad hasta 3 veces, y esta extensión se revierte cuando el Daf-16 está mutado6. En las moscas, la sobreexpresión de dFOXO es suficiente para aumentar la longevidad7,8. El papel de los factores FOXO en la longevidad de los mamíferos se está estudiando. Los ratones que carecen ya sea del receptor de la insulina o del receptor-1 del factor de crecimiento insulina-similar, pueden vivir hasta un 30% más que los ratones de tipo salvaje, lo que sugiere que los factores FOXO podrían estar involucrados en la longevidad de los mamíferos1. Además, los genes objetivos de FOXO implicados específicamente en la resistencia al estrés se conservan entre los invertebrados y mamíferos, lo que sugiere que la función de FOXO en la resistencia al estrés y la longevidad de los organismos está conservada evolutivamente.
En las células hepáticas el factor de transcripción FOXO parece aumentar la expresión de la PEPCK y del glucógeno-6-fosfatasa (las mismas enzimas que están bloqueadas por la vía de metformina/AMPK/SHP). El bloqueo de este factor de transcripción ofrece una oportunidad para nuevas terapias para la diabetes mellitus9. En las células alfa pancreáticas FoxO1 es importante para la regulación de la expresión prepro-glucagón10. En las células beta-pancreáticas FoxO1 media los efectos del péptido-1 glucagón-similar sobre la masa de las células beta-pancreáticas11.
Sobre la base de nuestro estudio anterior del gen Klotho y algunos marcadores metabólicos12, analizando la misma población, el objetivo de este estudio fue investigar la posible relación entre los genotipos de FoxO1 y los marcadores bioquímicos aceptados como indicadores del estado metabólico-nutricional en pacientes de mayor edad ingresados para cuidados geriátricos.
Material y métodosSujetos del estudioEste fue un estudio prospectivo transversal en conformidad con la Declaración de Helsinki, las directrices para la Buena Práctica Clínica y las guías del Fortalecimiento de los informes de los estudios observacionales en epidemiología (STROBE) (disponibles en la web: http://www.strobe-statement.org/). La aprobación del estudio para los experimentos con sujetos humanos se obtuvo de los Comités de Ética locales para la experimentación humana. El consentimiento informado para la investigación se obtuvo de cada paciente o de familiares/tutores legales en el caso de pacientes con discapacidades relacionadas con la demencia. Todos los pacientes ingresados de manera consecutiva en la Unidad de Geriatría del hospital «Casa Sollievo della Sofferenza», entre enero de 2005 y diciembre de 2007 fueron seleccionados para la inclusión en el estudio. Para cada paciente se realizó mediante una entrevista estructurada, una evaluación clínica y el examen de los registros médicos de los pacientes en general. También fueron recogidos los siguientes parámetros: fecha de nacimiento, sexo, historia clínica, las afecciones actuales y la historia de los tratamientos. Todos los sujetos examinados eran caucásicos, para la mayoría con ascendencia del centro y sur de Italia, y se excluyeron las personas de origen judío, de Europa del Este o de descendencia del norte de África.
Criterios de inclusión/exclusiónLos criterios de inclusión fueron: 1) edad ≥65 años, 2) capacidad de proporcionar un consentimiento informado y la voluntad de participar en el estudio, y 3) disponibilidad de análisis de sangre obtenidos durante la hospitalización. Se excluyeron los pacientes con un diagnóstico de cáncer y enfermedades hematológicas malignas.
Análisis de laboratorioLos niveles séricos de colesterol total (CT), triglicéridos (TG), glucosa en ayunas se determinaron en muestras de sangre fresca utilizando métodos de química clínica de rutina. Los niveles séricos de colesterol de lipoproteínas de alta densidad (HDL-C) y de lipoproteína de baja densidad (LDL-C) se analizaron enzimáticamente utilizando el ensayo Roche de tercera generación HDL-C y el ensayo de segunda generación LDL-C en el analizador químico clínico automatizado Modular® P de Roche (Roche/Hitachi Diagnostics, GmbH, Mannheim, Alemania).
GenotiposEl ADN genómico fue purificado a partir de muestras de sangre fresca o congelada siguiendo el método de salting-out13. En este estudio se analizaron los 3 polimorfismos de nucleótido único (SNP, del inglés single-nucleotide polymorphisms) rs2721069, rs4943794 y rs7981045, que abarca un bloque de 65kb en el locus FoxO1 (13q14) (41143720-41209236 bases de PTER).
El SNP rs2721069 (41143720 bases de PTER), es decir, el C102,015→T, en la región intrónica del gen FoxO1, se determinó por medio del ensayo de la discriminación de los alelos con tecnología TaqMan. El ADN genómico se analizó con el sistema ABI PRISM® 7700 Sequence Detector (Life Technologies Corporation, Carlsbad, CA, EE. UU.), utilizando el ensayo c_15926664_10 de acuerdo con las instrucciones del fabricante.
El SNP rs4943794 (41173408 bases de PTER), es decir, el T72,327→G, en la región intrónica del gen FoxO1, se determinó por medio del ensayo de la discriminación de los alelos utilizando la tecnología TaqMan. El ADN genómico se analizó con el sistema ABI PRISM® 7700 Sequence Detector (Life Technologies Corporation, Carlsbad, CA, EE. UU.), utilizando el ensayo c_30366093_20 de acuerdo con las instrucciones del fabricante.
El SNP rs7981045 (41209236 bases de PTER), es decir, el C36,499→T, en la región intrónica del gen FoxO1, se determinó por medio de la prueba de la discriminación de los alelos utilizando la tecnología TaqMan. El ADN genómico se analizó con el sistema ABI PRISM® 7700 Sequence Detector (Life Technologies Corporation, Carlsbad, CA, EE. UU.), utilizando el ensayo c_30886685_10 de acuerdo con las instrucciones del fabricante.
Análisis estadísticoEl equilibrio de Hardy-Weinberg se verificó para todos los polimorfismos investigados. El método de conteo genético se utilizó para estimar las frecuencias relativas de los alelos14. Para las variables dicotómicas, las diferencias entre los grupos fueron evaluadas mediante la prueba exacta de Fisher. Este análisis se realizó mediante el análisis de la tabla de contingencia de 2 vías disponible en las páginas de cálculo interactivas de estadística (disponible en la web: http://statpages.org/). Las variables continuas de distribución normal se verificaron por la prueba de normalidad de Shapiro-Wilk y la prueba de una muestra de Kolmogorov-Smirnov. Para las variables de distribución normal, las diferencias entre los grupos fueron evaluadas por la prueba t a 2 muestras de Welch, o con el análisis de varianza bajo el modelo lineal general, y el análisis de covarianza (ANCOVA). Para los que no tenían las variables distribuidas normalmente, las diferencias entre los grupos fueron evaluadas por la prueba de la suma de rangos de Wilcoxon con la corrección de la continuidad o la prueba de suma de rango de Kruskal-Wallis. Estos análisis se realizaron con SAS® versión 9.1 (SAS Institute, Cary, NC, EE. UU.). Todos los demás análisis se realizaron con el paquete de la versión 2.11.1 del software R (el proyecto de investigación para el cálculo estadístico. Disponible en la web: http://www.r-project.org/). Se declararon significativos los resultados de las pruebas en las que el valor de p fue menor que la proporción de error de tipo 1 de 0,05.
ResultadosLas características demográficas, clínicas y bioquímicas de los pacientes se basan en los valores de nuestro artículo publicado anteriormente12. Las características genéticas de los sujetos en función del sexo se resumen en la tabla 1.
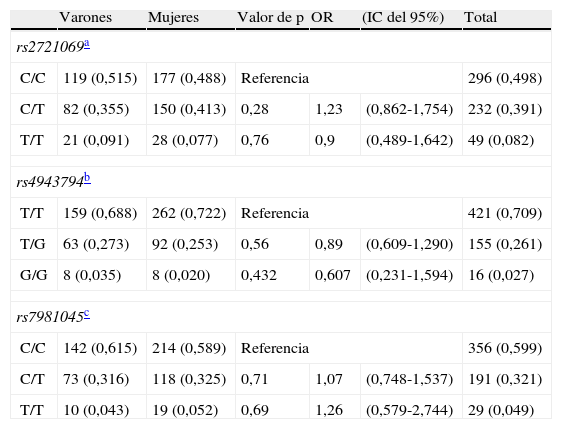
Distribución de los genotipos de FoxO1 rs2721069, rs4943794, rs7981045 según el sexo en pacientes hospitalizados mayores
| Varones | Mujeres | Valor de p | OR | (IC del 95%) | Total | |
| rs2721069a | ||||||
| C/C | 119 (0,515) | 177 (0,488) | Referencia | 296 (0,498) | ||
| C/T | 82 (0,355) | 150 (0,413) | 0,28 | 1,23 | (0,862-1,754) | 232 (0,391) |
| T/T | 21 (0,091) | 28 (0,077) | 0,76 | 0,9 | (0,489-1,642) | 49 (0,082) |
| rs4943794b | ||||||
| T/T | 159 (0,688) | 262 (0,722) | Referencia | 421 (0,709) | ||
| T/G | 63 (0,273) | 92 (0,253) | 0,56 | 0,89 | (0,609-1,290) | 155 (0,261) |
| G/G | 8 (0,035) | 8 (0,020) | 0,432 | 0,607 | (0,231-1,594) | 16 (0,027) |
| rs7981045c | ||||||
| C/C | 142 (0,615) | 214 (0,589) | Referencia | 356 (0,599) | ||
| C/T | 73 (0,316) | 118 (0,325) | 0,71 | 1,07 | (0,748-1,537) | 191 (0,321) |
| T/T | 10 (0,043) | 19 (0,052) | 0,69 | 1,26 | (0,579-2,744) | 29 (0,049) |
Número de sujetos investigados:
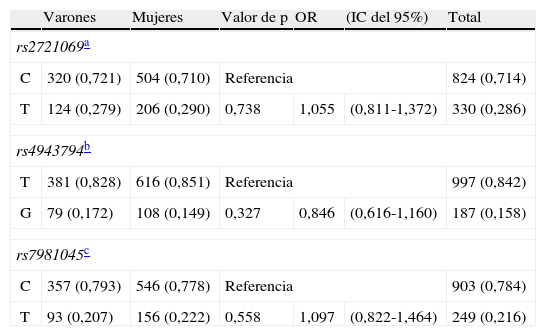
En el análisis de las frecuencias genotípicas de los 3 SNP no se observaron diferencias significativas en la distribución de los genotipos de rs2721069, rs4943794 y rs7981045. Como era de esperar de las frecuencias de genotipo, no se observaron diferencias en las frecuencias de alelos entre varones y mujeres (tabla 2).
Alelos de FoxO1 rs2721069, rs4943794, rs7981045 según el sexo en pacientes hospitalizados mayores
| Varones | Mujeres | Valor de p | OR | (IC del 95%) | Total | |
| rs2721069a | ||||||
| C | 320 (0,721) | 504 (0,710) | Referencia | 824 (0,714) | ||
| T | 124 (0,279) | 206 (0,290) | 0,738 | 1,055 | (0,811-1,372) | 330 (0,286) |
| rs4943794b | ||||||
| T | 381 (0,828) | 616 (0,851) | Referencia | 997 (0,842) | ||
| G | 79 (0,172) | 108 (0,149) | 0,327 | 0,846 | (0,616-1,160) | 187 (0,158) |
| rs7981045c | ||||||
| C | 357 (0,793) | 546 (0,778) | Referencia | 903 (0,784) | ||
| T | 93 (0,207) | 156 (0,222) | 0,558 | 1,097 | (0,822-1,464) | 249 (0,216) |
Número de sujetos investigados:
Las asociaciones de genotipos FoxO1 con los parámetros bioquímicos de los pacientes fueron analizados asumiendo distintos modelos genéticos. Para rs2721069 se observó una asociación significativa con los niveles séricos de la glucosa en ayunas (p=0,034) en el modelo dominante. También el análisis de rs4943794 libre de modelos genéticos, reveló una asociación con los niveles séricos de la glucosa en ayunas (p=0,039), que fue confirmada en el modelo aditivo (p=0,012) y en el recesivo (p=0,012). El análisis de rs7981045 no reveló ninguna asociación significativa con los parámetros metabólicos analizados.
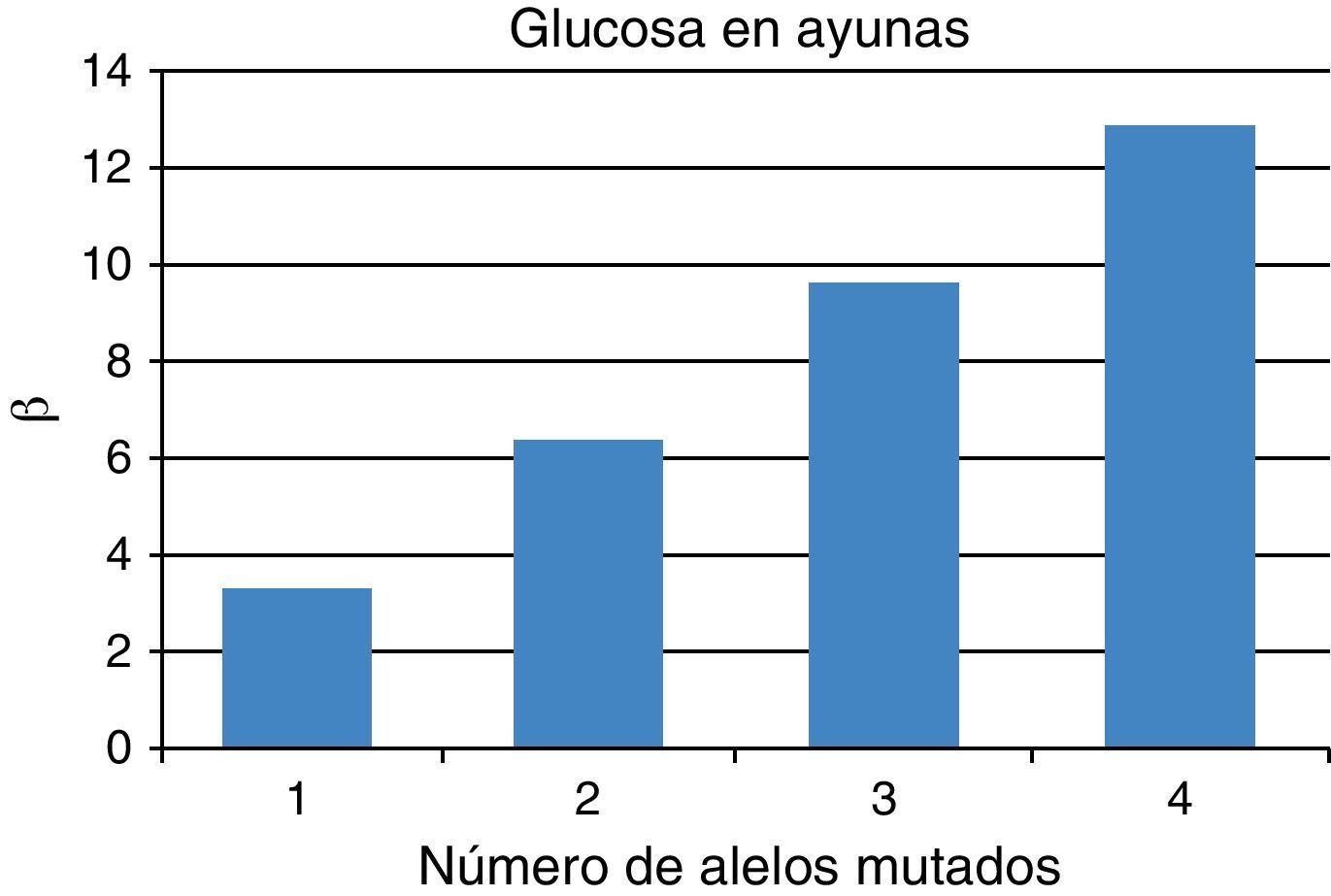
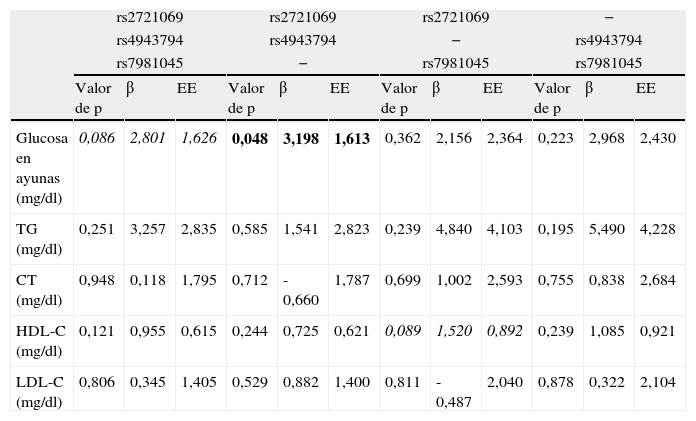
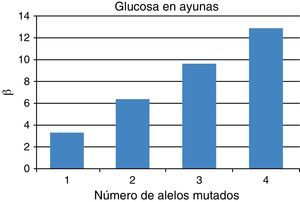
La asociación de variantes genéticas de FoxO1 con características bioquímicas de los pacientes evaluados por medio del análisis de Genotype Score se resumen en la tabla 3. Al considerar al mismo tiempo rs2721069, rs4943794 y rs7981045, un aumento progresivo de cambio de nucleótidos en estos SNP muestra una tendencia a aumentar los niveles de glucosa en ayunas, pero no es estadísticamente significativa (p=0,086; β=2,801). También se calculó el Genotype Score teniendo en cuenta 2 SNP a la vez. Al considerar solo rs2721069 y rs4943794, la asociación con los niveles de glucosa en ayunas resultó significativa (p=0,048) con un aumento de los niveles de glucosa en ayunas para cada variación añadida (β=3,198) (fig. 1). Teniendo en cuenta los 2 SNP rs4943794-rs7981045 y rs2721069 rs7981045, no se observaron asociaciones.
Asociación de genotipos FoxO1 con algunos parámetros de laboratorio de acuerdo con distintos puntajes de riesgo genético en todos los pacientes de edad avanzada hospitalizados
| rs2721069 | rs2721069 | rs2721069 | − | |||||||||
| rs4943794 | rs4943794 | − | rs4943794 | |||||||||
| rs7981045 | − | rs7981045 | rs7981045 | |||||||||
| Valor de p | β | EE | Valor de p | β | EE | Valor de p | β | EE | Valor de p | β | EE | |
| Glucosa en ayunas (mg/dl) | 0,086 | 2,801 | 1,626 | 0,048 | 3,198 | 1,613 | 0,362 | 2,156 | 2,364 | 0,223 | 2,968 | 2,430 |
| TG (mg/dl) | 0,251 | 3,257 | 2,835 | 0,585 | 1,541 | 2,823 | 0,239 | 4,840 | 4,103 | 0,195 | 5,490 | 4,228 |
| CT (mg/dl) | 0,948 | 0,118 | 1,795 | 0,712 | -0,660 | 1,787 | 0,699 | 1,002 | 2,593 | 0,755 | 0,838 | 2,684 |
| HDL-C (mg/dl) | 0,121 | 0,955 | 0,615 | 0,244 | 0,725 | 0,621 | 0,089 | 1,520 | 0,892 | 0,239 | 1,085 | 0,921 |
| LDL-C (mg/dl) | 0,806 | 0,345 | 1,405 | 0,529 | 0,882 | 1,400 | 0,811 | -0,487 | 2,040 | 0,878 | 0,322 | 2,104 |
β: coeficiente beta de remisión; CT: colesterol total; HDL-C: colesterol de lipoproteínas de alta densidad; LDL-C: colesterol de lipoproteínas de baja densidad; EE: error estándar; TG: triglicéridos.
En negrita se presentan los datos que resultan estadísticamente significativos. En cursiva, los datos que no son estadísticamente significativos pero muestran una tendencia al límite de la significatividad. El análisis fue ajustado por la edad y el sexo, y la presencia de diabetes mellitus, enfermedades neurodegenerativas, enfermedades cerebrovasculares y las enfermedades cardiovasculares, así como para el uso de estatinas, la hormona tiroidea, antiinflamatorios y fármacos hipoglucemiantes.
En este estudio se observó una influencia de los genotipos FoxO1 en los niveles séricos de la glucosa en ayunas bajo distintos modelos genéticos. En particular, la influencia de la glucosa en ayunas fue confirmada por un enfoque genotype risk-score (GRS).
La subfamilia FOXO de factores de transcripción tiene un papel muy conservado en la regulación de la duración de la vida, la progresión del ciclo celular, la apoptosis, el metabolismo de la glucosa y la resistencia al estrés mediante la integración de la información relativa a la abundancia de nutrientes y factores de crecimiento15.
En el presente estudio, se observó una asociación significativa entre el SNP rs4943794 con niveles de glucosa en ayunas que se confirmó cuando rs4943794 fue emparejado con rs2721069 en el modelo de GRS, y cuando los 3 SNP fueron considerados en conjunto, se observó una tendencia asociada con el aumento de los niveles de glucosa en ayunas. Probablemente, rs4943794 puede desempeñar un papel importante en lo que respecta al locus FoxO1 en su totalidad.
FoxO1 desempeña un papel en la promoción de la adaptación al ayuno. Además de regular la masa muscular, FoxO1 está involucrado en alterar su función. Promueve la expresión de genes implicados en el metabolismo de energía resultante en la transición de la oxidación de carbohidratos y la oxidación lipídica en condiciones, tales como el ayuno y el ejercicio16. En el hígado, FoxO1 media la expresión de genes implicados en el metabolismo lipídico y de la glucosa. En condiciones de ayuno, donde FoxO1 se localiza en el núcleo celular, impulsa la expresión de las enzimas de la gluconeogénesis17-20. Se ha demostrado que FoxO1 predominantemente media los efectos de la insulina en el metabolismo, incluyendo sus efectos en la producción hepática de glucosa21. Los ratones que sobreexpresan FoxO1 en la glándula hepática y en las células β pancreáticas tienen mayor resistencia a la hiperglucemia en ayunas y la insulina hepática, que conduce al desarrollo de la diabetes de una manera dependiente de la edad18,22,23. Esta asociación ha sido ampliamente discutida, en particular las variantes genéticas en FoxO1 se asociaron con los niveles de glucosa, y con una tendencia a la aparición temprana de diabetes tipo 2 en un estudio caso-control y un análisis de haplotipos formado por los participantes de mediana edad y mayores24,25. En estos estudios, los haplotipos en FoxO1 de los 3 SNP rs2721069, rs4943794 y rs7981045 se asociaron con el aumento de los niveles de la glucosa, y con una tendencia a desarrollar diabetes. Sin embargo, se concluyó que en los seres humanos FoxO1 puede influir en el metabolismo de la glucosa, y contribuir a la predisposición a la diabetes, lo que lleva a una mayor mortalidad.
En nuestro estudio el análisis GRS demostró que un aumento en el número de alelos de riesgo en los 3 SNP se asoció con una tendencia creciente de los niveles de glucosa en ayunas, en particular para el SNP rs4943794 y rs2721069, la tendencia de la asociación llegó a ser estadísticamente significativa, mostrando un mayor valor de coeficiente β de remisión. Estos resultados del modelo GRS confirman estudios recientes que demuestran que polimorfismos de FOXO se asociaron con diversos niveles de glucosa en ayunas.
Recientemente se ha descrito FoxO1 como un factor genético importante para determinar el proceso de envejecimiento y el desarrollo del fenotipo envejecimiento, pero la regulación de la vida de un organismo es compleja y no solo depende de múltiples factores genéticos, epigenéticos y ambientales, sino también de la interacción entre ellos. En este estudio, se utilizó un enfoque de genes candidatos, que se basa en la predicción de la identidad del gen o de los genes correctos, sobre la base de hipótesis biológicas, o la ubicación de los candidatos en una región de linkage determinada previamente. Este enfoque, sin embargo, identifica solo una fracción de los factores genéticos que contribuyen al fenotipo complejo. Un enfoque complementario sería un estudio de asociación de genoma completo para investigar la mayor parte del genoma por un conjunto de variantes genéticas causales.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.