Diversos estudios han evidenciado una resistencia cruzada entre isoniacida y etionamida, 2de los fármacos utilizados en el tratamiento de la tuberculosis multirresistente. El objetivo del presente estudio fue determinar la resistencia cruzada entre ambos fármacos en aislados de Mycobacterium tuberculosis obtenidos en un hospital de Lima (Perú), con alta proporción de pacientes con tuberculosis. Se calculó la frecuencia de mutaciones asociadas con la resistencia a la isoniacida (INH) evaluando el gen katG y la región promotora inhA mediante la prueba molecular Genotype MTBDRplus v2.0. El método gold standard conocido como agar proporciones en placa (APP) permitió la identificación de resistencia a INH y etionamida. De 107 aislamientos resistentes a INH, 54 fueron multirresistentes (identificados mediante la prueba Genotype MTBDRplus) y 49 (es decir, el 45,8% del total) también fueron resistentes a etionamida por el método APP. En los aislamientos resistentes a INH, se encontraron mutaciones en el gen katG en el 50,5% (54/107); en la región promotora inhA en el 23,3% (25/107), y un 14,0% (15/107) presentaron mutaciones en ambos. Un 12,1% (13/107) fueron resistentes a INH por ausencia de banda wild type y banda de mutación. La mutación C-15T en la región promotora inhA presentó una fuerte asociación con la resistencia a etionamida y alcanzó el 73,4% (36/49) de los aislamientos resistentes a dicho fármaco. Los resultados del presente estudio sugieren que la identificación de mutaciones relacionadas con resistencia a INH, sobre todo en la región promotora inhA, podría ser de gran utilidad para identificar la resistencia cruzada a etionamida y mejorar el tratamiento de las personas afectadas por tuberculosis.
Several studies have shown cross-resistance between isoniazid and ethionamide, 2of the drugs used in the treatment of multidrug-resistant tuberculosis. The objective of this study was to determine the cross-resistance between both drugs in Mycobacterium tuberculosis isolates from a hospital with high incidence of tuberculosis in Lima, Peru. The frequency of mutations to isoniazid in the katG gene and the inhA promoter region was identified by the Genotype MTBDRplus v2.0 molecular test. The gold standard Agar Proportion method (APM) allowed todetect resistance to isoniazid and ethionamide. Of 107 isoniazid-resistant isolates (54 multidrug-resistant isolates identified by the Genotype MTBDRplus test, 45.8% (49/107) were also resistant to ethionamide by the APM. Mutations were found in the katG gene in 50.5% (54/107), in the promoter region inhA in 23.3% (25/107) and 14.0% (15/107) that share both mutations in the resistant isolates to INH. The absence of the wild type and mutation bands indicated that 12.1% (13/107) of the isolates were resistant to INH. The mutation C-15T in the inhA promoter region showed a strong association with resistance to ethionamide in 73.4% (36/49) of the isolates analyzed. The results of the present study suggest that the identification of mutations related to resistance to isoniazid, especially in the inhA promoter region, could be very useful to identify cross-resistance to ethionamide and improve the treatment of individuals suffering from this disease.
La tuberculosis (TB) es en la actualidad una enfermedad 100% curable y prevenible, pese a ello, es una de las infecciones contagiosas que ocasionan mayor morbimortalidad en el mundo4,6,12,33.
Perú es uno de los países con más alta carga de TB en América Latina y líder en el número de casos de TB multirresistente (TB-MDR). La ciudad de Lima concentra el 60% de casos de TB, asociados principalmente a la pobreza y el hacinamiento de las personas2,19. En 2017 se reportaron 27.578 casos nuevos de TB, con una tasa de incidencia de 116 casos por 100.000 habitantes, y 1.335 fueron casos nuevos de TB-MDR. A pesar de que los casos de TB en general han ido disminuyendo, los casos de TB-MDR han ido en aumento año tras año35. Este escenario crea la necesidad de implementar nuevas metodologías de diagnóstico e identificación de resistencia a fármacos basadas en pruebas moleculares, las cuales en pocas horas pueden detectar TB-MDR o TB extremadamente farmacorresistente (TB-XDR). Desde hace algunos años, en algunos lugares especializados del Perú, como el Instituto Nacional de Salud, están en uso para la detección de TB-MDR las pruebas moleculares Genotype MTBDRplus (Hain Lifesciences, Alemania) y GeneXpert MTB/RIF (Cepheid, Estados Unidos). Sus costos, que incluyen infraestructura, recursos humanos, insumos y equipos, van desde los 25 y los 40 dólares por muestra, respectivamente10,13,24,28.
La resistencia cruzada a antibióticos usados en el tratamiento de la TB está muy bien documentada, tanto para fármacos de primera línea como de segunda línea8,17,23,29,30. Las micobacterias se tornan resistentes a los fármacos debido a la adquisición de mutaciones o a la activación de bombas de eflujo, característica propia de Mycobacterium tuberculosis (MTB), a diferencia de otras bacterias, que adquieren resistencia muy comúnmente a través de plásmidos o transposones31. El tratamiento de la TB incluye medicamentos de primera y de segunda línea, según el diagnóstico microbiológico, y dosis estándares recomendadas por la Organización Mundial de la Salud.
En pacientes con TB sensible, el tratamiento comprende el uso de rifampicina (RIF), isoniacida (INH), etambutol y pirazinamida. En pacientes con diagnóstico de TB-MDR, se aplican los fármacos levofloxacino, kanamicina, cicloserina y etionamida (ETH)20.
La INH es un antibiótico que inhibe la síntesis de la pared de la micobacteria. La resistencia a INH involucra hasta 4genes: katG, ahpC, oxyR y la región promotora inhA5. La ETH es un fármaco de segunda línea que presenta una tioamida estructural análoga a la INH. Las cepas con bajo nivel de resistencia a INH frecuentemente muestran también bajo nivel de resistencia a ETH, mientras que las cepas con alto nivel de resistencia a INH típicamente permanecen sensibles a ETH21,34. Las mutaciones presentes en cepas resistentes a ETH están relacionadas con los genes ethA y ethR9. Ambos fármacos son profármacos activados por la catalasa-peroxidasa de la micobacteria, codificada por el gen katG, y actúan inhibiendo la actividad de la enzima enoil-ACP-reductasa, responsable de la síntesis del ácido micólico de la pared celular. Por tanto, las mutaciones en la región promotora inhA, causantes de una sobreexpresión de esta enzima, conducen a una resistencia cruzada entre estos 2fármacos antituberculosos14. En Perú, se han realizado varios estudios acerca de la identificación de multirresistencia mediante secuenciación genética a partir de aislamientos y de muestras de esputo3,11.
En el presente estudio se analizaron 107 aislamientos de M. tuberculosis recuperados en un hospital de Lima con alta proporción de pacientes con TB, con la finalidad de identificar la resistencia cruzada entre los fármacos INH y ETH, y establecer una asociación en relación con el gen katG y la región promotora inhA.
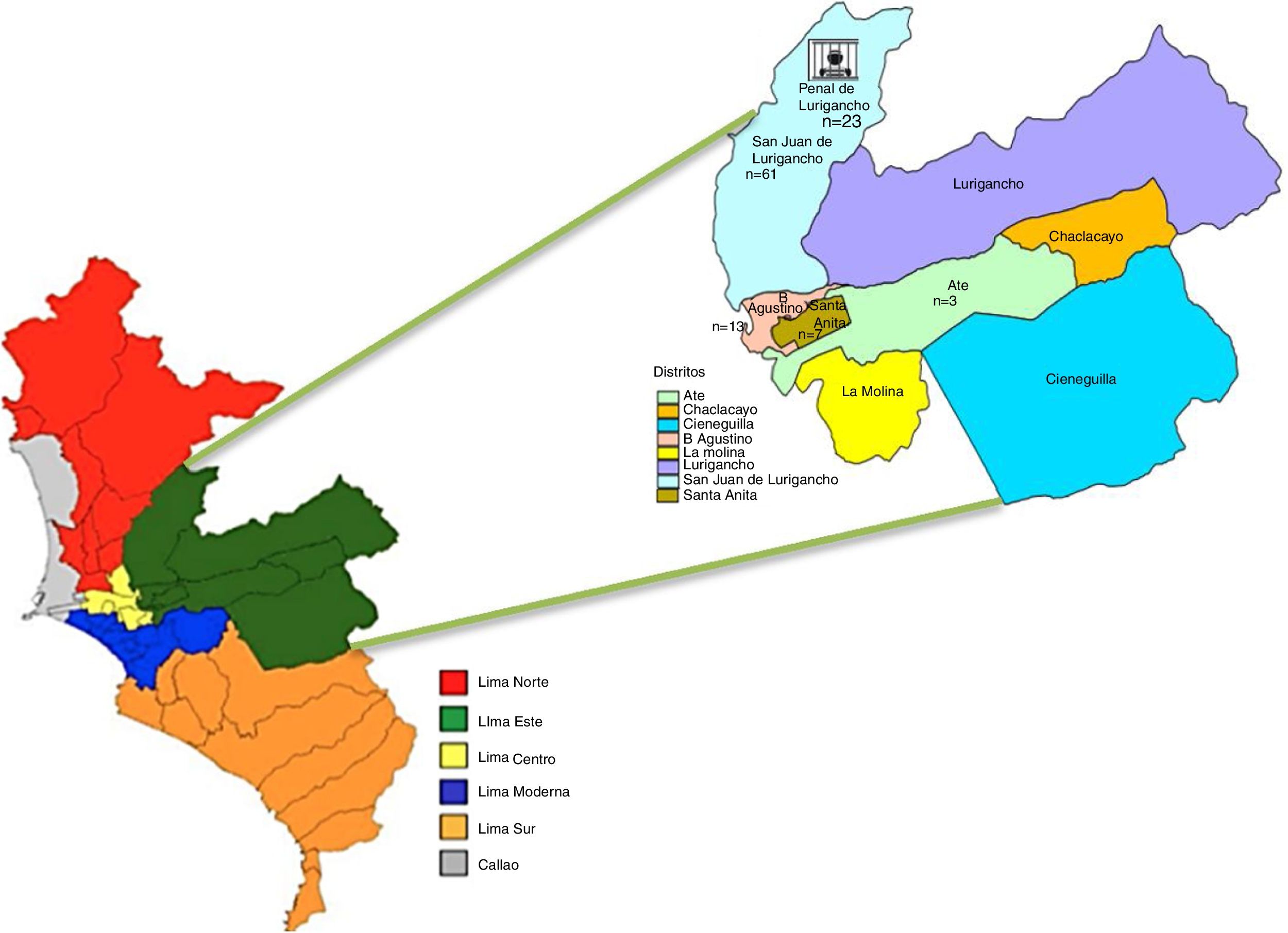
Materiales y métodosMuestras clínicas, sensibilidad a fármacos y extracción de ADNLas muestras biológicas fueron cepas y esputos obtenidos de diferentes pacientes procedentes de los distritos de Lima Este (fig. 1).
Las cepas fueron aisladas en medio Lowenstein Jensen y los esputos incluidos fueron positivos a baciloscopia como mínimo 1+. La recolección de muestras fue por un período de 6 meses (agosto del 2015-febrero del 2016).
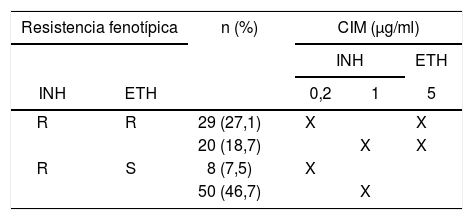
De un total de 584 muestras analizadas mediante la prueba Genotype MTBDRplus v2.0, se seleccionaron 107 aislamientos resistentes a INH por APP. De estos, 52 fueron resistentes a RIF y 49 a ETH. En la determinación de la sensibilidad a fármacos mediante APP, se usaron las siguientes concentraciones: RIF: 1μg/ml; INH: 0,2μg/ml y 1μg/ml, y ETH: 5μg/ml. El perfil de sensibilidad a INH y ETH, según la concentración inhibitoria mínima del crecimiento bacteriano, se muestra en detalle en la tabla 1.
Perfil de sensibilidad a isoniacida (INH) y etionamida (ETH) de 107 aislamientos de Mycobacterium tuberculosis en un hospital de Lima
| Resistencia fenotípica | n (%) | CIM (μg/ml) | |||
|---|---|---|---|---|---|
| INH | ETH | ||||
| INH | ETH | 0,2 | 1 | 5 | |
| R | R | 29 (27,1) | X | X | |
| 20 (18,7) | X | X | |||
| R | S | 8 (7,5) | X | ||
| 50 (46,7) | X | ||||
R: resistente; S: sensible; CIM: concentración inhibitoria mínima.
Las placas se incubaron a 37°C durante 21 días. Las lecturas se realizaron mediante el conteo de colonias en la placa control, que se comparó con el efectuado en las placas que contenían los fármacos. Si la proporción crítica fue mayor del 1%, se informó como resistente, y si fue menor del 1%, se informó como sensible. Se utilizó el kit Genolyse (Hain Lifesciences, Alemania) para la extracción de ADN a partir de las muestras descontaminadas, según recomendaciones del fabricante27,36.
PCR multiplex e hibridación reversaEn la amplificación génica se siguieron los procedimientos moleculares según lo indicado en el manual del fabricante37. Se consideró el número de ciclos específicos para muestras de esputo y cepas. Asimismo, la hibridación se realizó según las indicaciones del fabricante35.
Identificación de mutaciones en el gen katG, la región promotora inhA y el gen rpoBLa identificación de mutaciones relacionadas con la resistencia a INH y RIF se llevó a cabo con el kit Genotype MTBDRplus v2.0 (Hain Lifesciences, Alemania). Se utilizó ADN de la cepa H37Rv como control positivo sensible en cada corrida. La ausencia de bandas wild type, la presencia de bandas de mutación o la combinación de ambas se consideraron indicativas de resistencia al fármaco en estudio.
Análisis estadísticoSe calcularon la odds ratio (OR) y sus intervalos de confianza (IC) para identificar mutaciones estadísticamente significativas, responsables de conferir resistencia cruzada entre INH y ETH. El valor de p<0,05 fue considerado estadísticamente significativo. Los análisis estadísticos se realizaron utilizando el software SPSS versión 24 (IBM, Estados Unidos).
Aspectos éticosEl estudio fue aprobado por el Comité de Ética e Investigación del Hospital Nacional Hipólito Unanue, Lima, Perú. Este comité autorizó el uso de las muestras de ADN almacenadas en el Criobanco del Laboratorio de Micobacterias del Hospital Nacional Hipólito Unanue.
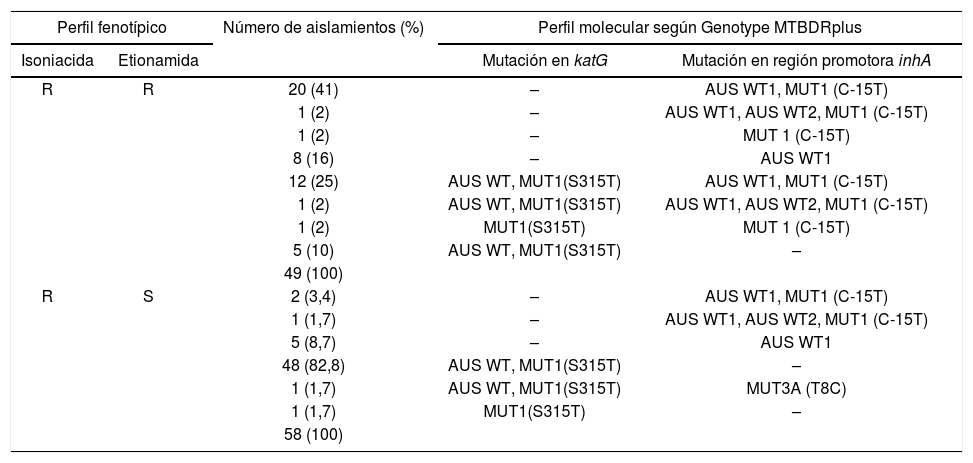
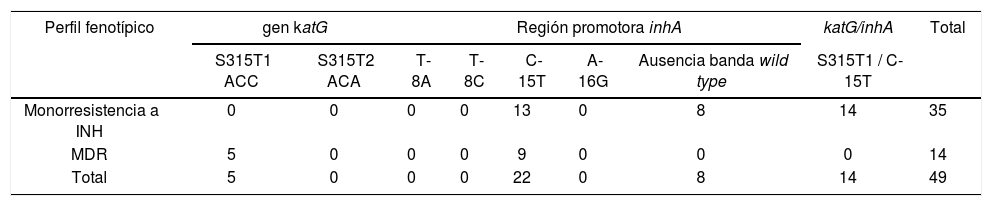
ResultadosIdentificación de mutaciones en el gen katG, la región promotora inhA y gen rpoBDe 107 aislamientos resistentes a INH, el 48,6% (52/107) fueron resistentes a RIF y el 45,8% (49/107) a ETH por APP. Se encontró en un 50,5% de estos aislamientos (54/107) presencia de mutación en el codón S315T1 del gen katG, mientras que en la región promotora inhA de un 23,4% de los aislamientos (25/107) se identificó la mutación C-15T. En un 14% (15/107) se encontraron ambas mutaciones (14 aislamientos con perfil S315T1/C-15T y un aislamiento con perfil S315T1/T-8C). Asimismo, un 12,1% (13/107) fue resistente a INH por ausencia de banda wild type o banda de mutación. Cabe indicar que todo este grupo de muestras presentó resistencia solamente a la concentración de INH de 0,2μg/ml. En lo referido a la resistencia a RIF (gen rpoB), en el 14% (15/107) de los aislamientos resistentes a INH se identificó la mutación D516V; en el 0,9% (1/107), la mutación H526D; en el 28% (30/107), la mutación S531L, y hubo un 7,5% (8/107) resistente por ausencia de banda wild type. En la tabla 2 se indica el perfil fenotípico y genotípico de los 107 aislamientos analizados en este estudio.
Resistencia cruzada a isoniacida y etionamida en 107 aislamientos de Mycobacterium tuberculosis en un hospital de Lima
| Perfil fenotípico | Número de aislamientos (%) | Perfil molecular según Genotype MTBDRplus | ||
|---|---|---|---|---|
| Isoniacida | Etionamida | Mutación en katG | Mutación en región promotora inhA | |
| R | R | 20 (41) | – | AUS WT1, MUT1 (C-15T) |
| 1 (2) | – | AUS WT1, AUS WT2, MUT1 (C-15T) | ||
| 1 (2) | – | MUT 1 (C-15T) | ||
| 8 (16) | – | AUS WT1 | ||
| 12 (25) | AUS WT, MUT1(S315T) | AUS WT1, MUT1 (C-15T) | ||
| 1 (2) | AUS WT, MUT1(S315T) | AUS WT1, AUS WT2, MUT1 (C-15T) | ||
| 1 (2) | MUT1(S315T) | MUT 1 (C-15T) | ||
| 5 (10) | AUS WT, MUT1(S315T) | – | ||
| 49 (100) | ||||
| R | S | 2 (3,4) | – | AUS WT1, MUT1 (C-15T) |
| 1 (1,7) | – | AUS WT1, AUS WT2, MUT1 (C-15T) | ||
| 5 (8,7) | – | AUS WT1 | ||
| 48 (82,8) | AUS WT, MUT1(S315T) | – | ||
| 1 (1,7) | AUS WT, MUT1(S315T) | MUT3A (T8C) | ||
| 1 (1,7) | MUT1(S315T) | – | ||
| 58 (100) | ||||
R: resistente; S: sensible; AUS WT: ausencia banda wild type; MUT: presencia de banda de mutación.
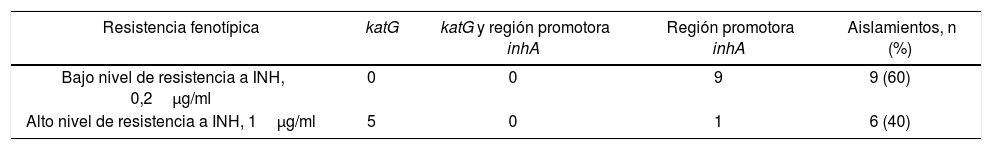
De los 52 aislamientos TB-MDR analizados, un 71,2% (37/52) fue sensible a ETH y el 28,8% (15/52) fue resistente a dicho agente. Entre estos últimos 15 aislamientos (TB-MDR y resistentes a ETH), hubo 9 que mostraron bajos niveles de resistencia a INH, en relación con mutaciones en la región promotora inhA, mientras que los 6 restantes presentaron altos niveles de resistencia a INH en relación con mutaciones en el gen katG (5 de ellos) o en la región promotora inhA (uno de ellos) (tabla 3). Cuando se analizó el tipo de mutación asociada con la resistencia a ETH, se observó que la mutación más frecuente fue la C-15T, presente en un 66,7% de los casos (10/15). De igual manera, cuando se analizaron los aislamientos resistentes a INH y a su vez resistentes a ETH, se observó que la mutación C-15T fue la más frecuente; esta representó un 73,4% de los casos (36/49). Se observó una fuerte asociación (OR ꞊ 50,7 [IC del 95%: 13,5-190,8], p <0,0001) entre la presencia de la mutación C-15T y la resistencia cruzada a ETH. El único aislado que presentó la mutación T-8C no mostró resistencia a ETH. Asimismo, no se detectaron mutaciones en la región promotora inhA, pero sí en el gen katG en un 10,2% (5/49) de aislamientos resistentes a ETH (tabla 4).
Relación entre presencia de mutaciones y niveles de resistencia a INH en 15 aislamientos multifarmacorresistentes, con resistencia a ETH, en un hospital de Lima
| Resistencia fenotípica | katG | katG y región promotora inhA | Región promotora inhA | Aislamientos, n (%) |
|---|---|---|---|---|
| Bajo nivel de resistencia a INH, 0,2μg/ml | 0 | 0 | 9 | 9 (60) |
| Alto nivel de resistencia a INH, 1μg/ml | 5 | 0 | 1 | 6 (40) |
Gen katG: mutación S315T presente; región promotora inhA: mutación C-15T presente.
Mutaciones específicas en gen katG y región promotora inhA en 49 aislamientos resistentes a INH y ETH
| Perfil fenotípico | gen katG | Región promotora inhA | katG/inhA | Total | |||||
|---|---|---|---|---|---|---|---|---|---|
| S315T1 ACC | S315T2 ACA | T-8A | T-8C | C-15T | A-16G | Ausencia banda wild type | S315T1 / C-15T | ||
| Monorresistencia a INH | 0 | 0 | 0 | 0 | 13 | 0 | 8 | 14 | 35 |
| MDR | 5 | 0 | 0 | 0 | 9 | 0 | 0 | 0 | 14 |
| Total | 5 | 0 | 0 | 0 | 22 | 0 | 8 | 14 | 49 |
ETH: etionamida; INH: isoniacida; MDR: multifarmacorresistente.
La TB representa en Perú uno de los mayores problemas en salud pública. Si bien es cierto que se están realizando todos los esfuerzos necesarios para disminuir las tasas de incidencia y mortalidad, los casos de TB-MDR van en aumento cada año, asociados principalmente al bajo seguimiento de los contactos intradomiciliarios de los pacientes afectados por cepas farmacorresistentes. Esta situación hace necesaria la implementación de metodologías de diagnóstico rápidas, a fin de que los pacientes puedan recibir el tratamiento necesario oportunamente1,18.
La monorresistencia a fármacos antituberculosos está presente en distritos con alta carga de TB en Perú. En un estudio observacional que incluyó a 1.292 pacientes peruanos, se encontró un 8,2% de monorresistencia a INH, asociada al uso ilícito de fármacos y a un alto riesgo de una pobre respuesta al tratamiento, mientras que se observó un 2,3% de monorresistencia a RIF, asociada a la infección por el HIV y un alto riesgo de muerte32.
Este es el primer estudio realizado en Perú que examina la prevalencia de las mutaciones en el gen katG y en la región promotora inhA y la resistencia cruzada con ETH en aislamientos procedentes de Lima Este, un lugar en el que la TB es endémica y que incluye 5grandes distritos (Santa Anita, Chosica, Chaclacayo, El Agustino y San Juan de Lurigancho) con elevada incidencia de TB.
Se encontró una marcada correlación entre la presencia de la mutación C-15T dentro de la región promotora inhA y la resistencia a ETH. Este hallazgo sugiere que hay 50 veces más posibilidades de encontrar un aislamiento resistente a ETH y que, a su vez, presente la mutación C-15T. Por otra parte, es necesario mencionar que la prevalencia de la mutación C-15T en Lima Este fue alta, a diferencia de lo que indican estudios previos realizados en Lima Oeste (Callao), que reportan una mayor prevalencia de la mutación del codón S315T en el gen katG3,11.
Los estudios previos acerca de la resistencia cruzada entre INH y ETH muestran resultados heterogéneos. En Corea, Lee et al. analizaron 24 aislamientos multirresistentes por secuenciación del gen inhA, de los cuales 12 (50%) fueron resistentes a ETH. Se identificó la mutación del codón C94T en el 100% de los aislamientos resistentes a ambos fármacos y en un 16,7% de los asilamientos resistentes solo a INH15. En Tailandia, Boonaiam et al.7 analizaron 160 aislamientos resistentes a INH, de los cuales el 79,4% presentó mutación en el codón 315. De 24 aislamientos resistentes a ETH, el 54,2% mostró mutaciones en el gen ethA y solo 6 (25%) aislamientos presentaron mutaciones en inhA, pero fueron sensibles a ETH. Ese estudio demostró que no existía una asociación entre mutaciones en inhA y resistencia a ETH.
En Francia, Brossier et al.8 analizaron 87 aislamientos y encontraron mutaciones en los genes ethA, ethR einhA y en la región promotora de este último en 38 de 47 (81%) aislamientos resistentes a ETH. En 24/47 (51,1%), la mutación C-15T estuvo presente. Machado et al.16, en Portugal, reportaron que la mutación C-15T se encontró en 16 de los 17 aislamientos (94,1%) multifarmacorresistentes analizados, con alto nivel de resistencia a INH y resistencia cruzada a ETH. Asimismo, identificaron los linajes Lisboa 3 y Q1 mediante 24 MIRU-VNTR.
Niehaus et al.22, en Sudáfrica, analizaron 994 aislamientos de M. tuberculosis con el kit Genotype MTBDRplus y reportaron una prevalencia de mutación en inhA y katG del 14,8 y el 10,3%, respectivamente. Asimismo, en 165 aislamientos multirresistentes, estos autores observaron un 75% de frecuencia de la mutación C-15T.
Rueda et al.25,26 realizaron 2estudios en Colombia sobre resistencia cruzada a INH y ETH. En el primero de ellos, al analizar 30 aislamientos multirresistentes y resistentes a ETH, observaron que un 40% de estos tenían resistencia cruzada por mutaciones en la región promotora inhA y el 30% estuvo asociado a la mutación C-15T. El segundo estudio analizó 64 aislamientos e incluyó los genes ethA, ethR, mshA y ndh para la identificación de resistencia por secuenciación. En 57 aislamientos multirresistentes y resistentes a ETH encontraron un 33,3% de resistencia cruzada por mutaciones en la región promotora inhA, la más frecuente fue la mutación C-15T (19,3%).
Dado este escenario, es necesaria la investigación a nivel molecular de la resistencia cruzada entre fármacos cuyos mecanismos de activación involucran enzimas en común. Debido a la alta tasa de resistencia cruzada entre INH y ETH observada en este estudio, la inclusión de ETH en el tratamiento de regímenes de segunda línea no tendría ningún beneficio en los pacientes y deberían ser consideradas otras combinaciones terapéuticas.
Los resultados expuestos sugieren que hay diferencias entre los aislamientos procedentes de Lima Este y el Callao. Estos hallazgos plantearían la nueva pregunta de qué tan diferentes son las cepas resistentes a INH por ubicación geográfica en Lima. Asimismo, podrían explicar la falla en el tratamiento por no contemplar la resistencia de bajo nivel a INH y la resistencia cruzada a ETH en las cepas circulantes en esta región de Lima, que incluye distritos con alta carga de TB. La presencia de mutaciones en la región promotora inhA podría ser usada como un criterio para evaluar resistencia cruzada a ETH. Nuestros resultados demuestran que la frecuencia de mutación C-15T presente en la región promotora inhA es alta en la zona de Lima Este y que, a su vez, se encuentra asociada a resistencia simultánea a ETH.
A nivel local, no existen estudios que permitan hacer comparaciones estadísticamente relevantes acerca de la resistencia cruzada entre fármacos antituberculosos, y esto es muy necesario, ya que la frecuencia de mutaciones específicas en cepas multirresistentes podrían ser distintas y generar un perfil genotípico variable según las áreas geográficas. Es importante conocer las frecuencias de mutaciones asociadas con la resistencia a INH en función de la distribución geográfica en la ciudad de Lima, sobre todo en los distritos con alta carga de TB, para evaluar la posible exclusión de tratamiento con ETH en casos con diagnóstico de TB-MDR. Además, consideramos que es necesario realizar más estudios para poder entender la resistencia de bajo nivel a INH y conocer el valor clínico que este hallazgo puede tener en el tratamiento de TB-MDR.
Finalmente, podemos concluir que se evidencia una alta resistencia cruzada entre INH y ETH en los aislamientos analizados. Las mutaciones en el gen katG y en la región promotora inhA están relacionadas con la resistencia cruzada entre ambos fármacos, además de existir un fuerte grado de asociación entre la mutación C-15T de la región promotora inhA y los aislamientos resistentes a ETH. Se hace necesario diseñar y realizar nuevos estudios que permitan determinar el papel de otros genes involucrados en la resistencia cruzada a estos 2fármacos.
Conflicto de interesesLos autores declaran no tener conflicto de intereses.
Los autores agradecen a todo el personal de salud del Laboratorio de Micobacterias del Centro de Excelencia para el Control de la Transmisión de la Tuberculosis (CENEX) del Hospital Nacional Hipólito Unanue (Lima, Perú), ya que sin su apoyo no hubiese sido posible realizar este estudio.