Arthrobacter agilis UMCV2 es una bacteria rizosférica que promueve el crecimiento vegetal de plantas leguminosas proveyéndoles hierro soluble. Un segundo mecanismo de promoción se da a través de la producción de compuestos volátiles que estimulan los mecanismos de absorción de hierro. Adicionalmente, A. agilis UMCV2 tiene la capacidad de inhibir el crecimiento de organismos fitopatógenos. En el presente trabajo se emplea una combinación de las técnicas de reacción en cadena de la polimerasa cuantitativa e hibridación in situ con fluorescencia para detectar y cuantificar la presencia de la bacteria en los tejidos internos de la planta leguminosa Medicago truncatula. Nuestros resultados demuestran que A. agilis UMCV2 se comporta como una bacteria endófita de M. truncatula especialmente en medios donde el hierro está disponible.
Arthrobacter agilis UMCV2 is a rhizosphere bacterium that promotes legume growth by solubilization of iron, which is supplied to the plant. A second growth promotion mechanism produces volatile compounds that stimulate iron uptake activities. Additionally, A. agilis UMCV2 is capable of inhibiting the growth of phytopathogens. A combination of quantitative polymerase chain reaction and fluorescence in situ hybridization techniques were used here to detect and quantify the presence of the bacterium in the internal tissues of the legume Medicago truncatula. Our results demonstrate that A. agilis UMCV2 behaves as an endophytic bacterium of M. truncatula, particularly in environments where iron is available.
La rizosfera es un ambiente único formado por la interfaz de las raíces de las plantas y el suelo circundante. Miríadas de microorganismos viven, compiten y prosperan en este ambiente. Dichos microorganismos pueden influir en el crecimiento y desarrollo de las plantas, de manera perjudicial o benéfica. Un subconjunto de las bacterias rizosféricas son capaces de colonizar y prosperar en el interior de los tejidos vegetales (raíz y tejidos aéreos), a estas bacterias se les denomina endófitas, a condición de que no tengan un efecto negativo en el crecimiento de la planta, y se asume que su efecto en la planta es más bien de simbiosis o de protocooperación3.
Las poblaciones endofíticas, al igual que las poblaciones rizosféricas, están condicionadas por factores bióticos y abióticos, pero en todo caso el entorno endofítico es mejor controlado por las plantas que el entorno rizosférico, y de este modo las plantas ejercen una mayor selección sobre las poblaciones endofíticas que sobre las poblaciones rizosféricas6.
Las bacterias con efectos promotores del crecimiento vegetal han sido ampliamente empleadas como bioinoculantes, con diferentes resultados. Mientras que en algunas ocasiones los bioinoculantes han tenido un impacto significativo en el aumento de la producción vegetal, en otras ocasiones su impacto ha sido más bien discreto o nulo3. Una de las razones que explican estas variaciones en los resultados es la diferente habilidad de las bacterias inoculadas para competir y sobrevivir dentro de las comunidades bacterianas autóctonas de las rizosferas donde son introducidas3.
Las plantas leguminosas (orden Fabales) son el segundo grupo más importante de plantas cultivadas para la alimentación humana y de ganado, solamente superada por las gramíneas4, de allí la importancia de su estudio.
En un trabajo previo se aisló la cepa Arthrobacter agilis UMCV2 de la rizosfera de plantas de Zea mays (maíz)11. A. agilis UMCV2 es una actinobacteria rizosférica capaz de promover el crecimiento vegetal por distintos mecanismos, que incluyen el suministro de hierro (Fe) soluble a las plantas11 y la emisión de dimetilhexadecilamina, un compuesto orgánico volátil que estimula el crecimiento de la raíz y de los tejidos aéreos de Medicago sativa12, además de estimular los mecanismos de absorción de Fe de la planta9. Este mismo compuesto tiene actividad inhibitoria sobre el crecimiento de los organismos fitopatógenos Botrytis cinerea y Phytophthora cinnamomi13.
Las capacidades como promotores del crecimiento vegetal antes descritas hacen de A. agilis UMCV2 un agente microbiano con potencial para ser usado como bioinoculante. Una cuestión relevante para este propósito es determinar el nivel de asociación de A. agilis UMCV2 con la planta inoculada, como un modo de inferir sus posibilidades de sobrevivencia dentro de la comunidad bacteriana asociada a la planta, independientemente del tipo de suelo y de sus condiciones nutricionales.
El objetivo del presente trabajo fue determinar si A. agilis UMCV2 es capaz de colonizar los tejidos internos de la planta leguminosa modelo Medicago truncatula, cultivar Jemalong A17 en diferentes condiciones de disponibilidad de Fe, y empleamos para ello una combinación de las técnicas de reacción en cadena de la polimerasa cuantitativa e hibridación in situ con fluorescencia.
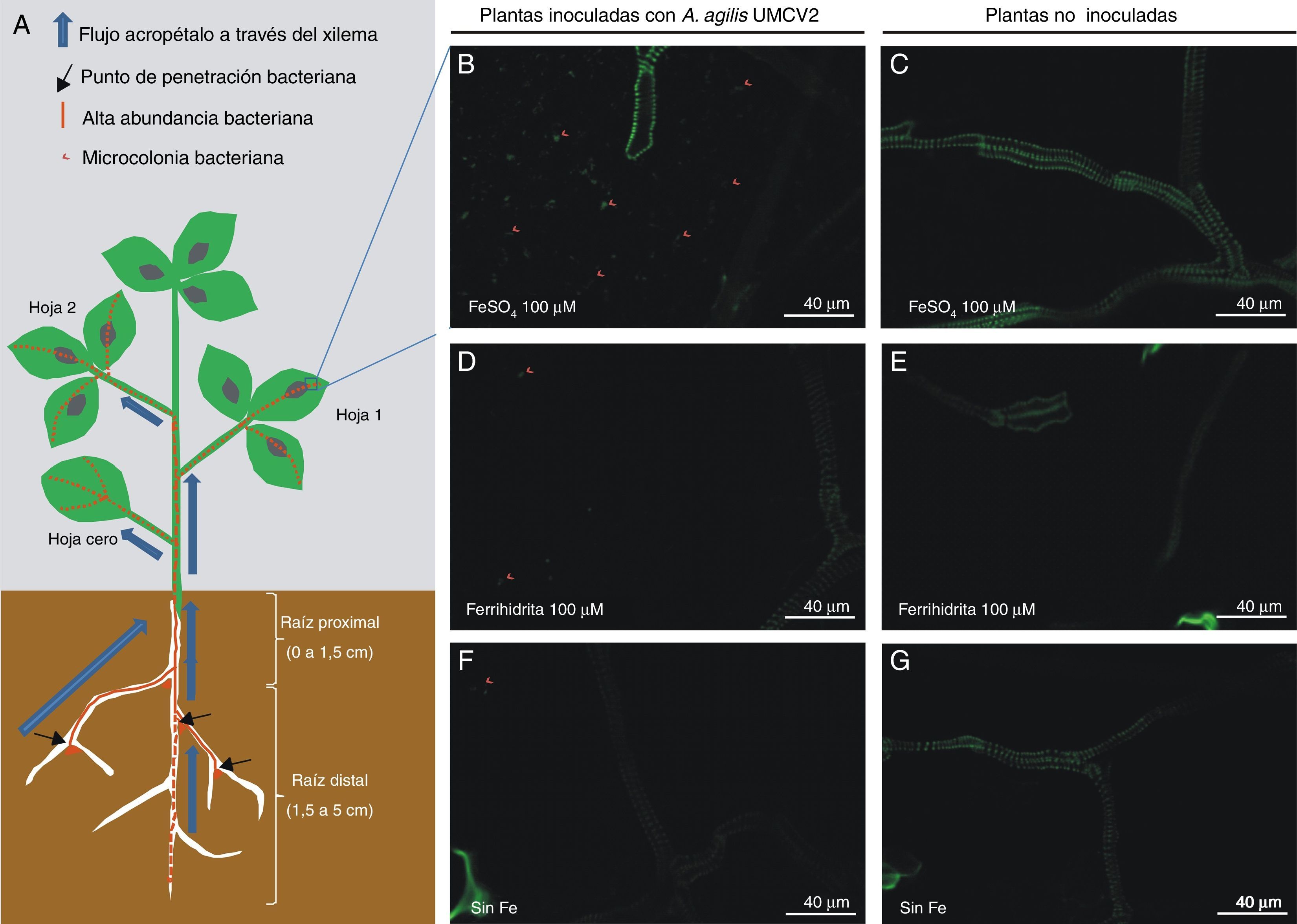
A. agilis fue crecida en agar nutritivo a 25°C durante 48h antes de su utilización. Las semillas de M. truncatula fueron desinfectadas, germinadas e incubadas según se ha descrito previamente9. Plántulas de M. truncatula, a 3d de su germinación, fueron colocadas en frascos de 170ml de capacidad, con 30ml de medio basal Hoagland solidificado con 6g/l de agar de micropropagación y sin Fe, con 100μM de Fe insoluble adicionado como ferrihidrita, o bien con 100μM de FeSO4. Algunas plantas se mantuvieron axénicas (controles axénicos), mientras que otras fueron inoculadas por goteo de 10μl de una suspensión bacteriana de A. agilis UMCV2 con DO590 de 0,1 (equivalente a 1,1×106UFC medidas por cuenta viable) sobre la raíz. Todos los tratamientos, incluidos los controles, tuvieron entre 3 y 8 réplicas independientes. Después de 3 semanas de incubación, se colectaron por separado la hoja cero (unifoliada), la hoja 1, la hoja 2, la raíz proximal (desde el hipocótilo hasta el punto de surgimiento de la primera raíz lateral, a aproximadamente 1,5cm del hipocótilo) y la raíz distal (el resto de la raíz de aproximadamente 3,5cm) (fig. 1A).
(A) Presencia y distribución de A. agilis UMCV2 en plantas de M. truncatula. El panel A muestra un esquema del sistema experimental, señalando las secciones de la planta analizadas y en las que se encontró A. agilis UMCV2 21 días después de la inoculación sobre la raíz. Los paneles B a G muestran fotografías representativas tomadas bajo un haz de luz de excitación de 488nm y una ventana de emisión de 505 a 550nm de longitud de onda, en un plano de 60μM por debajo de la superficie de la hoja 1 de plantas de M. truncatula inoculadas con A. agilis UMCV2 y crecidas en medio adicionado con FeSO4 (B), ferrihidrita (D) o sin Fe en el medio (F), o bien no inoculadas y crecidas en medio adicionado con FeSO4 (C), ferrihidrita (E) o sin Fe en el medio (G). Las fotografías muestran la fluorescencia esperadas de los elementos del xilema, pero solo las plantas inoculadas muestran microcolonias bacterianas señaladas con puntas de flecha.
Tanto las hojas como las raíces de las plantas fueron desinfectadas superficialmente y la ausencia de bacterias viables fue corroborada como se ha descrito previamente8. Se extrajo el ADN de las muestras de tejido vegetal y de las bacterias empleando un método adecuado para estos 2 tipos de materiales biológicos7. La concentración de ADN fue determinada empleando un espectrómetro para micromuestras NanoDrop 2000c (Termo Scientific Nanodrop, Wilmington Delaware, EE. UU.).
Considerando la secuencia del segmento de ADN del espaciador intergénico existente entre los genes del ARNr del 16S y 23S de A. agilis UMCV2 (número de acceso GenBank EF101871.1), se diseñaron los oligonucleótidos 2CV2F (5′-GTCCTGGTGGCATTC-3′) y 2CV2R (5′-ACGCAGAACAAGAGC-3′) utilizando el programa Beacon Disigner 4.02 (Primer Biosoft International, 3786 Corina Way, Palo Alto California, EE. UU.).
Para la detección de A. agilis UMCV2 y la cuantificación de su ADN dentro de los tejidos de M. truncatula, se realizaron pruebas de reacción en cadena de la polimerasa cuantitativa empleando un termociclador StepOne (Applied Biosystems. Foster City, California, EE. UU.). Las curvas de calibración cubrieron un intervalo de 20ng a 51,2fg de ADN de A. agilis UMCV2 como templado, con un coeficiente de determinación (r2) de 0,997, y fueron seguidas de una curva de disociación con un solo máximo a los 83°C, lo cual muestra un patrón de amplificación lineal con un solo amplicón. La cuantificación del ADN de A. agilis UMCV2 en tejidos de M. truncatula con base en las curvas de calibración se hizo de manera similar, utilizando como templado 20ng de ADN proveniente de las muestras de tejidos vegetales.
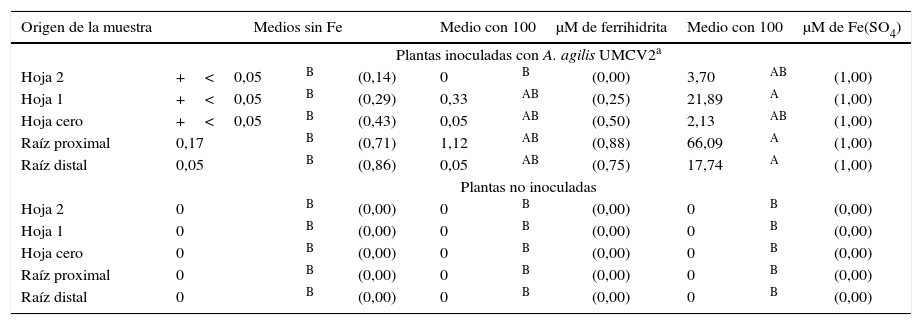
De esta forma se detectó a A. agilis UMCV2 en cada una de las secciones de las plantas inoculadas en donde se buscó (tabla 1), si bien no siempre se detectó en todas las réplicas. En plantas que fueron crecidas en un medio suplementado con Fe disponible (FeSO4) se encontraron las mayores cantidades de ADN bacteriano, en todos los órganos muestreados y en todas las réplicas, mientras que en las plantas crecidas en medio carente de Fe se registraron las menores cantidades de ADN bacteriano y no en todas las muestras (tabla 1). Un caso intermedio sucedió con las plantas crecidas en medios con Fe adicionado en forma insoluble (100μM de ferrihidrita), que tuvieron cantidades de ADN bacteriano numéricamente mayores aunque estadísticamente similares a las encontradas en plantas crecidas en medios sin Fe. En ninguna de las secciones de las plantas control, de ninguno de los tratamientos, se detectó ADN bacteriano (tabla 1).
Cantidad de ADN de A. agilis UMCV2 (pg) encontrada en 20ng de ADN extraídos de tejidos de M. truncatulaa
| Origen de la muestra | Medios sin Fe | Medio con 100μM de ferrihidrita | Medio con 100μM de Fe(SO4) | ||||||
|---|---|---|---|---|---|---|---|---|---|
| Plantas inoculadas con A. agilis UMCV2a | |||||||||
| Hoja 2 | +<0,05 | B | (0,14) | 0 | B | (0,00) | 3,70 | AB | (1,00) |
| Hoja 1 | +<0,05 | B | (0,29) | 0,33 | AB | (0,25) | 21,89 | A | (1,00) |
| Hoja cero | +<0,05 | B | (0,43) | 0,05 | AB | (0,50) | 2,13 | AB | (1,00) |
| Raíz proximal | 0,17 | B | (0,71) | 1,12 | AB | (0,88) | 66,09 | A | (1,00) |
| Raíz distal | 0,05 | B | (0,86) | 0,05 | AB | (0,75) | 17,74 | A | (1,00) |
| Plantas no inoculadas | |||||||||
| Hoja 2 | 0 | B | (0,00) | 0 | B | (0,00) | 0 | B | (0,00) |
| Hoja 1 | 0 | B | (0,00) | 0 | B | (0,00) | 0 | B | (0,00) |
| Hoja cero | 0 | B | (0,00) | 0 | B | (0,00) | 0 | B | (0,00) |
| Raíz proximal | 0 | B | (0,00) | 0 | B | (0,00) | 0 | B | (0,00) |
| Raíz distal | 0 | B | (0,00) | 0 | B | (0,00) | 0 | B | (0,00) |
Se muestran valores promedio.
La notación+< 0,05 indica muestras positivas para la prueba de detección de ADN bacteriano pero en una cantidad inferior al límite de cuantificación (0,05pg).
Los valores entre paréntesis expresan la proporción de muestras positivas. Las letras mayúsculas muestran diferencias significativas (p<0,05) según un análisis de varianza en rangos de Kruskal-Wallis seguido de una comparación múltiple por método de Dunns.
Para corroborar que el ADN bacteriano detectado provenía de bacterias con crecimiento endofítico y no de restos de bacterias adheridas a las superficies de las diferentes secciones vegetales, se realizaron fotografías de las superficies y planos interiores de los tejidos clarificados utilizando un microscopio confocal (FV1200, Olympus Corporation, Tokio, Japón). Las bacterias inoculadas fueron localizadas utilizando hibridación in situ con fluorescencia, de acuerdo al protocolo descrito por Lo Piccolo et al. (2010)5 empleando la sonda fluorescente HGC dirigida específicamente contra el ARNr de 23S de actinobacterias10 y unida al fluoróforo verde de rodanina (Uniparts SA de C.V., Galileo 92, México D.F, México). De esta manera fue evidente la presencia de la bacteria dentro de los tejidos de las plantas inoculadas, pero en ningún caso pudieron detectarse bacterias ni en la superficie de los tejidos ni en los tejidos internos de las plantas control (fig. 1).
Por otro lado, la sección de la planta con mayor cantidad de ADN bacteriano en todos los tratamientos fue la raíz proximal, que fue la parte de la planta sobre la cual se goteó la suspensión bacteriana al momento de la inoculación y su probable punto de penetración, seguida de la raíz distal y la hoja 1, y al final estuvieron la hoja cero y la hoja 2.
El anterior patrón de distribución del ADN bacteriano es compatible con la hipótesis de que las bacterias pudieron haberse distribuido por los tejidos aéreos de M. truncatula pasivamente a través del tejido vascular, más probablemente por el xilema, con un movimiento acropétalo (a favor de la corriente de transpiración), según ha sido descrito para el caso de Burkholderia phytofirmans y partículas de látex en Vitis vinicola2. Los vasos xilemáticos se comunican unos con otros en sus extremos a través de perforaciones con diámetros inferiores al diámetro bacteriano, lo que supone un obstáculo al paso de las bacterias de un vaso a otro2,14. En un estudio realizado en M. sativa14, se mostró que la mayoría de los vasos xilemáticos terminan en los nodos del tallo y solo una proporción muy pequeña ramifica desde los elementos vasculares del tallo hasta los pecíolos. De acuerdo a lo anterior, es explicable que las bacterias se encuentren impedidas de entrar pasivamente por el xilema hasta las hojas y que solo una pequeña fracción de la cantidad de ADN de A. agilis UMCV2 que encontramos en la raíz proximal fuera encontrada en la hoja 2, y solo en algunas réplicas.
Por otro lado, la presencia de ADN bacteriano en las raíces distales, teniendo en cuenta la escasa probabilidad de que las bacterias puedan penetrar pasivamente en el floema y ser llevadas por este en un flujo basipétalo (contra la corriente de transpiración), sugiere que la bacteria fue capaz de colonizar activamente los tejidos de la raíz y no que simplemente fue llevada por la corriente de transpiración, si bien en virtud de la cantidad de ADN hallada se concluye que la cantidad de bacterias que se movieron contra la corriente de transpiración fue entre 3 y 20 veces menor que las que se encontraron en la raíz proximal (tabla 1).
Fue muy interesante observar que la mayor cantidad de bacterias fue encontrada en los tejidos de plantas que crecieron en medios con Fe disponible, seguida de la observada en plantas crecidas con Fe insoluble y finalmente en plantas crecidas en medios sin Fe.
Se conoce que A. agilis UMCV2 es capaz de reducir Fe en la rizosfera, haciéndolo más soluble y por tanto más accesible a las plantas11, y que estimula los mecanismos de absorción y movilización de Fe en las plantas de M. truncatula9. Con base en estos antecedentes es posible hipotetizar que A. agilis UMCV2 y M. truncatula establecen una interacción positiva basada en la economía del Fe, donde la planta se vería beneficiada con la presencia de la bacteria en la rizosfera si el Fe estuviera en forma insoluble en el medio externo11, y se beneficiaría con la colonización endofítica si el Fe estuviera disponible en el interior de la planta. Ya en el interior de las raíces, parte del Fe debe ser movilizado a los tejidos aéreos, donde se conoce que A. agilis UMCV2 puede coadyuvar a la homeostasis de dicho metal1. En tales circunstancias es posible que las plantas favorezcan su colonización endofítica.
En conclusión, este trabajo muestra que A. agilis UMCV2 es capaz de colonizar y prosperar como bacteria endófita en M. truncatula, y que esta capacidad es mayor cuando el Fe se encuentra más disponible para la planta.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Agradecemos al fondo SEP-Conacyt-231585 (México) por el financiamiento de este trabajo y al Dr. León Ruíz-Herrera por haber tomado las fotografías en el microscopio confocal.