Alterações moleculares no endométrio eutópico de mulheres com endometriose podem estar envolvidas na infertilidade associada à doença. Este estudo objetivou comparar os genes diferencialmente expressos (DEG) no endométrio eutópico de mulheres inférteis com endometriose, controles inférteis (CI; fator masculino e/ou tubário) e controles férteis (CF) durante a janela de implantação, através de RNA‐Seq.
Material e métodosBiópsias endometriais foram obtidas de 17 pacientes (seis inférteis com endometriose, seis CI e cinco CF) durante a janela de implantação. O RNA total foi extraído e o RNA‐Seq foi feito na plataforma Illumina HISEQ 2500, high output, paired end. A normalização dos dados e a expressão diferencial foram conduzidas no ambiente estatístico R através do pacote DESeq2.
ResultadosOs grupos CI e CF foram semelhantes. Nenhum DEG foi identificado quando comparados os grupos CF e endometriose (independentemente do estágio da doença). Cinco DEGs (SCUBE1, CCL20, LGALS9C, TRIM 29 e WNT11) foram identificados no grupo endometriose avançada (EIII/IV) e um DEG (KANSL1‐AS1) no grupo endometriose inicial (EI/II), quando comparados com o CF. Dois DEGs (KANSL1‐AS1 e VGLL3) foram identificados com a comparação de EI/II e EIII/IV.
ConclusõesOs dados sugerem que o endométrio eutópico de mulheres inférteis com endometriose, especialmente na doença avançada, seja molecularmente diferente do endométrio eutópico de mulheres férteis durante a janela de implantação.
Molecular alterations in the eutopic endometrium of women with endometriosis may be involved in the endometriosis‐related infertility. This study aimed to compare the differentially expressed genes (DEG) in eutopic endometrium of infertile women with endometriosis, infertile controls (IC; male and/or tubal factor) and fertile controls (FC) through RNA‐Seq.
Material and methodsEndometrial biopsies were obtained from 17 patiens (6 infertile women with endometriosis, 6 IC and 5 FC) during the implantation window. The RNA was extracted and the RNA‐Seq was performed at a HISEQ 2500 Illumina Platform, high output, paired end. Standardization and differential expression were conducted in the statistical R environment using DESeq2 package.
ResultsThe groups IC and FC were similar. No DEG has been identified comparing CF and endometriosis groups. Five DEGs (SCUBE1, CCL20, LGALS9C, TRIM 29 e WNT11) were identified in the advanced endometriosis (EIII/IV) group, and 1 (KANSL1‐AS1) in the initial endometriosis (EI/II) group compared to FC. Two DEGs (KANSL1‐AS1 and VGLL3) were identified by comparing EI/II and EIII/IV groups.
ConclusionsThese data suggest that the eutopic endometrium of infertile women with endometriosis, especially those with advanced disease, may be molecularly different from those of fertile women during the implantation window.
A endometriose, caracterizada por implante e crescimento de tecido endometrial (glândulas e/ou estroma) fora da cavidade uterina,1,2 é uma doença de alta prevalência entre mulheres em idade reprodutiva,1 considerada um problema atual de saúde pública.3 Dentre suas manifestações clínicas mais frequentes, destaca‐se a infertilidade, há evidência de redução da fecundidade em grande parte de suas portadoras.4–7 Todavia, apesar de diversos estudos e hipóteses nos últimos anos, ainda não existe um consenso acerca dos mecanismos envolvidos no comprometimento da fertilidade natural em pacientes com endometriose, principalmente nos casos de doença inicial, em que não são observadas alterações anatômicas passíveis de justificar a infertilidade.8 Nesse sentido, diferentes autores têm sugerido que defeitos funcionais do endométrio possam estar envolvidos no comprometimento da implantação embrionária e participar da patogênese da infertilidade relacionada à doença.6,9–11
A implantação é um processo altamente controlado que depende de um adequado desenvolvimento embrionário e da chegada do embrião a um endométrio receptivo.12–14 O endométrio humano sofre mudanças histológicas, moleculares e funcionais ao longo do ciclo menstrual,15–17 está receptivo apenas durante determinado período da fase secretora média, definido como janela de implantação, é influenciado por uma variedade de moléculas sinalizadoras, com importante papel no estabelecimento da receptividade.16–20
A literatura apresenta diversos estudos que evidenciam alterações na expressão e/ou transcrição de moléculas no endométrio eutópico de mulheres com a doença.10,12,21 No entanto, a análise isolada de moléculas específicas não reflete necessariamente o conjunto de alterações que regem o comportamento do endométrio durante a janela de implantação. Nesse sentido, o sequenciamento de RNA, conhecido como RNA‐Seq, em que todos os transcritos de uma amostra são identificados,19,22 torna‐se particularmente adequado para a detecção de transcritos diferencialmente expressos em tecidos complexos, como o endométrio.19 Entretanto, até o presente, não há estudos que avaliem o endométrio eutópico de mulheres inférteis com endometriose através dessa metodologia, comparadas tanto com controles inférteis como férteis, o que possibilitaria um entendimento mais completo da janela de implantação dessas pacientes e contribuiria com a elucidação dos mecanismos envolvidos na etiopatogênese da infertilidade associada à doença.
Assim, o objetivo deste estudo foi avaliar o perfil diferencial de transcritos em endométrio eutópico de pacientes inférteis com endometriose (inicial e avançada) e controles inférteis e férteis durante a janela de implantação, através de RNA‐Seq.
Material e métodosDesenho do estudoFez‐se um estudo caso‐controle prospectivo junto ao Setor de Reprodução Humana do Departamento de Ginecologia e Obstetrícia da FMRP‐USP. Este estudo foi aprovado pelo Comitê de Ética em Pesquisa do Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto, Universidade de São Paulo (HCRP n° 6383/2011). As pacientes que preencheram os critérios de inclusão e manifestaram o desejo de participar do projeto assinaram o termo de consentimento livre e esclarecido previamente à inclusão no estudo.
De novembro de 2011 a novembro de 2014, as pacientes submetidas previamente aos procedimentos de videolaparoscopia diagnóstica ou laqueadura tubária no HCRP foram avaliadas de acordo com os critérios de elegibilidade e as consideradas elegíveis foram entrevistadas. As pacientes que aceitaram participar tiveram uma amostra endometrial coletada durante a janela de implantação.
Pacientes – Critérios de elegibilidadeForam consideradas elegíveis as pacientes entre 18 e 45 anos e com ciclos menstruais regulares (intervalos de 24 a 32 dias±três dias; dois a sete dias de duração e fluxo de até 80mL por ciclo)23 pelo menos nos três meses antes da coleta programada. Foram consideradas não elegíveis as pacientes com índice de massa corporal (IMC) maior ou igual a 30kg/m2, irregularidade menstrual, presença de doenças sistêmicas (diabetes mellitus ou outras endocrinopatias, doença cardiovascular, lúpus eritematoso sistêmico e outras doenças reumatológicas), infecção pelo vírus HIV ou qualquer infecção ativa, tabagismo, alcoolismo ou drogadição, uso de medicações hormonais e de anti‐inflamatórios hormonais e não hormonais nos três meses anteriores à inclusão no estudo. No grupo endometriose, foram incluídas as pacientes com infertilidade associada exclusivamente à doença, diagnosticada e classificada por videolaparoscopia (segundo os critérios da American Society for Reproductive Medicine, 1997). O grupo controle infértil foi composto por pacientes com infertilidade por fator masculino e/ou tubário submetidas à videolaparoscopia diagnóstica como procedimento de rotina para investigação da infertilidade conjugal, foi excluída a presença de endometriose. O grupo controle fértil foi composto por pacientes laqueadas, comprovadamente férteis (pelo menos um filho vivo), sem possíveis fatores endometriais associados.
Coleta e processamento das amostrasAs pacientes que fizeram os procedimentos de laqueadura tubária ou videolaparoscopia diagnóstica no HCFMRP‐USP tiveram seus prontuários avaliados de acordo com os critérios de elegibilidade e as consideradas elegíveis foram contatadas para o convite e a entrevista inicial. Ao concordar em participar do estudo, as pacientes tiveram as biópsias endometriais programadas para serem feitas durante a janela de implantação. Para a uniformização dos dados, o dia da ovulação foi considerado como o 14° dia de um ciclo menstrual de 28 dias e a janela de implantação foi considerada entre o 20° e 24° dia do ciclo. As coletas foram feitas no Laboratório de Ginecologia e Obstetrícia do HCFMRP‐USP, por médico com experiência em biópsia endometrial, com o uso de Pipelle de Cornier.
Imediatamente após a coleta, as amostras de endométrio foram divididas em pequenos fragmentos, uma parte foi destinada à análise histológica, mantida em solução de formaldeído 10% e posteriormente enviada ao Serviço de Patologia do HCFMRP‐USP para confecção das lâminas, e outra destinada à extração de RNA, mantida a 4°C por 24hs em criopreservador RNA‐Later (Ambion Life Technologies, Warrington, UK) e posteriormente armazenada a ‐80 C° até a extração do RNA total.
Datação endometrialPara a confirmação de que as biópsias endometriais foram feitas no período da janela de implantação, todas as amostras foram submetidas à análise histológica clássica com coloração hematoxilina‐eosina (HE)24 para as datações segundo critérios de Noyes25 por patologista com vasta experiência em datação endometrial (FMC).
Extração do RNA totalAs amostras foram lavadas em solução de PBS (1x) (NaCl 8,50g/L; Na2HPO4 1,11g/L; Na2HPO4.12H2O 2,81g/L; KH2PO4 0,20g/L pH7,0) para retirar o criopreservador dos tecidos e homogeneizadas com um homogeneizador de tecidos (Polytron). Em seguida, o RNA total (50mg de tecido) foi extraído com o kit RiboPure® (Ambion) e tratado com DNase (DNA free® kit – Ambion) de acordo com as instruções do fabricante. As concentrações de RNA total foram determinadas em espectrofotômetro NanoDrop à densidade óptica de 260nm. O RNA permaneceu armazenado à ‐800C até os procedimentos seguintes.
Expressão diferencial dos transcritosIntegridade do RNAA integridade do RNA total extraído foi avaliada no aparelho Agilent® Technologies 2100 Bioanalyzer™ (Agilent) segundo instruções do fabricante. Consideraram‐se adequadas as amostras de RNA com RNA Integrity Number (RIN) ≥ 7. As amostras íntegras foram quantificadas no fluorômetro Qubit® (Invitrogen) para verificar se apresentavam, ao menos, 1μg de RNA, quantidade mínima necessária para preparo das bibliotecas.
Preparo da biblioteca e sequenciamento (RNA‐Seq)Para o preparo das bibliotecas de RNA‐Seq usou‐se o kit TruSeq® RNA Sample Preparation v2 (Illumina), conforme instruções do fabricante.
O sequenciamento foi feito com o kit comercial TruSeq SBS kit v5 (Illumina Inc.), conforme instruções do fabricante. No total, 17 bibliotecas foram distribuídas em três lanes e sequenciadas paired end (PE 2×101pb) na Plataforma HISEQ2500 Illumina, através da corrida High Output.
Análise por bioinformáticaToda a análise foi feita no sistema operacional CentOS release 6.7.
Os arquivos com as sequências nucleotídicas e a descrição de qualidade das bases gerados pela plataforma Illumina HiSeq2500 (Illumina Inc.) foram analisados com o programa FastQC v0.10.1.26 A qualidade de cada lane da flow cell foi checada para cada amostra. Posteriormente, o mapeamento dos fragmentos gerados (reads) foi feito com TopHat v2.0.10,27 com o uso do GRCh37.2 como genoma de referência e a Release 104 do NCBI como anotação gênica. Para essa análise, os parâmetros usados foram: –max‐multihits 1, –splice‐mismatches 1 e–microexon‐search. Após o mapeamento, os arquivos com as informações finais do alinhamento das reads no formato binário bam foram convertidos para o formato sam com o uso do SAMtools v0.1.19‐44428cd.28
A quantificação das reads presentes nos genes foi feita com o HTSeqCount, selecionaram‐se apenas reads com qualidade de mapeamento maior do que 30.29 A normalização e a expressão diferencial entre os grupos a partir das contagens brutas foi feita no ambiente estatístico R (R Development Core Team, 2008) com o uso do pacote do DESeq2.30 No mesmo ambiente, foi feita a análise de componente principal (PCA) entre os grupos para checar o agrupamento das amostras conforme sua expressão global. Foram considerados genes diferencialmente expressos aqueles com valores de log2 (FoldChange)>1 ou<‐1 e p‐valor de<0,05.
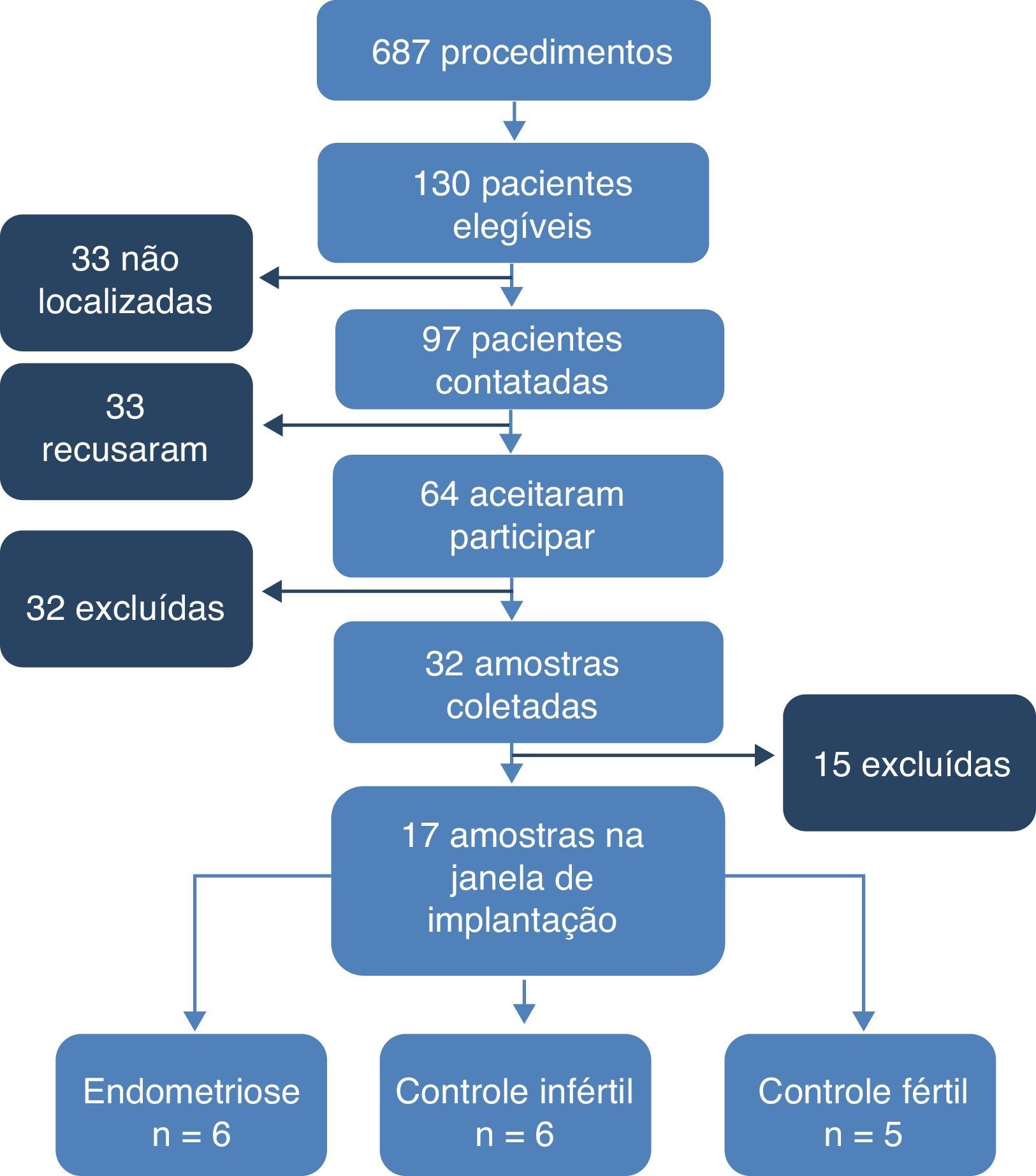
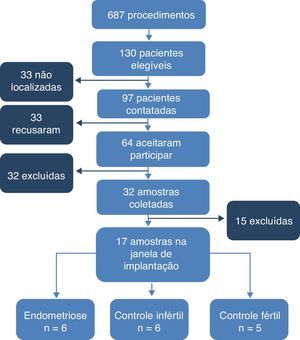
ResultadosFluxograma. Foram revisados 1.335 prontuários. Desses, 687 pertenciam a pacientes submetidas à videolaparoscopia diagnóstica (n=629) ou laqueadura tubária (n=58), foram consideradas elegíveis 130 pacientes. Dessas, 97 foram contatadas e 33 não foram localizadas. Das contatadas, 33 não aceitaram participar do estudo e 32 foram excluídas (nove por alterações no ciclo menstrual, 14 por fazerem uso de método contraceptivo hormonal, seis por receberem medicação hormonal e três por impossibilidade de comparecer à coleta na data programada). Assim, 32 mulheres tiveram amostras coletadas, 11 com endometriose (quatro grau I, dois grau II, dois grau III e três grau IV), 13 controles inférteis e oito controles férteis. Dessas, 15 amostras foram excluídas após a datação endometrial. Assim, 17 amostras foram consideradas na janela de implantação e incluídas no estudo, seis com endometriose (duas grau I, ima grau II, uma grau III e duas grau IV), seis controles inférteis e cinco controles férteis (fig. 1).
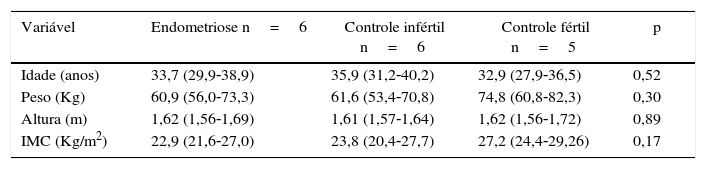
Características clínicas das pacientesOs grupos endometriose, controle infértil e controle fértil foram semelhantes em relação a idade, peso, altura e IMC (tabela 1).
Idade, peso, altura e IMC das pacientes inférteis com endometriose, controles inférteis e controles férteis
| Variável | Endometriose n=6 | Controle infértil n=6 | Controle fértil n=5 | p |
|---|---|---|---|---|
| Idade (anos) | 33,7 (29,9‐38,9) | 35,9 (31,2‐40,2) | 32,9 (27,9‐36,5) | 0,52 |
| Peso (Kg) | 60,9 (56,0‐73,3) | 61,6 (53,4‐70,8) | 74,8 (60,8‐82,3) | 0,30 |
| Altura (m) | 1,62 (1,56‐1,69) | 1,61 (1,57‐1,64) | 1,62 (1,56‐1,72) | 0,89 |
| IMC (Kg/m2) | 22,9 (21,6‐27,0) | 23,8 (20,4‐27,7) | 27,2 (24,4‐29,26) | 0,17 |
Nota: dados expressos como mediana (desvio interquartil).
IMC, índice de massa corporal. Teste de Kruskal‐Wallis. Nível de significância: 5%.
RNA‐Seq. Foram mapeadas 85% das reads, com phred‐score>30. Das reads mapeadas, 1,5% foram singleton e 1% tiveram múltiplos alinhamentos, as quais foram removidas da análise. A uniformidade das reads mapeadas em todas as amostras foi considerada boa.
A montagem dos transcritos e a normalização dos dados foram feitas com os métodos HTSeq e DESeq2 e uma média de 20.200 transcritos foi encontrada em cada amostra.
Nenhum transcrito diferencialmente expresso foi evidenciado quando comparados os grupos endometriose, controle infértil e controle fértil.
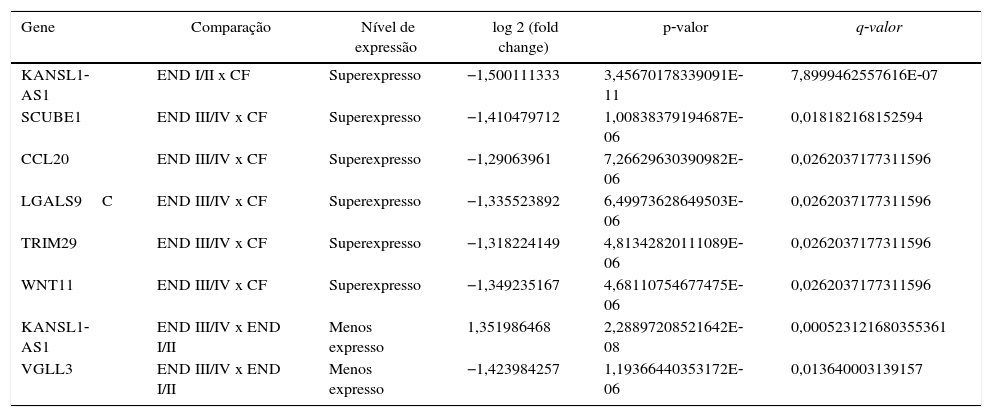
Ao se subdividir o grupo endometriose em estágios iniciais (I/II) e avançados (III/IV) e compará‐los com o grupo controle fértil, foram identificados sete genes diferencialmente expressos (DEGs) (tabela 2). Dois DEGs foram identificados ao serem comparados os grupos endometriose I/II e endometriose III/IV (tabela 2).
Genes diferencialmente expressos identificados em endométrio eutópico de mulheres inférteis com endometriose inicial (END I/II), endometriose avançada (END III/IV) e controles férteis (CF) na janela de implantação
| Gene | Comparação | Nível de expressão | log 2 (fold change) | p‐valor | q‐valor |
|---|---|---|---|---|---|
| KANSL1‐AS1 | END I/II x CF | Superexpresso | −1,500111333 | 3,45670178339091E‐11 | 7,8999462557616E‐07 |
| SCUBE1 | END III/IV x CF | Superexpresso | −1,410479712 | 1,00838379194687E‐06 | 0,018182168152594 |
| CCL20 | END III/IV x CF | Superexpresso | −1,29063961 | 7,26629630390982E‐06 | 0,0262037177311596 |
| LGALS9C | END III/IV x CF | Superexpresso | −1,335523892 | 6,49973628649503E‐06 | 0,0262037177311596 |
| TRIM29 | END III/IV x CF | Superexpresso | −1,318224149 | 4,81342820111089E‐06 | 0,0262037177311596 |
| WNT11 | END III/IV x CF | Superexpresso | −1,349235167 | 4,68110754677475E‐06 | 0,0262037177311596 |
| KANSL1‐AS1 | END III/IV x END I/II | Menos expresso | 1,351986468 | 2,28897208521642E‐08 | 0,000523121680355361 |
| VGLL3 | END III/IV x END I/II | Menos expresso | −1,423984257 | 1,19366440353172E‐06 | 0,013640003139157 |
A pesquisa de vias e dos termos do Gene Ontology referentes aos DEGs foi feita para identificar potenciais processos biológicos associados com receptividade endometrial. De acordo com a análise de enriquecimento, os DEGs identificados estão envolvidos nos processos biológicos de proliferação celular, vascularização, resposta inflamatória e imune e destino celular e nas vias de sinalização de quimiocinas, sinalização de citocinas e pluripotência.
DiscussãoApesar da forte associação entre endometriose e infertilidade, os mecanismos envolvidos na sua etiopatogênese, principalmente nos casos de doença mínima e leve, ainda não foram precisamente elucidados.8 A receptividade endometrial comprometida em pacientes com a doença tem sido considerada um possível fator envolvido na infertilidade apresentada por aproximadamente metade das suas portadoras. Como a implantação é um processo altamente controlado, a alteração na expressão de diferentes genes importantes para a modulação da receptividade endometrial comprometeria a fertilidade natural em mulheres com a doença. A literatura apresenta evidências de expressão anormal de diferentes moléculas envolvidas no processo implantacional nessas mulheres. No entanto, não dispomos de estudo que tenha avaliado o perfil diferencial de transcritos, através de sequenciamento de nova geração, em endométrio eutópico dessas pacientes, com a comparação com controles inférteis e férteis, o que seria essencial para uma compreensão mais ampla dos mecanismos etiopatogênicos da infertilidade associada à endometriose. Assim, propusemos uma avaliação abrangente do endométrio eutópico de mulheres inférteis com doença inicial e avançada durante a janela de implantação, é o primeiro estudo a usar a tecnologia de RNA‐Seq para essa finalidade.
Os resultados obtidos indicam não haver expressão diferencial de qualquer transcrito quando comparados os grupos controle infértil e controle fértil durante a janela de implantação, descartou‐se qualquer efeito isolado da infertilidade sobre a expressão gênica no endométrio eutópico das pacientes com e sem endometriose. Esse dado permitiu a comparação entre os grupos endometriose e controle fértil e afastou quaisquer dúvidas acerca da relação entre a doença e possíveis alterações encontradas no endométrio eutópico.
Quando comparadas mulheres inférteis com endometriose e controles férteis, não foram encontrados genes diferencialmente expressos entre os grupos, o que sugere, a princípio, que o endométrio eutópico dessas pacientes seja molecularmente semelhante ao de mulheres saudáveis. Esses dados corroboram estudo recente de Garcia‐Velasco et al.,31 que não evidenciou diferença no perfil receptivo de mulheres inférteis com endometriose quando comparadas com mulheres inférteis sem a doença, através da plataforma de microarray ERA.
No entanto, no presente estudo, a fim de se avaliar o efeito da progressão da doença sobre a fertilidade de suas portadoras, as pacientes inférteis com endometriose foram subdivididas entre os estágios iniciais (endometriose I/II) e avançados (endometriose III/IV), de modo que essa comparação identificou a existência de genes diferencialmente expressos quando comparadas com controles férteis e entre si, o que sugere que a doença tenha efeito distinto no endométrio eutópico dessas mulheres de acordo com seu estágio. O fato de se terem identificado DEGs quando subdividido o grupo endometriose em estágios iniciais e avançados e não terem se identificado diferenças quando avaliados conjuntamente, possivelmente se deve a um mascaramento dos resultados, uma vez que as diferenças entre eles podem ter se sobreposto. Um dado interessante é que mulheres com endometriose III/IV apresentaram maior número de genes diferencialmente expressos do que as pacientes com endometriose I/II quando comparadas com controles férteis, o que evidencia o efeito deletério do avanço da doença na receptividade endometrial. Assim, sugere‐se que Garcia‐Velasco et al.,31 possivelmente, não encontraram diferenças entre mulheres inférteis com e sem a doença por não terem subdividido o grupo endometriose de acordo com sua gravidade. Em estudo semelhante, Aghajanova et al.32 compararam o transcriptoma do endométrio eutópico entre mulheres com endometriose leve e avançada durante as fases proliferativa, secretora inicial e secretora média, através de microarray (plataforma Affimetrix), evidenciou‐se desregulação em transcritos no endométrio de pacientes com doença avançada na fase secretora média, alguns deles relacionados à receptividade endometrial, o que poderia comprometer a implantação em mulheres com endometriose avançada. Apesar de os autores terem incluído pacientes com dor pélvica e sem infertilidade associada, de não avaliarem exclusivamente a janela de implantação e de os transcritos relacionados serem distintos dos evidenciados no presente estudo, os resultados aqui encontrados corroboram esses autores no que se refere à existência de diferenças no endométrio eutópico de mulheres inférteis com endometriose, sobretudo nos estágios avançados.
De acordo com os presentes achados, quando comparado o grupo endometriose I/II com o grupo controle fértil, apenas o gene KANSL1‐AS1 foi diferencialmente expresso, sua expressão foi maior nas pacientes com a doença inicial. Apesar de já ter sido identificado e validado em outros estudos, a função, as vias e os processos biológicos desse gene ainda não foram descritos. Porém, ele parece ter uma participação importante no endométrio de mulheres com a doença, uma vez que também aparece mais expresso nessas pacientes em comparação ao grupo endometriose III/IV. Futuros estudos que elucidem a função, as vias e os processos biológicos com os quais esse gene esteja envolvido possibilitarão um melhor entendimento dessa alteração encontrada no endométrio de mulheres com doença inicial.
Já quando comparado o grupo endometriose III/IV com o grupo controle fértil, cinco DEGs foram identificados (SCUBE1, CCL20, LGALS9C, TRIM 29 e WNT11). Embora não pertençam a uma mesma via, esses genes parecem estar envolvidos na patogênese da doença e podem ter importante papel na receptividade endometrial, uma vez que, na sua maioria, são relacionados a processos biológicos de proliferação celular, vascularização, resposta inflamatória e imunológica e destino celular, estão envolvidos em vias de sinalização de quimiocinas e citocinas. Nesse sentido, sabe‐se que os sistemas imune e endócrino têm papel chave no controle da função endometrial, os hormônios ovarianos influenciam fatores imunológicos no endométrio a fim de protegê‐lo de infecções e prepará‐lo para a implantação.33 Os eventos endometriais chave para esse processo são a modificação da matriz extracelular, o aumento da permeabilidade vascular,33 a aumentada expressão de quimiocinas e citocinas, a decidualização e a presença aumentada de leucócitos,34 que, juntamente com fatores de crescimento, interagem para propiciar um ambiente adequado à implantação embrionária que envolvem moléculas pró e anti‐inflamatórias e medeiam a aposição, adesão e invasão do endométrio pelo blastocisto.33 Assim, a manutenção de um estado inflamatório adequado é fundamental para o sucesso da implantação, de modo que uma resposta inflamatória exacerbada ou comprometida pode levar a problemas de fertilidade.33 Dessa forma, a análise individual dos genes alterados nas pacientes com doença avançada sugere um potencial comprometimento da receptividade endometrial, com base na sua relação com esses eventos. No entanto, essa hipótese precisa ser confirmada em estudos com metodologias apropriadas.
Quando comparados os grupos EI/II e EIII/IV, o gene KANSL1‐AS1 mostrou‐se menos expresso e o gene VGLL3 mais expresso nas pacientes com doença avançada. O gene KANSL1‐AS1 ainda não foi descrito na literatura, enquanto que o VGLL3 tem poucos dados com relação ao seu papel em células normais ou câncer.35 Sabe‐se que, em mamíferos, o VGLL3 está relacionado com o cofator de transcrição vestigial (originalmente descrito em Drosophila melanogaster), codifica cofatores da família de fatores de transcrição TEAD35 e parece estar envolvido em vias de supressão tumoral em câncer de ovário.36 Apesar de serem poucas as informações a respeito da função biológica desse gene e de não haver nada descrito com relação ao endométrio, considerando seu possível papel como um cofator de transcrição, pode‐se questionar se sua expressão aumentada em pacientes com doença avançada comparadas com pacientes com doença inicial indica um potencial envolvimento no estabelecimento e avanço da doença e na regulação da expressão de outros genes, talvez aqueles encontrados com expressão aumentada no grupo endometriose III/IV em comparação com o controle fértil.
A principal limitação deste estudo foi a pequena casuística avaliada em decorrência dos critérios de elegibilidade restritivos adotados, o que limita a generalização dos resultados aqui obtidos. Por outro lado, os critérios de elegibilidade rigorosos, que eliminaram fatores potencialmente relacionados com comprometimento da receptividade endometrial, foram necessários para aumentar a validade interna do estudo.
Resumidamente, a comparação entre os grupos endometriose, controle infértil e controle fértil não evidenciou genes diferencialmente expressos no endométrio eutópico dessas pacientes durante a janela de implantação. Isso sugere que o endométrio eutópico de mulheres inférteis por fator tubário/masculino (controle infértil) não influencia a expressão de genes endometriais e que, quando avaliado o endométrio eutópico de mulheres inférteis com endometriose sem considerar a progressão da doença, esse se apresenta semelhante ao de mulheres saudáveis. No entanto, os dados preliminares obtidos a partir da subdivisão do grupo endometriose em estágios iniciais e avançados sugerem que haja alterações moleculares significativas, evidenciadas por diferenças no perfil de expressão de alguns genes, no endométrio eutópico dessas pacientes, sobretudo na endometriose avançada. Esses achados reforçam a importância de, em estudos futuros que investiguem o papel do comprometimento da receptividade endometrial na etiopatogênese da infertilidade relacionada à endometriose, se avaliar separadamente essas mulheres de acordo com a severidade da doença. Embora não pertençam a uma mesma via, os genes diferencialmente expressos identificados no endométrio eutópico de pacientes inférteis com doença avançada parecem estar envolvidos na patogênese da doença e podem ter importante papel na receptividade endometrial. Caso, em estudos com metodologias apropriadas, seja comprovado que esses genes participam do processo de implantação embrionária, o comprometimento da receptividade uterina poderá ser elucidado como um dos mecanismos envolvidos na etiopatogênese da endometriose III/IV.
FinanciamentoConselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq: 471943/2012‐6; 140137/2015‐7) e Fundação de Amparo à Pesquisa do Estado de São Paulo (Fapesp: 2011/17614‐6).
Conflitos de interesseOs autores declaram não haver conflitos de interesse.
Estudo vinculado à Universidade de São Paulo (USP), Faculdade de Medicina de Ribeirão Preto, Ribeirão Preto, SP, Brasil.