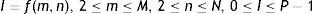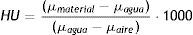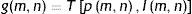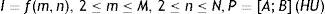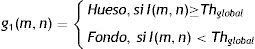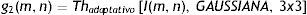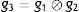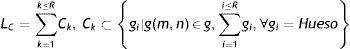El tejido óseo es el elemento principal del esqueleto. Sirve de soporte a las partes blandas, protege órganos vitales y constituye un sistema de palancas que amplifica las fuerzas generadas durante la contracción muscular.
La descripción del comportamiento mecánico del tejido óseo, mediante el empleo de modelos discretos, pasa por diferentes etapas de análisis, las cuales van desde el procesamiento digital de la imagen hasta la especificación de las propiedades físicas del tejido al modelo discreto, siendo la descomposición de estos modelos en sus partes constitutivas un elemento clave.
En la presente investigación se discute un método para la descripción geométrica de los huesos a partir de una secuencia de imágenes de tomografía computarizada y basado en la combinación de la umbralización global y adaptativa.
Los resultados obtenidos demostraron que este método constituye una propuesta efectiva al problema del volumen parcial y la separación de los huesos en las articulaciones.
Bone is the main element of the skeleton. It support soft tissues, protects vital organs and constitutes a lever system that amplifies forces generated during muscular contraction.
A description is presented of the mechanical behavior of hard tissues by means of discrete models going through various stages of analysis, which range from digital image processing until the specification of physical properties of tissue to the discrete model. The decomposition of these models into their constituent parts being a key element.
In this paper, we discuss a method for the geometric description of bones from a sequence of computed tomography images, combining global and adaptive thresholding to determine the geometric domain of bones in each slice.
Resultsobtained showed that this method constitutes an effective proposal for the problem of partial volume and separation of bones on joints.
El tejido óseo es el elemento principal del esqueleto. Sirve de soporte a las partes blandas, protege órganos vitales y constituye un sistema de palancas que amplifica las fuerzas generadas durante la contracción muscular.
En función de su densidad, se clasifica en tejido óseo compacto (cortical) y tejido óseo esponjoso (reticulado, trabecular).
El tejido óseo cortical se encuentra en la superficie de los huesos, y es muy denso y fuerte; como resultado, tiene un mayor coeficiente de absorción electromagnética que los tejidos óseos esponjosos y cancerosos.
En las imágenes de tomografía computarizada (TC) el tejido óseo cortical generalmente se observa como un borde brillante que define el contorno exterior del hueso. En el interior de este, los tejidos óseos esponjosos y cancerosos inducen un determinado patrón de textura que provoca variaciones de la intensidad1.
Si bien existen modelos tridimensionales que reproducen con precisión los detalles visuales de las estructuras anatómicas (aceleran la compresión de la forma y posición espacial), estos distan de ser útiles para el análisis numérico por elementos finitos, al carecer de la definición de las estructuras anatómicas como entidades geométricas únicas.
Entre los campos de la investigación médica donde es necesaria la definición del dominio geométrico se encuentran: la visualización tridimensional2, las mediciones volumétricas3,4, la cirugía dirigida por imagen5-8 y la detección de cambios anatómicos con el tiempo9.
En la última década se han propuesto múltiples métodos para la obtención del dominio geométrico de estructuras óseas a partir de un conjunto de imágenes médicas.
Hirano y Hata10 proponen la aplicación de un sistema experto basado en lógica Fuzzy para la segmentación y la determinación de los huesos y sus fragmentos en fracturas del pie, considerando para ello 2 tipos de conocimiento: el de la unión y el de la distancia.
Hoad y Martel8 utilizan el crecimiento de regiones y el filtrado morfológico para segmentar la espina lumbar en imágenes de resonancia magnética (RM).
Sebastian et al.11 combinan técnicas de contornos activos, crecimiento y competición de regiones para la segmentación de los huesos del carpo en imágenes TC.
Mastmeyer et al.12 introducen una técnica semiautomática para la segmentación y delimitación de las vértebras con la selección manual de las vértebras en las imágenes TC, asumiendo que las mismas son cilindros elípticos, ajustados mediante la aplicación de operaciones booleanas hasta definir las vértebras con el empleo del modelo de balón en el dominio 3D, propuesto por Cohen13,14.
Liu et al.15 proponen una técnica de separación interactiva de los huesos del pie (en el plano). Se empieza con la selección manual de un píxel semilla en cada hueso a segmentar, y posteriormente aplican la técnica de graph-cut para obtener los contornos de los huesos en cuestión.
Liu et al.16 emplean la segmentación livewire para determinar los huesos.
Kose et al.17 utilizan métodos de histograma, segmentación estadística y una plantilla triangular que aplican a la imagen para la detección de los desgarros del menisco en imágenes RM.
Zhang et al.18 proponen el empleo de umbrales adaptativos 3D para la identificación de los huesos. Posteriormente, mediante el crecimiento de regiones en 3D se extrae la región específica del hueso.
Schmid et al.19 determinan los huesos presentes en imágenes RM de baja calidad mediante la comparación de modelos ideales de la región de análisis con las imágenes originales.
Calder et al.1 utilizan conjuntos de nivel para la segmentación de imágenes del pie, la muñeca y el tobillo.
Los principales problemas aún sin resolver para la correcta definición del dominio geométrico de estructuras anatómicas son:
- 1.
El efecto del volumen parcial, debido a la insuficiente resolución espacial del equipo de adquisición, al movimiento del paciente o a la presencia de artefactos.
- 2.
La separación de los huesos adyacentes.
- 3.
El ruido presente en la imagen.
El efecto de volumen parcial se observa cuando el nivel de intensidad de algunos píxeles representa a 2 o más tipos de tejido.
Como consecuencia, existen píxeles que no pueden ser clasificados, en función de su intensidad, en un tipo de tejido específico, lo que incide en la correcta delimitación de los huesos adyacentes.
Para dar solución a este problema, se propone un algoritmo semiautomático basado en la combinación de la umbralización global y la adaptativa. Esto permite la extracción de los volúmenes óseos, como entidades geométricas únicas, para el estudio biomecánico.
Materiales y métodoImagen DICOMEl formato DICOM (Digital Imaging and Communications in Medicine) es empleado para producir, mostrar, almacenar, enviar, procesar, obtener, consultar e imprimir imágenes médicas. Cada imagen obtenida (ecuación 1) representa un corte de la parte del cuerpo analizada; en estas, cada píxel denota el grado de atenuación del haz radiológico sobre el tejido humano. La distancia entre píxeles (Dp) y entre cortes (Dc) son atributos que están presentes como un elemento de dato en los ficheros DICOM y hacen posible cualquier medición en distancias reales20.
En las imágenes TC, la intensidad de la señal espacial está dada por el coeficiente de atenuación lineal μ. Este coeficiente es específico de cada sustancia o materia y expresa la atenuación que sufre un haz de rayosX al atravesar una determinada longitud de una sustancia dada.
La unidad de Hounsfield (HU) es una transformación lineal del coeficiente de atenuación lineal μ, donde se considera 0HU la radiodensidad del agua destilada (a presión y temperatura estándar, STP), mientras que la radiodensidad del aire a STP es –1.000HU. Cada material posee un coeficiente de atenuación lineal único, y su valor en HU está dado por la ecuación 2:
Recientemente se ha demostrado que el nivel de HU de un material está en función de la intensidad de las radiaciones emitidas por el equipo durante el proceso de adquisición de la imagen21-23. Por tanto, mientras mayor es la energía emitida por el equipo, menores serán los valores de HU para un mismo material.
UmbralizaciónPara la determinación de los contornos que representan los huesos es necesario etiquetar y agrupar los píxeles cuyas intensidades están en un rango o función matemática determinada; esto es conocido como umbralización.
De forma general, la umbralización se define como una operación en la que se evalúa, en una funciónT, el nivel de intensidad de cada píxel con respecto a una condición de umbralización p (ecuación 3), obteniéndose como resultado una nueva imagen g.
Las técnicas de umbralización han sido ampliamente utilizadas en la segmentación de imágenes debido al nivel de información que pueden ofrecer, pero al mismo tiempo son subjetivas en su aplicación, no tienen en cuenta las características espaciales de la imagen y son sensibles a inhomogeneidades de la intensidad y al ruido. Se dividen en 2 clases: global y local (adaptativa).
En la umbralización global se emplea un mismo valor de umbral para toda la imagen. Como resultado se obtiene una imagen binaria con los píxeles cuyo nivel de intensidad cumple con el criterio de umbralización.
La umbralización adaptativa consiste en subdividir la imagen en pequeñas áreas (subimágenes), y se utiliza una función estadística para calcular el nuevo valor del píxel central en cada subimagen. Como resultado, se realzan los contornos presentes en la imagen.
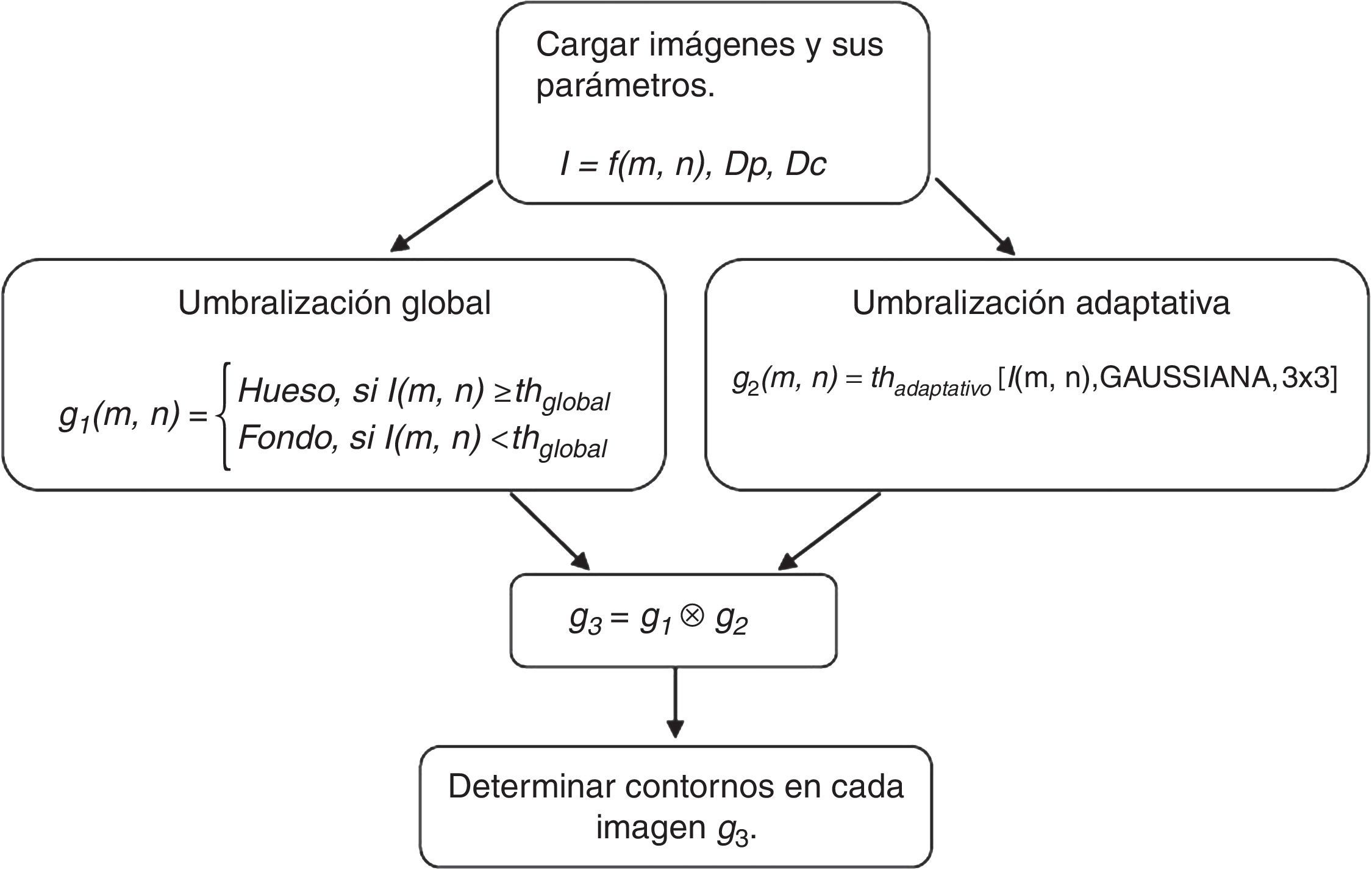
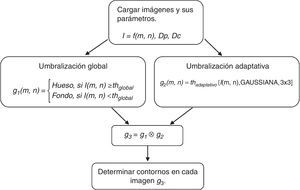
Propuesta de umbralizaciónSe propone la combinación de la umbralización global y adaptativa seguida de la detección de contornos (fig. 1).
El algoritmo tiene como entrada el conjunto de imágenes DICOM (ecuación 4) que representan la información correspondiente a la región anatómica de análisis, donde el intervalo [A; B] es el rango del coeficiente de atenuación (HU) obtenido durante el proceso de adquisición de la imagen.
Para clasificar cada píxel como hueso o fondo se aplica a cada imagen la umbralización global (ecuación 5), obteniéndose una nueva imagen g1.
La especificación del valor de umbralización global (Thglobal) es un aspecto importante en la identificación de los píxeles que representan los huesos. Un nivel de umbral demasiado bajo produce la unión de regiones que pertenecen a huesos distintos, mientras que si es demasiado alto fallará la región del hueso debido a las inhomogeneidades de la intensidad (no se obtienen contornos cerrados).
Para realzar los contornos presentes en la imagen I se aplica la umbralización adaptativa (ecuación 6). El nuevo valor de intensidad de cada píxel se calcula mediante la distribución gaussiana, con un tamaño de subimagen de 3×3, obteniéndose la imagen g2.
La imagen binaria g3, que define el área que ocupa cada hueso en la imagen, es el resultado de la intersección (ecuación 7) de las imágenes g1, g2.
La ventaja de combinar ambas umbralizaciones radica en que si un píxel es identificado como hueso en la umbralización global pero no es parte de un contorno, este no aparecerá al aplicar la umbralización adaptativa; por ende, al multiplicar ambas imágenes este píxel no aparece en la imagen resultante.
La descripción geométrica del exterior de los huesos (contornos) se define mediante el algoritmo propuesto por Suzuki24, obteniéndose por cada imagen segmentada una lista de contornos (ecuación 8).
donde: Ck contorno.R dimensiones de la imagen R=M·N.
Imágenes empleadasLas imágenes utilizadas en la presente investigación fueron obtenidas mediante TC, y secorresponden con las regiones anatómicas de la rodilla, el hombro y la pelvis:
- •
Hombro, 43 cortes de 512×512píxeles, con un tamaño de vóxel de 0,97×0,97×1,5mm, con un rango de intensidad de [–1.024,0; 1.607] (HU), adquiridos utilizando un escáner Siemens Sensation 16 con una energía de 120Kvp y 238mA.
- •
Cadera, 355 cortes de 512×512píxeles, con un tamaño de vóxel de 0,66×0,66×0,7mm, con un rango de intensidad de [–1.024,0; 1.607] (HU), adquiridos utilizando un escáner Philips Mx8000 IDT 16 con una energía de 140Kvp y 272mA.
- •
Pie: 110 cortes de 512×512píxeles con un tamaño de vóxel de 0,74×0,74×1,0mm3, con un rango de intensidad de [–1,024,0; 1.238,0] (HU), adquiridos mediante un escáner Siemens Sensation 16 con una energía de 120Kvp y 345mA.
Se implementaron 2 aplicaciones: la primera basada en el algoritmo propuesto en el epigráfe «Propuesta de umbralización» y la segunda omitiendo los pasos de umbralización adaptativa e intersección de imágenes. Como librería se utilizó Visualization Toolkit (VTK)25, y para la manipulación de los modelos geométricos, pyFormex26.
El hardware utilizado fue el procesador Core I7 2.67GHz con 4GB de RAM.
La construcción tridimensional de cada isosuperficie (cerrada y definida por una malla de triángulos) se realiza empleando el algoritmo Marching Cubes27, teniéndose en cuenta, para una correcta representación espacial, los valores de la distancia entre cortes Dc y la distancia entre píxeles Dp.
Los estudios anatómicos fueron procesados por ambas aplicaciones, obteniéndose los dominios geométricos correspondientes; como métrica de análisis se consideraron los huesos adyacentes identificados correctamente.
Para clasificar cada píxel como hueso y no hueso, el valor de umbralización global (Thglobal) se especificó manualmente, fijándose un nivel de confianza del 95% partiendo de la selección de un intervalo de valores, en la escala de Hounsfield, dentro del cual se encontraban los huesos.
Para cada estudio los intervalos escogidos fueron: articulación del hombro [80; 120], articulación de la cadera [80; 120] y pie [150; 200].
Al valor óptimo de Thglobal se arribó mediante aproximaciones sucesivas hasta obtener una correcta definición del hueso; en cada aproximación la variación fue de 10HU.
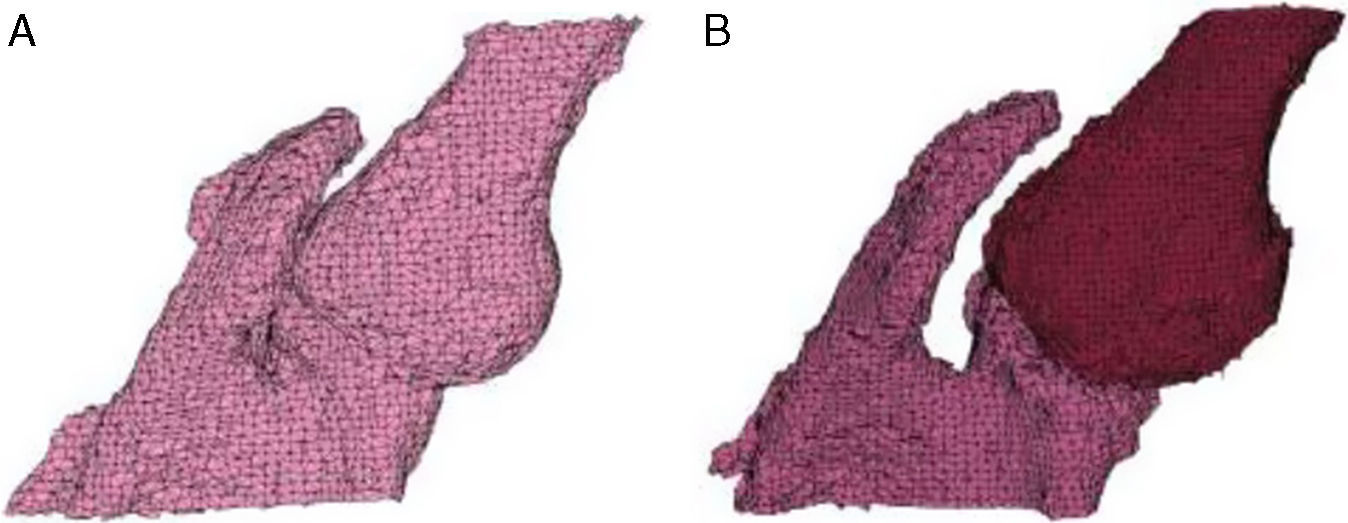
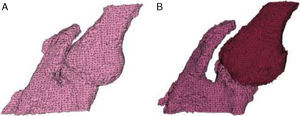
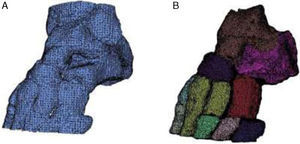
Discusión de los resultadosEn el estudio del hombro, al aplicar solamente la umbralización global (fig. 2a) no fue posible la separación del húmero de la escápula.
La combinación de ambas técnicas de umbralización (fig. 2b) permitió la obtención de la articulación completa. En ambos casos, Thglobal=100HU.
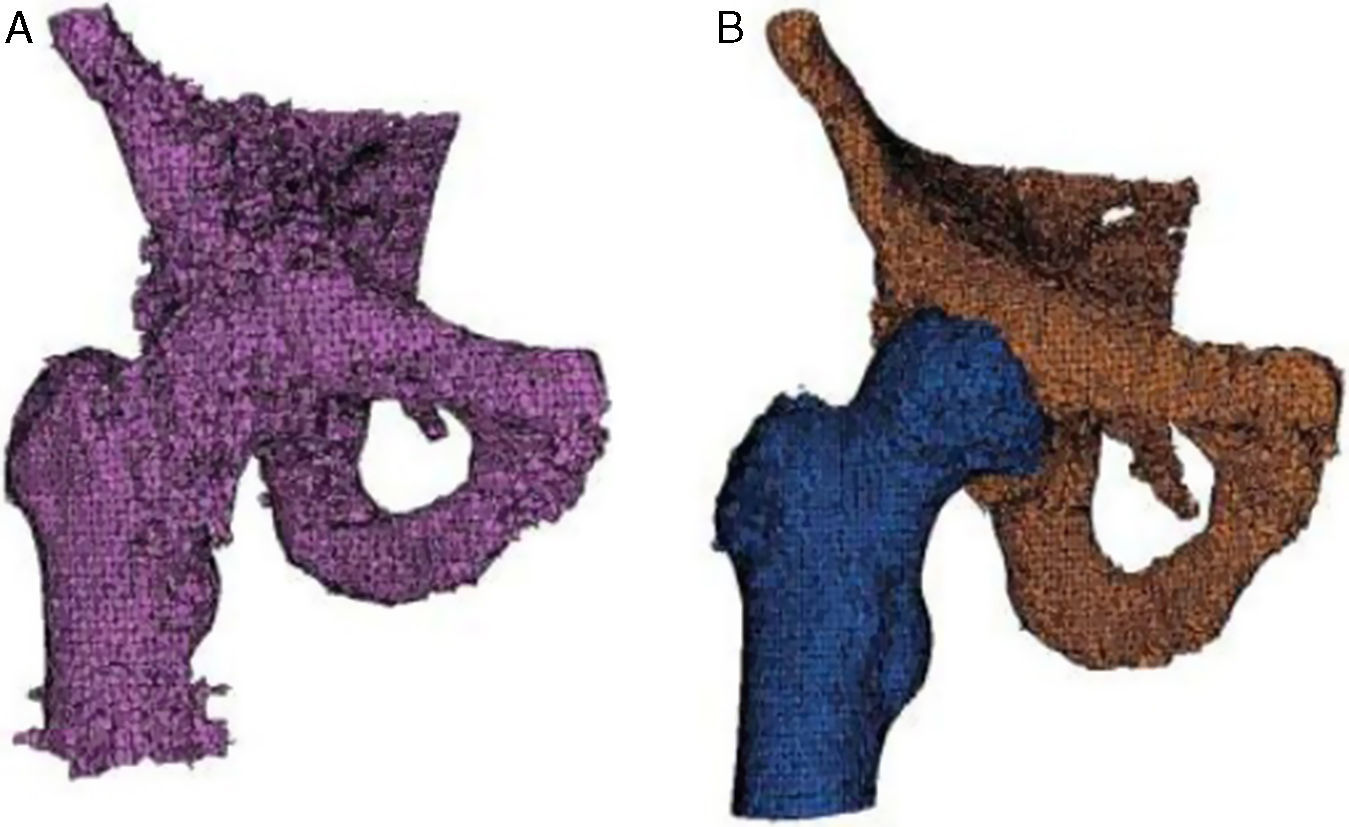
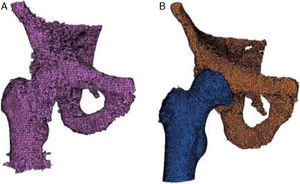
Para la obtención del dominio geométrico de la cadera, Thglobal=80HU. En este caso, al igual que el anterior, aplicando solamente la umbralización global no fue posible obtener los huesos de esta articulación (fig. 3a).
Al combinar de ambas técnicas se obtuvo por separado el fémur y el hueso iliaco (fig. 3b).
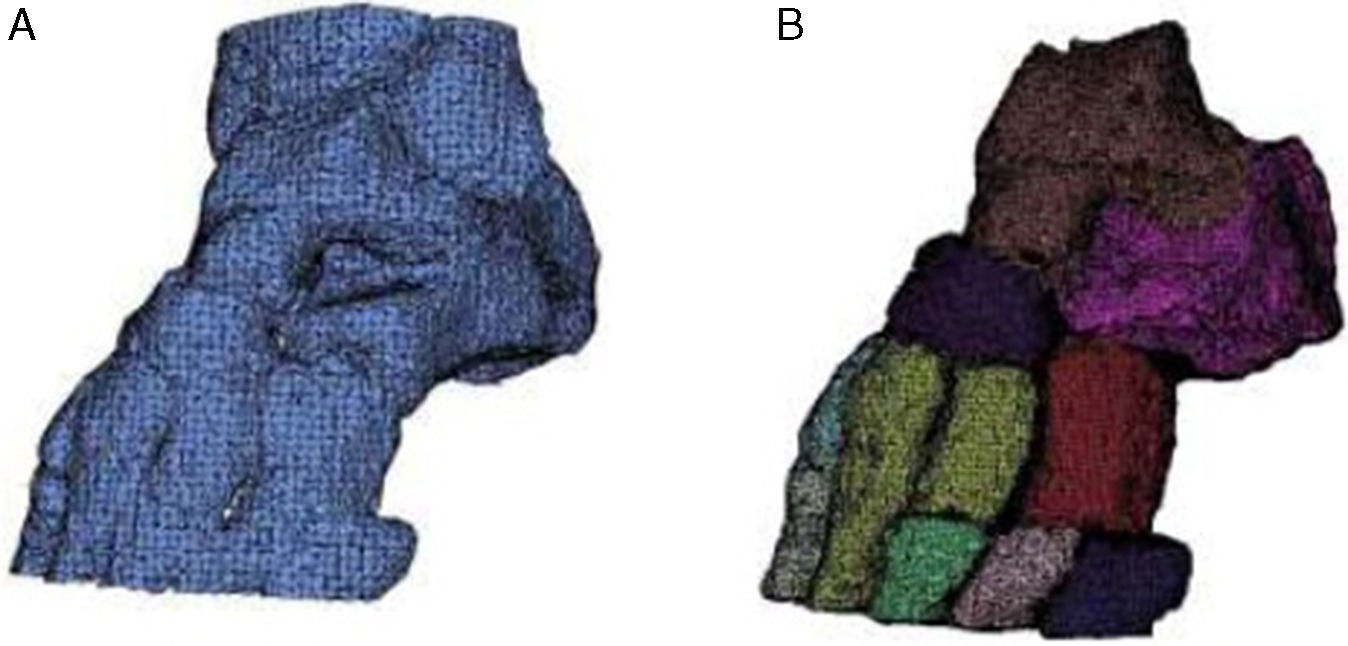
En el caso del pie, al aplicar solamente la umbralización global (fig. 4a) no se obtuvo ningún hueso por separado.
Sin embargo, a pesar de la pequeña separación de los huesos en el pie, la aplicación del algoritmo propuesto obtuvo buenos resultados (fig. 4b). Se definieron todos los huesos del tarso, excepto el segundo y tercer cuneiforme además del segundo metatarso. En ambos casos, Thglobal=180HU.
La ambigüedad en la definición de los contornos de los huesos (y por consiguiente de los volúmenes óseos) generalmente ocurre debido a la baja resolución de las imágenes o a cambios degenerativos que pueden provocar la anquilosis (fusión espontanea articular), o sea, se unen las articulaciones y en las imágenes de TC varios huesos aparecen como una sola entidad28. Esta fusión puede ser realizada de forma quirúrgica (artrodesis).
Se demostró que la combinación de la umbralización global y la adaptativa constituye un método efectivo para dar solución al problema del volumen parcial y la separación de los huesos en las articulaciones.
El desempeño del algoritmo propuesto se encuentra en función de la amplitud del rango de Hounsfield de la imagen. Las imágenes con mayor amplitud implican la presencia de implantes metálicos, que son complejos de atenuar o eliminar, por lo que la interacción manual aún continúa aportando robustez y eficacia.
En cuanto al tiempo de procesamiento, este fue de 5min como promedio por cada estudio.
Los resultados obtenidos validan la efectividad del algoritmo propuesto para ser empleado como núcleo funcional en la obtención del dominio geométrico de estructuras anatómicas.
Una línea de investigación futura es la determinación del nivel de umbral global en función de las características del sistema de adquisición de imagen.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.