Los microorganismos del género Desulfovibrio son bacilos gramnegativos anaerobios estrictos, curvados y espirilados, con capacidad de reducir los compuestos con azufre a sulfuro de hidrógeno. Presentes en el medioambiente y forman parte de la flora del tracto gastrointestinal humano1. Se han descrito, raramente, casos de bacteriemias primarias (traslocación intestinal) y de infecciones intraabdominales, habitualmente, por Desulfovibrio desulfuricans (D. desulfuricans) y Desulfovibrio fairfieldensis2. Son microorganismos de crecimiento muy lento en anaerobiosis y con resistencia intrínseca a antimicrobianos betalactámicos, incluyendo piperacilina/tazobactam3. Desulfovibrio porci (D. porci) es una nueva especie descrita recientemente (https://lpsn.dsmz.de/species/desulfovibrio-porci) en el tracto gastrointestinal del cerdo4, no habiendo casos descritos de infecciones en humanos.
Presentamos 2 casos de bacteriemia por D. porci en pacientes con enterocolitis y sepsis.
Caso 1. Mujer de 65 años trasplantada hepática en tratamiento con micofenolato y tacrolimus, que acude a urgencias en agosto de 2023 tras 2-3 días de diarreas sin productos patológicos, y vómitos, con antecedentes de trasplante hepático en diciembre de 2014. A la exploración se objetiva que no presenta fiebre pero sí deshidratación y taquipnea, abdomen blando y depresible, a la vez que presenta dolor abdominal tipo cólico y oligoanuria, sin datos de peritonismo, acidosis metabólica e insuficiencia renal aguda. En la TAC hay signos claros de enterocolitis. Se inicia en urgencias fluidoterapia y antibioterapia con piperacilina/tazobactam. Desarrolla shock séptico de origen abdominal, ingresando en UCI con mala evolución con fallo multiorgánico y, finalmente, éxitus, diagnosticándose como shock séptico por enterocolitis infecciosa, falleciendo en menos de 24 horas desde el ingreso hospitalario.
Caso 2. Varón de 64 años, sin antecedentes de interés, que acude a urgencias en enero de 2022 por dolor abdominal y diarrea sin productos patológicos de 5 días de evolución, un episodio de rectorragia autolimitada e insuficiencia renal aguda. No refiere fiebre. A la exploración, se aprecia dolor difuso abdominal, sin sangrado activo en el tacto rectal, deshidratación, taquipnea, abdomen distendido sin peritonismo, anuria y acidosis metabólica. En la TAC hay signos claros de enterocolitis. Desarrolla shock séptico de origen abdominal, instaurándose tratamiento con piperacilina/tazobactam y evolucionando mal con hipotensión refractaria, anuria, fallo hemodinámico y cardiaco derivando en éxitus en menos de 24 horas a pesar del tratamiento antibiótico en observación de urgencias, por shock séptico refractario y enterocolitis.
En ambos casos, en los hemocultivos extraídos en urgencias (sistema BACTEC FX, Becton-Dickinson, 2 sets que constan de un frasco aerobio, BACTEC Plus Aerobic/F y uno anaerobio, BACTEC Lytic/10 Anaerobic/F) hubo detección de crecimiento en un frasco anaerobio en el 5.° día de incubación, observándose en la tinción de Gram bacilos gramnegativos curvados, con poca afinidad por el contracolorante. Se subcultivaron en placas de agar sangre Columbia y agar chocolate (ThermoFisher) a 35°C en atmósfera aerobia con 5% de CO2 y placas de Schaedler (ThermoFisher) a 35° en atmósfera anaeróbica (Genbox anaer, BioMerieux), creciendo en las placas de anaerobiosis a las 72 horas de incubación, unas colonias muy pequeñas puntiformes, transparentes, de bordes lisos y no hemolíticas. La identificación con espectrometría de masas (MALDI-TOF, Bruker), no proporcionó ningún resultado.
En los 2 casos se hizo estudio microbiológico de las heces, en uno cultivo bacteriológico y en el otro detección molecular mediante PCR múltiplex (QIAstat-Dx Gastrointestinal Panel 2, Qiagen), no detectándose ningún patógeno.
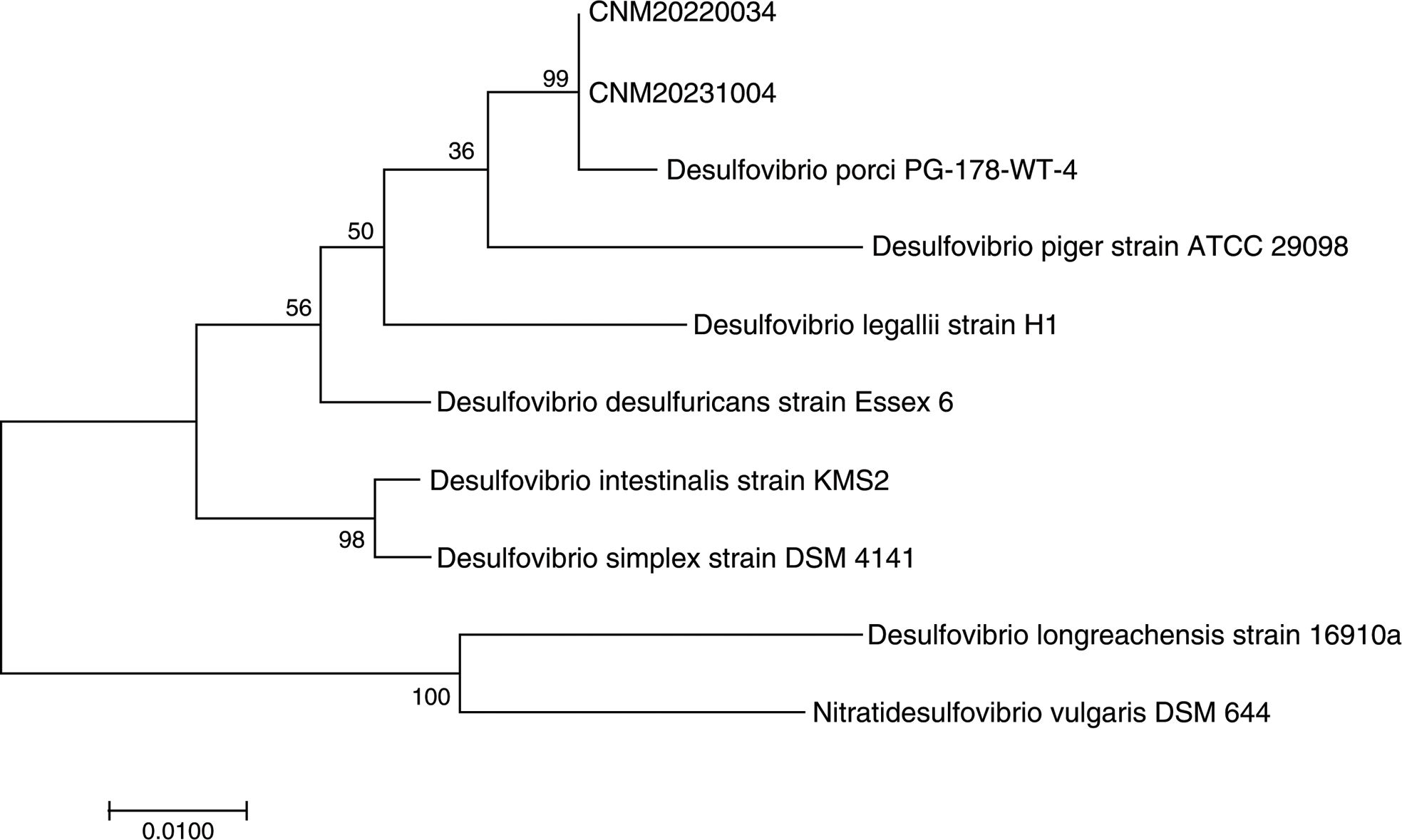
Para su asignación taxonómica se remitió la cepa al Laboratorio de Referencia e Investigación en Taxonomía del Centro Nacional de Microbiología (Instituto de Salus Carlos III). La identificación de las cepas se lleva a cabo mediante análisis de la secuencia del gen 16S rARN utilizando E781 y U1115 como primers y el secuenciador Applied Biosystems DNA Analyzer 3730xl. Para la cepa CNM20220034 (caso 1), se obtiene una secuencia de 1304pb que, al ser comparada con las secuencias de las cepas tipo depositadas en la base de datos de GeneBank (https://blast.ncbi.nlm.nih.gov/Blast.cgi), presenta un porcentaje de identidad del 99,2% con la cepa tipo de D. porci (PG-178-WT-4). Para la cepa CNM20231004 (caso 2), se obtiene una secuencia de 1409pb con un porcentaje de identidad del 99,2% (Figura). El alineamiento de las secuencias de CNM20220034, CNM20231004 y de la cepa tipo de D. porci utilizando Clustal W (DNAStar Lasergene 15), muestra un porcentaje de identidad del 100% entre las dos cepas.
Árbol filogenético realizado con las secuencias del 16S rARN (1245 bp) de las cepas CNM20220034 (caso 1) y CNM20231004 (caso 2) y con respecto a las cepas tipo de las diferentes especies relacionadas del género Desulfovibrio (MEGA7, método de Maximum Likelihood basado en el modelo de Tamura-Nei, 1000 réplicas, longitud de las ramas calculada según el número de sustituciones por posición). Los números sobre las ramas indican la frecuencia de agrupamiento de los taxones.
El antibiograma se realizó mediante método epsilométrico (E-Test, Bio-Merieux) en agar Brucella HK solo en el primero de los casos, detectándose resistencia a penicilina (CMI>32mg/L), amoxicilina/clavulánico (CMI>256mg/L), piperacilina/tazobactam (CMI>256mg/L) y sensibilidad a imipenem (CMI=1mg/L), clindamicina (CMI=0,5mg/L) y metronidazol (CMI<=0,016mg/L). Se siguieron los criterios del CLSI (M100, Ed. 33) para la interpretación clínica de las CMI.
Los dos casos son pacientes con enterocolitis graves con ingreso hospitalario, desarrollando shock séptico y falleciendo en menos de 24 horas. El tratamiento antimicrobiano con piperacilina/tazobactam, muy habitual en infecciones intraabdominales, no sería adecuado para infecciones por Desulfovibrio spp., intrínsecamente resistente a este.
Para el diagnóstico de infecciones por Desulfovibirio spp. es importante recordar la necesidad de extraer frascos de hemocultivos en anaerobiosis, en caso de enterocolitis, incubarlos al menos 5 días, realizar tinción de Gram a todos los frascos de hemocultivo con detección de crecimiento, dejar en incubación durante 72 horas las placas de agar en atmósfera anaeróbica tras haber observado los microorganismos en la tinción y realizar la asignación taxonómica mediante dianas moleculares en los microorganismos no identificados por métodos habituales (pruebas bioquímicas, espectrometría de masas…). Añadir que, las técnicas comerciales de PCR múltiples tampoco identificarían estos microorganismos por no tenerlos incluidos en sus dianas.
D. porci es un microorganismo anaerobio de muy lenta y difícil detección en hemocultivos que podría estar implicado en casos graves de enterocolitis infecciosas con sepsis. La implicación clínica de este microorganismo podría estar subestimada si no se procesan correctamente los hemocultivos, debido a su lento crecimiento en medios líquidos y sólidos, su difícil visualización en tinción de Gram y la difícil identificación al no estar incluidos en las bases de datos de los sistemas comerciales, requiriendo la aplicación de dianas moleculares con valor taxonómico como el gen 16S rARN.
En general, la bacteriemia por microorganismos del género Desulfovibrio se produce por las especies D. desulfuricans y D. fairfieldensis, por translocación bacteriana del tracto gastrointestinal en pacientes con patología colónica, siendo la evolución buena en la mayoría de los casos5.
En nuestros casos, la bacteriemia por D. porci, se asocia con enterocolitis (sin poder asegurar si la infección por D. porci es la causa de la enterocolitis, o la bacteriemia es la consecuencia de la patología intestinal), sepsis grave y fallecimiento de los dos pacientes por shock séptico.
FinanciaciónNo existe financiación para este estudio.