SRAG
En octubre de 2004, la OMS publicó dos documentos que suponen la última recopilación de normas, directrices y recomendaciones para la vigilancia del síndrome respiratorio agudo grave (SRAG) (www.who.int/csr/resources/publications/SARSNEWGUIDANCE), y para establecer el riesgo y la preparación de la respuesta frente a esta infección (www.who.int/csr/resources/publications/ WHO_CDS_CSR_ARO_2004_2/en/).
Estos documentos suponen una actualización de los anteriormente publicados (14 de agosto de 2004).
Etiología
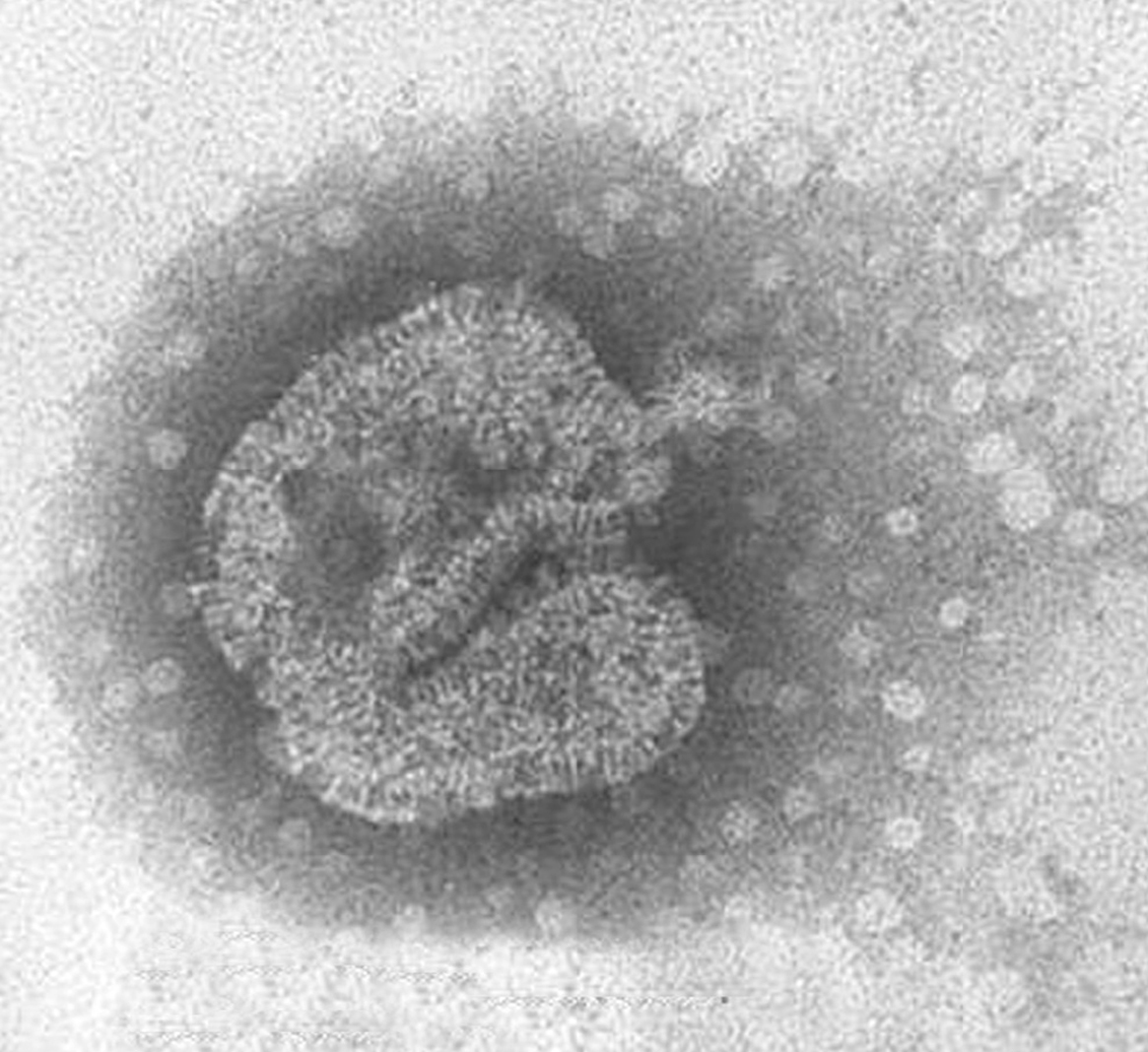
El SRAG es una enfermedad asociada a un nuevo coronavirus (CoV-SRAG). El 22 de marzo del año 2003, a partir de una biopsia pulmonar de un paciente con SRAG, se consiguió aislar en cultivos celulares un virus que se identificó por sus características morfológicas mediante microscopia electrónica como un coronavirus 1-3. Los estudios sobre su biología, su origen y su evolución siguen realizándose activamente en la actualidad.
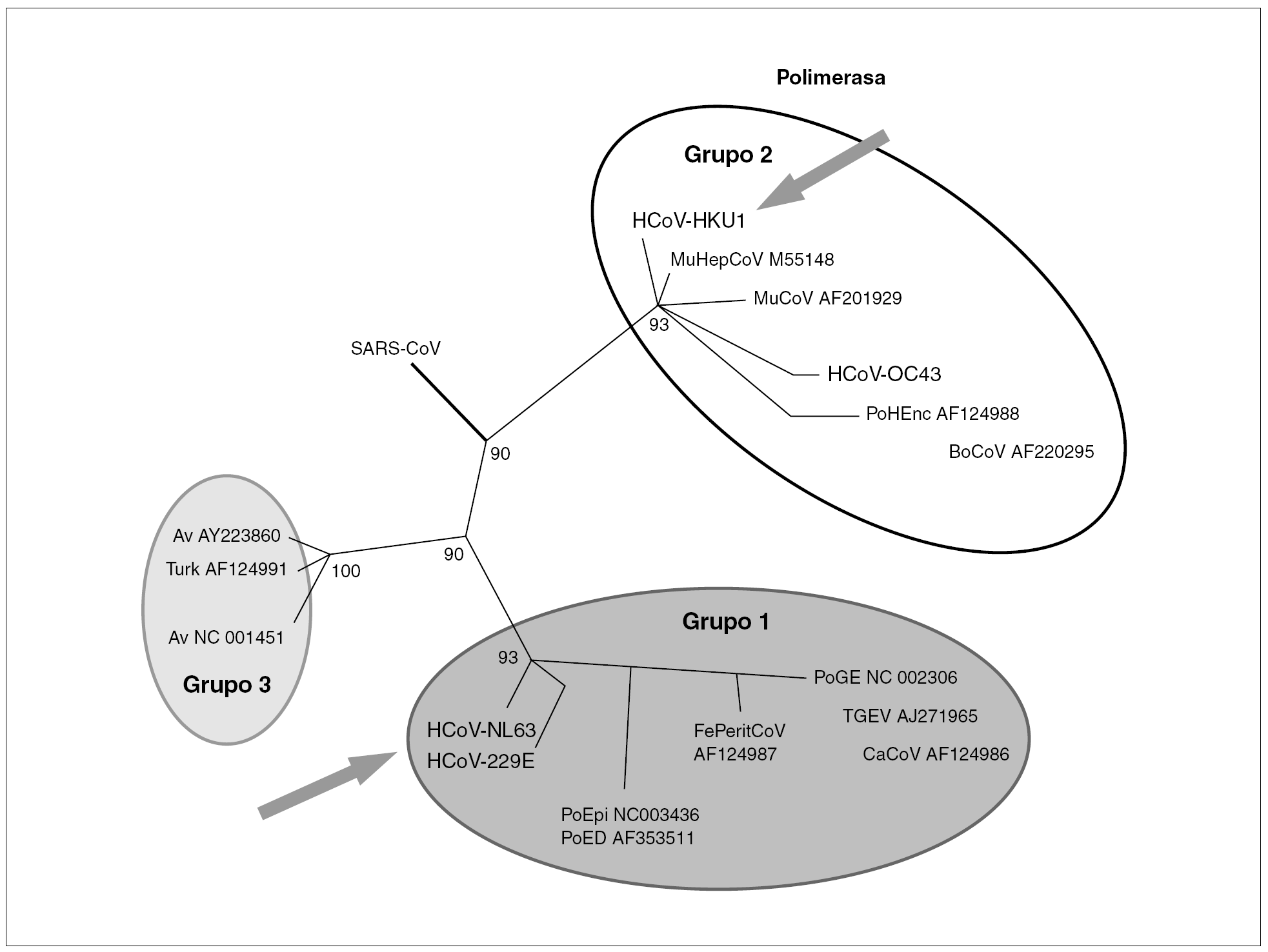
Los coronavirus (orden Nidovirales, familia Coronaviridae, género Coronavirus) son un grupo diverso de virus. Se han definido tres grupos, siendo los grupos 1 y 2 característicos de mamíferos mientras que el grupo 3 lo es de aves. Específicos del ser humano son el CoV-229E del grupo 1 y el CoV-OC43 del grupo 2. Recientemente se han descubierto dos nuevos coronavirus en muestras de pacientes: CoV-NL63 del grupo 1 y el CoV-HKU1 del grupo 2 (fig. 1).
Figura 1. Relaciones filogenéticas de los coronavirus humanos en un fragmento del gen que codifica para la polimerasa viral. Se diferencian tres grupos de virus. En el grupo 1 y 2 se encuentran los virus que infectan al ser humano. Con flecha se señalan los nuevos coronavirus humanos recientemente descritos.
Las partículas virales son grandes y presentan una envuelta. Su genoma está formado por una molécula de ARN de polaridad positiva, con un tamaño de aproximadamente 31.000 nt, lo que supone el genoma más grande de los virus ARN conocidos 4. Este genoma se presenta muy conservado, aunque se han descrito mutaciones que contribuyen a la diversidad genética del virus 5-7. El orden de los genes es el siguiente: 59 -replicasa-espícula (S)-envuelta (E)-membrana (M)-nucleocápsida (N)-39. Dos tercios del genoma lo forma el gen que codifica para la replicasa o polimerasa que está dividido en 2 ORF. Cada uno de los ORF (ORF1 y ORF2) codifica 2 poliproteínas mediante un proceso cotraduccional. El resto de los ORF codifican para las proteínas estructurales que se definen de la misma manera en todos los coronavirus (S, E, M y N). En el CoV-SRAG no se ha encontrado el gen de la hemaglutinina-esterasa, presente en el grupo 2 y en algunos coronavirus del grupo 3.
Epidemiología
En noviembre de 2002, empezaron a aparecer en Guandong, provincia de China, los primeros casos de SRAG, una nueva enfermedad que no fue reconocida hasta el mes de febrero de 2003. En esa fecha se habían producido numerosos casos, varias muertes y algunos de los pacientes habían viajado a otras regiones de China o al extranjero, extendiendo la enfermedad de manera inadvertida. El 12 de marzo, la OMS declaró la alerta mundial del SRAG y tomó la dirección de la investigación. En pocos días el número de casos se multiplicó en Hong Kong y, a causa de los viajes internacionales, aparecieron casos también en Vietnam, Singapur y Canadá. El 5 de julio la OMS declaró que la cadena de infección se había interrumpido. Según datos del 31 de diciembre de 2003, aproximadamente 8.096 personas se vieron afectadas en 26 países y se contabilizaron 774 muertes, la mayoría en China con afectación del personal sanitario que ascendió a 1.706 personas. Desde julio de 2003, el SRAG ha reaparecido en cuatro ocasiones, tres de las cuales fueron accidentes de laboratorio (Singapur, Taipei y Beijing). Solamente en uno de estos accidentes hubo transmisión secundaria ajena al laboratorio. El cuarto incidente se extendió a 4 personas en Guangzhou, Provincia de Guangdong (China), que se atribuyeron a una exposición con un animal infectado o por fuentes medioambientales.
La transmisión nosocomial es una de las características más importantes del brote de SRAG. La mayoría de los casos se han descrito en personas adultas y se puede indicar que los niños estuvieron afectados de forma esporádica. El período de incubación es de 5 días, con un rango entre 2 y 10 días, aunque existen publicaciones en donde se detallan períodos más largos. No existe evidencia de transmisión de la infección antes de desarrollar los síntomas.
Basándose en los cuadros clínicos de los pacientes de Canadá, China, Hong Kong, Singapur, Vietnam y Estados Unidos el porcentaje de mortalidad varía entre el 0 y el 50%, dependiendo del grupo de edad. Se ha asociado mayor mortalidad en hombres que en mujeres.
Tras la contención del brote mundial de SRAG en el verano de 2003, la posibilidad de la reaparición de la enfermedad sigue siendo una gran preocupación para los sistemas sanitarios. Los orígenes de una posible nueva circulación viral son: su persistencia en la población humana, bien mediante una transmisión indetectable, o a través de portadores, y el salto de la barrera interespecie desde un reservorio animal. Esta última hipótesis está siendo investigada en estos momentos muy activamente.
Recientemente la OMS ha definido cuatro fases de actuación frente a la posible existencia de casos de infección que tienen como objeto la contención de la enfermedad. Estas fases condicionan los niveles de actividad y se definen como: fase 0, ausencia de la circulación del CoV-SRAG en poblaciones humanas; fase 1, detección de casos esporádicos en humanos; fase 2, cadenas de transmisión entre humanos a nivel local o a nivel de provincia; fase 3, evidencia de una extensión de la enfermedad a nivel internacional; fase 4, período postepidémico con disminución del número de casos, y fase 5, no circulación del CoV-SRAG (tabla 1).
Patogenia
Se cree que el CoV-SRAG es un virus animal que cruzó la barrera interespecie y fue capaz de infectar a seres humanos debido a cambios ecológicos o a cambios en el comportamiento de los humanos que incrementaron las posibilidades para que se produjera una exposición al virus o una adaptación de este, permitiendo una transmisión entre humanos 8. Aunque el reservorio natural no se ha identificado todavía, el virus se ha detectado en diferentes especies de pequeños mamíferos, como las civetas, que se venden para consumo humano en los mercados de alimentos de animales vivos en Guandong 9. Estos mercados constituyen una interfase para la transmisión interespecie del virus desde los animales al hombre. Además, el virus se ha detectado en gatos, siendo posible la infección de estos animales de forma controlada. El objeto de establecer la infección en modelos animales es evaluar la eficacia de las estrategias de prevención y de tratamiento del SRAG. Se ha demostrado que el CoV-SRAG replica en ratones BALB/c, hámsteres y mono verde africano, tras la administración a través del tracto respiratorio. La replicación del virus alcanza pronto un máximo de carga viral que se aclara rápidamente en ausencia de enfermedad clínica. Esta replicación viral está acompañada por enfermedad pulmonar en los hámsteres y en el mono verde africano. Se está tratando de identificar modelos animales que desarrollen enfermedad clínica asociada a la replicación, de forma consistente. Los ratones infectados, hámsteres y primates no humanos desarrollan una respuesta de anticuerpos neutralizantes en suero y están protegidos de la reinfección 28 días después de la infección primaria. Estos modelos se utilizarán para investigar aspectos todavía no claros de la patogenia del SRAG.
Como en otras infecciones respiratorias virales, la gran variación encontrada en la patogenia del SRAG puede ser el resultado de una respuesta proinflamatoria del huésped frente al virus. En los casos leves la respuesta del interferón es alta en las primeras 48 h y va disminuyendo durante el curso de los siguientes 8 días.
Atendiendo al cuadro clínico, el sistema respiratorio es la primera diana del CoV-SRAG, siendo los pulmones los órganos principalmente afectados 3,10,11. Pocos grupos han publicado la presencia del virus en el aparato digestivo a pesar de que se determinó su presencia en heces desde los primeros momentos de las investigaciones. Recientemente se está trabajando en la afectación del aparato digestivo 12.
Manifestaciones clínicas
Definición de caso (tabla 2)
Primera semana de infección. Los pacientes inicialmente desarrollan un cuadro clínico semejante a una infección gripal. Los síntomas más comunes son: fiebre, mialgia, dolor de cabeza y rigidez. No existe un síntoma o grupos de síntomas específicos, aunque la fiebre es el síntoma más común, pero puede no presentarse.
Segunda semana de infección. Se caracteriza por una instauración de un cuadro respiratorio con tos, que en un principio es seca, disnea y diarrea. En los casos más graves los síntomas respiratorios se agravan y existe una desaturación de oxígeno del 20% que requiere el ingreso del paciente en la UCI. Existe linfopenia y afectación del hígado, por lo que se observa una elevación de las aminotransferasas hepáticas. Hasta el 70% de los pacientes desarrollan diarrea acuosa con gran volumen de sangre y moco. Se ha comprobado que durante esta semana se produce la transmisión a personas sanas.
Casos geriátricos, pediátricos y embarazo. Tanto en niños como en ancianos las situaciones crónicas y la mayor visita al médico de cabecera y hospitales, enmascararon la transmisión nosocomial al principio del brote de SRAG. En general, en ancianos y niños se produce un cuadro atípico, ya que no se evidenció instauración de fiebre. En concreto, en los ancianos la infección se enmascaró con infecciones secundarias bacterianas y neumonía. En los niños, el SRAG es una infección de gravedad media 13. Existen muy pocos estudios en mujeres embarazadas pero se sugiere que existe riesgo de aborto si la infección se produce al principio del embarazo y mortalidad de la madre si se produce al final.
Hallazgos radiológicos. En general, la radiografía de tórax al inicio de los síntomas (3-4 días) no demuestra una afectación respiratoria. Según avanza la infección se visualiza una consolidación que empieza con una lesión periférica unilateral que progresa a múltiples lesiones. Las observaciones durante los estados más avanzados incluyen neumotórax y fibrosis subpleural.
SRAG en médicos y en trabajadores sanitarios. Con objeto de conocer si existe o no una infección por CoV-SRAG se requiere una información de diferentes fuentes: historia clínica, examen clínico, monitorización del paciente en cama, investigación radiológica, investigación hematológica, investigación bioquímica, investigación microbiológica y virológica, respuesta al tratamiento, información epidemiológica tanto del individuo como del centro médico en donde trabaja. Se debe realizar una búsqueda exhaustiva del origen de la infección y de las posibles causas de la trasmisión basadas en la monitorización de todo el equipo médico.
Diagnóstico
Definición de caso confirmado: una persona que presenta algún resultado de laboratorio positivo a CoV-SRAG por cualquiera de las técnicas validadas que se comentan seguidamente y realizadas en un laboratorio que tenga capacidad de realizar este tipo de análisis.
Diagnóstico diferencial
Como en el resto de las infecciones respiratorias, los síntomas clínicos no permiten establecer un diagnóstico de la infección debido a que son inespecíficos. El diagnóstico diferencial de la infección de SRAG incluye patógenos comunes de la infección respiratoria como son los virus de la gripe, el virus respiratorio sincitial (VRS), parainfluenzavirus, adenovirus y otros coronavirus humanos (CoV-229E y CoV-OC43). Además, se investiga la presencia de bacterias incluidas en los protocolos de diagnóstico de una neumonía. Así, hay que descartar la presencia de Streptococcus pneumoniae y Haemophilus influenzae como agentes causales de la neumonía "típica" que se acompaña de los mismos síntomas clínicos (fiebre, cuadro respiratorio, tos y disnea) y que, en general, responde bien al tratamiento antibiótico. No hay que olvidar que el SRAG se definió en un principio como un cuadro de neumonía "atípica", cuadro de presentación clínica no usual con tos no productiva y seca. Los microorganismos más comunes incluyen Mycoplasma pneumoniae, Chlamydia spp., Legionella pneumophila y Coxiella burnetii.
Diagnóstico de laboratorio
A lo largo de estos últimos años (2003-2005) se ha avanzado en la consolidación del diagnóstico etiológico del SRAG. Han aparecido nuevos ensayos y las técnicas iniciales se han modificado para ser más sensibles y más específicas, dado que el conocimiento del virus cada vez es mayor.
Las aproximaciones fundamentales son: a) detección de ARN específico del CoV-SRAG para alcanzar una detección temprana y rápida de la infección en los casos sospechosos, y b) análisis serológicos para la confirmación de la presencia de inmunoglobulinas M y G (IgM e IgG) específicas que permitan establecer estudios de seroprevalencia en una población, en un centro médico o en una residencia.
El aislamiento del virus en cultivo celular es muy difícil y es necesario realizar todo el proceso en laboratorios de seguridad biológica de nivel 3. Además, las dificultades para el crecimiento del virus siguen constituyendo un obstáculo importante para el diagnóstico mediante aislamiento viral. Por esta razón los métodos de diagnóstico se centran en la detección molecular y en la serología.
El diagnóstico debe cumplir las siguientes recomendaciones:
1.Se debe utilizar un método validado de amplificación genómica (RT-PCR). Un resultado positivo será informado si: a) es positivo en dos muestras clínicas diferentes (p. ej., exudado nasofaríngeo, heces, plasma), o b) utilizando la misma muestra clínica pero tomada en diferentes días de la infección, o c) ensayos diferentes de un mismo método de RT-PCR usando ARN extraído en dos veces diferentes a partir de una única muestra clínica.
2.Se deben analizar muestras de suero tomadas en la fase aguda y en la fase convaleciente de la infección o se debe obtener un aumento de título de anticuerpos de cuatro veces (seroconversión) entre la fase aguda y la convaleciente.
Se han desarrollado ensayos comerciales tanto para la detección de ARN viral en muestras clínicas como para la determinación de anticuerpos IgG frente a CoV-SRAG en suero.
Interpretación de los resultados de laboratorio
Es muy importante saber que un diagnóstico de laboratorio nunca se basa en un resultado positivo obtenido por un único método de diagnóstico, o en una única muestra en una única determinación.
El tipo de muestra clínica condiciona el diagnóstico, ya que los métodos dependen crucialmente de la muestra clínica, tanto de la toma como de la rapidez del transporte al laboratorio. La posibilidad de detectar ARN viral en las muestras clínicas mediante RT-PCR debe ser un proceso organizado en el que todos los profesionales sanitarios y no sanitarios trabajen en equipo.
La existencia de seroconversión o de un aumento del título de anticuerpos en un paciente puede no ir acompañado de un resultado positivo de RT-PCR por haber habido un aclaramiento viral o por un mal almacenamiento de la muestra.
Tratamiento
El primer tratamiento antibiótico de los pacientes con sospecha de SRAG se instaura a la recepción del paciente en el hospital con objeto de tratar la posible neumonía asociada a la comunidad o la neumonía atípica, con las que se puede confundir. Si no existe una respuesta positiva a este tratamiento se evidencia una sospecha aún mayor del caso. No existe un tratamiento específico.
Profilaxis
Gracias a la experiencia en la preparación de vacunas de coronavirus animales, el desarrollo de las vacunas frente a SRAG ha partido de una posición muy ventajosa. Desde hace un tiempo, se están comenzando las primeras pruebas en animales de experimentación con una vacuna prototipo atenuada. Sin embargo, una alternativa a estas vacunas que en definitiva utilizan virus vivo, son las basadas en la introducción de modificaciones en el genoma viral para evitar el peligro de reversión al "virus salvaje". También se está trabajando en el uso de virus de bajo poder patogénico para la producción masiva de virus recombinantes que posean determinados genes seleccionados que codifiquen para proteínas de superficie implicadas en la producción de la respuesta inmunitaria 14. Esta última alternativa permite la preparación de vacunas no patogénicas y con un alto nivel de seguridad. Los principales esfuerzos se centran actualmente en la glucoproteína de la cápsida del CoV-SRAG, proteína S o de la espícula, que se une al receptor celular ACE2 en el inicio de la infección, por lo que se considera un antígeno prometedor para el desarrollo de una vacuna anti-SRAG.
Gripe aviar
El subtipo H5N1 del virus de la gripe A se ha considerado siempre como un patógeno exclusivo de las aves hasta que en 1997 se detectaron los primeros casos de infección en seres humanos, en un brote ocurrido en Hong Kong. En aquella ocasión fueron hospitalizadas 18 personas con síntomas de infección respiratoria aguda y seis de ellas fallecieron. Los estudios epidemiológicos y moleculares que se realizaron demostraban por primera vez que la transmisión del virus aviar se había producido directamente desde aves a personas. La capacidad del subtipo H5N1 para infectar directamente al ser humano se ha podido demostrar recientemente en otras dos ocasiones. Por una parte, en febrero de 2003 se detectó un brote que infectó a dos miembros (probablemente a tres) de una familia de Hong Kong después de haber realizado un viaje a China. Por otra, en enero de 2004 las autoridades sanitarias de Vietnam y Tailandia informan de los primeros casos humanos de infección por el virus de la gripe aviar H5N1. Hasta ahora es el brote más reciente y el que, hasta la fecha, puede ser considerado el más devastador en términos económicos y el que más alarma ha generado en salud pública. Hasta comienzos de febrero de 2005 se habían confirmado un total de 55 casos de infección en seres humanos (37 casos en Vietnam, 17 en Tailandia y uno en Camboya) de los cuales 42 fallecieron, la mayor parte de ellos niños y adultos jóvenes. Estas cifras, que se corresponden con el número de casos oficialmente reconocidos por la OMS, con toda probabilidad no representan la verdadera magnitud de los casos de infección.
Todos los casos humanos descritos hasta ahora han estado precedidos por brotes de gripe aviar H5N1 altamente patogénica existentes en las explotaciones avícolas del entorno. Ya desde mediados de diciembre de 2003 se sabía de la existencia de brotes de gripe aviar altamente patogénica en varias granjas de Corea del Sur, una enfermedad desconocida hasta entonces en este país. Durante los primeros meses de 2004 los brotes de gripe aviar se extendieron a las explotaciones avícolas de otros siete países asiáticos (Vietnam, Tailandia, Japón, China, Laos, Indonesia y Camboya), y con posterioridad se sumaba Malasia, que no refirió brotes hasta agosto de 2004. Hasta entonces, nunca se habían visto afectados tantos países por brotes de gripe aviar en aves en su forma más virulenta. La mayoría de los expertos opinan que el control del brote de gripe aviar por virus H5N1 altamente patogénico en las aves del sudeste asiático llevará varios años o puede incluso que permanezca de manera endémica en las aves de la zona. Obviamente, cuanto más se prolongue la presencia del subtipo H5N1 en las aves domésticas, tanto mayores serán las oportunidades para que ocurran más casos de infección en seres humanos, así como para que aparezca un virus con potencial pandémico. En este escenario sin precedentes el riesgo de aparición de una cepa de virus influenza pandémico es evidente, y la OMS ya está alertando de ello.
Potencial pandémico del virus H5N1
Un virus de la gripe A pandémico puede generarse de dos maneras. Una de ellas sería mediante el intercambio (del inglés reassortment) de material genético entre dos subtipos diferentes (en este caso entre el subtipo H5N1 y cualquiera de los dos subtipos que infectan habitualmente al hombre, H1N1 o H3N2) cuando coinfectan una misma célula, tal y como ya ocurrió en las pandemias de 1957 y 1968. Este intercambio de material genético puede realizarse en células de un ser humano, puesto que puede infectarse directamente con subtipos aviares, o en las células epiteliales del tracto respiratorio superior del cerdo, que poseen receptores de hemaglutinina de subtipos humanos y aviares. La otra forma sería por mutaciones puntuales en el genoma del subtipo H5N1 en su proceso de adaptación a nuevos hospedadores, sea el ser humano u otros mamíferos.
Para el comienzo de una nueva pandemia por el virus de la gripe se han identificado tres prerrequisitos. Primero, un nuevo subtipo debería ser transmitido al ser humano. Segundo, el nuevo subtipo debería poder replicarse en el ser humano y causar enfermedad. Y tercero, el nuevo virus debería poder transmitirse persona-persona de manera efectiva. Los dos primeros requisitos ya se han producido en varias ocasiones desde 1997. El tercero, afortunadamente, aún no se ha observado.
Hasta ahora prácticamente todos los casos de infección en seres humanos se han atribuido a la transmisión directa del virus desde las aves al hombre, bien por contacto directo con aves infectadas, bien con material contaminado por sus excreciones, descartándose la transmisión efectiva de persona a persona. Las sospechas de la posible transmisión del virus de persona a persona siempre han arreciado cuando los casos ocurren muy próximos en el tiempo entre personas que comparten un mismo lugar. A lo largo de 2004 han sido varias las ocasiones en que los casos se producían en miembros de una misma familia; sin embargo, no se han documentado casos de infección por H5N1 en profesionales sanitarios, a pesar de la existencia de un contacto estrecho con pacientes gravemente enfermos. El objetivo último de la investigación epidemiológica llevada a cabo en los brotes familiares es decidir si los miembros de una misma familia habían estado expuestos a aves o a sus excretas como fuente común de infección, que suele ser lo más probable. Descartada esta posibilidad sólo queda como explicación más plausible atribuir la fuente de infección a la transmisión persona a persona, circunstancia que hasta ahora sólo se ha producido en un único brote familiar 15. Por supuesto, los casos puntuales de transmisión persona a persona no deberían entenderse como ejemplos de transmisión efectiva entre personas, que se reflejará como la existencia de varias cadenas de transmisión sostenidas en el tiempo causando brotes comunitarios relativamente amplios en cuanto al número de personas afectadas.
La transmisión no efectiva de persona a persona era algo que ya ponían de manifiesto los estudios serológicos que se realizaron después del brote de Hong Kong en 1997, donde se observaba una mayor tasa de seropositivos específicos para el subtipo H5 en familiares de personas infectadas 16, así como en los profesionales sanitarios expuestos a pacientes infectados 17.
Etiología
Los virus de la gripe pertenecen a la familia Orthomyxoviridae, y se caracterizan por contener un genoma segmentado constituido por ocho fragmentos de ARN en el caso de los tipos A y B y siete en el tipo C, y de polaridad negativa. Los virus de la gripe tipos A y B causan epidemias de gripe prácticamente cada año, sin embargo, el tipo C, que causa infección respiratoria leve, no produce epidemias. El tipo A se clasifica en subtipos en función de las variaciones antigénicas de dos glucoproteínas de superficie denominadas hemaglutinina y neuraminidasa. Hasta el momento se han identificado 16 hemaglutininas y 9 neuraminidasas diferentes, que se combinan entre sí generando una amplia variedad de subtipos virales. En la actualidad dos subtipos, H1N1 y H3N2, son los que infectan habitualmente al ser humano, aunque recientemente el espectro se ha ampliado con los casos de infección por los subtipos H5N1, H7N7 (Holanda), H7N3 (Canadá) y H9N2 (China).
Epidemiología
Todos los subtipos conocidos del virus de la gripe A están presentes en aves acuáticas, en particular en patos silvestres, que constituyen su hospedador natural. Estas aves están perfectamente adaptadas a los diferentes subtipos del virus de la gripe de manera que no muestran ningún síntoma de enfermedad cuando están infectadas, limitándose únicamente a excretar virus en sus heces. La conducta migratoria de estos animales y la gran resistencia que muestran los virus de la gripe en el agua desempeñan un papel muy importante en la dispersión de los virus y en la infección, por contacto directo o indirecto, de las aves de explotaciones avícolas, principalmente pollos y pavos, que son especialmente susceptibles. Las aves domésticas infectadas excretan grandes cantidades de virus en las heces y en secreciones nasales y oculares. La diseminación del virus de una explotación a otra se produce con una enorme facilidad, bien sea a través de movimientos de aves infectadas, del personal de una granja a otra o por utilización de equipos y utensilios contaminados. Cuando la densidad de animales es elevada la transmisión aérea también puede llegar a ser importante.
Según la gravedad de la enfermedad observada en las aves domésticas, el virus de la gripe aviar puede ser de alta virulencia (HPAI, del inglés highly pathogenic avian influenza), causante de una enfermedad aguda con una elevada tasa de mortalidad en aves, o de baja virulencia (LPAI, del inglés low pathogenic avian influenza), bastante más común y que causa, en general, una enfermedad leve, que puede incluso pasar desapercibida si no se realiza una vigilancia activa con regularidad. Cualquiera de los subtipos de virus de la gripe A, incluidos H5 y H7, pueden infectar a las aves domésticas causándoles una enfermedad leve. No obstante, todas las variantes altamente patogénicas identificadas pertenecen a los subtipos H5 y H7. Hasta ahora se pensaba que no había un reservorio natural para las variantes HPAI, y que estas surgían por mutaciones producidas en variantes LPAI después de haber infectado a las aves domésticas a lo largo de un período de tiempo impredecible. No obstante, en el brote del sudeste asiático se ha demostrado por primera vez la existencia de aves acuáticas y otras aves migratorias infectadas con la variante HPAI del virus sin mostrar signos de enfermedad, sugiriendo que la evolución y mantenimiento de estas cepas virulentas en esta región podría estar cambiando, con tendencia a establecerse de manera permanente en determinadas zonas de Asia.
Sin duda, uno de los hallazgos más sorprendentes es que el virus H5N1 ha ampliado su rango de animales con susceptibilidad de ser infectados a determinadas especies de mamíferos, como tigres 18 y cerdos 19 infectados de manera natural, así como ratones 20 y gatos 21, que han podido ser infectados de manera experimental. En el caso de los tigres, en los que se observó fiebre y neumonía, la infección se produjo después de ser alimentados con cadáveres de pollos que habían sido previamente sacrificados. No hubo evidencia de transmisión tigre a tigre. No obstante, cuando fueron infectados experimentalmente otros felinos se pudo evidenciar la existencia de transmisión gato a gato. La infección natural de cerdos con el subtipo H5N1 del virus de la gripe es un hallazgo preocupante en tanto que estos poseen receptores en las células epiteliales de su tracto respiratorio superior que los hace susceptibles a la infección por virus aviares y humanos, pudiendo entonces hacer de "coctelera" en caso de que un cerdo sea infectado simultáneamente por un virus aviar y otro humano y facilitando el intercambio de material genético entre ambos.
Patogenia
En aves, la diferencia principal entre una cepa LPAI y otra HPAI es la capacidad de infección que tienen una y otra. Así, las cepas LPAI se limitan a producir una infección localizada, normalmente en el tracto digestivo, en tanto que las cepas HPAI se caracterizan por provocar una infección sistémica en la que prácticamente todos los órganos se ven afectados. Los mecanismos moleculares por los que se originan cepas HPAI, a partir de cepas LPAI, y que explican estas diferencias en el tropismo por los tejidos, afectan a más de un gen y no se conocen completamente. No obstante, se sabe que determinadas mutaciones producidas en el gen de la hemaglutinina, que afectarán a su posterior activación mediante proteólisis, están entre los factores responsables. Podemos diferenciar dos tipos de mutaciones, aquellas que implican una inserción de aminoácidos básicos (Lys y Arg) en la región conocida con el nombre de péptido de conexión, y que es el lugar donde se produce la escisión proteolítica de la hemaglutinina, y aquellas que implican la desaparición de una cadena carbohidratada que en la estructura secundaria de la proteína se ubica en la vecindad del péptido de conexión 22,23.
Existen dos clases de proteasas capaces de escindir la hemaglutinina nativa (HA0) en los dos fragmentos (HA1 y HA2) que hacen que la proteína sea funcional. El grupo de proteasas similares a la tripsina, que son las únicas con capacidad para realizar la proteólisis de la hemaglutinina de cepas LPAI, se encuentran en un número muy reducido de células localizadas en el tracto respiratorio de los mamíferos o en el tracto intestinal de las aves. Por este motivo, estas cepas sólo causarían infecciones localizadas. Sin embargo, en la activación de la hemaglutinina de las cepas HPAI, además de intervenir proteasas del grupo de la tripsina, intervienen proteasas del grupo de la subtilisina, bastante más ubicuas y que posibilitan la infección sistémica.
Manifestaciones clínicas
Las manifestaciones clínicas asociadas con la infección por H5N1 pueden abarcar desde una infección asintomática hasta neumonía o fallo multiorgánico. Aceptando de antemano que no es fácil precisar el número de casos en los que la infección es asintomática, lo cierto es que la tasa de mortalidad entre los casos confirmados sintomáticos es bastante elevada. Si en Hong Kong en 1997 llegó a ser del 33% (6/18), en el brote del sudeste asiático la cifra llega a ser alarmante y alcanza el 76% (42/55). Según las series publicadas de pacientes con infección por H5N1 de Hong Kong 24 y Vietnam 25 se sabe que en las fases iniciales de la infección no es posible establecer diferencias con la gripe debida a infección con los subtipos habituales H1N1 o H3N2, observándose fiebre superior a 38 °C, tos, disnea, mialgias y dolores articulares. El tiempo estimado entre la exposición al virus y el inicio de los síntomas sugiere un período de incubación que oscila entre 2 y 4 días. Los grupos de edad más afectados son los niños y adultos jóvenes. Curiosamente, sólo en la serie de Hong Kong los casos leves se asociaban con los niños menores de 12 años. En los casos graves, la progresión clínica de los pacientes hacia neumonía es muy rápida. La neumonía es primaria, no se debe a sobreinfección bacteriana. Entre los datos de laboratorio destaca la marcada linfopenia que presentan los pacientes con infección grave. El recuento de linfocitos puede utilizarse como un marcador de progresión de la enfermedad, de manera que sólo los pacientes que muestran un aumento en el recuento de linfocitos se recuperan. Los niveles elevados de transaminasas en suero así como las concentraciones anormales de creatinina observados apuntan hacia un deterioro de las funciones hepáticas y renal.
El espectro clínico de enfermedades en las que cabe sospechar infección por H5N1 podría ser más amplio. Se han descrito casos que presentan fiebre y síntomas gastrointestinales como únicas manifestaciones clínicas en las fases iniciales de la infección 26,27. En uno de estos casos se pudo incluso aislar un virus viable de las heces, hecho que tiene una enorme repercusión en cuanto a que amplía las posibilidades de vías de transmisión y dificulta el control de la infección.
Diagnóstico
Las recomendaciones de la OMS para realizar el diagnóstico de infección por H5N1 están disponibles en la página web http://www.who.int/csr/disease/avian_influenza/guidelines/labtests/en/. Las muestras óptimas son el aspirado o lavado nasofaríngeo obtenido dentro de los 3 días después del comienzo de los síntomas, aunque también son muestras válidas los frotis faríngeos, nasales y nasofaríngeos. Es recomendable la extracción de suero en las fases aguda y convaleciente para la confirmación serológica de resultados positivos utilizando otras técnicas. El envío de las muestras hasta el laboratorio debe realizarse de manera segura en el triple contenedor homologado, cuyas características se pueden revisar en la página web http://www.who.int/csr/disease/avian_influenza/guidelines/transport/en/.
El subtipo H5N1 puede ser cultivado en células Madin Darby de riñón de perro, que son las que se utilizan habitualmente para el aislamiento de virus de la gripe. El efecto citopático aparece 4-5 días después de inoculado y se debe confirmar realizando una RT-PCR específica del cultivo o fluorescencia utilizando anticuerpos monoclonales específicos de subtipo H5. Estas dos técnicas pueden ser utilizadas también para realizar un diagnóstico rápido mediante detección directa del virus a partir de la muestra clínica. El protocolo utilizado de RT-PCR debe haberse optimizado para obtener la máxima sensibilidad cuando se realiza partiendo directamente de muestra clínica en vez de cultivo celular. Por último, también se puede diagnosticar la infección por H5N1 mediante ensayos serológicos específicos para anticuerpos anti-H5.
Es necesario recordar que el cultivo celular o cualquier otra técnica que requiera la propagación del virus debe realizarse en laboratorios con un nivel 3 de bioseguridad (BSL3), para el resto de técnicas es suficiente con un laboratorio BSL2.
Tratamiento
Existen dos clases de antivirales específicos para el tratamiento de la gripe. El subtipo H5N1 ha demostrado ser resistente a los inhibidores de la M2, amantadina y rimantadina, que son los antivirales clásicos. Es de suponer que esta resistencia podría ser transmitida a un posible virus pandémico. La segunda clase de antivirales son los inhibidores de la neuraminidasa, oseltamivir y zanamivir, bastante más caros en cuanto a su producción, que es limitada, y almacenamiento. Oseltamivir (75 mg, 2 veces al día, 5 días) ha demostrado ser efectivo en los casos de gripe aviar en el ser humano en Vietnam y Tailandia, siempre y cuando sea administrado dentro de los primeros 2 días después del inicio de los síntomas. Este mismo antiviral está siendo utilizado también con fines profilácticos (75 mg/día, 7 días) en familiares de enfermos y personal sanitario expuesto a enfermos.
Profilaxis
En aves, la medida de control de la gripe aviar más efectiva continúa siendo el sacrificio masivo de aves infectadas y la desinfección de las explotaciones afectadas. No obstante, se puede utilizar la vacunación como medida suplementaria y es una decisión que debe ser tomada por el gobierno del país afectado. La utilización de vacunas para controlar la gripe aviar es una cuestión muy debatida. Las vacunas disponibles en la actualidad, que se elaboran con virus inactivados, reducen la gravedad y la mortalidad de la infección, pero las aves infectadas continúan eliminando virus, de manera que pueden contribuir a que la infección se haga endémica en una determinada región. Además, en ocasiones no es posible diferenciar las aves naturalmente infectadas de aquellas que han sido vacunadas. Otro problema que presentan las vacunas actuales es la falta de inmunogenización cruzada entre unas cepas virales y otras, un hecho que obliga a utilizar como cepa inmunógena la misma cepa causante del brote. Por otro lado, la vacunación actual presenta serios problemas de administración puesto que debe ser inyectada a todas y cada una de las aves de una explotación. Las vacunas de nueva generación o recombinantes, aún no disponibles comercialmente, resolverán todos estos problemas.
Por ahora no existe una vacuna específica que pueda prevenir la infección por gripe H5N1 en el ser humano. Aunque se han elaborado vacunas específicas utilizando la cepa que ocasionó el brote de Hong Kong en 1997, la eficacia de las mismas es, al menos, cuestionable para prevenir la infección con las cepas que actualmente circulan en el sudeste asiático teniendo en cuenta las variaciones antigénicas observadas entre unas y otras cepas 28. Se recomienda no obstante la vacunación de personal de riesgo, como trabajadores de explotaciones avícolas, trabajadores implicados en el sacrificio de aves o personal sanitario de las zonas afectadas, con la vacuna de la gripe de la temporada en curso, de manera que, aunque sólo protegerá de infecciones ocasionadas por los subtipos H1 y H3 del virus influenza circulante, se minimizan las posibilidades de coinfección con una cepa humana y otra aviar.
En el siglo pasado la carta de presentación de las pandemias de gripe era, en mayor o menor medida, la aparición repentina de un enorme número de casos acompañados de unas tasas de mortalidad y morbilidad muy elevados, provocando además alarma social, enormes pérdidas económicas y el colapso de los sistemas sanitarios en todo el mundo. Esta vez la situación es diferente y existen muchas evidencias que apuntan a que las posibilidades de padecer una nueva pandemia de gripe de manera inminente son muy elevadas. Por decirlo utilizando otras palabras, esta vez estamos siendo avisados. No puede predecirse si estamos en el preludio de la primera pandemia de gripe del siglo xxi, pero de ocurrir no debería llegar sin que estemos preparados.
Infecciones por metapneumovirus humano
Etiología
En el año 2001 se descubrió un nuevo virus respiratorio tras el estudio de muestras respiratorias de niños en Holanda 29. Este virus nuevo se asignó a la familia Paramyxoviridae, subfamilia Pneumovirinae, género Metapneumovirus, en función de estudios morfológicos de la partícula viral por microscopia electrónica y por estudios moleculares. Se denominó metapneumovirus humano (HMNV). La especie tipo del género es el metapneumovirus aviar, anteriormente conocido como el virus de la rinotraqueítis del pavo.
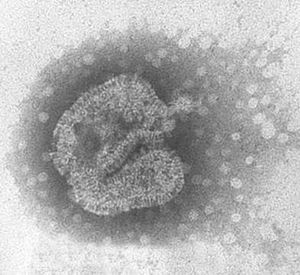
La morfología del virión (fig. 2) es aquella definida en general para los paramyxovirus: partículas pleomórficas, con un tamaño de 150-600 nm, con una envuelta que presenta proyecciones cortas o espículas de 13-17 nm y con un genoma constituido por una única molécula de ARN de polaridad negativa con una longitud de 13.378 nt. Además, no presentan actividad hemaglutinante con eritrocitos de pavo, pollo y cobayas y su ciclo de replicación depende de la presencia de tripsina.
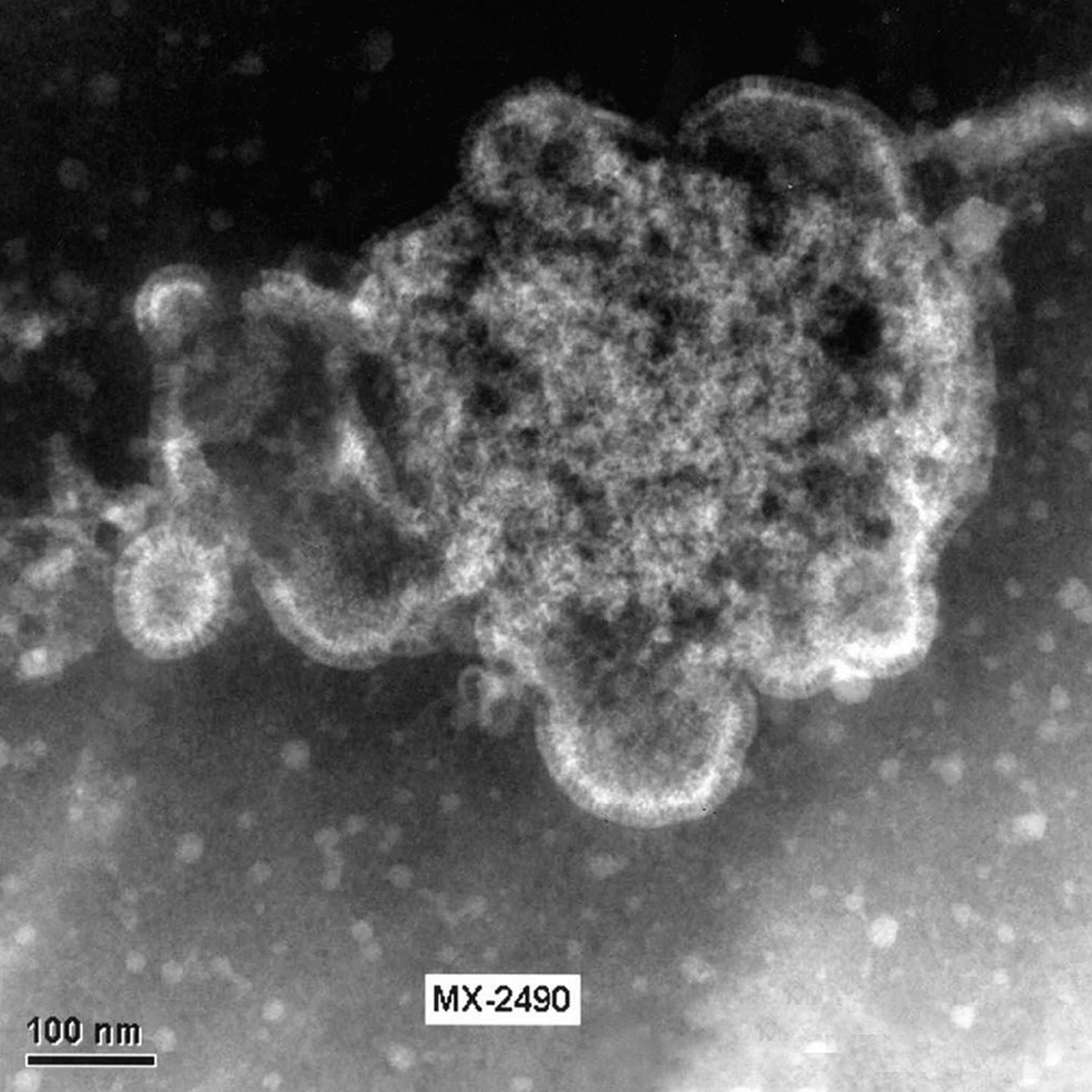
Figura 2. Partícula viral de HMNV de morfología pleomórfica cultivada en células LLC-MK2. Laboratorio de Gripe y Virus Respiratorios (CNM, Instituto de Salud Carlos III). (Foto: Servicio de Microscopia Electrónica; CNM, Instituto de Salud Carlos III.)
La diferenciación de la subfamilia Pneumovirinae en 2 géneros (Metapneumovirus y Pneumovirus) se debe a que el género metapneumovirus carece de proteínas no estructurales NS1 y NS2 y a que el orden de los genes es diferente. La especie tipo del género pneumovirus es el VRS que presenta un orden genómico: 39 NS1-NS2-N-P-M-SH-G-F-M2-L-59; sin embargo, el HMNV presenta el siguiente orden: 39 N-P-M-F-M2-SH-G-L-59 (N: nucleoproteína; P: fosfoproteína; M: proteína de la matriz; F: proteína de fusión; M2: proteína de matriz 22K; SH: proteína hidrofóbica pequeña; G: glucoproteína de anclaje con el receptor celular; L: polimerasa).
Se han definido 2 genotipos A y B que a su vez se subdividen en cuatro subgenotipos, A1, A2 y B1, B2, en función de las secuencias de los genes que codifican para las proteínas F y G.
Epidemiología
Originariamente el virus se aisló de muestras respiratorias de 28 pacientes no relacionados epidemiológicamente en Holanda, que presentaban una infección respiratoria. Estas muestras se recogieron desde 1981. En este trabajo, la edad de los niños era inferior a 5 años siendo inferior a un año en 13 de ellos. Actualmente se sabe que el HMNV produce una infección respiratoria aguda no sólo en niños sino también en ancianos y en pacientes inmunodeprimidos. La aparición de casos asociados a HMNV es estacional en los países con clima templado y se ha definido desde el invierno a la primavera (noviembre-mayo). Se ha demostrado la coinfección entre el VRS y el HMNV lo que supone un aumento de la gravedad del cuadro respiratorio de los niños infectados 30. Actualmente existen trabajos de descripción de casos de bronquiolitis asociada a HMNV en Alemania, Dinamarca, Australia, Japón, Grecia, España, Estados Unidos, Reino Unido, Finlandia, Hong Kong.
Patogenia
Cuando el HMNV fue descubierto una de la primeras preguntas a contestar fue si el virus era realmente aviar y había infectado a humanos o si por el contrario era propiamente humano. Para demostrarlo los autores holandeses inocularon diferentes especies de aves y monos. Como resultado se obtuvo infección respiratoria en los monos y no en las aves, demostrando la afinidad por los primates. Se cree que el HMNV lleva circulando en la especie humana más de 50 años. No se conoce el receptor celular por el momento.
Manifestaciones clínicas
En general, los niños infectados por HMNV presentan un cuadro clínico semejante al de la infección respiratoria del tracto inferior asociada al VRS, desde una infección de intensidad media hasta tos grave, bronquiolitis y neumonía, con frecuencia con presentación de fiebre muy alta, mialgia y vómitos. Muchos de los pacientes son hospitalizados y requieren ventilación asistida. Si se comparan los cuadros clínicos asociados a VRS y a HMNV se pueden encontrar ligeras diferencias, ya que la infección por HMNV es ligeramente menos grave y se presenta con un cuadro asmático (16%), menor disnea, menor dificultad para comer, menor hipoxemia, menor porcentaje de pacientes que requieren administración de oxígeno y mayor frecuencia de instauración de administración de antibióticos (60%).
Diagnóstico
Aislamiento en cultivos celulares
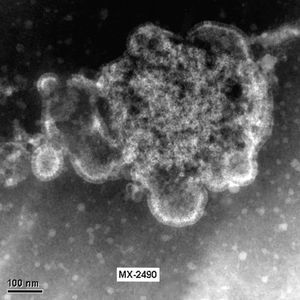
El HMNV se ha identificado recientemente por un crecimiento difícil en células permisivas, en concreto células terciarias de riñón de mono. En la actualidad se ha podido comprobar que el aislamiento del virus se puede realizar en líneas continuas celulares como son LLC-MK2 (células de riñón de mono Rhesus), VERO (células de riñón de mono verde africano) y HEp-2. En las figuras 2 y 3 se muestran virus liberados y virus en fase de liberación cultivado a partir de células LLC-MK2 en el Laboratorio de Gripe y Virus Respiratorios del CNM (Instituto de Salud Carlos III).
Figura 3. Célula LLC-MK2 infectada con HMNV. Distintas fases de la liberación de las partículas virales al exterior celular. Laboratorio de Gripe y Virus respiratorios (CNM, Instituto de Salud Carlos III). (Foto: Servicio de Microscopia Electrónica; CNM, Instituto de Salud Carlos III.)
Detección molecular mediante amplificación genómica
Se han utilizado diferentes muestras respiratorias como son exudados nasofaríngeos y lavados bronquioalveolares y se ha encontrado un buen rendimiento de diagnóstico. Los métodos desarrollados hasta la fecha no son comerciales y los diferentes grupos de investigación han desarrollado técnicas moleculares en distintos genes diana: F, N, L, M, G. Se considera que los métodos de amplificación genómica son de gran utilidad, ya que se pueden generar resultados positivos de una posible infección en un tiempo mucho menor que el aislamiento en cultivos celulares. Dado que el aislamiento de HMNV es muy difícil, los métodos de amplificación genómica representan la única alternativa junto con la investigación de los antígenos virales en las células epiteliales presentes en las muestras clínicas mediante inmunofluorescencia directa. Al no existir anticuerpos monoclonales específicos de HMNV suficientemente extendidos comercialmente, la RT-PCR supone una muy buena alternativa para el diagnóstico. Recientemente la PCR a tiempo real va siendo instaurada como método de diagnóstico molecular para algunos virus en los diferentes laboratorios del Sistema Nacional de Salud. Seguramente se instaurarán en dichos laboratorios este tipo de métodos después de haber sido validados. Existen trabajos en los que se describen métodos para la detección específica de HMNV en tiempo real 31.
Serología
Ninguno de los métodos desarrollados sirve para análisis seroepidemiológicos a gran escala porque hasta el momento no han sido validados completamente. No existen métodos comerciales que faciliten el diagnóstico, aunque se han desarrollado sistemas de análisis de inmunoabsorción ligado a enzimas (ELISA) para la detección de los anticuerpos frente a HMNV. En estos ensayos se han utilizado diferentes antígenos virales obtenidos tras el cultivo viral en células (lisado celular) o antígenos virales obtenidos mediante recombinación: proteína de la matriz M y nucleoproteína N 32. Realmente, los sistemas de análisis de los anticuerpos de los pacientes son métodos de inmunofluorescencia en donde se utilizan las células infectadas fijadas en un porta de cristal.
Tratamiento
Se han iniciado estudios para producir anticuerpos monoclonales y policlonales que puedan neutralizar al HMNV y ser administrados como tratamiento de la infección tal y como se está llevando a cabo con el VRS.
Profilaxis
Se está intentando producir vacunas vivas atenuadas de HMNV en diferentes laboratorios de Estados Unidos, Canadá, Europa y Australia. Hasta la fecha no se han emitido las licencias y se puede considerar que nos encontramos en las primeras fases de investigación de estas vacunas. Otra alternativa es la producción mediante genética reversa de quimeras que expresen en su superficie diferentes proteínas virales 33. Estas quimeras podrían expresar de manera bivalente proteínas de diferentes virus (VRS y HMNV) por los que la inmunización sería doble.
NOTA
Los artículos publicados en la sección "Formación Médica Continuada" forman parte de grupos temáticos específicos (antibiograma, antimicrobianos, etc.). Una vez finalizada la publicación de cada tema, se irán presentando al Sistema Español de Acreditación de la Formación Médica Continuada (SEAFORMEC) para la obtención de créditos.
Una vez concedida la acreditación, esta se anunciará oportunamente en la Revista y se abrirá un período de inscripción gratuito para los socios de la SEIMC y suscriptores de la Revista, al cabo del cual se iniciará la evaluación, durante un mes, que se realizará a través de la web de Ediciones Doyma.
RELACIÓN DE SERIES ACREDITADAS:
"Avances en el tratamiento de la infección por el virus VIH"
Disponible en: http://www.doyma.es/eimc/formacion
1 junio / 31 diciembre 2005