Hasta fechas muy recientes eran muy pocos los genomas de Chlamydia trachomatis disponibles, a pesar de su importancia en salud pública. Actualmente se están secuenciando 66 genomas completos de C.trachomatis. Esta revolución genómica está permitiendo comprender su biología, mejorar la sensibilidad y especificidad en el diagnóstico o desarrollar herramientas epidemiológicas no solo de C.trachomatis sino también de especies relacionadas, como C.pneumoniae o C.psittaci. El diagnóstico basado en cultivo celular, serología o microinmunofluorescencia está siendo progresivamente sustituido por técnicas de amplificación de ácidos nucleicos, al superarse los inconvenientes de escaso rendimiento o reacciones cruzadas y mejorar la estandarización entre laboratorios. Por otra parte, el desarrollo de técnicas de tipificación (MLST y VNTR) aplicadas a Chlamydiae ha aumentado el conocimiento de la epidemiología local y global aportando información sobre cómo esas bacterias evolucionan, causan brotes o adquieren mecanismos de resistencia. Esta revisión se centra en los grandes avances alcanzados en el conocimiento de las diferentes especies de Chlamydia, en parte debido a la innovación tecnológica aplicada a la genómica como una aproximación para explicar la revolución que, tanto en el diagnóstico como en su epidemiología, se ha observado en este grupo de bacterias en los últimos años.
Until recently the number of completed genomes belonging to Chlamydia trachomatis was very low, despite its importance in Public Health. Now, there are currently sixty-six completed genomes of C.trachomatis sequenced in different parts of the world. This genomic revolution has helped in understanding its biology, as well as improved the sensitivity and specificity in the diagnosis, and the development of epidemiological tools, not only for in C.trachomatis, but also for related species such as C.pneumoniae and C.psittaci. The diagnosis based on cell culture, serology and microimmunofluorescence is gradually being replaced by molecular techniques based on PCR or real-time PCR. This is because these molecular tests do not have cross-reactions problems and the procedures are easily standardised between laboratories. Moreover, molecular epidemiology tools described recently, such as Multi-Locus Sequence Typing (MLST) and Variable Number Tandem Repeat (VNTR), have increased our knowledge on local and global epidemiology. This article focuses on the impact of the genomics advances achieved over the last few years as applied to the diagnosis, epidemiology and biology of the family Chlamydiaceae family and related species.
Las especies patógenas para el hombre de la familia Chlamydiaceae, Chlamydia trachomatis, C.pneumoniae y C.psittaci, aunque presentan una pared celular típica de gramnegativos, pertenecen a una división bacteriana muy divergente del resto del árbol evolutivo de las eubacterias. Constituyen, junto con los micoplasmas, las especies bacterianas con los genomas más pequeños, entre 1,04 y 1,23Mb (25% del genoma de Escherichia coli), posiblemente debido a que son bacterias intracelulares obligadas desde hace mucho tiempo, lo que les ha permitido perder genes considerados esenciales (como FtsZ) o ciertas rutas metabólicas (como la biosíntesis de aminoácidos o de la fermentación anaeróbica)1.
Pese a las grandes cifras mundiales de infecciones humanas por clamidias, las pruebas microbiológicas necesarias para la precisa detección e identificación de estas bacterias en muestras humanas han sido tradicionalmente difíciles, tediosas y requerían personal altamente preparado. Sin embargo, las alarmas de salud pública internacional ocurridas en 2003 como consecuencia de un brote de linfogranuloma venéreo (LGV) inicialmente descrito en Holanda2 pero rápidamente extendido a otros países europeos, Estados Unidos y Australia3, o la identificación y rápida diseminación en Suecia de una nueva variante de C.trachomatis en 2006 que no era detectada por los sistemas comerciales más ampliamente empleados4, generaron la necesidad de disponer de sistemas de diagnóstico y genotipificación rápidos y precisos, ya que las dificultades para la detección en el laboratorio podrían estar ligadas a un retraso en el diagnóstico y, consecuentemente, a la diseminación de nuevas infecciones5.
La disponibilidad de técnicas de secuenciación masiva que permiten descifrar genomas bacterianos completos ha supuesto una verdadera revolución en la taxonomía bacteriana, especialmente entre las bacterias de difícil manejo en el laboratorio, como las clamidias6. Hoy en día disponemos de secuencias de genomas completos de C.trachomatis7, C.pneumoniae8 o C.psittaci9 y especies relacionadas10. Si bien es cierto que la existencia de especies relacionadas con Chlamydia es conocida desde hace tiempo11, los análisis bioinformáticos de secuencias completas de especies del orden Chlamydiales han permitido inferir una biodiversidad mayor de la esperada. Gracias a la enorme capacidad actual de secuenciación, en los últimos años este grupo de especies relacionadas con clamidias ha aumentado significativamente, identificándose hasta 7 familias pertenecientes al orden Chlamydiales: Clavochlamydiaceae, Criblamydiaceae, Parachlamydiaceae, Piscichlamydiaceae, Rhabdochlamydiaceae, Simkaniaceae y Waddliaceae. Algunas de estas familias engloban especies patógenas para el hombre, como Parachlamydia acanthamoebae, Simkania negevensis o Rhabdochlamydia spp.12, y el número de nuevas especies descritas sigue creciendo. Los análisis bioinformáticos sorprendentemente también han servido para rebatir el paradigma de la escasa tasa de recombinación en este grupo de bacterias, sospechado por lo particular de su ciclo biológico, que sugería escasas oportunidades de intercambio genético13. La existencia de recombinación tiene su relevancia clínica en la descripción de la cepa hipervirulenta de LGV, L2c, relacionada con el brote de LGV14, resultado de la recombinación entre serovares L2b y D de C.trachomatis.
Los serovares de C.trachomatis están tradicionalmente asociados a la seroespecificidad de la principal proteína de membrana externa, MOMP. Sin embargo, hoy en día no existe relación entre la secuencia del gen ompA (que codifica para MOMP) y el tropismo celular o la virulencia15, por lo que la genotipificación basada en el gen ompA como biomarcador no es válida, aunque ha sido ampliamente usada. Varios marcadores genéticos (pmpH, inc, tarp, T3SS…) han sido identificados como biomarcadores útiles en el estudio de los mecanismos biológicos implicados en la infección y el tropismo16. Los intentos para caracterizar y establecer relaciones entre los aislados de LGV en diferentes partes del mundo relanzó el desarrollo de herramientas de epidemiología molecular, ya descritas en otras especies bacterianas pero poco exploradas en Chlamydia. En 2007 se diseñaron las primeras propuestas de multilocus sequence typing (MLST) para la caracterización epidemiológica de especies de Chlamydia17, y un año más tarde se describió el sistema variable number of tandem repeats (VNTR). Posteriormente, en parte debido a la baja variabilidad observada y en parte a la accesibilidad a los sistemas de secuenciación masiva, se inició la secuenciación de genomas completos7.
Todos estos cambios son suficientes para realizar una actualización de esta familia bacteriana, donde C. trachomatis es responsable de más de 100millones de infecciones de transmisión sexual y de 90millones de casos de tracoma en el mundo18,19. Por su parte, C.pneumoniae podría ser responsable de un número importante de neumonías asociadas a la comunidad20 y de otras patologías crónicas, como aterosclerosis, miocarditis o cáncer de pulmón. Finalmente, aunque la incidencia de las infecciones por C.psittaci en la población humana es muy escasa, probablemente infravalorada por la dificultad en el diagnóstico, brotes de psitacosis son descritos todos los años en Europa21.
Estudios genómicos: implicaciones evolutivas y taxonómicasNuestra percepción de la diversidad de las clamidias cambió sustancialmente cuando en 1997 se describieron nuevas especies Chlamydia-like, llamadas clamidias ambientales22. El descubrimiento de especies de clamidias que no son patógenos primarios para el hombre abrió nuevas posibilidades para investigar la evolución y adaptación de este particular grupo de bacterias. De hecho, la diversidad ahora conocida es solamente la punta del iceberg, pues entre las 7 familias conocidas, algunas de ellas están constituidas por un solo representante23. Este orden Chlamydiales se separó hace ∼2000millones de años desde un ancestro común con las especies de vida libre Verrucomicrobia y Lentisphaera24 y hace ∼700millones de años se diferenció la familia Chlamydiaceae de las otras familias del mismo orden25, que se comportan como simbiontes de amebas de vida libre y con quienes comparte casi la mitad de su genoma (560genes son comunes en todas las especies del orden). De hecho, especies de la familia Chlamydiaceae como C.pneumoniae también sobreviven y se multiplican dentro de amebas, sugiriendo que especies de Acanthamoeba han podido servir como el principal reservorio y vehículo de dispersión de las clamidias25.
La clasificación taxonómica de la familia Chlamydiaceae ha sido muy discutida en los últimos años. En 1999, estudios basados en el análisis de las subunidades 16S y 23S del ADNr establecieron los géneros Chlamydia (como Chlamydia trachomatis) y Chlamydophila (como Chlamydophila pneumoniae o Chlamydophila psittaci)26. Esta nueva nomenclatura no ha sido ampliamente aceptada, pues se considera que puede llevar a confusiones. El International Committee on Sistematics of Prokaryotes ha recomendado la fusión de los 2géneros en uno, Chlamydia27. La edición de 2011 del Manual Bergey se hace eco de esta recomendación1, pero el debate permanece abierto28.
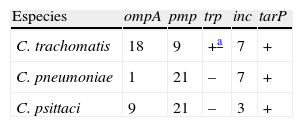
El tamaño del genoma de las especies de Chlamydia (que oscila entre 1,042Mb en C.trachomatis a 1,230 en C.pneumoniae) es 2-3 veces más reducido que el de otras especies de familias pertenecientes al mismo orden (que oscila entre 2,116Mb en Waddlia chondrophila y 3,072Mb en P.acanthamoebae)10, sugiriendo que el proceso de adaptación desde formas de vida libre hacia patógenos humanos ha supuesto también una reducción genómica. Un buen ejemplo se obtiene de la comparación del genoma de C.pneumoniae aislada de koalas australianos con aislados de origen humano, que permite inferir que C.pneumoniae de origen humano es una zoonosis donde la cepa se adaptó a través de un proceso de pérdida de material genético29. El primer paso evolutivo desde formas de vida libre hacia formas patógenas eucariotas fue la adquisición de un sistema de secreción tipo iii (solo presente en proteobacterias), sistema esencial para la infección de la célula eucariota. Progresivamente los miembros de la familia Chlamydiaceae fueron adquiriendo otros factores de virulencia relacionados con la adherencia e invasión celular, aunque todavía no podemos definir con certeza todos los factores de virulencia que han influido en los diferentes tropismos bacterianos (tabla 1). A medida que alcancemos un mayor grado de conocimiento sobre la prevalencia de determinados factores de virulencia que determinen el tropismo celular, podremos definir con mayor precisión técnicas moleculares que permitan genotipar las diferentes variantes o biotipos de una especie15.
Relación de los factores de virulencia más comunes descritos en Chlamydiae
| Especies | ompA | pmp | trp | inc | tarP |
| C. trachomatis | 18 | 9 | +a | 7 | + |
| C. pneumoniae | 1 | 21 | – | 7 | + |
| C. psittaci | 9 | 21 | – | 3 | + |
ompA (el gen que codifica para la proteína principal de membrana externa, MOMP); pmp (proteínas polimórficas de membrana externa); trp (ruta biosintética del triptófano); inc (inclusión), y tarP (fosfoproteína de reclutamiento de actina) son proteínas efectoras del sistemas de secreción tipo iii.
La ruta biosintética del triptófano está ausente en los serovares relacionados con tracoma. Puede observarse la mayor similitud en factores de virulencia entre C.pneumoniae y C.psittaci que ha vuelto a abrir nuevamente el debate de la reclasificación de clamidias en 2géneros: Chlamydia y Chlamydophila28. Los números indican el número de serovares o genes que existen en esa especie bacteriana.
Así, los estudios de genómica comparada de C.trachomatis han permitido inferir que solo los genes pmpH, pmpF, incD e incE agrupan las cepas en función de su tropismo: ocular, genital no-invasivo o genital invasivo30. Estos genes podrían emplearse para la genotipificación del LGV, si bien el gen más comúnmente utilizado hoy en día es pmpH. Similares aproximaciones se han iniciado en C.pneumoniae31. Esto es, a nuestro parecer, lo que progresivamente puede ir marcando el diagnóstico de clamidias en el futuro. Es decir, el empleo de técnicas moleculares no solo de cribado, sino también de genotipificación.
Detección en el laboratorio de las principales especies de Chlamydia relacionadas con patología en humanosHasta finales del siglo xx, el cultivo celular de Chlamydia ha sido el estándar de referencia frente al cual se han comparado todas las demás pruebas. Pero debido principalmente a la aparición de nuevos métodos diagnósticos más fáciles de implementar, rápidos y sensibles, el cultivo celular ha quedado relegado a laboratorios de referencia, con utilidad en estudios epidemiológicos y/o forenses. Progresivamente las técnicas de amplificación de ácidos nucleicos se están convirtiendo en las herramientas de diagnóstico más habituales en los laboratorios de diagnóstico, al superar los inconvenientes del escaso rendimiento en el cultivo, o de las reacciones cruzadas observadas en técnicas de microinmunofluorescencia.
La aseveración anteriormente expuesta podría tener tintes más realistas en C.trachomatis, donde la implantación generalizada de las técnicas moleculares para el diagnóstico diferencial de esta especie, desde 2007, ha aumentado significativamente la tasa de incidencia publicada de las infecciones relacionadas con C.trachomatis en Europa y Estados Unidos32. En los próximos años podremos valorar si un impacto similar ocurrió en C.pneumoniae, si bien recientes publicaciones sobre la incidencia de casos detectados por técnicas moleculares ha sido de 0,1% en un período de 10años33. Para una revisión más extensa de las técnicas diagnósticas de Chlamydia recomendamos la lectura del procedimiento número44 de la SEIMC34.
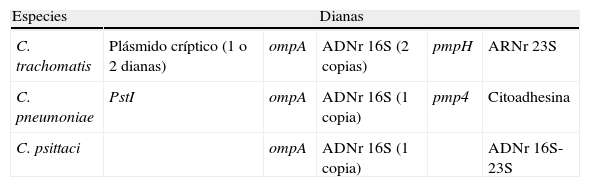
El grado de desarrollo y universalidad de estas técnicas de amplificación de ácidos nucleicos es distinto según las especies de clamidias (tabla 2). El mayor grado de desarrollo se ha alcanzado en C.trachomatis, donde por su elevada sensibilidad y especificidad han desplazado al resto de técnicas, convirtiéndose en el método de referencia actual. De hecho, las recomendaciones diagnósticas del CDC sugieren usar como cribado técnicas moleculares de amplificación35. La disponibilidad de métodos moleculares ha incrementado sensiblemente el potencial diagnóstico de estas bacterias en los laboratorios de microbiología. En C.pneumoniae las técnicas moleculares son preferentemente empleadas en las fases tempranas, mientras que las pruebas de serología son más útiles en fases más avanzadas o de confirmación. Finalmente, en C.psittaci no existen técnicas moleculares disponibles comercialmente y las técnicas serológicas son las pruebas de referencia para el diagnóstico de la psitacosis debido a la escasa prevalencia y agudeza de la enfermedad.
Dianas empleadas para el diagnóstico molecular de las principales especies de Chlamydia relacionadas con patología humana
| Especies | Dianas | ||||
| C. trachomatis | Plásmido críptico (1 o 2 dianas) | ompA | ADNr 16S (2 copias) | pmpH | ARNr 23S |
| C. pneumoniae | PstI | ompA | ADNr 16S (1 copia) | pmp4 | Citoadhesina |
| C. psittaci | ompA | ADNr 16S (1 copia) | ADNr 16S-23S | ||
La estrategia en C.trachomatis para aumentar la sensibilidad se basa en la detección de genes presentes en alto número de copias, como el plásmido críptico (∼10copias/genoma) o algunas técnicas caseras basadas en el ADNr 16S (2copias/genoma). En Europa, la mayoría de los sistemas comerciales se basan en la detección del plásmido críptico, alcanzando una sensibilidad entre 300-350copias/ml. El inconveniente de esta aproximación es que el 1% de las cepas carecen del plásmido, por lo que los sistemas que solo detectan esta diana darían resultados falsos negativos en estas cepas. Por otra parte, en 2006 se detectó en Suecia una nueva variante con una deleción de 377pb en la región CDS1 del plásmido empleada como diana diagnóstica36. Esta misma deleción había sido detectada previamente en 1990 en C.pneumoniae37. La falta de sensibilidad de los sistemas disponibles dio lugar a la diseminación de esta variante, que llegó a representar el 30% de los casos en Suecia, y un incremento significativo de los casos de C.trachomatis en los años posteriores. Esta variante comenzó a diseminarse por los países vecinos, e incluso se detectaron casos en Rusia, Escocia y Francia. Como consecuencia se diseñaron técnicas que empleaban 2dianas distintas con objeto de detectar esta variante (bien 2dianas en el mismo plásmido o bien una diana en plásmido críptico y otra en ompA), resultando en un descenso de los casos por esta variante desde el 30 al 6% en 201138. En Estados Unidos, las técnicas basadas en la captura e hibridación de los ácidos nucleicos mediante el empleo de sondas específicas constituyen el sistema más distribuido. Tiene una alta sensibilidad al emplear como diana los miles de transcritos de ARNr23S, su sensibilidad es mayor que las técnicas moleculares de primera generación, pero requiere el doble de tiempo39. Un inconveniente de las técnicas moleculares disponibles comercialmente es que no han sido aprobadas para su empleo con muestras de origen rectal, si bien múltiples ensayos han sido publicados a raíz del brote de LGV descrito en 2003, demostrando su utilidad.
Los ensayos serológicos para el diagnóstico de C.trachomatis, como el enzimoinmunoensayo (EIA) o los que emplean anticuerpos monoclonales marcados con fluoresceína (DFA) frente a la proteína MOMP o frente al lipopolisacárido (LPS), o frente a la proteína HSP60, están en claro retroceso en los laboratorios de microbiología, pues aunque son técnicas rápidas o incluso algunas comercializadas como técnicas «point-of-care» (∼30min), tienen baja sensibilidad y especificidad clínica. La detección del LPS da muchos falsos positivos por reacciones cruzadas con el LPS de otras enterobacterias y la proteína MOMP de C.pneumoniae puede reaccionar inespecíficamente con la MOMP de C.trachomatis. También pueden observarse problemas de reacciones cruzadas con P.acanthamoebae cuando se emplean anticuerpos específicos frente a la HSP60, por lo que no deben emplearse en muestras con alta carga bacteriana, como rectales o faríngeas40.
Diagnóstico de Chlamydia pneumoniaeLas técnicas moleculares para la detección de C.pneumoniae usan como diana ADNr16S o el gen ompA (una copia en cada caso), adaptándose en los últimos años a técnicas de PCR en tiempo real en formato multiplex (técnicas que simultáneamente detectan otros patógenos respiratorios, fundamentalmente Mycoplasma pneumoniae y Legionella pneumophila), desplazando a las técnicas de PCR anidada, debido a los problemas de contaminación asociados con esta técnica. Anteriormente se ha descrito la conveniencia de emplear técnicas moleculares para el diagnóstico de infecciones agudas, porque la PCR puede resultar positiva incluso antes de aparecer los síntomas, mientras que los valores serológicos solo empiezan a positivizarse a partir de la cuarta semana para la IgM y de las 6-8 semanas para la IgG41. Este retraso en la respuesta serológica condiciona que en muchos casos el diagnóstico se realice de forma retrospectiva. Por el contrario, en infecciones crónicas o procesos crónicos relacionados con C.pneumoniae la carga bacteriana puede ser baja y la PCR puede resultar negativa, mientras que los valores serológicos se mantienen positivos, como la IgE en pacientes asmáticos42. La dificultad para definir la fase clínica de la infección puede dar lugar a resultados a veces contradictorios en la sensibilidad entre PCR y serología. Así, se encuentran en la literatura oscilaciones en la prevalencia de infecciones por C.pneumoniae de entre 0-44%, dependiendo del grupo de edad, de la fase de la infección, de la localización geográfica y de la técnica empleada, pero en general los ensayos de PCR rinden tasas más bajas (≤1%) de infecciones del tracto respiratorio inferior asociadas a C.pneumoniae que los test serológicos43. La posible sobreestimación de los casos, basándose en ensayos serológicos, podría estar condicionada por la larga vida media de la IgG de semanas o meses, teniendo en cuenta que la seroprevalencia en población adulta es ∼70%. Así, pacientes mayores o con enfermedad obstructiva crónica pueden tener títulos altos de IgG sin relación con la clínica. Otras explicaciones de la posible sobreestimación son problemas de reacción cruzada entre diferentes especies de Chlamydia. Por ello, en caso de infecciones crónicas la estimación de IgA (vida media de 5-7días) es un buen marcador de infección persistente. Debido a la alta prevalencia de anticuerpos en la población, el diagnóstico serológico de la infección requiere la disponibilidad de sueros pareados. La técnica de referencia es la microinmunofluorescencia (MIF)44, capaz de detectar IgM, IgG e IgA. Los criterios usados para identificar una infección por C. pneumoniae son: IgM≥1:16 o incremento de 4veces el título de IgG o IgG≥1:512.
Las técnicas de EIA frente al LPS o MOMP son específicas de género o especie, respectivamente, y al igual que en el caso de C.trachomatis son rápidas y fáciles de implementar, pero pueden dar lugar a falsos positivos por problemas de reacciones cruzadas. Estos ensayos deben validarse respecto a la técnica de referencia, la MIF, con una sensibilidad del 58-96% y una especificidad del 95-100%45.
Diagnóstico de Chlamydia psittaciLas técnicas serológicas constituyen la base del diagnóstico de la psitacosis. Según el CDC, se considera caso confirmado si existe un cuadro clínico compatible y se evidencia por fijación del complemento (FC) o MIF la presencia de IgM específicas con un título ≥1/16 o una seroconversión (>4veces) en sueros pareados entre la fase aguda y convaleciente, obtenidas con una diferencia entre 2-4semanas. Será caso probable si existe sospecha epidemiológica o se evidencia en suero único un título ≥1/32. Las técnicas de EIA son menos sensibles que la FC y la MIF. La MIF, o inmunofluorescencia indirecta (IFI), es probablemente la técnica más sensible y específica. Sin embargo, puede dar reacciones cruzadas con otras especies de Chlamydia. Para aumentar la fiabilidad de las técnicas se recomienda que los estudios serológicos se realicen al mismo tiempo y en el mismo laboratorio. Emplea antígenos de superficie de C.psittaci. El sistema comercial de Focus Diagnostics contiene antígenos para C.pneumoniae, C.trachomatis y C.psittaci. Tiene un elevado valor predictivo negativo (98%) y una alta sensibilidad. Otros sistemas detectan independientemente IgA, IgM e IgG para C.pneumoniae, C.trachomatis y C.psittaci.
Se han utilizado distintas aproximaciones basadas en PCR. Los ensayos de touchdown enzyme time release-PCR (TETR-PCR) amplifican regiones del 16S y regiones intergénicas ADNr16S-23S46. Esta aproximación tiene como característica realizar una PCR de hasta 60ciclos. También se han descrito métodos de PCR a tiempo real que amplifican el gen ompA o envB, empleando sondas específicas o SYBR green con distintos tipos de muestras47,48. Estas técnicas resultan positivas rápidamente, a partir del tercer día de síntomas, lo que supone una ventaja respecto a la serología. Sin embargo, no están disponibles comercialmente, por lo que se requiere una optimización y validación previa en cada laboratorio.
Diagnóstico de infecciones por nuevos géneros del orden ChlamydialesSe han desarrollado técnicas de amplificación de ácidos nucleicos, no comerciales, con fines epidemiológicos y diagnósticos de las infecciones por P.acanthamoebae, S.negevensis y W.chondrophila. De hecho, el hallazgo de las primeras especies relacionadas con Chlamydia se describió por la amplificación genérica (pan-clamidia) del ADNr16S y posterior secuenciación en muestras de origen clínico49. Recientemente se ha desarrollado una nueva técnica de PCR en tiempo real, también basada en ADNr16S pero específica para miembros de las familias Parachlamydiaceae, Simkaniaceae y Waddlia50,51. También se han desarrollado técnicas de EIA para S.negevensis, pero presentan reacción cruzada con C.pneumoniae. Asimismo, técnicas de MIF desarrolladas para C.pneumoniae se han empleado para P.acanthamoebae, con resultado positivo. Estas bacterias se han logrado cultivar en las mismas líneas celulares empleadas para el resto de clamidias.
Tipificación molecular en ChlamydiaLa tipificación molecular es de gran importancia en la caracterización de brotes o nodos de transmisión de C.trachomatis en redes sexuales. La aplicación de las técnicas moleculares mejora nuestra comprensión de la inmunopatogénesis de las infecciones por Chlamydia52. El genoma core, común a todos los miembros de la familia Chlamydiaceae, llega a ser ∼90% y comúnmente dispuesto en el mismo orden (sintenia). Esto supone una dificultad añadida al desarrollar sistemas de tipificación. Existen 2estrategias mayoritariamente aceptadas para la tipificación de cepas de Chlamydia. En 2007 se describió el MLST, basado en la secuenciación de 5regiones genómicas: CT046 (hctB), CT682 (pbpB), CT058, CT144, CT17253. En 2008 se describió el sistema VNTR, definido como diferencias en regiones repetidas de un mismo nucleótido o motivo. Las variaciones en el número de repeticiones da lugar a diferente longitud del segmento genómico, lo que permite diferenciar cepas. En esas regiones es fácil sospechar que la ADN-polimerasa sea más propensa a cometer errores. Se eligieron 3 regiones: CT1335, CT1299 y CT129154. El poder discriminativo de esta técnica aumenta al secuenciar también el gen ompA, técnica denominada Multilocus-VNTR-ompA (MLVA). Por MLVA se ha alcanzado gran poder de discriminación en cepas estudiadas de distintos serotipos de C.psittaci y C.trachomatis de diferentes orígenes geográficos. Esta estrategia muestra un índice Simpson de diversidad del 0,94, muy próximo al índice de diversidad óptimo recomendado en los sistemas de tipificación55 y similar al índice de diversidad obtenido con MLST17. Existe una segunda propuesta de MLST basada en 7genes, pero el índice de discriminación no es superior al MLST de 5genes. Por ello, el subcomité sobre la taxonomía de las clamidias sugirió, en su reunión de 2009, que al menos 5genes housekeeping deberían ser usados y que los criterios de distancia génica deberían ser comparados con análisis filogenéticos, cuando exista un número suficientemente alto de secuencias conocidas. La caracterización de cepas de C.psittaci por MLST ha demostrado su utilidad para la clasificación de genotipos de acuerdo con la especie del huésped. Sin embargo, esta estrategia tiene escaso poder de discriminación en cepas de C.pneumoniae.
Recientemente se ha publicado un estudio basado en la amplificación de una región variable de ompA con un sistema de PCR a tiempo real, seguido de un análisis del amplicón por high resolution melting (HRM). El sistema, sencillo y rápido, demostró su eficiencia en la detección y tipificación de C.psittaci y C.trachomatis56.
Conflicto de interesesLos autores declaran no tener conflicto de intereses.
Este trabajo está incluido dentro de Subprograma de Inmigración y Salud perteneciente al programa DAPET en CIBERESP (Biomedical Research in Network in Epidemiology and Public Health).