El cultivo de micobacterias es la prueba de referencia, y la más sensible, en el diagnóstico microbiológico de la tuberculosis y de las infecciones por micobacterias no tuberculosas. Sin embargo, requiere al menos 2-3 semanas para ser positivo. La tinción, rápida, aunque poco sensible, actúa como un complemento. Las pruebas de amplificación genética tienen una sensibilidad intermedia y obtienen resultados en 1-2 días. Estas últimas están indicadas cuando el grado de sospecha es moderado o alto. En pacientes infectados por el VIH con inmunodepresión severa (<200 CD4) puede ser útil la detección de antígeno lipoarabinomanano en orina.
La identificación de los aislamientos de los cultivos positivos es imprescindible para evaluar la significación clínica y la orientación terapéutica. Actualmente se dispone de un abanico de técnicas de identificación que facilitan resultados en periodos de solo 1-4 días.
El futuro del diagnóstico pasa por un mayor desarrollo de las técnicas de amplificación genética y por potenciar la búsqueda de biomarcadores que permitan un nuevo enfoque del diagnóstico de estas infecciones.
Mycobacterial culture has a high sensitivity and is the test of choice for the microbiological diagnosis of tuberculosis and nontuberculous mycobacterial infections. However, the results of this culture require at least 2-3 weeks to obtain positivity. Staining is rapid and can be used as a complementary study, although its sensitivity is low. Gene amplification tests have an intermediate sensitivity and obtain results in 1-2 days. These last tests are indicated in cases with moderate or high clinical suspicion. In HIV patients with severe immunodeficiency (<200 CD4), lipoarabinomannan antigen detection in urine may be useful.
The identification of isolates from positive cultures is essential to evaluate the clinical significance of the culture results and consider the therapeutic options available. At present, there is a wide range of identification techniques available, which provide results within just 1-4 days.
The future of diagnostic techniques in tuberculosis and nontuberculous mycobacterial infections lies in greater development of gene amplification techniques and promoting the search for biomarkers which enable a new approach to the diagnosis of these infections.
En la actualidad, hay descritas alrededor de 170 especies del género Mycobacterium1. La más importante es Mycobacterium tuberculosis, agente de la tuberculosis (TB), que causa 1,6 millones de muertes/año2. El último informe de casos comunicados en España indica que la incidencia de TB en 2014 fue de 10,8/100.000h3. Las infecciones por micobacterias no tuberculosas (MNT) han aumentado en los últimos años, sobre todo en pacientes con enfermedades pulmonares crónicas. La frecuencia de aislamientos presenta una distribución geográfica no homogénea en los diferentes países, relacionada con las condiciones ambientales en que se desarrollan las MNT4. En nuestro entorno suponen alrededor del 35-40% de los aislamientos clínicos de micobacterias. En la presente revisión se revisan los métodos de diagnóstico microbiológico de la TB y las infecciones por MNT. Se comentan las pruebas básicas para el diagnóstico directo, así como las utilizadas en la identificación de los cultivos positivos (tablas 1 y 2).
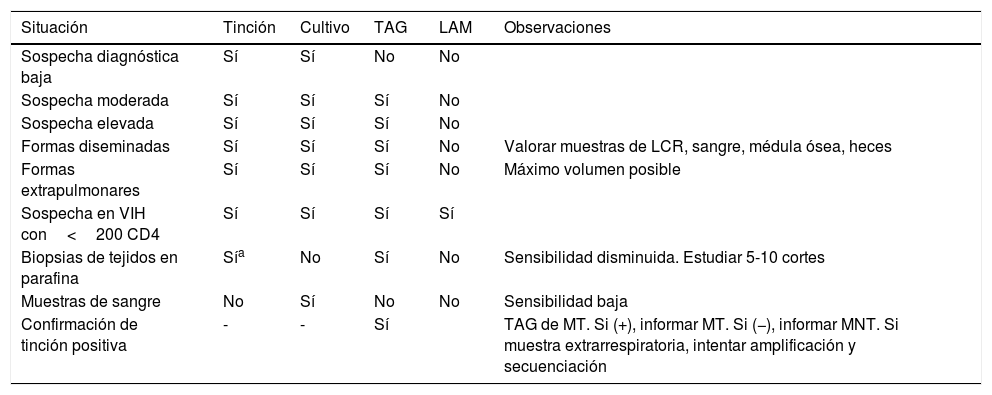
Utilización de las pruebas de diagnóstico directo en muestras clínicas
| Situación | Tinción | Cultivo | TAG | LAM | Observaciones |
|---|---|---|---|---|---|
| Sospecha diagnóstica baja | Sí | Sí | No | No | |
| Sospecha moderada | Sí | Sí | Sí | No | |
| Sospecha elevada | Sí | Sí | Sí | No | |
| Formas diseminadas | Sí | Sí | Sí | No | Valorar muestras de LCR, sangre, médula ósea, heces |
| Formas extrapulmonares | Sí | Sí | Sí | No | Máximo volumen posible |
| Sospecha en VIH con<200 CD4 | Sí | Sí | Sí | Sí | |
| Biopsias de tejidos en parafina | Sía | No | Sí | No | Sensibilidad disminuida. Estudiar 5-10 cortes |
| Muestras de sangre | No | Sí | No | No | Sensibilidad baja |
| Confirmación de tinción positiva | - | - | Sí | TAG de MT. Si (+), informar MT. Si (−), informar MNT. Si muestra extrarrespiratoria, intentar amplificación y secuenciación |
AG: técnicas de amplificación genética; LAM: lipoarabinomanano en orina; LCR: líquido cefalorraquídeo; MNT: micobacterias no tuberculosas; MT: Mycobacterium tuberculosis; VIH: virus de la inmunodeficiencia humana.
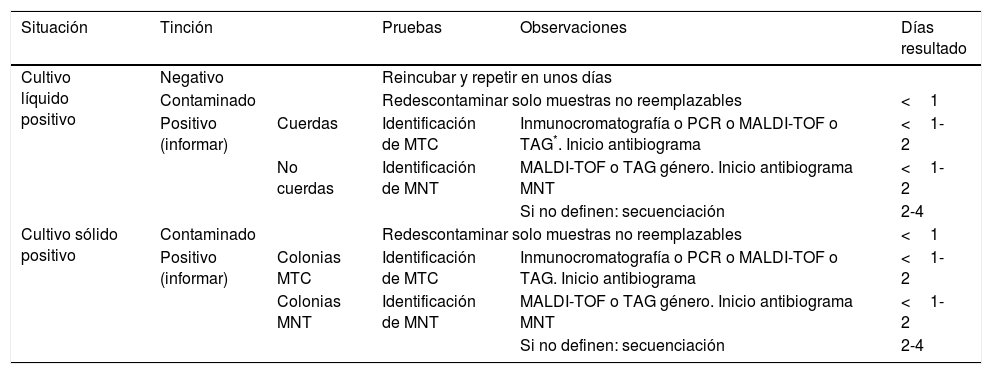
Algoritmo de utilización de las pruebas de identificación de los cultivos positivos
| Situación | Tinción | Pruebas | Observaciones | Días resultado | |
|---|---|---|---|---|---|
| Cultivo líquido positivo | Negativo | Reincubar y repetir en unos días | |||
| Contaminado | Redescontaminar solo muestras no reemplazables | <1 | |||
| Positivo (informar) | Cuerdas | Identificación de MTC | Inmunocromatografía o PCR o MALDI-TOF o TAG*. Inicio antibiograma | <1-2 | |
| No cuerdas | Identificación de MNT | MALDI-TOF o TAG género. Inicio antibiograma MNT | <1-2 | ||
| Si no definen: secuenciación | 2-4 | ||||
| Cultivo sólido positivo | Contaminado | Redescontaminar solo muestras no reemplazables | <1 | ||
| Positivo (informar) | Colonias MTC | Identificación de MTC | Inmunocromatografía o PCR o MALDI-TOF o TAG. Inicio antibiograma | <1-2 | |
| Colonias MNT | Identificación de MNT | MALDI-TOF o TAG género. Inicio antibiograma MNT | <1-2 | ||
| Si no definen: secuenciación | 2-4 | ||||
MNT: micobacterias no tuberculosas; MTC: Mycobacterium tuberculosis complex; PCR: reacción en cadena de la polimerasa; TAG: técnicas de amplificación genética.
Por último, se mencionan algunas de las técnicas que pueden adquirir relevancia diagnóstica en el futuro.
Diagnóstico directo en muestras clínicasMuestrasDeben ser representativas de la localización y de la sintomatología5,6. La localización más frecuente en la TB y las infecciones por MNT es la pulmonar. En pacientes que expectoran son adecuadas 2-3 muestras seriadas de esputo matinal. En ausencia de expectoración, están indicadas muestras obtenidas por broncoscopia, como broncoaspirado o lavado broncoalveolar. En niños o en situaciones sin acceso a broncoscopia, el aspirado gástrico puede ser una alternativa. Los líquidos biológicos, como el cefalorraquídeo, el articular o el pleural, estarán indicados en sus presentaciones clínicas, siempre que no sea posible obtener una biopsia relacionada. En la enfermedad renal se usan muestras seriadas de orina matinal. En afectaciones de tejidos u órganos, las muestras se obtendrán por punción o preferiblemente por biopsia. Si se sospecha infección diseminada, pueden ser útiles los cultivos de sangre, médula ósea y heces. En general, exceptuando la inmunodepresión avanzada, las infecciones por micobacterias son poco bacteriémicas.
Deben considerarse las siguientes cuestiones básicas para lograr una sensibilidad óptima5:
1. Obtención y recipientes estériles: la contaminación bacteriana disminuye la sensibilidad.
2. Volumen mínimo adecuado: 5ml en muestras respiratorias sin diluir o 7-10ml en diluidas. Al menos 1ml en líquido cefalorraquídeo; 5-10ml en otros líquidos biológicos. Biopsia en lugar de punción.
3. Transporte: como norma general, las muestras deben enviarse al laboratorio en las 2h siguientes a su obtención. Las biopsias y tejidos deben mantenerse húmedos con agua destilada estéril. Es muy importante evitar el formol utilizado en el análisis anatomopatológico, ya que es letal para todos los microorganismos. Las muestras parafinadas pueden utilizarse para detección génica, aunque el proceso de desparafinización y de digestión del tejido disminuye notablemente la sensibilidad. El pH ácido del jugo gástrico es nocivo para las micobacterias, por lo que es importante el transporte rápido o usar neutralizantes.
4. La calidad de las muestras, es decir, que provengan del foco infeccioso, indicado por la presencia de células inflamatorias.
5. La petición de múltiples pruebas atomiza las muestras y disminuye la sensibilidad. Es crucial un diagnóstico diferencial previo y un diálogo con el laboratorio para decidir cuáles son las pruebas más adecuadas a realizar en cada caso.
Métodos basados en detección directa del microorganismoTinciones directamente sobre muestrasEl elevado porcentaje de lípidos de la pared de las micobacterias es responsable de la dificultad de penetración de los colorantes, así como de su posterior salida. Esta propiedad, conocida como ácido-alcohol resistencia, es la causa de que no se tiñan bien con la tinción de Gram y de que existan tinciones específicas. Estas se dividen en 2 tipos, según se observen con microscopio óptico (MO) o de fluorescencia. Entre las primeras, la más conocida es la tinción de Ziehl-Neelsen, que colorea de rojo a las micobacterias y de azul las estructuras restantes. Las segundas resaltan las micobacterias en color fluorescente sobre un fondo oscuro. La más utilizada es la tinción de auramina-rodamina6.
La especificidad de la tinción es elevada, con escasas excepciones distinguibles por su morfología (Nocardia, Rhodococcus, Tsukamurella, Cryptosporidium, Cyclospora e Isospora), ya que la mayoría de los microorganismos no se tiñen de rojo o fluorescente6.
La sensibilidad de la tinción está condicionada por factores como la extensión de la enfermedad, el volumen de la muestra, la concentración de micobacterias y, sobre todo, por el tipo de muestra. El límite de sensibilidad estaría entre 50.000 y 100.000 micobacterias/ml. La sensibilidad global puede oscilar entre 20-80% de las muestras procesadas, de acuerdo con su tipo y origen anatómico5. Es más elevada en muestras respiratorias, sobre todo en lesiones cavitadas, y en tejidos, como adenopatías. En nuestro entorno se sitúa entre el 50 y el 80%. En líquidos biológicos es muy poco sensible por la baja concentración de micobacterias. La calidad de la tinción no es adecuada en muestras de sangre o muy hemáticas, por lo que no se recomienda efectuarla. La tinción realizada del concentrado después de descontaminar la muestra para el cultivo es de un 11 a un 26% más sensible que si se realiza directamente de la muestra5. Del mismo modo, se admite que las tinciones fluorescentes son un 10% más sensibles que las basadas en MO5, aunque en general se recomienda confirmar los positivos realizando tinción de MO6 o una técnica de amplificación génica. Las tinciones deben observarse detenidamente, al menos 300 campos en MO y 30 para microscopio de fluorescencia, antes de emitir un informe de negatividad. La positividad se reporta semicuantificada de 1+ a 4+6. El resultado, principalmente en las formas pulmonares, proporciona información sobre la extensión de la afectación, así como de la contagiosidad y el periodo de aislamiento, en el caso de la TB. También es útil para evaluar la respuesta terapéutica.
Las infecciones por MNT son cada vez más frecuentes. Sin embargo, cuando la tinción directa es positiva, en general no permite diferenciar claramente entre estas y Mycobacterium tuberculosis complex (MTC).
Métodos de cultivoEl cultivo es el método diagnóstico más sensible. Se estima que su límite de detección está entre 10 y 100 bacterias/ml5. Su principal inconveniente es el largo periodo de incubación, desde varios días hasta 6 semanas antes de emitir un informe definitivo de negatividad. Ello es debido a que la velocidad de división de las micobacterias, entre 18 y 24h, es 40 veces más lenta que en la mayoría de las bacterias. Por esta misma razón, en las muestras con presencia de flora o de otras bacterias, como las de origen respiratorio, debe realizarse una eliminación previa de estas, a través de un proceso de descontaminación química, preservando las micobacterias. Existen varios métodos de descontaminación. El más conocido se basa en el uso de NaOH y N-acetil-L-cisteína (método de Kubica). Se aconseja no utilizarlo en muestras habitualmente estériles, ya que no es totalmente inocuo para las micobacterias. Por otra parte, una proporción de cultivos se contaminarán a pesar de la descontaminación. Se considera aceptable entre un 3 y un 5%, aunque dependerá del tipo de muestras de cada laboratorio. Las muestras que contengan Pseudomonas aeruginosa, bacterias capsuladas, levaduras u hongos, entre otros, tendrán más posibilidades de contaminar los cultivos. Hay métodos de descontaminación dirigidos a este tipo de muestras6.
La mayoría de las especies de micobacterias suelen ser nutricionalmente exigentes, por lo que se utilizan medios de cultivo específicos. No obstante, la mayoría de ellas pueden crecer en medios enriquecidos comunes, como agar sangre, agar chocolate o los utilizados en los hemocultivos, aunque más lentamente que en los propios específicos. Actualmente, para los aislamientos primarios a partir de muestras clínicas se recomienda el uso combinado de un cultivo líquido y un cultivo sólido. Los medios sólidos pueden estar basados en huevo coagulado o en agar. Los más conocidos de ambos tipos son Löwenstein-Jensen y Middlebrook 7H11, respectivamente. Otros medios que contienen huevo son Trudeau, Coletsos, ATS o Petragnani. Middlebrook 7H10 sería otro medio basado en agar. En algunas áreas geográficas se utilizan medios selectivos que incluyen antibióticos (7H11S o 7H10S) para los aislamientos primarios. Existen numerosos medios líquidos, como Middlebrook 7H9, Dubos, Youmans o Proskauer-Beck, entre otros. El más usado actualmente es el primero. La mayoría de los medios líquidos usados en diagnóstico están semiautomatizados, basados en el uso de incubadores capaces de detectar el crecimiento por consumo de O2 o producción de CO2, a través de un sistema de monitorización continua de las botellas o tubos que contienen el medio de cultivo y la muestra. Suelen utilizar Midlebrook 7H9 como medio base, complementado con suplementos de enriquecimiento y soluciones antibióticas para neutralizar microorganismos que hayan resistido la descontaminación. Tres métodos son los más usuales: VersaTREK® (Thermo Fisher Scientific, Oakwood Village, OH, US), BacT ALERT 3D® (BioMerieux, Marcy l’Etoile, Francia) y MGIT960® (Becton Dickinson, Sparks, MD, US). El primero contiene unas esponjas de celulosa incluidas en el medio líquido, que también permiten ofrecer una superficie de cultivo sólida para el crecimiento. El incubador dispone de sensores de presión capaces de detectar crecimiento a partir del consumo de O2. BacT ALERT 3D® posee sensores que registran cambios de color ocasionados por la producción de CO2 en un indicador contenido en el fondo de cada botella de cultivo. MGIT960® utiliza tubos que contienen un fondo de silicona en el que está incluido un indicador que emite fluorescencia ante cambios en el consumo de O2 en el tubo. Los cultivos para la mayoría de las micobacterias se incuban a 35-37°C. Las especies causantes de enfermedad cutánea y de tejidos blandos, como M. ulcerans, M. marinum, M. haemophilum y M. chelonae, crecen mejor a 25-30°C. Contrariamente, M. xenopi crece mejor a 42°C y M. thermoresistibile a 52°C. Algunas especies necesitan suplementos específicos para crecer en los cultivos. Los ejemplos más claros son M. haemophilum, que precisa hemina, hemoglobina o citrato amónico férrico, o M. genavense. Este último crece muy lentamente, hasta 3-6 meses, generalmente solo en medios líquidos con pH acidificado, como el utilizado para el antibiograma de pirazinamida en el sistema MGIT960® o en medios sólidos suplementados con micobactina J, carbón activado o sangre.
Los cultivos se monitorizan de forma continua en los equipos de cultivos líquidos, detectando los potenciales positivos cuando se producen. Los cultivos sólidos se inspeccionan 1-2 veces/semana durante las 3 primeras semanas y una vez durante las 3 siguientes. La sensibilidad de los medios de cultivo está relacionada con la carga bacteriana de la muestra, a su vez vinculada al tiempo de evolución de la enfermedad en cada paciente y a la localización clínica. Cuando el grado de sospecha sea elevado o moderadamente elevado, cursar 2-3 muestras seriadas aumentará la sensibilidad. En nuestro medio, en muestras de origen respiratorio puede oscilar entre el 70 y el 85%, al igual que en tejidos sólidos como adenopatías. En localizaciones con líquidos biológicos, el rendimiento es menor, no alcanzando más de 35-50% en las mejores situaciones. El mejor rendimiento se observa con el uso de cultivos líquidos automatizados7. En cuanto al periodo de detección de positivos, la mayoría se positivizan entre la primera y la tercera semana. La media está en 14 días. En detalle, para muestras con baciloscopia positiva el periodo de positividad desciende a 13 días para aislamientos de MTC y a 11 días en MNT. Cuando la baciloscopia es negativa, se sitúa en 17 y 16 días, respectivamente, para MTC y MNT. Los cultivos sólidos se positivizan entre la segunda y la cuarta semana. Globalmente, casi el 100% de los cultivos, sólidos y líquidos, se positivizan en las 4 primeras semanas. En muestras de seguimiento, después de iniciar el tratamiento, este porcentaje se alcanzaría a las 5 semanas. Por otra parte, la recuperación en medios líquidos es netamente superior a la de los sólidos, sobre todo para el aislamiento de MNT.
Métodos basados en la detección de material genético o antígenosMétodos genéticos aplicados a muestra directa en el diagnóstico de Mycobacterium tuberculosis complex (MTC)Las pruebas de detección molecular en muestra clínica están basadas en la amplificación y la posterior detección de ácidos nucleicos. Cada vez se utilizan más para diagnosticar la TB debido a la rapidez con que se obtienen los resultados, y a la sensibilidad en comparación con la microscopia. Se han desarrollado gran cantidad de técnicas basadas en la amplificación de secuencias específicas de MTC, principalmente 16S ARNr, IS6110 o el gen rpoB, y muchas de ellas se han comercializado. Hay publicaciones recientes en las que se detallan8–10. En la presente revisión se comentan algunas de ellas. Es importante tener en cuenta que la sensibilidad de las técnicas de amplificación es inferior a la del cultivo en aquellas muestras con microscopia negativa, por lo tanto, no se pueden utilizar para descartar la enfermedad. Para aumentar la sensibilidad de estas pruebas y lograr resultados similares al cultivo, se deberían eliminar eficazmente los inhibidores que se hallen en la muestra para detectar≤10-100 micobacterias/ml, el límite inferior de detección del cultivo11, y además para su aplicación en la rutina es importante la consistencia y sistematización de la técnica, de forma que se manipule lo menos posible12. Por este motivo, se considera más adecuada la utilización de técnicas comerciales frente a las caseras. Ya en 2010, apareció el sistema Xpert® MTB/RIF (Cepheid, Sunnyvale, CA, Estados Unidos) como un método totalmente automatizado, con una mínima manipulación previa a la introducción de la muestra en el cartucho donde se desarrolla toda la reacción. La técnica consiste en una nested PCR a tiempo real del gen rpoB, que codifica para la subunidad de la ARN polimerasa. Utiliza 5 sondas genéticas del tipo molecular beacon, cada una marcada con un fluoróforo distinto. Cubre totalmente la zona de 81 pares de bases, entre los codones 426 y 452 del gen rpoB (507 y 533 referidos a la nomenclatura del gen en Escherichia coli), determinante de resistencia a la rifampicina. De esta manera, informa simultáneamente de la presencia de MTC y de las mutaciones más frecuentes en el gen rpoB, prediciendo la resistencia a rifampicina, que en la mayoría de los casos va ligada a multirresistencia. Según una reciente revisión de la Cochrane13, tiene una sensibilidad del 89%, siendo del 68% para muestras con baciloscopia negativa, y una especificidad del 98%. Su rapidez y extrema sencillez hace que sea muy atractivo, habiendo sido impulsado por la OMS como herramienta para diagnosticar rápidamente tanto la TB como las cepas multirresistentes en las zonas con altas tasas14 (http://www.who.int/tb/publications/xpert-mtb-rif-assay-diagnosis-policy-update). En los países desarrollados el mayor inconveniente es el precio, aunque en un estudio muy reciente se expone que su uso en todos los pacientes podría resultar coste-efectivo mejorando el diagnóstico diferencial15. La baja sensibilidad de este sistema en las muestras paucibacilares ha impulsado a la misma empresa a desarrollar un sistema Xpert® MTB/RIF Ultra que recoge un volumen de muestra mayor que el modelo anterior, y añade 2 nuevas dianas que se encuentran en mayor número de copias en el genoma de MTC (IS6110 y IS1081). Ambos cambios logran aumentar la sensibilidad considerablemente, lo que ha llevado a la OMS a proponer su utilización especialmente en los casos de pacientes infectados por el VIH y pacientes pediátricos16. No obstante, es necesario realizar estudios que avalen los resultados de especificidad de este nuevo sistema.
Otros métodos disponibles en el mercado de nuestro entorno para la detección directa son los ideados por HAIN Lifescience: Fluorotype® MTB y GenoQuick® MTB (HAIN Lifescience, Nehren, Alemania). Fluorotype® MTB es un método semiautomatizado comercializado recientemente, basado en PCR a tiempo real. Las primeras evaluaciones17 indican una especificidad del 98,9%, con una sensibilidad del 100% para muestras con microscopia positiva y del 56,3% para microscopia negativa. GenoQuick® MTB aplica una tecnología GenoQuick® para el diagnóstico del MTC directamente de muestra pulmonar y extrapulmonar. Puede realizarse en pequeños laboratorios, ya que únicamente es necesario un termociclador específico del sistema. Realiza en 2-3h la extracción y amplificación de los ácidos nucleicos y desnaturaliza para hibridar con sondas específicas marcadas con oro; utilizando un sistema de amortiguación de flujo lateral, se unirá a un sitio específico en la tira de hibridación del ensayo18.
Por otra parte, los test comerciales desarrollados para la detección de mutaciones de resistencia también tienen utilidad en el diagnóstico de TB debido a que en las versiones más recientes han introducido mejoras en la sensibilidad que permiten aplicarlos sobre muestra clínica. FluoroType® MTBDRplus (HAIN Lifescience, Nehren, Alemania), TB Resistance módulos (AID Diagnostika, Strassberg, Alemania)19 y AnyplexTM plus MTB/NTM/MDR-TB (Seegene, Seoul, Corea)20 son algunos de los disponibles.
Con el propósito de aumentar la sensibilidad en el diagnóstico la literatura reciente recoge diversas propuestas con posible implantación en el futuro. Una de ellas es el sistema XtracTB12. Se ha diseñado para su inclusión en una plataforma automatizada en un laboratorio central. Combina varias dianas de ADN con varios lavados para reducir la presencia de los inhibidores de qPCR con la posterior amplificación de 2 dianas específicas de MTC, IS6110 y senX3-regX3, para aumentar la sensibilidad y especificidad de la prueba y minimizar la probabilidad de falsos negativos. Los primeros ensayos realizados muestran una sensibilidad de 5 copias genómicas/mL en esputo que rivalizan con la sensibilidad del cultivo. Este sistema no detecta genes de resistencia, pero la tecnología podría utilizarse en este sentido.
Por último, aunque la tendencia es el uso de test comerciales automatizados o semiautomatizados, la OMS21 ha recomendado el uso de la técnica loop-mediated isothermal amplification o LAMP en su estrategia de diagnóstico rápido de la TB; consiste en una amplificación isotérmica que utiliza 6 primers, uniéndose a 8 dianas distintas. Puede realizarse con tecnología mínima y lectura manual con una lámpara UV, por lo que es costo-efectiva y potencialmente asequible en numerosos países. Datos aportados recientemente por la OMS21 cifran su especificidad en un 98%, con una sensibilidad del 96,6% para muestras con microscopia positiva y del 42,2% cuando la microscopia es negativa.
Métodos genéticos aplicados a muestra directa en el diagnóstico de las Micobacterias no tuberculosas (MNT)El diagnóstico molecular de las MNT directamente en muestra clínica ha centrado menor atención que el diagnóstico de la TB, probablemente por razones de incidencia y de importancia epidemiológica, aunque en los últimos años está adquiriendo un interés creciente. La diversidad de especies dificulta un enfoque específico, por lo que la mayoría de las técnicas en la literatura se basan en métodos caseros y generalmente dirigidos al diagnóstico de género, sobre todo amplificando el gen ARNr 16S. Una aproximación ejercida en algunos laboratorios es secuenciar el producto de amplificación para intentar el diagnóstico de especie. Existen algunos test comerciales aplicables al diagnóstico de las MNT. Así, Anyplex plus MTB/NTM MDR-TB combinaría la identificación de MTC y de Mycobacterium sp. en una única amplificación a tiempo real. Otra aproximación sería el test GenoType® (GT) NTM-DR (HAIN Lifescience, Nehren, Alemania), capaz de detectar mutaciones de resistencia a macrólidos y aminoglucósidos, al mismo tiempo que puede identificar algunas especies de MNT que se aíslan con frecuencia en muestras clínicas, como M. chelonae, M. avium, M. intracellulare y M. chimaera, así como las 3 subespecies de M. abscessus (sub. abscessus, sub. bolletii, sub. massiliense). Este test se utiliza sobre todo en la caracterización de aislados clínicos. No se posee aún experiencia contrastada respecto a su uso directamente sobre muestras clínicas.
Detección de antígeno lipoarabinomanano en orinaLa detección directa del antígeno de pared lipoarabinomanano (LAM) en la orina es una técnica innovadora. LAM es un glucolípido termoestable específico liberado por las micobacterias que es filtrado por el riñón y está presente en la orina de los pacientes con TB activa. Inicialmente fue analizado en el suero, pero la formación de complejos inmunes dificultaba su detección. El test Determine TB LAM Ag® (Alere Inc., Waltham, MA, EE.UU) está basado en inmunocromatografía y utiliza anticuerpos de captura específicos frente al antígeno LAM. El antígeno presente en orina es capturado por un conjugado de anticuerpo y oro coloidal formando un inmunocomplejo, que a su vez se une a anticuerpos anti-LAM fijados en una membrana de nitrocelulosa. La detección se hace visible gracias a las partículas de oro coloidal con las que está marcado el complejo. La sensibilidad de esta técnica oscila entre un 38 y un 50,7% para TB y la especificidad entre un 87,8 y un 89%, frente a la microscopia, el cultivo en medio sólido y/o líquido. En pacientes con VIH e inmunodepresión severa, CD4<200, la sensibilidad aumenta significativamente hasta el 62%5. Presenta varias ventajas que lo hacen muy útil en determinadas circunstancias: la orina es una muestra más fácil de recoger que el esputo, puede ser menos variable en calidad y es más segura de manejar. No requiere de infraestructura, puede recogerse a pie de cama del paciente, ni de equipamiento, con un coste inferior a 3$. Se han publicado recientemente varios estudios comparativos entre Determine TB LAM Ag® y Xpert MTB/RIF® en países de alta incidencia de tuberculosis y VIH22,23. El test LAM es más sensible en pacientes con niveles bajos de CD4, llegando a mostrar una disminución de un 4% en la mortalidad a las 8 semanas en los pacientes que fueron diagnosticados al ingreso en hospital mediante este test23. Con estos resultados, se recomienda su utilización en hospitales donde los pacientes ingresan con inmunodepresión severa y sin capacidad de expectoración. Aparte de la sensibilidad, otra limitación reconocida por el propio fabricante es que no diferencia entre el MTC y otras micobacterias.
Estrategia de uso de las pruebas de diagnóstico directoLas características clínicas de la TB y las infecciones por MNT conllevan que formen parte de la sospecha diagnóstica en un gran número de procesos infecciosos de curso subagudo o crónico, muchos de los cuales acaban teniendo un diagnóstico distinto, por lo que es aconsejable un uso racional de las pruebas, acorde al grado de sospecha clínica (tabla 1). El cultivo debe realizarse siempre en todas las muestras en que interese descartar micobacterias. Es aconsejable también que se procesen 2-3 muestras seriadas, sobre todo en la localización pulmonar. La tinción microscópica es la prueba más rápida y barata, aunque también la menos sensible. En líquidos biológicos tiene una sensibilidad muy baja y probablemente debería reevaluarse su indicación en estas muestras.
Las pruebas basadas en amplificación genética solo están indicadas si el grado de sospecha es elevado o medianamente elevado y pensando casi siempre en TB. En muestras ganglionares en niños o en pacientes inmunodeprimidos estará indicada la amplificación de MTC y la de género Mycobacterium. En muestras cutáneas, la etiología más frecuente será M. marinum u otra MNT, por lo que estará indicada la amplificación de género, con o sin amplificación de MTC. En pacientes con enfermedad pulmonar crónica y clínica de reagudización, cuando se plantee el uso de técnicas de amplificación, estas deben estar sobre todo dirigidas a MNT.
Siempre que estén disponibles, es ideal utilizar métodos comerciales, ya que garantizan el control de calidad y la consistencia de las pruebas. Así mismo, es aconsejable que requieran la menor manipulación posible. Si la sospecha es elevada, utilizar las pruebas sobre muestras seriadas aumentará la sensibilidad. Una aplicación importante de la amplificación genética será confirmar o descartar como MTC las muestras con tinción positiva. Es aconsejable que se realice en las siguientes horas a la tinción para mantener o anular el aislamiento respiratorio y para orientar el tratamiento.
En las localizaciones extrapulmonares, es importante disponer del volumen de muestra necesario, después de un diagnóstico diferencial lo más exhaustivo posible e intentando reducir el abanico de pruebas a realizar, así como tener en cuenta el diagnóstico cuando se toman biopsias o tejidos quirúrgicamente, preservando una parte de estas muestras libres de formol.
Identificación de los aislamientosLa identificación de los aislamientos puede realizarse desde varias aproximaciones (tabla 2). Una estrategia común es comprobar con una tinción microscópica que el crecimiento corresponde a micobacterias. Para la identificación de especie pueden utilizarse métodos basados en la detección de antígenos, como la inmunocromatografía; en la amplificación genética, con todas sus posibles variantes; o en la matrix assisted laser desorption ionization time-of-flight espectrometría de masas (MALDI-TOF). Todos estos métodos ofrecen mayor precisión y rapidez que la identificación basada en pruebas bioquímicas, que históricamente ha sido el pilar de la identificación, pero que actualmente está relegada a ser coadyuvante en situaciones puntuales. Los actuales métodos moleculares también han ocasionado que técnicas basadas en la cromatografía líquida de alta presión o en la cromatografía de gas, que en su momento fueron una alternativa a los métodos bioquímicos, tengan un uso muy minoritario.
Morfología de las colonias y tincionesCuando los cultivos se positivizan debe comprobarse que lo que está creciendo son micobacterias. Los incubadores de cultivos líquidos indican cuándo el cultivo de una muestra alcanza el crecimiento estipulado como punto de corte para ser considerado positivo. La inspección visual permite una orientación, ya que MTC y otras especies de MNT, no todas, no enturbian el medio, como sí lo hacen las de crecimiento rápido y las bacterias convencionales.
En los cultivos sólidos se observa el crecimiento macroscópico de colonias. Dependiendo de la especie, la morfología es distinta. MTC crece habitualmente con colonias planas, rugosas y de aspecto seco, de color entre el blanco y el amarillo claro. En las restantes especies pueden observarse distintas morfologías, y algunas son pleomorfas. Alrededor de 10 especies son pigmentadas, estimuladas o no por la luz (fotocromógenas o escotocromógenas). Cuando los cultivos sólidos se contaminan, alteran el pH del medio o lo digieren en parte y generan colonias distintas, por lo que son claramente evidentes.
Realizar una tinción de Ziehl-Neelsen del medio, sólido o líquido, confirma la positividad, descarta la contaminación del cultivo o cultivos mixtos, así como falsos positivos. La morfología de tinción que adoptan las micobacterias crecidas en los cultivos, sobre todo los líquidos, permite distinguir con cierta precisión si se trata de MTC o de MNT. La agrupación de varios centenares de bacilos formando cuerdas es muy indicativa de MTC. Durante años se ha considerado un patrón único para MTC, aunque recientemente se ha observado en cultivos de M. abscessus y se ha descrito también en otras especies de MNT24. Otras especies, como M. avium complex, M. kansasii o M. xenopi, suelen tener morfologías muy características en la tinción25. Esta primera aproximación permite emitir un informe de positividad e indicar las pruebas adecuadas para la identificación de la especie aislada.
Identificación por inmunocromatografíaPara identificar y diferenciar rápidamente MTC de otras micobacterias se han desarrollado varias pruebas comerciales basadas en la detección del antígeno MPT64, que fue detectado en los filtrados de cultivos de MTC26. Este test sencillo, rápido y sin apenas manipulación ofrece resultados en menos de 15min y sin necesidad de ningún equipamiento, por lo que los costes son reducidos. Utiliza una membrana de nitrocelulosa con el anticuerpo monoclonal anti-MPT64 de ratón fijado en ella. La prueba debe realizarse a partir de un cultivo positivo. En el caso de cultivo en medio líquido se aplica directamente. A partir de medio sólido se deben resuspender las colonias en un tampón de extracción que aporta el comercial. Al enfrentar la suspensión bacteriana y los anticuerpos, en el caso de existir el antígeno excretado al medio, se aprecia una línea de color. La sensibilidad y especificidad están muy cercanas al 100%. Sin embargo, esta técnica puede presentar falsos negativos, ya que algunas cepas del complejo (como se ha visto en algún aislamiento de M. bovis BCG) no logran producir esta proteína debido a polimorfismos del gen que codifica el antígeno MPT64. El gen mpt64 (Rv1980c) se encuentra en una región diferencial llamada RD227. La región RD2 fue delecionada en cepas de M. bovis BCG entre los años 1927 y 1931, coincidiendo con los informes de atenuación de la vacuna. Afecta a varios subtipos de BCG, entre ellos BCG Pasteur, BCG Danish y BCG Glaxo, mientras que no afectaría a BCG Japón y Moreau, entre otros28.
Existen diversos productos comerciales que utilizan esta metodología como diagnóstico (SD BIOLINE TB Ag MPT64 Rapid Test® kit, Alere Healthcare SLU, Hospitalet de Llobregat, España; Capilia TB®, TAUNS Laboratories Inc., Numazu, Shizouka, Japón; BD MGIT TBc Identification test®, Becton Dickinson Diagnostics, Sparks, MD, EE.UU).
Identificación con métodos de PCRSe dirige sobre todo a la identificación de MTC. Se basa en la utilización de métodos de amplificación, comerciales o caseros, que tienen como diana genes específicos de MTC, como la IS6110. Potencialmente todos los métodos diseñados para la detección directa de MTC pueden usarse como test confirmatorio de la identificación.
Identificación con métodos de Line Probe AssayLos métodos de Line Probe Assay (LPA) se basan en amplificación específica y posterior hibridación en fase sólida con sondas genéticas inmovilizadas sobre tiras de nitrocelulosa. La hibridación se hace evidente utilizando un revelado enzimático. Existen distintos test comerciales, entre ellos INNO-LiPA® MYCOBACTERIA (Innogenetics, Ghent, Bélgica) y GT Mycobacterium (HAIN Lifescience, Nehren, Alemania). El primero de ellos, realiza una amplificación del espacio intergénico 16S-23S, y posteriormente hibrida sobre una tira de nitrocelulosa que tiene 22 sondas que permiten identificar el MTC y otras 16 MNT, de relevancia clínica. El test GT Mycobacterium amplifica el gen 23S ARNr y existen 3 diferentes formatos: el GT MTBC, que identifica las distintas especies integrantes del MTC; el formato GT CM, que identifica MTC y 13 especies de MNT, y por último, la presentación GT AS, que identifica 16 especies más, por lo que en conjunto pueden identificar 32 especies, incluidas entre las más frecuentemente aisladas.
Identificación con espectrometría de masas MALDI-TOFEsta técnica se está implantando rápidamente en los laboratorios de microbiología, sobre todo para la identificación de los aislamientos bacterianos, aunque se extiende a otros microorganismos como hongos y parásitos. Se trata del análisis del perfil proteico de los microorganismos a través de un espectrómetro de masas y la comparación con una librería de espectros de perfiles previamente identificados para las diferentes especies. En los últimos años ha revolucionado la identificación bacteriana, ya que puede conseguir resultados en minutos a partir de cultivos positivos (tabla 2). Las muestras a analizar pueden ser cultivos sólidos o líquidos. Para el análisis, alrededor de 1μl de la muestra se mezcla con una matriz que facilita su ionización tras ser bombardeada por un haz láser, que provoca una disrupción y una migración de las proteínas a lo largo del tubo del espectrómetro de masas, de acuerdo con su carga eléctrica y tamaño, determinándose un time of flying para cada una de ellas y creándose un perfil para cada muestra. El equipo posee una librería de perfiles para comparar con el de la muestra. El grado de similitud se traduce en un valor numérico o score. El software del equipo indica los intervalos de interpretación de los scores obtenidos, clasificando las muestras por un porcentaje o una categoría de fiabilidad de la identificación. Actualmente están comercializados 2 equipos de espectrometría de masas MALDI-TOF, el MALDI Biotyper (Bruker Daltonics GmbH, Bremen, Alemania) y el VITEK® MS (bioMérieux, Marcy l’Etoile, Francia), si bien el primero parece haber alcanzado una mayor difusión. Aunque los protocolos de uso pueden variar ligeramente, los resultados obtenidos parecen equivalentes en ambos. Hay 2 aspectos fundamentales en el uso de MALDI-TOF, como son una correcta extracción y, por otra parte, disponer de una librería de patrones lo más amplia posible. La complejidad y la resistencia de la pared micobacteriana implican realizar una lisis intensa con métodos físico-químicos y una extracción previa de las proteínas, que no son necesarias para las bacterias convencionales. Esta preparación debe ser sumamente cuidadosa para obtener la mejor extracción posible. La librería de espectros es el segundo aspecto crucial. Exceptuando la secuenciación, no existe ningún otro método en el mercado que permita identificar más especies que MALDI-TOF. Por ejemplo, el equipo Biotyper de Bruker ha desarrollado recientemente una versión (v4) que contiene 880 patrones, de los que 450 son aislados clínicos y corresponden a 159 especies, de las aproximadamente 170 especies que componen el género. Esta versión coexiste con la anterior (v3), capaz de identificar hasta 149 especies29. El número de espectros de cada especie determinará también la precisión de la identificación. Algunas especies son muy difíciles de diferenciar debido a que sus espectros guardan mucha similitud. Tal es el caso de M. intracellulare y M. chimaera o de las 3 subespecies de M. avium (sub. avium, sub. silvaticum, sub. paratuberculosis) o las 3 de M. abscessus, o entre M. abscessus y M. chelonae, o entre M. fortuitum y M. porcinum, entre otros. Además de la librería, aspectos operativos como el tiempo de incubación de los cultivos, o la lectura manual dirigiendo los disparos del láser sobre la muestra, en lugar de hacerlo automáticamente, pueden aumentar la eficacia de la identificación. Quedan por estandarizar aspectos como unificar la extracción, concretar con mayor exactitud los criterios de interpretación de los perfiles y los puntos de corte utilizados y establecer el número de perfiles de cada especie necesario para considerar la identificación altamente fiable. En la actualidad existe en Europa un grupo de trabajo dirigido a esta estandarización.
MALDI-TOF se está afianzando como un método importante en la identificación de las micobacterias, con un espectro de especies identificadas muy superior a cualquier otra técnica que no sea la secuenciación. Los laboratorios que deseen introducirlo han de tener en cuenta que es un método complejo y que será necesario un proceso de aprendizaje y manejo que puede requerir varios meses.
Identificación por secuenciaciónLa secuenciación está considerada la técnica de referencia para la identificación de las micobacterias. El gen más utilizado es el ARNr 16S, aunque también se utilizan hsp65, rpoB y la 16S-23S internal transcriber spacer y a menudo una combinación de 2 o más de ellos. La realización técnica de la secuenciación es ampliamente conocida, consistente en una primera amplificación con los iniciadores del gen a estudiar, seguida de una segunda amplificación de cada una de las hebras de la cadena de ADN con nucleótidos marcados con moléculas fluorescentes. Este segundo producto, una vez purificado se analiza con el secuenciador, que emite un resultado en forma de secuencia escrita y de electroferograma. El patrón resultante se introduce en una base de datos que contiene multitud de secuencias a las que se compara. Para que una identificación sea aceptable el porcentaje de similitud con las secuencias identificadas de la base de datos debe ser al menos del 99%, e idealmente del 99,5%. Existen algunas bases de datos pertenecientes a compañías o instituciones, como MicroSEQ® ID Analysis, RIDOM o EzTaxon, que contienen patrones que han sido sometidos a un control de identificación. Otras bases de datos permiten el libre acceso para introducir secuencias con unos requisitos mínimos y sin demostrar la identificación, lo que no ofrece garantías de control de que las secuencias introducidas sean lo que dicen ser. Sin embargo, estas últimas contienen un número muy elevado de secuencias, algunas de ellas de más de 20 millones, por lo que en la práctica ofrecen una fiabilidad considerable. Las más conocidas son EMBL y Genbank. Numerosos laboratorios utilizan secuenciación del gen ARNr 16S y como apoyo hsp65 en la identificación de las micobacterias de crecimiento lento y rpoB en las de crecimiento rápido30. La relativa difusión de la secuenciación permite diferentes escenarios operativos, de manera que si no se dispone de un equipo de secuenciación propio, puede derivarse el producto de amplificación a laboratorios externos especializados, con costes y periodos de resultados cortos.
La secuenciación tiene también limitaciones en la interpretación. Algunas especies son difíciles de diferenciar, como son los componentes del MTC, indistinguibles entre ellos, la distinción entre M. intracellulare y M. chimaera, las subespecies de M. avium o las de M. abscessus.
Estrategia de uso de la identificaciónLos aislamientos positivos deben identificarse siempre a nivel de especie o al menos a nivel de complejo. La especie mayoritaria en nuestro entorno es MTC, aunque los aislamientos de MNT son cada vez más frecuentes. Probablemente la técnica más rápida y costo-efectiva es la tinción de Ziehl-Neelsen de los aislamientos. Confirma que se trata de Mycobacterium spp. y en los cultivos líquidos permite en muchos casos orientar si se trata de MTC por la formación de cuerdas, o de MNT. La sospecha de MTC puede confirmarse utilizando una prueba de inmunocromatografía y obtener en el mismo día la identificación. Alternativamente se puede utilizar cualquier método basado en PCR o MALDI-TOF. Teóricamente es posible tener el resultado el mismo día, aunque las condiciones de rutina probablemente lo retrasarán 1-2 días. La diferenciación de las especies de MTC generalmente no es necesaria debido a que la gran mayoría de los aislamientos corresponden a M. tuberculosis, siendo las demás muy poco frecuentes. Estaría indicada en los siguientes casos: 1) aislamiento resistente a pirazinamida (M. bovis y M. bovis BCG son resistentes a este fármaco); 2) sospecha de M. bovis, por datos epidemiológicos, o de M. bovis BCG usado en el tratamiento de neoplasia de vejiga urinaria, y 3) sospecha de M. africanum en paciente de África del Oeste.
Si las pruebas iniciales no confirman MTC o la tinción orienta claramente a MNT, pueden utilizarse las técnicas de LPA. Si se dispone de equipo MALDI-TOF, en el momento actual es una alternativa a las técnicas de LPA, aunque también puede utilizarse cuando estas no consiguen identificar los aislamientos. Ambos tipos de técnicas precisan de unas horas de dedicación manual. La secuenciación es la técnica de referencia, utilizable cuando no se consiga la identificación con las demás o cuando esta sea dudosa. En estos casos a menudo será necesario secuenciar 2 o más genes distintos. Es importante que el laboratorio emita informes parciales que ayuden en el manejo clínico. Así, en un primer momento es importante saber cuanto antes si se trata de MTC o no, por las condiciones de aislamiento epidemiológico del paciente y por tener un tratamiento distinto. Si se trata de MNT, será importante saber si es de crecimiento lento o de crecimiento rápido, ya que el esquema de tratamiento empírico variará. Del mismo modo, si la infección es significativa y la identificación inicial es M. abscessus, es importante saber si los macrólidos se podrán usar (M. abscessus sub. massiliense y algunas cepas de las otras 2 subespecies). En este caso, el uso de la prueba LPA GenoType NTM DR permitirá confirmar o descartar el gen erm que codifica una metilasa inducible de los macrólidos. Dependiendo de la especie y de las técnicas utilizadas, la identificación definitiva de MNT puede demorar entre 2-7 días, aunque una primera aproximación estará disponible en 1-2 días.
Futuro del diagnósticoEl diagnóstico de las micobacteriosis (TB e infecciones por MNT) presenta 3 tipos de dificultades esenciales: en primer lugar, la escasa sensibilidad de las pruebas en las localizaciones extrapulmonares; en segundo lugar, la lentitud de la prueba más sensible, el cultivo, que obliga a diagnósticos de presunción y tratamientos empíricos, y en tercer lugar, la dificultad de obtener muestras adecuadas en algunas localizaciones. La prueba ideal debería poder realizarse en muestras universales, como sangre u orina, ser rápida y ser sensible.
Las pruebas clásicas, microscopia y cultivo, probablemente han alcanzado su máximo y difícilmente incrementarán su rendimiento. Las pruebas de amplificación genética tienen aún espacios de mejora, tanto en el rendimiento de la amplificación como en lograr una extracción de ácidos nucleicos más eficiente. También debe explorarse la utilidad, observada en algunos estudios previos, de detectar ADN eliminado a través de la vía urinaria. La secuenciación masiva dispone de una perspectiva de futuro en el diagnóstico. Su potencialidad es enorme, sobre todo en la caracterización de las mutaciones y otros determinantes de la resistencia. Sin embargo, en la actualidad tiene una expansión limitada debido a la sensibilidad de la amplificación, aún subóptima para desarrollar su potencial. No obstante, se requieren otros enfoques más allá de detectar el microorganismo o su ADN. En los últimos años se están realizando esfuerzos en la búsqueda de biomarcadores dirigidos sobre todo a la TB. Este es un camino aún incipiente y poco desarrollado. Se pueden apuntar algunos detalles31. Por ejemplo, en relación con biomarcadores del paciente, en estudios que analizan numerosos metabolitos en sangre, se ha observado un incremento de algunos de ellos en pacientes con TB activa, en comparación con personas sanas. Otros estudios evidencian que podría haber un incremento de producción de interferón gamma en pacientes con TB latente a punto de evolucionar a TB activa. Respecto a marcadores del microorganismo, se ha indicado que el antígeno LAM detectado en orina podría mejorar su sensibilidad utilizando métodos de visualización del resultado que no sean enzimáticos. Una mención especial merece la detección de compuestos aromáticos. Analizar el aire espirado para el diagnóstico de TB es una alternativa atractiva, no invasiva y que no provoca dolor al paciente. Se sabe que determinados pacientes pueden liberar algunos olores en función de su estado. Mediante técnicas cromatográficas se han identificado un gran número de componentes orgánicos volátiles de muestras clínicas que podrían servir como marcadores para monitorizar diferentes estadios de enfermedades. Los avances en la tecnología de odor-sensing y la inteligencia artificial están investigando en este tema, y se han diseñado narices artificiales que identifican cultivos de M. tuberculosis con casi un 100% de aciertos. Después de un estudio piloto llevado a cabo en Bangladesh con resultados muy exitosos32, se procedió al desarrollo de una nariz electrónica portátil manejable y con batería basada en sensores metal-óxidos con posibilidad de producirse de forma industrial con bajo coste (Aenose®, eNose Company, Zutphen, Países Bajos). Un reciente estudio ha utilizado Anose® en pacientes con cultivo positivo frente a gente sana y ha mostrado una sensibilidad y especificidad del 91 y 93%, respectivamente, teniendo aceptación entre los participantes en el estudio y siendo de fácil manejo. Probablemente serán necesarios estudios más amplios y con más variables a incluir, como la valoración de diferentes enfermedades simultáneas33. En la misma línea, también se han presentado los resultados de sensores de gases en arrays sobre cuarzo, alcanzando unas cifras de sensibilidad y especificidad similares34.
En conclusión, el futuro en el diagnóstico probablemente esté en combinar el máximo rendimiento de las pruebas existentes, mejorar las técnicas de amplificación e invertir tiempo y financiación en la investigación de biomarcadores. La prueba ideal debería detectar simultáneamente mutaciones de resistencia a todos los fármacos y estar lo más automatizada posible, y a un coste asequible, además de poderse realizar en una muestra fácilmente obtenible.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.