En el trasplante renal pueden presentarse complicaciones quirúrgicas, infecciosas y rechazo al injerto; su detección oportuna a través de marcadores no invasivos es la clave para modificar la terapia evitando la biopsia.
ObjetivoDeterminar los perfiles de expresión de proteínas urinarias en pacientes con trasplante renal que desarrollaron complicaciones y detectar su variación al modificar la terapia.
Material y métodosSe recolectaron muestras de orina pretrasplante y de diversas fases del postrasplante; el análisis fue realizado por marcaje peptídico mediante isótopos isobáricos para la cuantificación relativa (iTRAQ®).
ResultadosSe incluyeron 22 pacientes, de los cuales 12 presentaron complicaciones en el postrasplante renal: 2 con rechazo al injerto (un hombre y una mujer) y 10 (6 hombres y 4 mujeres) en el grupo de infecciones sistémicas. A través de iTRAQ® se identificaron en el rechazo al injerto 15/345 proteínas, y en pacientes con infección, 28/113 proteínas, que cumplieron los criterios de aceptación.
ConclusionesLa albúmina fue la única proteína encontrada en ambos grupos; el resto de las proteínas fueron diferentes. Las 5 proteínas con mayor score en rechazo al injerto fueron alfa-1-microglobulina, 5’-nucleotidasa citosólica, proteína 4 de unión a retinol, proteína de membrana 4 palmitolada y serín carboxipeptidasa, mientras que en el grupo de infecciones fueron la acetil coenzima A sintetasa mitocondrial, adenosil homocisteinasa 2, proteína de dedo de cinc GLIS1, proteína putativa de la isoforma FAM157B y proteína de dedo de cinc 615. Queda por dilucidar la participación de cada una de estas en los pacientes con trasplante renal.
After renal transplant, surgical, infection complications, as well as graft rejection may occur; early detection through non-invasive markers is the key to change therapy and avoid biopsy.
ObjectiveThe aime of the study is to determine urine protein profiles in patients undergoing renal transplant with complications and detect its variation when therapy is modified.
Material and methodsUrine samples were collected from patients prior the transplant and various postoperative stages. Urinary protein profiles were obtained by peptide labeling using isobaric isotopes for relative quantification (iTRAQ®).
ResultsA total of 22 patients were included, of whom 12 developed post-transplant complication: 2 with graft rejection (one male and one female) and 10 (6 males and 4 females) in the group of post-transplant infections. Using iTRAQ® 15/345 and 28/113 proteins were identified and fulfilled the acceptance criteria, in graft rejection and post-transplant infections group, respectively.
ConclusionsAlbumin was the only protein found in both groups, the remaining proteins were different. The 5 proteins with higher scores in graft rejection were: alpha-1-microglobulin, 5’-nucleotidase cytosolic III, retinol-binding protein 4, membrane protein palmitoylated 4, and serine carboxypeptidase, while post-transplant infections were: mitochondrial acetyl-coenzyme A synthetase, putative adenosyl homocysteinase 2, zinc finger protein GLIS1, putative protein FAM157B, and zinc finger protein 615. It remains to elucidate the involvement of each of these in patients with renal transplantation.
El trasplante renal es la mejor opción disponible para el tratamiento de la enfermedad renal crónica terminal, y en comparación con la diálisis crónica, mejora la calidad de vida y disminuye la mortalidad de la mayoría de los pacientes. La escasez de donación de órganos es la mayor limitación de esta terapia y, por lo tanto, prolongar la vida útil de los injertos renales es la principal prioridad1,2.
El análisis mediante biopsia protocolizada del órgano trasplantado es el método de elección para calcular y predecir el riesgo de pérdida del injerto, aportando indicios sobre el posible mecanismo patogénico de su disfunción; así mismo, orienta la terapia inmunosupresora a seguir3. Existen diversos mecanismos etiológicos, como la toxicidad por anticalcineurínicos, el rechazo celular y humoral, y las infecciones, que generan una lesión tisular inicial y temporal que tiene como morfología final y común la presencia de fibrosis intersticial y atrofia tubular4. Por esta razón, actualmente se están generado nuevos abordajes diagnósticos que permitan detectar y seguir la evolución de la disfunción del injerto y sus diferentes subtipos histológicos para optimizar la información que aporta la histología actualmente, y cuyo objetivo final sea generar una herramienta no invasiva que sustituya a la biopsia del injerto renal5,6.
La proteómica ha sido aplicada ampliamente en la búsqueda de marcadores para el diagnóstico y pronóstico de diversas enfermedades, entre las que se incluyen: las cardiovasculares, del aparato reproductor femenino y masculino, enfermedades hepáticas y renales, y peritonitis relacionada a diálisis, entre otras7–10. Actualmente se han desarrollado otras alternativas cuantitativas con gran potencial, como el marcaje peptídico mediante isótopos isobáricos para la cuantificación relativa (iTRAQ®), que está jugando un papel determinante en la proteómica de expresión diferencial cuantitativa11.
En el presente trabajo se determinó el perfil de proteínas urinarias asociado a complicaciones del trasplante renal y se valoró su modificación con la terapia específica.
Material y métodosPoblación de estudioEste fue un estudio observacional, longitudinal, analítico y prospectivo, en el que se incluyó a pacientes que recibieron trasplante renal de donador cadavérico en el Hospital Universitario Dr. José Eleuterio González, Universidad Autónoma de Nuevo León, durante el periodo comprendido desde el 28 de enero de 2009 al 30 de mayo de 2013. Este protocolo fue aprobado por el Comité de Ética de nuestra institución y quedó registrado con el No. HI09-003.
Criterios de inclusión de pacientesPacientes mayores de 18 años que hayan acudido al Servicio de Trasplantes del Hospital Universitario Dr. José Eleuterio González y que decidieron participar en el protocolo, firmando la carta de consentimiento informado. Estos se clasificaron en 2 grupos: 1) grupo de pacientes con trasplante renal con rechazo agudo, donde se incluyeron aquellos que presentaron elevación de creatinina y confirmación del rechazo mediante biopsia, y 2) grupo de infecciones sistémicas con trasplante renal, con aquellos que presentaron infección demostrada mediante hemocultivo.
Criterios de no inclusión de pacientesLos pacientes menores de 18 años y los que no cumplieron con alguno de los criterios de inclusión.
Obtención de muestras biológicasA los pacientes que presentaron rechazo agudo se les realizó un seguimiento en las fases pretrasplante, a las 24, 48 y 72h, y a los 7, 15 y 30 días postrasplante, con tomas de muestra de sangre, una de 5ml para suero y otra de 3ml para sangre con EDTA, obtenidas por las técnicas convencionales de extracción; el suero se almacenó en alícuotas de 1ml a −20°C hasta su uso.
Para las muestras de orina se utilizó la técnica convencional de obtención de orina de micción media, mientras que en pacientes con sonda vesical permanente se realizó aspirando con aguja a través del cono de la sonda; se almacenó en 6 alícuotas de 4ml a −70°C hasta su uso. Estas muestras se recolectaron en las fases pretrasplante y cada tercer día hasta un mes postrasplante; a partir de ese momento, si el paciente no presentaba complicación, se descartaba. A partir del momento en que los pacientes presentaron alguna complicación, la recolección de muestras de orina se realizó de forma diaria durante los 4 días subsecuentes, y posteriormente fueron cada tercer día hasta los 30 días posteriores a la misma, mientras que las muestras de sangre fueron subsecuentes hasta el séptimo día, y después cada semana hasta los 30 días posteriores.
Determinación de parámetros bioquímicos y hematológicosLas pruebas bioquímicas de glucosa (mg/dl), nitrógeno de la urea (mg/dl), creatinina (mg/dl), cloro (mmol/l), sodio (mmol/l), potasio (mmol/l), calcio (mg/dl), fósforo (mg/dl) y magnesio (mg/dl) se realizaron en el equipo Vitros® DT60 II Ortho Chemistry Clinical Diagnostic (DTSC II, Johnson & Johnson Co., Rochester, EE. UU.). Para realizar la determinación de la citometría hemática, es decir, hemoglobina (g/dl) y leucocitos (K/ul) en las muestras de sangre con EDTA, se utilizó el equipo Cell Dyn 1700 (Abbott Co., IL, EE. UU.).
Análisis de orina por iTRAQ®Las muestras de orina fueron analizadas en pool de acuerdo con el grupo en que fueron catalogados los pacientes (rechazo al injerto y grupo de infecciones), tomando de cada uno de ellos 2ml, con los cuales se hicieron alícuotas de 4ml de cada grupo y de cada una de las fases de estudio: fase pretrasplante renal (F1), fase postrasplante renal previo a complicación (F2), fase postrasplante renal complicación en curso (F3) y fase postrasplante renal complicación postratamiento (F4) para su posterior concentración, precipitación, marcaje y análisis.
- a.
Concentración de muestras de orina: se utilizó una alícuota de 4ml de orina y se colocó en un filtro Ultracel centricon 10kDa, Millipore; se centrifugó a 9,500rpm a 20°C hasta alcanzar un volumen de 200μl. Se añadió 1μl de coctel de inhibidor de proteasas Amresco y se homogenizó la muestra. Para la precipitación de las proteínas se agregaron 2.4ml de acetona fría a −20°C y se dejaron precipitar a −20°C durante 12 a 16h. Se centrifugaron las muestras a 3,500rpm durante 15min. Se eliminó el sobrenadante y se agregaron 200μl de agua Milli-Q®.
- b.
Cuantificación de proteínas: se preparó una curva de calibración con estándares de 10, 20, 30, 40 y 50μg de albúmina bovina. Se utilizó un kit de cuantificación de proteínas Bio-Rad basado en la determinación de Bradford. Para la reducción, alquilación, digestión, desalado y liofilización de proteínas, los puentes disulfuro de estas se redujeron con ditiotreitol 10mM durante 30min a 56°C. Posteriormente, las muestras se incubaron con yodoacetamida 50mM durante 30min a temperatura ambiente bajo protección de la luz para permitir la alquilación de las cisteínas. Finalmente, se adicionó tripsina (Promega, Fitchburg, WI, EE. UU.) en una proporción 1:50 (enzima:substrato). La digestión enzimática se incubó durante 18h a 37°C. Finalmente las muestras se desalaron utilizando cartuchos Sep-Pack® C18 Vac (Waters) y se liofilizaron utilizando un Savant SpeedVac® (Thermo Fisher, San José, CA, EE. UU.).
- c.
Marcaje con el kit iTRAQ®: los viales de reactivos con los marcadores isobáricos 114, 115, 116 y 117 se dejaron a temperatura ambiente, se resuspendió el reactivo, se agregaron 70μl de etanol a cada reactivo del kit y se agitó durante un minuto. Se agregó un marcador isobárico a cada fase de estudio, la fase F1 se marcó con el reactivo 114, la F2, con el 115, la fase F3, con el marcador 116, y la F4, con el marcador 117. Las muestras y los reactivos se mezclaron y se incubaron una hora a temperatura ambiente. Finalmente se mezclaron todas las muestras. El sistema de cromatografía de líquidos-espectrometría de masas estuvo constituido por un cromatógrafo de líquidos de micro-flujo Accela (Thermo Ficher Co., San José, CA, EE. UU.) con spliter (1/20) y un espectrómetro de masas LTQ-Orbitrap Velos™ (Thermo-Ficher Co., San José, CA, EE. UU.) con sistema de ionización tipo nano-electrospray. La calibración del espectrómetro fue realizada con una solución (Calmix®) de 10 moléculas calibrantes, que permitió determinaciones con exactitudes mejores que 5 partes/millón. En la cromatografía de líquidos se utilizó un sistema gradiente de 10-100% de solvente B (acetonitrilo/ácido acético al 0.1%) por 120min sobre una columna capilar PicoFrit® ProteoPep™ II C18 75μm ID×50mm (New Objective Inc., Woburn, MA, EE. UU.). El flujo del sistema de cromatografía de líquidos fue de 400nanolitros/min. Para la fragmentación de los péptidos se utilizaron los métodos de disociación inducida por colisión y disociación por colisión de alta energía. Todos los espectros fueron adquiridos en modo de detección positivo. Los datos espectrométricos fueron analizados contra la base de datos de humano a través del programa Proteome Discoverer™ 1.3, como plataforma SEQUEST. Se colocaron los parámetros restrictivos, como tolerancia de error para los iones precursores y los iones hijos; y las modificaciones como: carbamidometilcisteína, marcaje por iTRAQ® en el extremo terminal y en lisinas (constantes), y oxidación en metioninas (variables).
Los resultados bioquímicos y hematológicos fueron promediados±desviación estándar, y fueron analizados con las pruebas de ANOVA de un factor. Para ello se utilizó el programa estadístico SPSS® versión 16.0. Se consideró estadísticamente significativo si el valor de p<0.05.
ResultadosEn el periodo de estudio 48 pacientes recibieron un trasplante renal de donación cadavérica, de los cuales 22 cumplieron los criterios de inclusión y aceptaron participar en el protocolo previa firma del consentimiento informado.
Se incluyeron 10 mujeres (45.5%) y 12 hombres (54.5%), con una edad promedio de 45±15 años. De los 22 pacientes incluidos, 12 (54.5%) presentaron complicaciones en la etapa postrasplante renal con un promedio de edad de 53±17 años; 10 (45.5%) de ellos del tipo infeccioso, con una edad promedio de 43±17 años, y 2 (9%) con rechazo agudo y una edad de 63 años en ambos casos.
La etiología de la enfermedad renal de los pacientes con rechazo agudo fue nefropatía diabética en ambos casos (100%), mientras que en el grupo con infección sistémica las etiologías fueron: 2 (20%) con glomerulonefritis membranosa, 2 (20%) con glomeruloesclerosis, 2 (20%) con nefropatía diabética, uno (10%) con nefropatía lúpica severa y 2 (20%) de etiología desconocida.
De los pacientes trasplantados con infecciones (n=10), 6 presentaron alguna complicación quirúrgica aparte de la infección: un caso por Staphylococcus aureus con pseudoaneurisma de la arteria; uno con Enterococcus faecalis e infección en herida quirúrgica; uno con infección por Staphylococcus sp. y fístula arteriovenosa; uno con Enterococcus faecalis y fibrosis del uréter; uno con infección por Pseudomonas aeruginosa con fístula urinaria, y otro caso con Escherichia coli con fístula urinaria; los 4 restantes evolucionaron con ausencia de complicaciones y se identificaron los siguientes agentes etiológicos, respectivamente: Escherichia coli, Clostridium difficile, Enterococcus faecalis y Staphylococcus aureus.
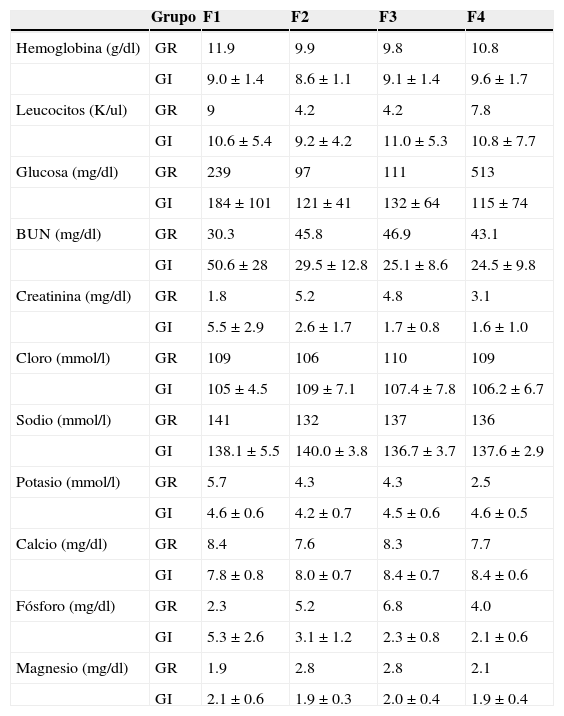
Los resultados de los parámetros bioquímicos y hematológicos de cada grupo de complicaciones en las diferentes fases del estudio se muestran en la tabla 1. En el caso del grupo de rechazo de injerto solo se muestran los promedios de los valores, ya que al tratarse solo de 2 casos no se pudo obtener la desviación estándar.
Parámetros bioquímicos y hematológicos de los grupos en las 4 fases del estudio
| Grupo | F1 | F2 | F3 | F4 | |
|---|---|---|---|---|---|
| Hemoglobina (g/dl) | GR | 11.9 | 9.9 | 9.8 | 10.8 |
| GI | 9.0±1.4 | 8.6±1.1 | 9.1±1.4 | 9.6±1.7 | |
| Leucocitos (K/ul) | GR | 9 | 4.2 | 4.2 | 7.8 |
| GI | 10.6±5.4 | 9.2±4.2 | 11.0±5.3 | 10.8±7.7 | |
| Glucosa (mg/dl) | GR | 239 | 97 | 111 | 513 |
| GI | 184±101 | 121±41 | 132±64 | 115±74 | |
| BUN (mg/dl) | GR | 30.3 | 45.8 | 46.9 | 43.1 |
| GI | 50.6±28 | 29.5±12.8 | 25.1±8.6 | 24.5±9.8 | |
| Creatinina (mg/dl) | GR | 1.8 | 5.2 | 4.8 | 3.1 |
| GI | 5.5±2.9 | 2.6±1.7 | 1.7±0.8 | 1.6±1.0 | |
| Cloro (mmol/l) | GR | 109 | 106 | 110 | 109 |
| GI | 105±4.5 | 109±7.1 | 107.4±7.8 | 106.2±6.7 | |
| Sodio (mmol/l) | GR | 141 | 132 | 137 | 136 |
| GI | 138.1±5.5 | 140.0±3.8 | 136.7±3.7 | 137.6±2.9 | |
| Potasio (mmol/l) | GR | 5.7 | 4.3 | 4.3 | 2.5 |
| GI | 4.6±0.6 | 4.2±0.7 | 4.5±0.6 | 4.6±0.5 | |
| Calcio (mg/dl) | GR | 8.4 | 7.6 | 8.3 | 7.7 |
| GI | 7.8±0.8 | 8.0±0.7 | 8.4±0.7 | 8.4±0.6 | |
| Fósforo (mg/dl) | GR | 2.3 | 5.2 | 6.8 | 4.0 |
| GI | 5.3±2.6 | 3.1±1.2 | 2.3±0.8 | 2.1±0.6 | |
| Magnesio (mg/dl) | GR | 1.9 | 2.8 | 2.8 | 2.1 |
| GI | 2.1±0.6 | 1.9±0.3 | 2.0±0.4 | 1.9±0.4 |
F1: fase pretrasplante renal; F2: fase postrasplante renal previo a complicación; F3: fase postrasplante renal complicación en curso; F4: fase postrasplante renal complicación tratada; GI: grupo de pacientes con infección; GR: grupo de pacientes con rechazo del injerto.
Se realizó la comparación de los parámetros bioquímicos y hematológicos entre las fases para el grupo de infecciones. Los valores estadísticamente significativos fueron glucosa al comparar la F1 (184±101mg/dl) vs. F2 (121±41mg/dl), p=0.009; nitrógeno de la urea entre F1 (50.6±28mg/dl) vs. F3 (25.1±8.6mg/dl), p=0.018, y F1 (50.6±28mg/dl) vs. F4 (24.5±9.8mg/dl), p=0.049; creatinina en F1 (5.5±2.9mg/dl) vs. F3 (1.7±0.8mg/dl), p=0.004, F1 (5.5±2.9mg/dl) vs. F4 (1.6±1.0mg/dl), p=0.013, y F2 (2.6±1.7mg/dl) vs. F3 (1.7±0.8mg/dl), p=0.020; y magnesio en F1 (2.1±0.6mg/dl) vs. F2 (1.9±0.3mg/dl), p=0.050.
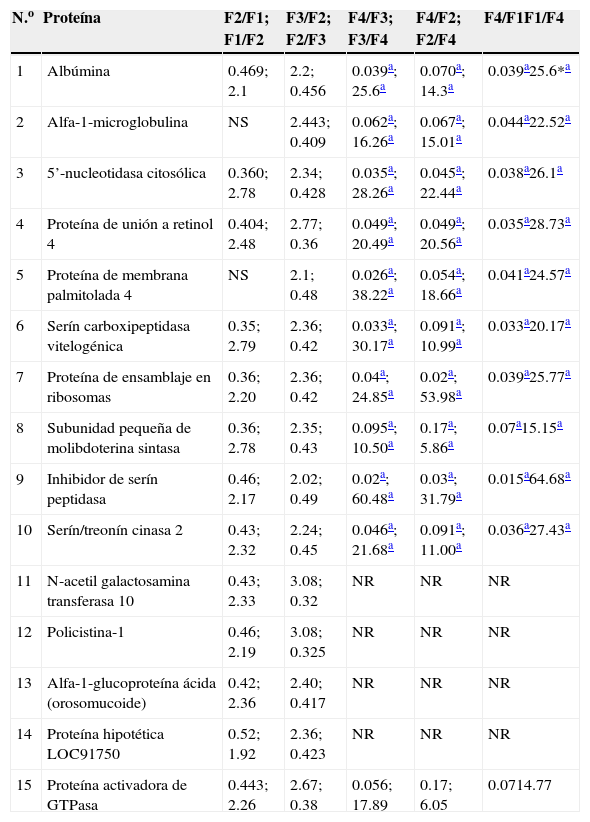
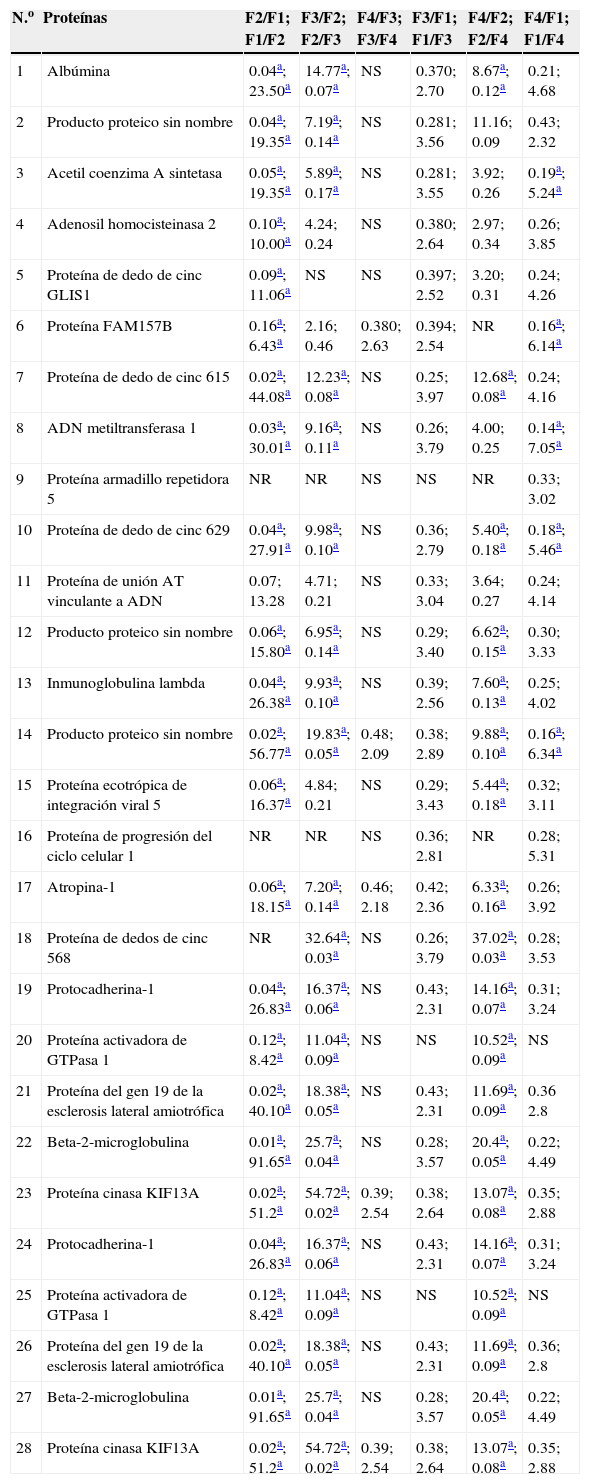
Con la aplicación de las técnicas de iTRAQ® mediante cromatografía de líquidos-espectrometría de masas, en el grupo con rechazo al injerto se encontraron 345 proteínas, de las cuales 15 cumplieron con los criterios de aceptación de la técnica (score>30, 2 o más péptidos identificados con el 95% de confianza) y todos correspondieron a péptidos humanos (tabla 2), mientras que en el grupo de pacientes con infección se encontraron 113 proteínas, de las cuales 28 cumplieron con los criterios de aceptación de la técnica (tabla 3). Se consideraron significativas las diferencias con 2 veces la abundancia de las proteínas entre las fases de estudio.
Proteínas en el grupo de pacientes con rechazo del injerto que cumplieron los criterios de aceptación
| N.o | Proteína | F2/F1; F1/F2 | F3/F2; F2/F3 | F4/F3; F3/F4 | F4/F2; F2/F4 | F4/F1F1/F4 |
|---|---|---|---|---|---|---|
| 1 | Albúmina | 0.469; 2.1 | 2.2; 0.456 | 0.039a; 25.6a | 0.070a; 14.3a | 0.039a25.6*a |
| 2 | Alfa-1-microglobulina | NS | 2.443; 0.409 | 0.062a; 16.26a | 0.067a; 15.01a | 0.044a22.52a |
| 3 | 5’-nucleotidasa citosólica | 0.360; 2.78 | 2.34; 0.428 | 0.035a; 28.26a | 0.045a; 22.44a | 0.038a26.1a |
| 4 | Proteína de unión a retinol 4 | 0.404; 2.48 | 2.77; 0.36 | 0.049a; 20.49a | 0.049a; 20.56a | 0.035a28.73a |
| 5 | Proteína de membrana palmitolada 4 | NS | 2.1; 0.48 | 0.026a; 38.22a | 0.054a; 18.66a | 0.041a24.57a |
| 6 | Serín carboxipeptidasa vitelogénica | 0.35; 2.79 | 2.36; 0.42 | 0.033a; 30.17a | 0.091a; 10.99a | 0.033a20.17a |
| 7 | Proteína de ensamblaje en ribosomas | 0.36; 2.20 | 2.36; 0.42 | 0.04a; 24.85a | 0.02a; 53.98a | 0.039a25.77a |
| 8 | Subunidad pequeña de molibdoterina sintasa | 0.36; 2.78 | 2.35; 0.43 | 0.095a; 10.50a | 0.17a; 5.86a | 0.07a15.15a |
| 9 | Inhibidor de serín peptidasa | 0.46; 2.17 | 2.02; 0.49 | 0.02a; 60.48a | 0.03a; 31.79a | 0.015a64.68a |
| 10 | Serín/treonín cinasa 2 | 0.43; 2.32 | 2.24; 0.45 | 0.046a; 21.68a | 0.091a; 11.00a | 0.036a27.43a |
| 11 | N-acetil galactosamina transferasa 10 | 0.43; 2.33 | 3.08; 0.32 | NR | NR | NR |
| 12 | Policistina-1 | 0.46; 2.19 | 3.08; 0.325 | NR | NR | NR |
| 13 | Alfa-1-glucoproteína ácida (orosomucoide) | 0.42; 2.36 | 2.40; 0.417 | NR | NR | NR |
| 14 | Proteína hipotética LOC91750 | 0.52; 1.92 | 2.36; 0.423 | NR | NR | NR |
| 15 | Proteína activadora de GTPasa | 0.443; 2.26 | 2.67; 0.38 | 0.056; 17.89 | 0.17; 6.05 | 0.0714.77 |
F1: fase pretrasplante renal; F2: fase postrasplante renal previo a complicación; F3: fase postrasplante renal complicación en curso; F4: fase postrasplante renal complicación tratada; GI: grupo de pacientes con infección; GR: grupo de pacientes con rechazo del injerto; NR: no realizado; NS: no significativo.
Proteínas en el grupo de pacientes con infecciones que cumplieron los criterios de aceptación
| N.o | Proteínas | F2/F1; F1/F2 | F3/F2; F2/F3 | F4/F3; F3/F4 | F3/F1; F1/F3 | F4/F2; F2/F4 | F4/F1; F1/F4 |
|---|---|---|---|---|---|---|---|
| 1 | Albúmina | 0.04a; 23.50a | 14.77a; 0.07a | NS | 0.370; 2.70 | 8.67a; 0.12a | 0.21; 4.68 |
| 2 | Producto proteico sin nombre | 0.04a; 19.35a | 7.19a; 0.14a | NS | 0.281; 3.56 | 11.16; 0.09 | 0.43; 2.32 |
| 3 | Acetil coenzima A sintetasa | 0.05a; 19.35a | 5.89a; 0.17a | NS | 0.281; 3.55 | 3.92; 0.26 | 0.19a; 5.24a |
| 4 | Adenosil homocisteinasa 2 | 0.10a; 10.00a | 4.24; 0.24 | NS | 0.380; 2.64 | 2.97; 0.34 | 0.26; 3.85 |
| 5 | Proteína de dedo de cinc GLIS1 | 0.09a; 11.06a | NS | NS | 0.397; 2.52 | 3.20; 0.31 | 0.24; 4.26 |
| 6 | Proteína FAM157B | 0.16a; 6.43a | 2.16; 0.46 | 0.380; 2.63 | 0.394; 2.54 | NR | 0.16a; 6.14a |
| 7 | Proteína de dedo de cinc 615 | 0.02a; 44.08a | 12.23a; 0.08a | NS | 0.25; 3.97 | 12.68a; 0.08a | 0.24; 4.16 |
| 8 | ADN metiltransferasa 1 | 0.03a; 30.01a | 9.16a; 0.11a | NS | 0.26; 3.79 | 4.00; 0.25 | 0.14a; 7.05a |
| 9 | Proteína armadillo repetidora 5 | NR | NR | NS | NS | NR | 0.33; 3.02 |
| 10 | Proteína de dedo de cinc 629 | 0.04a; 27.91a | 9.98a; 0.10a | NS | 0.36; 2.79 | 5.40a; 0.18a | 0.18a; 5.46a |
| 11 | Proteína de unión AT vinculante a ADN | 0.07; 13.28 | 4.71; 0.21 | NS | 0.33; 3.04 | 3.64; 0.27 | 0.24; 4.14 |
| 12 | Producto proteico sin nombre | 0.06a; 15.80a | 6.95a; 0.14a | NS | 0.29; 3.40 | 6.62a; 0.15a | 0.30; 3.33 |
| 13 | Inmunoglobulina lambda | 0.04a; 26.38a | 9.93a; 0.10a | NS | 0.39; 2.56 | 7.60a; 0.13a | 0.25; 4.02 |
| 14 | Producto proteico sin nombre | 0.02a; 56.77a | 19.83a; 0.05a | 0.48; 2.09 | 0.38; 2.89 | 9.88a; 0.10a | 0.16a; 6.34a |
| 15 | Proteína ecotrópica de integración viral 5 | 0.06a; 16.37a | 4.84; 0.21 | NS | 0.29; 3.43 | 5.44a; 0.18a | 0.32; 3.11 |
| 16 | Proteína de progresión del ciclo celular 1 | NR | NR | NS | 0.36; 2.81 | NR | 0.28; 5.31 |
| 17 | Atropina-1 | 0.06a; 18.15a | 7.20a; 0.14a | 0.46; 2.18 | 0.42; 2.36 | 6.33a; 0.16a | 0.26; 3.92 |
| 18 | Proteína de dedos de cinc 568 | NR | 32.64a; 0.03a | NS | 0.26; 3.79 | 37.02a; 0.03a | 0.28; 3.53 |
| 19 | Protocadherina-1 | 0.04a; 26.83a | 16.37a; 0.06a | NS | 0.43; 2.31 | 14.16a; 0.07a | 0.31; 3.24 |
| 20 | Proteína activadora de GTPasa 1 | 0.12a; 8.42a | 11.04a; 0.09a | NS | NS | 10.52a; 0.09a | NS |
| 21 | Proteína del gen 19 de la esclerosis lateral amiotrófica | 0.02a; 40.10a | 18.38a; 0.05a | NS | 0.43; 2.31 | 11.69a; 0.09a | 0.36 2.8 |
| 22 | Beta-2-microglobulina | 0.01a; 91.65a | 25.7a; 0.04a | NS | 0.28; 3.57 | 20.4a; 0.05a | 0.22; 4.49 |
| 23 | Proteína cinasa KIF13A | 0.02a; 51.2a | 54.72a; 0.02a | 0.39; 2.54 | 0.38; 2.64 | 13.07a; 0.08a | 0.35; 2.88 |
| 24 | Protocadherina-1 | 0.04a; 26.83a | 16.37a; 0.06a | NS | 0.43; 2.31 | 14.16a; 0.07a | 0.31; 3.24 |
| 25 | Proteína activadora de GTPasa 1 | 0.12a; 8.42a | 11.04a; 0.09a | NS | NS | 10.52a; 0.09a | NS |
| 26 | Proteína del gen 19 de la esclerosis lateral amiotrófica | 0.02a; 40.10a | 18.38a; 0.05a | NS | 0.43; 2.31 | 11.69a; 0.09a | 0.36; 2.8 |
| 27 | Beta-2-microglobulina | 0.01a; 91.65a | 25.7a; 0.04a | NS | 0.28; 3.57 | 20.4a; 0.05a | 0.22; 4.49 |
| 28 | Proteína cinasa KIF13A | 0.02a; 51.2a | 54.72a; 0.02a | 0.39; 2.54 | 0.38; 2.64 | 13.07a; 0.08a | 0.35; 2.88 |
F1: fase pretrasplante renal; F2: fase postrasplante renal previo a complicación; F3: fase postrasplante renal complicación en curso; F4: fase postrasplante renal complicación tratada; GI: grupo de pacientes con infección; GR: grupo de pacientes con rechazo del injerto; NR: no realizado; NS: no significativo.
De las 15 proteínas seleccionadas para el grupo que presentó rechazo al injerto, se encontró que el inhibidor de serín peptidasa, la proteína de membrana palmitolada 4, la serín carboxipeptidasa vitelogénica, la 5’-nucleotidasa citosólica y la subunidad pequeña de molibdoterina sintasa fueron las proteínas que presentaron los mayores cocientes entre las fases F3 y F4, mientras que las proteínas con mayor valor de score fueron la alfa-1-microglobulina, la 5’-nucleotidasa citosólica, la proteína de unión a retinol 4, la proteína de membrana palmitolada 4 y la serín carboxipeptidasa vitelogénica para este mismo grupo.
En el grupo de pacientes con infecciones de las 28 proteínas, las que presentaron los mayores cocientes entre las fases F2 y F3 fueron la proteína de procesamiento de ARN 12, el glipicano 4, la proteína de dedo de cinc 568, la proteína cinasa KIF13A y la protocadherina-1; y las de mayor score fueron la acetil coenzima A sintetasa mitocondrial, la adenosil homocisteinasa 2, la proteína FAM157B, la proteína de dedo de cinc 615 y la ADN metil transferasa 1.
DiscusiónEl trasplante renal es la mejor opción disponible para el tratamiento de la enfermedad renal crónica terminal, y en comparación con la diálisis crónica, mejora la calidad de vida y disminuye la mortalidad de la mayoría de los pacientes. La escasez de donación de órganos es la mayor limitación de esta terapia y, por lo tanto, prolongar la vida útil de los injertos renales es la principal prioridad1,2.
Actualmente se están generando nuevos abordajes diagnósticos no invasivos que permitan detectar y seguir la evolución de la disfunción del injerto9–11; la proteómica ha sido aplicada ampliamente en la búsqueda de marcadores para el diagnóstico y pronóstico de diversas enfermedades10,12–14. Recientemente se han desarrollado otras alternativas cuantitativas con gran potencial, como el iTRAQ®, que está jugando un papel determinante en la proteómica de expresión diferencial cuantitativa. Se prevé que la proteómica desempeñará un rol importante en el área de la nefrología a corto plazo y que este progreso exigirá un diálogo interactivo y la colaboración entre especialistas clínicos y analíticos15,16.
En el presente trabajo se describe la presencia de una serie de proteínas asociadas a diversas complicaciones postrasplante renal, como el rechazo del injerto e infecciones, y la modificación de estas proteínas con la terapia específica para cada una de ellas. Este estudio es el primero en nuestro país que describe qué proteínas se encuentran alteradas en los pacientes con trasplante renal que desarrollan rechazo del injerto y en aquellos que presentan complicaciones infecciosas.
Mediante el marcaje con la técnica de iTRAQ® y el análisis final por cromatografía de líquidos-espectrometría de masas se encontró en el grupo de rechazo del injerto un total de 345 proteínas, de las cuales solamente 15 cumplieron con los criterios de aceptación de la técnica descritos anteriormente (tabla 2), mientras que en el grupo de pacientes con infecciones, 28 de las 113 proteínas identificadas cumplieron con los criterios de aceptación de la técnica (tabla 3).
De las 15 proteínas encontradas en el grupo con rechazo al injerto, las 5 que presentaron los mayores cocientes entre las fases F3 vs. F4 fueron: el inhibidor de serín peptidasa, que se activa en respuesta a estrés celular y durante el daño por isquemia-reperfusión renal17,18, así mismo se ha reportado que los niveles del inhibidor se correlacionan con la gravedad del rechazo, siendo capaz de predecir la función posterior del injerto renal19,20; la proteína de membrana palmitolada 4, una proteína integral de membrana con actividad de guanilato cinasa, lo que le permite interactuar con el citoesqueleto y regular la proliferación celular y la transducción de señales21; la serín carboxipeptidasa vitelogénica, una proteasa de función desconocida que se caracterizó por primera vez en macrófagos humanos, se cree que se encuentra limitada en gran medida a la estirpe monocítica, aunque estudios recientes evidencian que también puede ser expresada por células fuera del sistema inmune22; la 5’-nucleotidasa citosólica, que está implicada en diversas funciones, como la comunicación célula-célula, la reparación de ácido nucleico, la síntesis de nucleótidos, la transducción de señales y el transporte de membrana23, y por último, la subunidad pequeña de molibdoterina sintasa, que en los seres humanos está implicada en la biosíntesis de cofactor de molibdeno, cuya deficiencia genética desencadena una enfermedad autosómica recesiva y generalmente fatal con síntomas neurológicos graves24. Los mayores cocientes entre las fases F3 vs. F4 de estas 5 proteínas indican su utilidad como candidatos a biomarcadores del mal funcionamiento del injerto renal. Además, se observó que en F4, con la modificación de la terapia inmunosupresora, se reduce de manera significativa la expresión de estas 5 proteínas, evidenciando su utilidad como potenciales biomarcadores de buen funcionamiento del injerto renal en respuesta a la terapia inmunosupresora.
Por otro lado, para este mismo grupo de pacientes, las 5 proteínas con mayor valor de score fueron: alfa-1-microglobulina, que es una lipocalina con propiedades inmunosupresivas25, la cual elimina radicales libres26, y es una de las proteínas más estudiadas como potencial biomarcador no invasivo para la detección temprana de anormalidades renales, además de su utilidad en la diferenciación de enfermedades nefrológicas y urológicas27; la 5 nucleotidasa citosólica, de la cual se ha reportado que su inhibición farmacológica aumenta las concentraciones de AMP y mejora la función renal en ratas en modelos experimentales de isquemia y reperfusión28; la proteína 4 de unión a retinol, de la que se ha descrito su elevación en pacientes con daño tubular intersticial29 y es secretada por los adipocitos, que se ha reportado aumentada en casos de resistencia a la insulina30; la proteína de membrana palmitolada 4, que se ha visto sobreexpresada en tejido renal sometido a procesos de isquemia-reperfusión31, y la serín carboxipeptidasa vitelogénica, que se expresa especialmente en corazón y riñones22. Al igual que en diversas publicaciones asociadas a la función renal, en este estudio se pudo constatar que estas 5 proteínas están sobreexpresadas durante el episodio de rechazo agudo del injerto y que su expresión es significativamente baja cuando los pacientes no presentan rechazo al injerto renal o responden a la modificación hecha a la terapia inmunosupresora.
En el grupo de pacientes con infecciones de las 28 proteínas, las que presentaron mayor cociente entre las fases F2 vs. F3 fueron: la proteína de procesamiento de ARN 12; el glipicano 4, de la familia de proteoglicanos de sulfato de heparán de la superficie celular que se expresa de una manera regulada por el desarrollo de un tejido específico32; la proteína de dedo de cinc 568, la cual se ha descrito que tiene un efecto protector en diversas condiciones de estrés, como shock térmico y estrés osmótico33; la proteína cinasa KIF13A, que son proteínas de la superfamilia de la cinesinas, las cuales son protagonistas en el sistema de transporte intracelular, que es esencial para la función celular y la morfología34; y la proteína protocadherina-1, familia de proteínas que se ha propuesto que juegan un papel en el desarrollo cerebral, la formación de hendidura glomerular, y también pueden actuar como supresores de tumores, aunque sus mecanismos de acción no se han dilucidado35.
Para este mismo grupo con infecciones, las proteínas de mayor score fueron: la isoforma 3 de la acetil coenzima A sintetasa mitocondrial, de la cual se ha descrito su importancia para mantener la temperatura corporal normal durante el ayuno y la homeostasis energética36; la adenosil homocisteinasa 2, cuya expresión aumenta notablemente durante la activación de células sanguíneas y de piel37; la proteína FAM157B, cuya función no ha sido reportada; la proteína de dedo de cinc 615, que participa en la regulación de la transcripción38, y la proteína ADN metil transferasa 1, que participa en la regulación de la expresión génica de acuerdo con un patrón de metilación39.
Por otro lado, la proteinuria masiva ha sido reconocida como un factor de riesgo para la pérdida del injerto a largo plazo40. Un estudio reciente indicó que la proteinuria de bajo grado fue un potente predictor, independiente de la pérdida del injerto a largo plazo41. En el presente estudio la albúmina se expresó en las 2 complicaciones evaluadas, por lo que no fue útil como un biomarcador específico de alguna de las complicaciones estudiadas, pero sí es un indicador de daño renal, ya que es de las proteínas mayormente expresadas previamente al trasplante; luego, al realizarse este, su expresión disminuye considerablemente, pero al presentarse cualquier tipo de complicación, se vuelve a sobreexpresar.
Como se ha descrito en el presente trabajo, diversas proteínas son candidatas a biomarcadores de buen funcionamiento del injerto renal, ya que su presencia en la orina puede ser utilizada para discriminar los pacientes que tienen buena función del injerto de aquellos que no; además, cada una de las complicaciones evaluadas presentó una serie de proteínas diferentes, a excepción de la albúmina. Este trabajo enfatiza la utilidad de la proteómica urinaria para identificar biomarcadores y para el futuro diseño de una herramienta diagnóstica no invasiva para detectar más oportunamente la disfunción del injerto renal trasplantado.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.