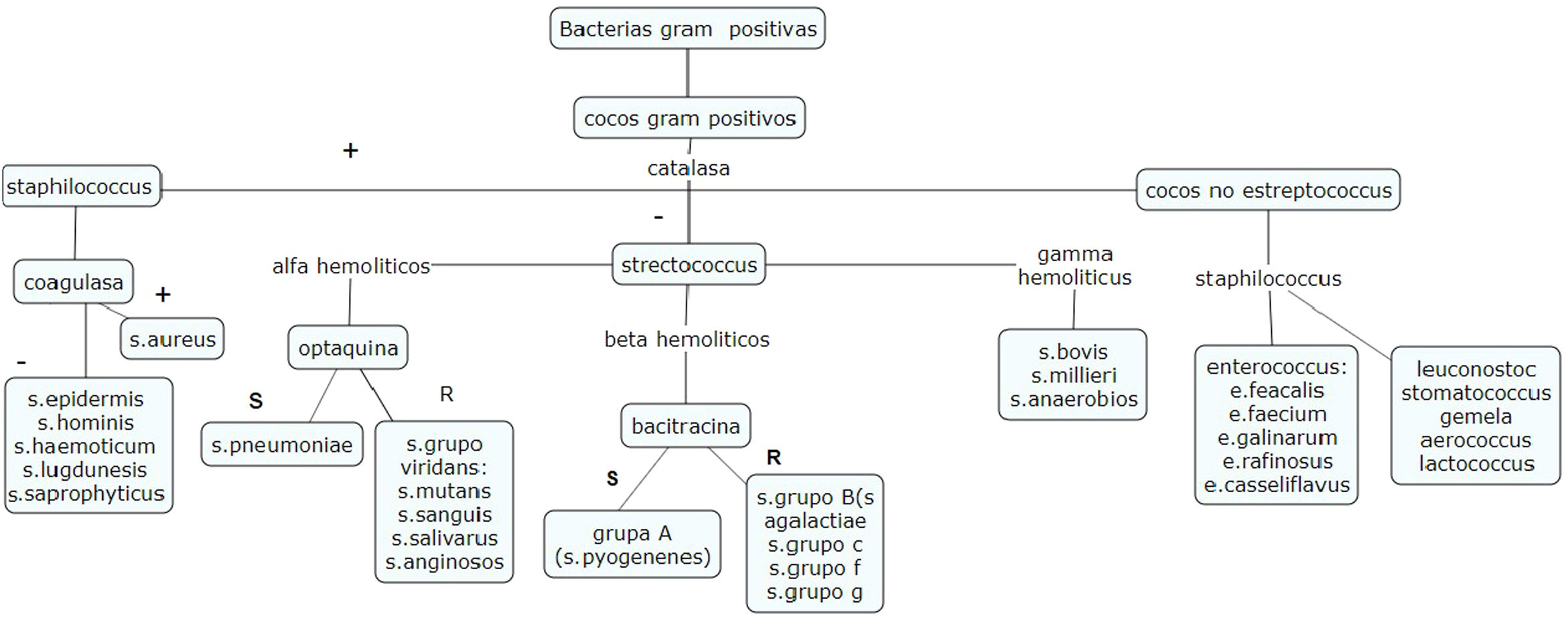
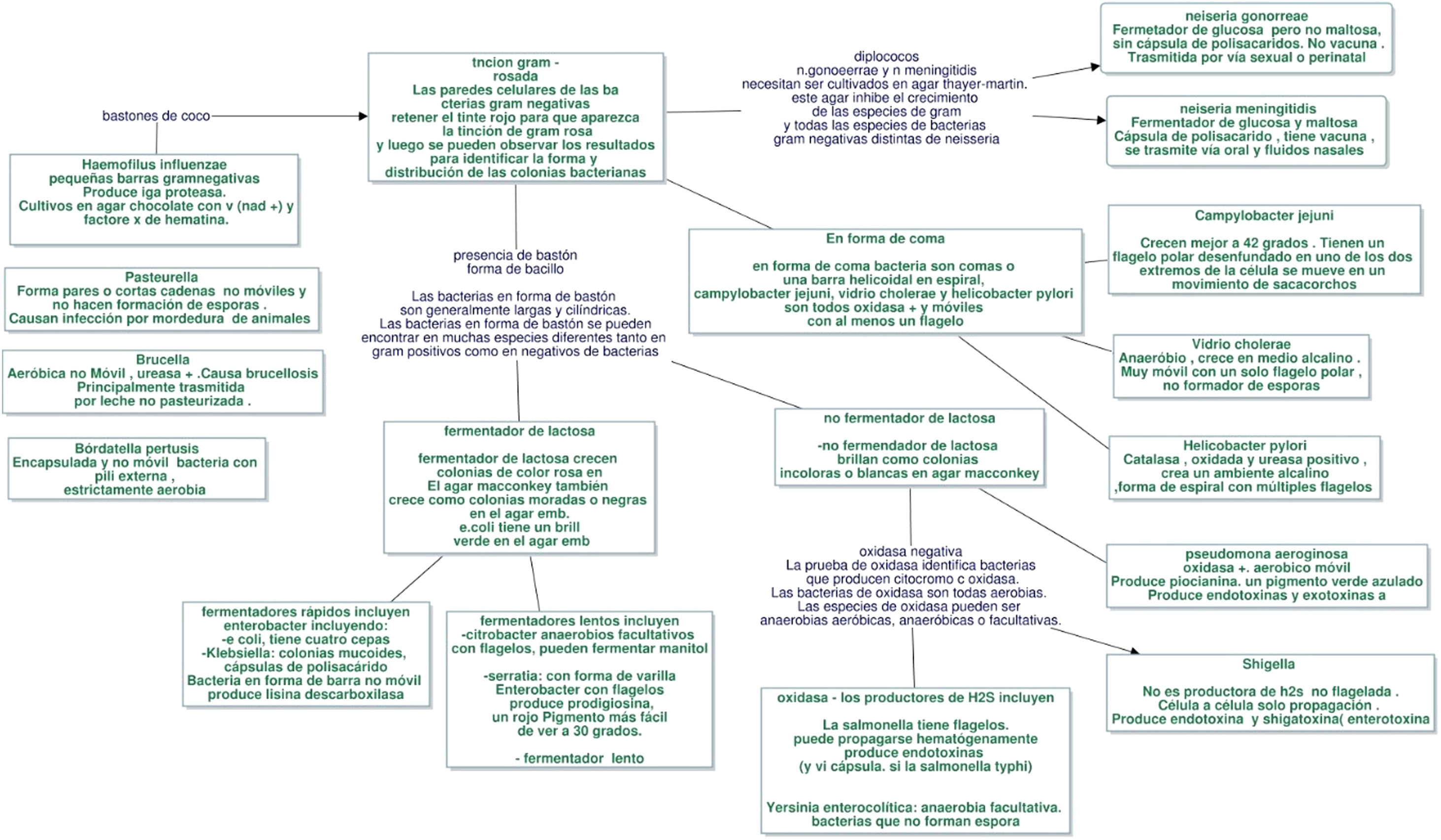
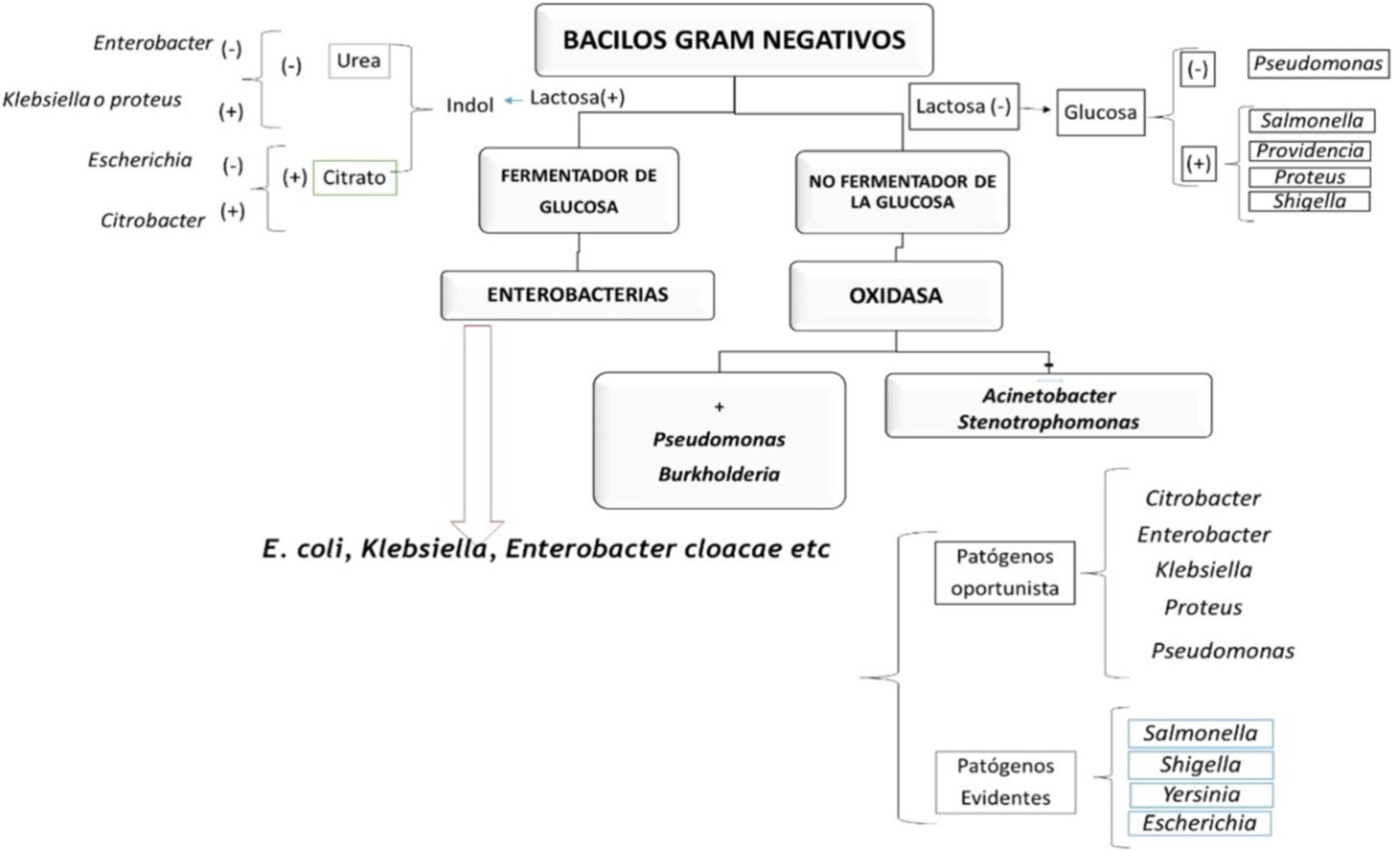
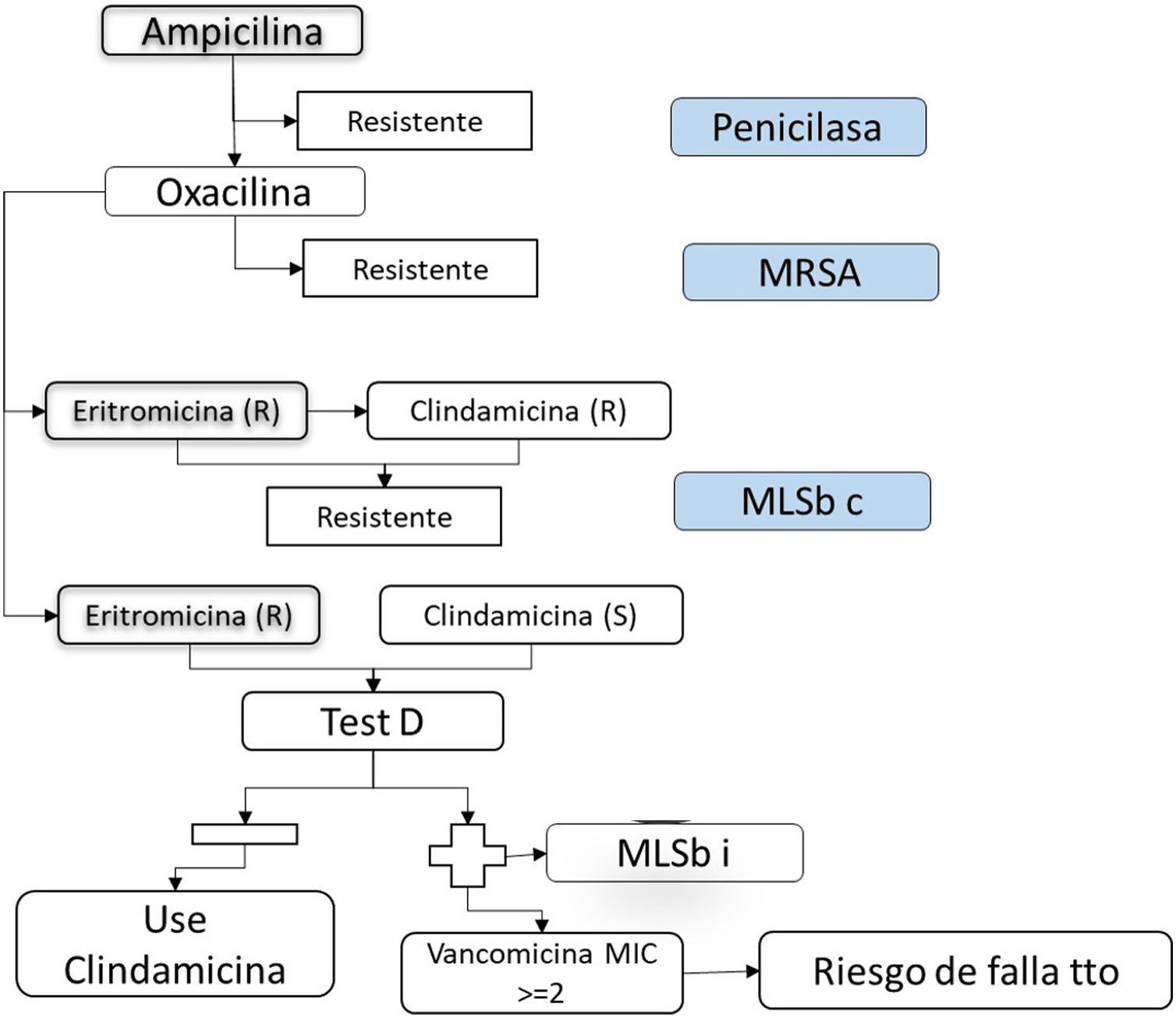
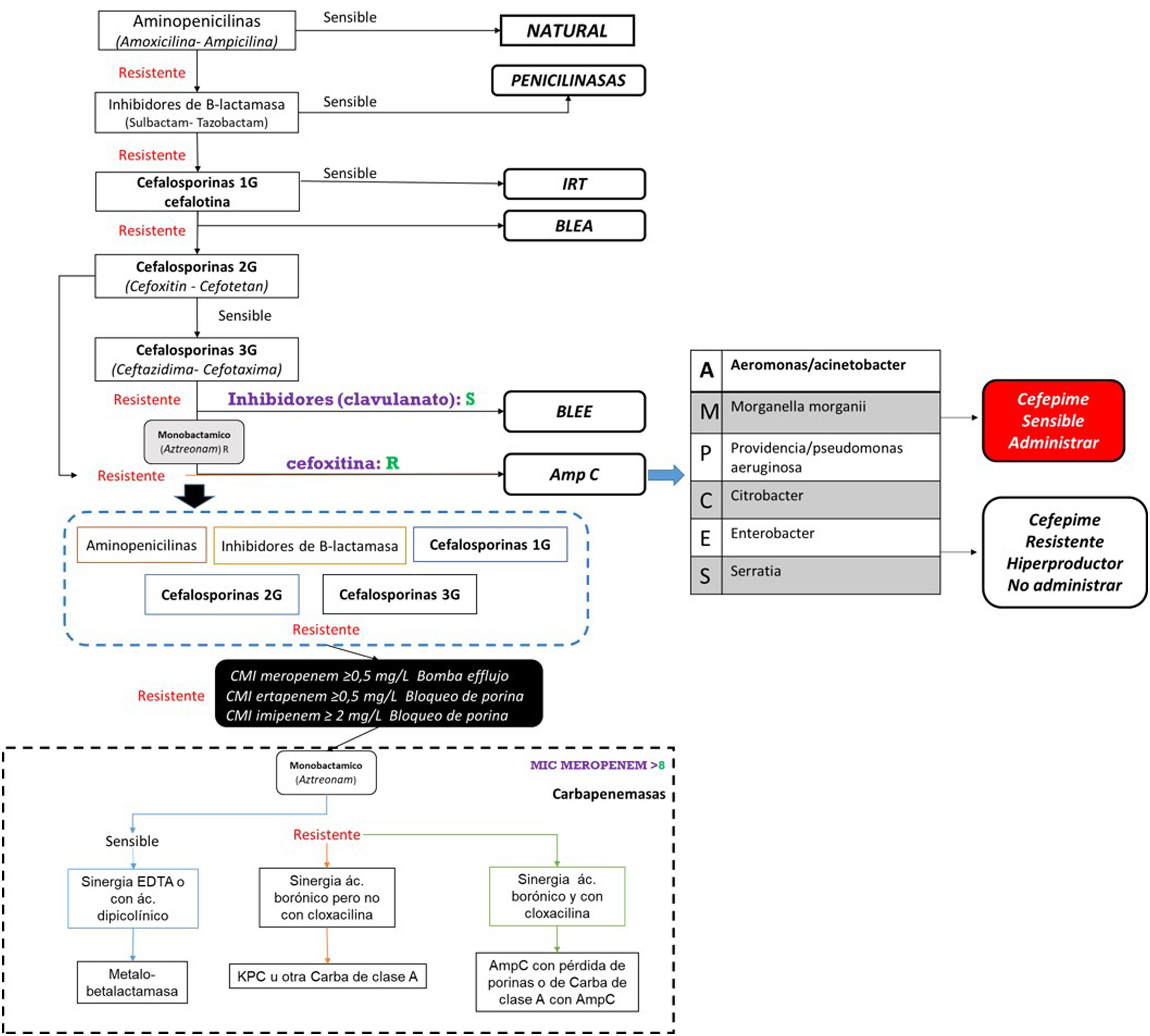
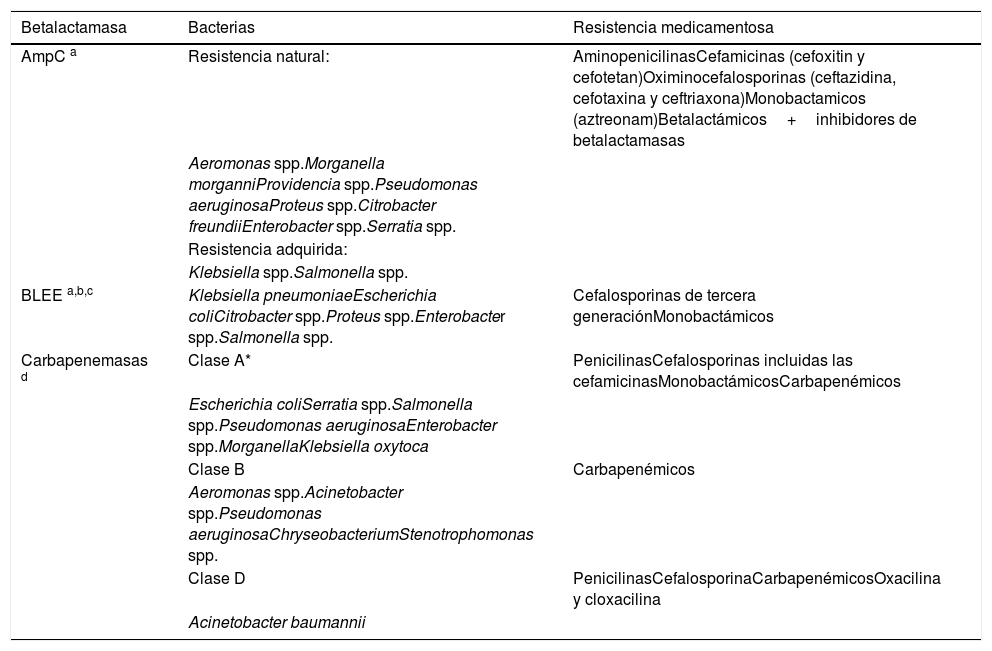
El antibiograma evalúa la susceptibilidad de un patógeno a un fármaco. Los resultados se expresan en las categorías sensible, intermedio o resistente, y se analizan teniendo en cuenta los puntos de cohorte establecidos por los distintos comités. Sin embargo, la lectura del antibiograma va más allá de la determinación del perfil de sensibilidad bacteriano. Actualmente se propone la lectura interpretada del antibiograma, la cual provee una aproximación al mecanismo de resistencia de la bacteria, la detección de patrones inusuales y finalmente un cambio en la conducta terapéutica. Este abordaje implica 5 pilares: el paciente, conocimiento de la especie del microorganismo, los fenotipos de resistencia de este, la epidemiología local y los fármacos marcadores.
Antibiotic sensitivity testing evaluates the susceptibility of a pathogen to an antibiotic. The results are expressed in the categories: sensitive, intermediate, or resistant, and are analysed taking into account the cohort points established by the different committees. However, the reading of antibiotic sensitivity testing goes beyond the determination of the bacterial sensitivity profile. The interpretive reading of antibiotic sensibility tests is proposed in this article, which will provide an estimation of the mechanism of resistance of the bacteria, the detection of unusual patterns, and finally a change in the therapeutic management. This approach involves 5 pillars: the patient, knowledge of the species of the microorganism, its resistance phenotypes, local epidemiology, and marker drugs.
Artículo
Socios de la Asociación de Medicina Crítica y Cuidado Intensivo
Para acceder a la revista
Es necesario que lo haga desde la zona privada de la web de la AMCI, clique aquí
Comprando el artículo el PDF del mismo podrá ser descargado
Precio 19,34 €
Comprar ahora