Uno de los elementos claves en la medicina de la segunda década del siglo XXI es el crecimiento exponencial de la información producida por los pacientes que se debe no solo a la transición hacia la digitalización de las historias clínicas, sino también a la aparición de nuevas fuentes de información y a la capacidad de análisis e interpretación de las ya existentes. Se estima que la cantidad de información médica se duplica cada 2 años, lo que supone que en 2020 existirá 50 veces más información que en 2011. En este contexto, el manejo adecuado de grandes cantidades de datos o big data debe traducirse en nuevas iniciativas que mejoren el diagnóstico, tratamiento y pronóstico de los pacientes en el camino hacia la medicina personalizada.
El concepto de personalización o de medicina de precisión cobra especial interés en la patología respiratoria crónica. En los últimos años la investigación en entidades como el asma, la EPOC, el cáncer o el SAHS se ha centrado en la identificación de alteraciones genómicas, moleculares, metabólicas y proteicas (biomarcadores). Su análisis mediante herramientas de análisis de big data permite dejar atrás los modelos centrados en la respuesta media al tratamiento, que a su vez resultan subóptimos para la mayoría, para dar paso a la respuesta individualizada. En este camino se incorpora la medicina de sistemas, que, integrando también datos clínicos y poblacionales, aporta una visión multidimensional de la enfermedad y facilita el establecimiento de asociaciones causales, la mayoría de las cuales solo son apreciables mediante análisis de big data.
One of the key elements of medicine in the second decade of the 21st century is the exponential growth of patient-produced information, due not only to the transition to the digitization of medical records, but also to the emergence of new sources of information and the capacity for analysis and interpretation of existing ones. The amount of medical information is expected to double every 2 years, which means that there will be 50 times more information available in 2020 than in 2011. In this setting, these large amounts of data or «big data» must be properly managed to implement new initiatives that improve the diagnosis, treatment, and prognosis of patients on the path to personalized medicine.
The concept of personalization or precision medicine is of special interest in chronic respiratory disease. In recent years, research in entities such as asthma, COPD, cancer, or SAHS has focused on the identification of genomic, molecular, metabolic, and protein changes (biomarkers). Big data analysis tools can be used to move on from models based on the mean response to treatment, which are suboptimal for most patients, to focus on the individualized response. Part of this journey involves systems medicine, which also integrates clinical and population data to provide a multidimensional view of the disease and help identify causal associations that are usually only evident on big data analysis.
«In GOD we trust, all others must bring data». W. Edwards Deming
IntroducciónEl big data puede definirse mediante 3 «V»: volumen, variedad y velocidad1–3. La primera hace referencia a la enorme cantidad de datos, de crecimiento exponencial, que, en el caso de la patología respiratoria, puede proceder de las historias clínicas electrónicas (HCE), de los dispositivos electrónicos que manejan los pacientes, de sensores, de los medios sociales y de otras bases de datos regionales o nacionales que aportan diferentes informaciones poblacionales. De la heterogeneidad de estos datos se extrae la segunda constante del big data, la variedad. Por último, la velocidad hace referencia a la rapidez con la que la información es producida y recogida, lo que permite analizar el estado de salud individual y poblacional, mejorar el abordaje temprano de cada paciente y tomar decisiones casi en tiempo real en cuanto a la distribución de los recursos. Teniendo en cuenta que en Europa el gasto asociado a la patología respiratoria supone el 6% del presupuesto sanitario4, mejorar el abordaje clínico, y la distribución y planificación de los recursos gracias al análisis de los datos generados por los pacientes repercutirá, no solo positivamente sobre estos, sino también sobre la eficiencia del sistema sanitario.
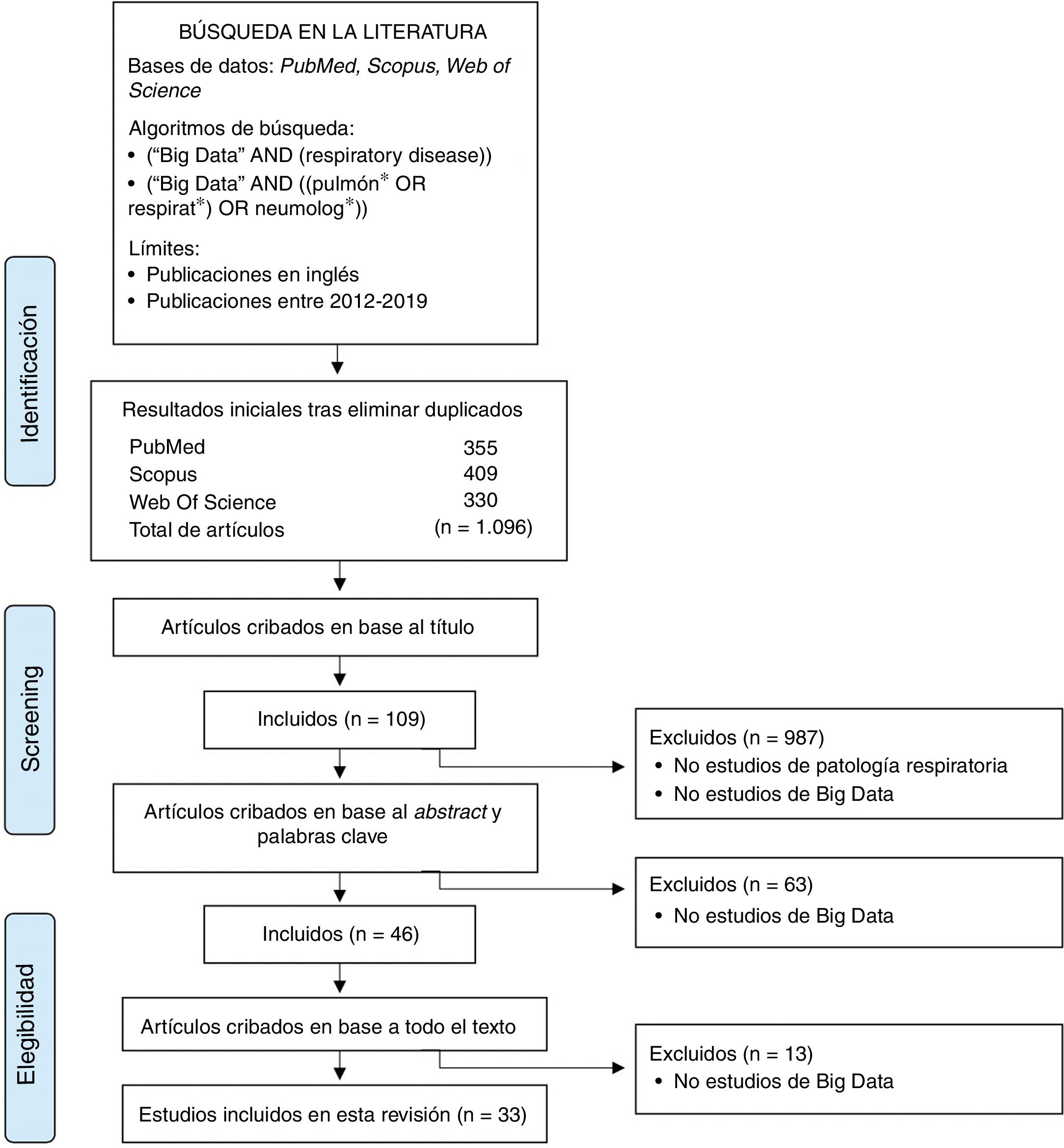
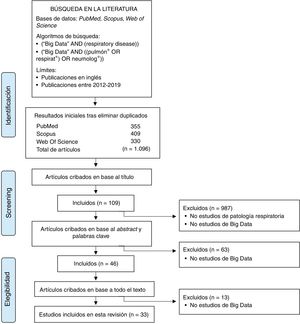
Con el fin de revisar el estado del arte del big data en patología respiratoria se ha realizado una búsqueda bibliográfica de artículos publicados entre 2012 y 2019 utilizando como motores de búsqueda Pubmed, Scopus y Web Of Science (fig. 1).
Aplicaciones del big data en la EPOCFrente a una metodología clásica, como fue la utilizada en el estudio ECLIPSE5,6, Serra-Picamal et al., en un estudio de big data llevado a cabo en Cataluña, seleccionaron a 17.555 pacientes hospitalizados por primera vez por EPOC7. Este estudio, gracias al análisis de macrodatos, permite describir 4 diferentes trayectorias del paciente EPOC tras la primera hospitalización (EPOC frágil, EPOC con reingreso precoz, EPOC con reingreso y EPOC sin reingreso), cada una de ellas con un perfil clínico y pronóstico propio. Otro de los trabajos big data más representativos hasta la fecha es el de Souliotis et al., que establece una cohorte EPOC a partir de los datos de Central Unit of Prescriptions Processing (KMES), la mayor aseguradora social de Grecia y que incluye a más del 90% de la población8. Este estudio proporciona los datos poblacionales necesarios para mejorar la prescripción farmacológica según la edad, el sexo y los tratamientos previos. Siguiendo esta idea, Kuilboer et al. publicaron los resultados de AsthmaCritic (Holanda), un sistema de apoyo para la toma de decisiones que tiene el objetivo de mejorar la práctica clínica en atención primaria para asma y EPOC mediante un algoritmo basado en los datos recogidos por el clínico en la HCE9. En otro campo, el análisis big data de Qiu et al. estudia el impacto de la contaminación ambiental en las exacerbaciones (hospitalizaciones) de EPOC (cohorte de 54.966 pacientes), encontrando una relación positiva para niveles elevados de PM25, PM10 y SO2 en los días previos al ingreso. La asociación en pacientes de edad avanzada (>80 años) fue mayor10. Además, este estudio recogió la temperatura media diaria, objetivando interacciones entre los niveles de contaminación y las temperaturas bajas, de manera que concentraciones altas de PM y temperaturas bajas se asociaron con el mayor riesgo de hospitalización entre la población EPOC10. Estos hallazgos concuerdan con los de Lee et al., en cuya cohorte big data de pacientes EPOC las bajas temperaturas, la humedad, las concentraciones elevadas de PM10 y las infecciones víricas se asociaron con el aumento de las exacerbaciones; sugiriendo además que el frío potencia el efecto de estas dos últimas11. En Europa (Polonia), Zakowska et al. han iniciado un estudio big data con análisis de datos demográficos, sociales, sanitarios, económicos y ambientales con el objetivo de identificar asociaciones múltiples entre estos factores y las exacerbaciones de EPOC12.
Recientemente se ha publicado el primer estudio que ha analizado la situación de la EPOC en España, utilizando metodología de big data, a partir de los datos capturados de registros clínicos electrónicos13. Con la popularización de esta tecnología es factible identificar los problemas reales en una población, implementar proyectos que ayuden a mejorar la práctica clínica y, simultáneamente, una monitorización continua de los resultados.
Aplicaciones del big data en el asmaEn el asma también existe heterogeneidad, con diferentes endotipos y fenotipos que modifican la respuesta al tratamiento14,15. Sin embargo, los estudios realizados hasta la fecha con el objetivo de identificar las alteraciones genéticas responsables de los endotipos no han obtenido resultados concluyentes, en parte porque se necesitan grandes bases de datos para poder establecer relaciones significativas, pero también porque las interacciones genéticas pueden ser múltiples, sin estar un solo gen asociado con un fenotipo concreto. A pesar de ello, un estudio sobre 1.173 pacientes sí relaciona el gen CDHR3 con un aumento de la susceptibilidad a asma en general y a las hospitalizaciones tempranas en particular16. El futuro del big data es prometedor en este campo. El análisis de grandes cantidades de datos no requiere la estratificación de los pacientes y es capaz de integrar información genética, biológica, medioambiental y fenotípica si se dispone de las herramientas computacionales necesarias para analizarlos. Este abordaje supone una ventaja importante en el estudio de las ómicas y de los biomarcadores. En el momento de esta revisión no se ha encontrado ningún estudio de big data realizado con dicho objetivo, pese a que sí existen estudios multicéntricos a nivel europeo como U-BIOPRED (Unbiased Biomarkers for the Prediction of Respiratory Disease), que emplean el abordaje de la medicina de sistemas para identificar los mecanismos subyacentes al asma17.
Aplicaciones del big data en el SAHSEl potencial del big data en el SAHS es enorme, ya que la información producida por los pacientes se puede recoger desde diversas fuentes: estudios de sueño, equipos de CPAP, dispositivos electrónicos, encuestas y formularios, HCE, biomarcadores y datos genéticos. Actualmente la base de datos genéticos más amplia (International Sleep Genetic Epidemiology Consortium) recoge aproximadamente 25.000 registros, pero se estima que esta muestra solo es la décima parte de la necesaria para identificar variantes genéticas poco frecuentes que expliquen la variabilidad interindividual3. Para obtener y manejar grandes volúmenes de datos genéticos que, además, deben integrarse con los datos clínicos para extraer conclusiones, el National Institute of Health (NIH) creó la iniciativa Big Data to Knowledge (BD2K), que pretende facilitar el acceso a la información y favorecer el desarrollo de los métodos analíticos18. De forma similar, en España, la SSN (Spanish Sleep Network) puso en marcha BIOSSAHS, un proyecto en el que se recogen datos clínicos y muestras biológicas con el objetivo de avanzar en la identificación de nuevos biomarcadores y en la caracterización fenotípica19.
Respecto a la asociación de las diferencias interindividuales con las comorbilidades del SAHS, Mohklesi et al. llevaron a cabo un estudio de big data en EE.UU. con 1.704.905 pacientes y un grupo control, concluyendo que la HTA, la diabetes mellitus, la cardiopatía isquémica, la depresión, la obesidad mórbida, las arritmias, la insuficiencia cardiaca congestiva y el IAM son comorbilidades más frecuentes en el SAHS que en la población general. Dichas asociaciones se conocían previamente, pero este estudio confirma la relación con mayor precisión y aporta, en contraposición con otras cohortes tradicionales, nuevas asociaciones específicas en subgrupos de pacientes poco estudiados hasta la fecha: mujeres y diferentes rangos de edad20. En Europa, a partir de la European Sleep Apnea Database (ESADA), también se trabaja en la identificación de fenotipos clínicamente relevantes y en su relación con las comorbilidades, destacando la caracterización de pacientes en función de los patrones de sueño (hipersomnolencia diurna vs. síntomas de insomnio)21.
Con el objetivo de encontrar factores predictivos de finalización del tratamiento con CPAP e identificar fenotipos de pacientes que pudieran beneficiarse de otros abordajes, Woehrle et al. recogieron los datos de 98.329 pacientes que iniciaron el tratamiento con CPAP entre 2009 y 2014 a partir del ResMed Healthcare, una de las empresas prestadoras de servicios de CPAP alemana. Se describe que el 12% abandonó el tratamiento en el primer año y se encontró una relación en «U» entre los pacientes, siendo que los más jóvenes (<30 años) y mayores (>80 años) tienen las tasas más altas de discontinuación. Otros factores relacionados con el cese de la CPAP fueron: el sexo femenino, la sanidad pública y que el primer tratamiento fuera CPAP22. Con objetivos similares, Turino et al. analizaron la heterogeneidad entre todos los pacientes con SAHS tratados con CPAP en Cataluña e identificaron 6 subgrupos diferentes en cuanto a comorbilidades asociadas, mortalidad y utilización de los recursos23. Es muy probable que la expansión de los biomarcadores y las mejoras en el análisis de datos procedentes de dispositivos electrónicos, estudios de sueño, CPAP, etc., todo ello en el contexto de la integración de big data, tengan un impacto significativo sobre el manejo de los pacientes en la medicina del sueño24.
Aplicaciones del big data en el cáncer del pulmónEsfuerzos similares dirigidos a optimizar el manejo de los pacientes mediante big data se han realizado también en otras entidades como el cáncer de pulmón. La base de estas intervenciones es que la agregación de múltiples parámetros (biomarcadores, pruebas de imagen u otros), HCE y protocolos de manejo permite el desarrollo de herramientas inteligentes que apoyen y faciliten la toma de decisiones, tal y como se objetiva con el modelo desarrollado por Wu et al.25. Continuando con la aplicación de esta herramienta en la práctica clínica, Murphy et al. presentan un algoritmo capaz de identificar red flags en los informes radiológicos y alertar al clínico cuando un paciente está sufriendo retrasos en el diagnóstico que pueden comprometer el pronóstico. La sensibilidad fue del 99%, la especificidad del 38%, el VPN del 97% y el VPP del 61%, concluyendo que el algoritmo es capaz de identificar, entre grandes cantidades de datos radiológicos e HCE, casi todos los retrasos diagnósticos y mejorar de forma coste-efectiva el manejo de los pacientes con imágenes sugestivas de malignidad26.
El volumen de big data producido por estos pacientes permite el desarrollo del aprendizaje automático o machine learning, cuyo cometido es analizar los datos mediante algoritmos computacionales que permitan clasificar, predecir y segmentar la información para extraer conclusiones no disponibles para el ojo humano27. Con este objetivo ya se han desarrollado sistemas CADe (computer-aided detection) y CADx (computer-aided diagnosis) que permiten cuantificar y calificar las apodadas como características radiómicas, para mejorar el diagnóstico por imagen. Actualmente existen otros sistemas de machine learning aplicables al diagnóstico histológico, tratamiento y pronóstico de los pacientes27.
Big data multidisciplinar en patología respiratoriaLa aparición de herramientas computacionales cada vez más potentes permite que el análisis de big data pueda agregar no solo información clínica o médica, sino también otra serie de datos no relacionados, como variables socio-ecológicas y ambientales28. Qiu et al. estudiaron durante un año (2015) el número de hospitalizaciones por causa respiratoria en 17 ciudades de Sichuan Basin (China), en relación con los niveles diarios de PM2.5, PM10, NO2 y SO2. Del total, 115.788 hospitalizaciones fueron debidas a infecciones del tracto respiratorio superior, 152.537 a neumonía, 207.770 a EPOC y 16.986 a asma. Se concluye que el aumento de las concentraciones de contaminantes incrementa el riesgo de ingreso por patología respiratoria, sin encontrarse diferencias entre sexos29. Estos resultados fueron consistentes con los obtenidos previamente por Scarinzi et al. en un estudio en 25 ciudades italianas30. Además, la inclusión de grupos poblacionales de edades extremas permitió corroborar que los niños y pacientes de edades avanzadas son más vulnerables a los efectos de los contaminantes ambientales29. En el estudio de Kuo et al. (cohorte de big data) se explora la interacción particular del asma en niños y la contaminación, concluyendo que el factor con mayor impacto en exacerbaciones es el O3 y que existen variaciones estacionales (pico de hospitalización en primavera)31. Los resultados de estos estudios nos indican que con la agregación de datos ambientales y clínicos se pueden extraer conclusiones que pueden mejorar el manejo y reducir las hospitalizaciones.
Otro ejemplo de análisis big data multidisciplinar es el de Dwyer-Lindgren et al., que con el empleo de datos socio-ecológicos, demográficos, geográficos y clínicos estudiaron las diferencias en mortalidad de las enfermedades respiratorias crónicas entre los diferentes estados de EE.UU.32. En esta línea Mannino y Sanderson realizan un análisis descriptivo de las asociaciones geográficas de cada una de las entidades con los patrones de mortalidad, de modo que la identificación de factores de riesgo permite implementar medidas de salud pública específicas en cada estado33.
Modelos predictivos y análisis en el mundo realLas estrategias encaminadas a mejorar el pronóstico de los pacientes con patología respiratoria convergen en intentar disminuir la tasa de exacerbaciones, el empeoramiento clínico y las complicaciones de la enfermedad. Los datos recogidos en múltiples fuentes de big data pueden contribuir en la identificación de «biomarcadores digitales»34 que sirvan como predictores de futuras exacerbaciones.
Revisando la literatura se han encontrado algunos modelos que reúnen las características descritas, dentro de los cuales cabe destacar el de Ram et al., un modelo predictivo para asma que introduce como nuevas fuentes de big data las búsquedas en Google, Twitter y sensores ambientales35, partiendo del antecedente de Google Flu Trends y su capacidad de estimar la actividad de la epidemia gripal36,37. Este modelo es capaz de predecir en tiempo real el número de pacientes con asma que acudirán a urgencias con un 70% de precisión35. Los autores ya han puesto en marcha un nuevo estudio para validar los resultados en un área geográfica más amplia y durante más tiempo.
Otros ejemplos son el de Fishe et al., que estudian variables clínicas, demográficas y socio-ecológicas para identificar características prodrómicas de asma, ACO y EPOC con el objetivo de mejorar el diagnóstico precoz de estas entidades en base a modelos predictivos28; o el de Lynch et al., que analizan las posibilidades de aplicación del machine learning a la predicción de la supervivencia de los pacientes con cáncer de pulmón38. De forma similar, el aprendizaje automático también se comienza a emplear en la predicción de la respuesta al tratamiento2,39,40. El potencial de estos modelos queda patente, pero también se intuye que la cantidad de variables necesarias para que la predicción sea eficaz excede en número a lo manejable en la práctica clínica41.
La práctica clínica genera interrogantes a una velocidad y complejidad tales que se hace necesaria una nueva forma de llevar a cabo la investigación médica. La reciente epidemia por el virus SARS-Cov-2 es un buen ejemplo de esta necesidad. Esta experiencia hace necesario un nuevo abordaje que sea compatible con la actividad asistencial y que, a su vez, dependa de la misma, todo ello con el objetivo de responder con la mayor evidencia y rapidez a preguntas centradas en el paciente. De este modo, la información del mundo real, la generada durante el proceso asistencial, debe ser recogida y analizada para proporcionar evidencia del mundo real42. Esta forma de trabajo, además de transformar la relación entre el clínico y la investigación, resulta la mejor fuente de evidencia posible para tomar decisiones clínicas. El volumen, la velocidad, la variedad y la veracidad de la información del mundo real que promete el big data permite diseñar estudios observacionales y experimentales como el CleanUP-IPF trial43, el único ensayo clínico con estas características en el campo de la enfermedad pulmonar intersticial. Así, en los próximos años, la creación de cohortes extensas permitirá comparar la respuesta al tratamiento, todo ello a un coste radicalmente menor que la investigación actual.
Nuevos abordajes: mHealth y eHealthA pesar de los avances terapéuticos, el pronóstico de los pacientes con enfermedades respiratorias crónicas sigue siendo pobre en comparación con otras entidades. Si bien se pueden identificar múltiples causas, la falta de adherencia al tratamiento es una de las principales34. Con el objetivo de mejorar dicha adherencia, se han desarrollado tecnologías digitales que permiten monitorizar al paciente, extraer resultados agregados y mejorar el tratamiento mediante el abordaje personalizado. Ejemplos de ello son los inhaladores electrónicos o smart inhalers, los servicios de mensajería, las herramientas para el autocontrol de la enfermedad, los nebulizadores con chip, los biosensores y los monitores remotos de parámetros fisiológicos, como el PEF o FeNO, contaminación ambiental, humedad, temperatura o de ejercicio físico. Estos dos últimos se encuadran dentro del término mHealth (Mobile Health), que hace referencia al uso de dispositivos móviles para mejorar la práctica clínica3,44. El potencial de esta tecnología contribuye a que empresas como Amazon, Apple o Google decidan introducirse en el sector sanitario con la creación de dispositivos médicos interconectados, centrados en las necesidades del consumidor y que, a día de hoy, ya pueden aplicarse en el control de la diabetes o enfermedades cardiovasculares45,46. En definitiva, existe potencial para identificar los comportamientos que requieren mejora, intervenir de forma efectiva y mejorar la adherencia y el pronóstico de los pacientes34.
ConclusiónEl big data aplicado a la patología respiratoria proporciona las herramientas necesarias para avanzar hacia la medicina personalizada, mejorar el pronóstico de los pacientes con enfermedades respiratorias crónicas, apoyar la labor clínica del médico, identificar grupos poblacionales en riesgo y para implementar medidas preventivas y actuaciones coste-eficientes a nivel regional y nacional. Sin embargo, es necesario comprender las implicaciones que tiene utilizar un análisis generador de hipótesis con el fin de tomar las medidas necesarias para que la evidencia generada sea clínicamente útil y significativa.
FinanciaciónBeca UAH 2019/00003/016/001/005, Universidad de Alcalá, España.
Conflicto de interesesLourdes Prats no refiere conflicto de intereses.
José Luis Izquierdo ha recibido honorarios por asesoramiento, proyectos o ponencias de AstraZeneca, Bayer, Boehringer Ingelheim, Chiesi, Glaxo, Grifols, Smith Kline, Menarini, Novartis, Orion, Pfizer, Sandoz, y Teva.