La existencia de pacientes colonizados es una de las principales vías de propagación de las bacterias multirresistentes, y su contención una prioridad asistencial y de salud pública. Los estudios de vigilancia son imprescindibles para una detección precoz de la colonización por estas bacterias. Este artículo aborda los diferentes métodos microbiológicos, basados en el cultivo y moleculares, para la detección del estado de portador de bacterias multirresistentes. Se incluyen aquellas especies de mayor interés debido a su impacto clínico/epidemiológico y a las dificultades terapéuticas que generan: Staphylococcus aureus resistente a meticilina, Enterococcus spp. resistente a los glucopéptidos, enterobacterias productoras de β-lactamasas de espectro extendido o β-lactamasas plasmídicas de tipo AmpC, enterobacterias productoras de carbapenemasas, Acinetobacter baumannii multirresistente y Pseudomonas aeruginosa multirresistente. La información recogida en este documento debe considerarse como una estructura matriz que deberá adaptarse a las necesidades específicas de cada centro.
The presence of colonised patients is one of the main routes for the spread of multiresistant bacteria, and its containment is a clinical and public health priority. Surveillance studies are essential for early detection of colonisation by these bacteria. This article discusses the different microbiological methods, both based on culturing and molecular methods, for detection of carriers of multiresistant bacteria. Those species with a high clinical/epidemiological impact or generating therapeutic difficulties are included: Methicillin-resistant Staphylococcus aureus, Enterococcus spp. resistant to glycopeptides, enterobacteriaceae producing extended spectrum β-lactamases and plasmid-mediated AmpC, carbapenemases producing enterobacteriaceae, Acinetobacter baumannii and multiresistant Pseudomonas aeruginosa. The information in this document should be considered as a structure matrix to be tailored to the specific needs of each centre.
La resistencia a múltiples antibióticos está aumentando en algunos de los principales patógenos bacterianos1–3, limitando las alternativas terapéuticas. La multirresistencia (MDR), definida como la ausencia de sensibilidad a al menos un antibiótico de 3 o más familias4, puede afectar a bacterias grampositivas y gramnegativas. Sin embargo, merece especial atención la diseminación de bacterias gramnegativas con resistencia extensa (XDR: ausencia de sensibilidad a al menos un antibiótico de todas las familias excepto una o 2) y panresistencia (PDR: ausencia de sensibilidad a todos los antibióticos de todas las familias habitualmente utilizadas en el tratamiento de la bacteria considerada)4, en especial Acinetobacter baumannii (A. baumannii), Pseudomonas aeruginosa (P. aeruginosa) y algunas especies de enterobacterias. Las bacterias MDR suelen presentar una alta capacidad de diseminación epidémica, no solo intrahospitalaria sino también inter- y extrahospitalaria1,5. La presencia de pacientes colonizados por estas bacterias es una de sus principales vías de propagación6,7, y su contención una prioridad asistencial y de salud pública reconocida por las principales instituciones nacionales e internacionales. Por tanto, son necesarios estudios de vigilancia microbiológica locales que permitan una detección precoz de los pacientes colonizados por este tipo de bacterias. EUCAST dispone de unas guías para la detección de mecanismos de resistencia de importancia clínica y epidemiológica8.
Recientemente la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC) ha publicado un procedimiento de microbiología clínica titulado «Métodos microbiológicos para la vigilancia del estado de portador de bacterias multirresistentes»9. El objetivo del presente artículo es resumir las aportaciones de este procedimiento. Dicho documento aborda las especies patógenas de mayor interés debido a su impacto clínico y epidemiológico: Staphylococcus aureus resistente a meticilina (SARM), Enterococcus spp. resistente a los glucopéptidos (ERG), enterobacterias productoras de β-lactamasas de espectro extendido (BLEE) o β-lactamasas plasmídicas de tipo AmpC (AmpC-p), enterobacterias productoras de carbapenemasas (EPC), A. baumannii multirresistente y P. aeruginosa multirresistente. La información general recogida en este documento debe considerarse como una estructura matriz que se beneficiará de una adecuada adaptación a las necesidades específicas establecidas por el equipo multidisciplinar de control de la infección nosocomial de cada centro.
Impacto clínico y epidemiológicoStaphylococcus aureus resistentes a la meticilina (SARM)S. aureus es un microorganismo que, además de colonizar habitualmente a los seres humanos, puede comportarse como un patógeno oportunista y ser la causa de una gran diversidad de infecciones. En 2013, los datos de la European Antimicrobial Resistance Surveillance Network (EARS-Net) mostraban una prevalencia de SARM en bacteriemias del 22,6% en España y del 18% en el total de los países europeos participantes. En España, existen variaciones importantes en su prevalencia dependiendo del área geográfica y del tipo de hospital. Las implicaciones clínicas y epidemiológicas obligan a establecer sistemas de detección adecuados10. El principal mecanismo de resistencia a la meticilina es la producción de una proteína de unión a la penicilina (PBP2a), codificada por el gen mecA, que presenta baja afinidad por los antibióticos β-lactámicos, a excepción de una nueva clase de cefalosporinas como es la ceftarolina o el ceftobiprole. En el año 2011 se describió un gen homólogo denominado mecC11. Se han aislado cepas de SARM-mecC en multiples países, incluido España, procedentes de humanos y de una gran variedad de animales. La detección del gen mec permite la diferenciación de otras cepas de S. aureus, muy poco frecuentes, con bajo nivel de resistencia a la meticilina debido tanto a alteraciones de las PBP (moderately resistant S. aureus [MODSA]) como a hiperproducción de β-lactamasa (bordeline oxacillin-resistant S. aureus [BORSA]). El protocolo que se desarrolla en este procedimiento se refiere a S. aureus que contiene el gen mec.
Enterococcus spp. resistente a los glucopéptidos (ERG)Enterococcus faecalis (∼80-90%) y Enterococcus faecium (∼5-10%) son las especies que causan la mayoría de las infecciones en humanos. Estos microorganismos son responsables de aproximadamente el 10% de las infecciones relacionadas con la asistencia sanitaria. Además, es importante su resistencia intrínseca a múltiples antibióticos, y su capacidad de desarrollar resistencias a otros antibióticos, bien por adquisición de genes localizados en plásmidos o transposones (ej.: resistencia a glucopéptidos) o por mutaciones cromosómicas (ej.: resistencia a fluoroquinolonas). En EE. UU. se produjo un rápido aumento de brotes nosocomiales de ERG que condujo a una situación endémica en ciertos hospitales. En contraste, en Europa, el ERG se aisló inicialmente de fuentes comunitarias (personas sanas y animales) debido al amplio uso de avoparcina, un glucopéptido utilizado como promotor de crecimiento de animales de granja hasta su prohibición en 1997. Los datos del EARS-Net en 2013 mostraban importantes diferencias en la prevalencia de ERG en los distintos países europeos. En España la prevalencia de E. faecium resistente a vancomicina fue del 0,9% en 2013. A pesar de este bajo porcentaje, con cierta frecuencia se describe la aparición de brotes hospitalarios.
La resistencia adquirida a glucopéptidos en enterococo está mediada por 9 operones diferentes denominados van (vanA, vanB, vanC, vanD, vanE, vanG, vanL, vanM, vanN). Dos de ellos, el vanA y el vanB, son los más frecuentes en E. faecalis y E. faecium con diferencia. El gen vanA confiere resistencia inducible de alto nivel a vancomicina y teicoplanina mientras que el gen vanB confiere resistencia inducible de alto o moderado nivel a vancomicina y permanece sensible a teicoplanina. Las especies de E. faecium o E. faecalis con resistencia a vancomicina (CMI >4mg/l) son las de mayor interés epidemiológico y requieren vigilancia microbiológica12.
Enterobacterias productoras de β-lactamasas de espectro extendido, β-lactamasas plasmídicas de tipo AmpC y carbapenemasasLa diseminación de enterobacterias resistentes a cefalosporinas de amplio espectro, principalmente BLEE, se ha convertido en una de las principales amenazas para el tratamiento antibiótico de las infecciones producidas por estas bacterias. En España, E. coli y K. pneumoniae productores de BLEE, sobre todo del tipo CTX-M-15 y SHV-12, se han extendido rápidamente en los últimos años y han alcanzado prevalencias globales de alrededor del 15%. Aunque menos frecuentes, las AmpC-p están emergiendo como una amenaza adicional a la actividad de las cefalosporinas de amplio espectro. Las principales tipos de AmpC-p son CIT y DHA, que afectan a principalmente a E. coli, K. pneumoniae y Proteus mirabilis. La frecuente asociación de la producción de BLEE y AmpC-p con resistencia a otros antibióticos no β-lactámicos ha generado un aumento en el consumo de antibióticos carbapenémicos y la aparición de cepas resistentes a ellos. En la actualidad, una de las mayores amenazas en el campo de la resistencia a antibióticos es la rápida diseminación de cepas MDR y XDR productoras de carbapenemasas. Estas enzimas son capaces de hidrolizar la casi totalidad de los antibióticos β-lactámicos, incluidos los carbapenémicos. La mayoría se agrupan, según la clasificación molecular de Ambler, en: 1) clase A, principalmente enzimas del tipo KPC; 2) clase B o metalo-β-lactamasas (MBL), principalmente enzimas del tipo VIM, IMP y NDM y 3) clase D, principalmente OXA-48. Un estudio multicéntrico realizado en 2013 (proyecto GEIH-GEMARA-REIPI) mostró que OXA-48 (71,5%) y VIM-1 (25,3%) fueron las carbapenemasas más frecuentes en enterobacterias en España y K. pneumoniae (74,4%) la especie más afectada1. Además, se detectó una amplia diseminación de algunos clones de alto riesgo de K. pneumoniae como ST11/OXA-48, ST15/OXA-48, ST405/OXA-48 y ST11/VIM-11. Aunque menos frecuentes, otras carbapenemasas como las KPC y NDM también están aumentando en España y generan importantes brotes intra- e interhospitalarios13. A finales de 2015, España presentaba un escenario de diseminación interregional de EPC13. Las alternativas terapéuticas en estos casos son muy escasas y casi nunca óptimas.
Mientras que la vigilancia de la colonización por enterobacterias productoras de BLEE y carbapenemasas debería abarcar cualquier especie, la vigilancia de las AmpC-p debería realizarse en aquellas especies que no poseen AmpC cromosómicas y han demostrado su capacidad de diseminar estas enzimas: fundamentalmente K. pneumoniae y P. mirabilis.
Acinetobacter baumannii multirresistente a antibióticosA. baumannii es un patógeno oportunista implicado en infecciones como neumonía asociada a ventilación mecánica, endocarditis, infecciones de la piel y partes blandas, meningitis e infecciones del tracto urinario, entre otras, especialmente entre los pacientes admitidos en las UCI.
El género Acinetobacter se compone de 30 especies con un nombre asociado y 9 especies genómicas definidas a través de estudios de hibridación ADN-ADN. Entre ellas, A. baumannii muestra una extraordinaria capacidad para adquirir resistencia a múltiples antibióticos. En el último estudio multicéntrico nacional (proyecto GEIH-GEMARA-REIPI-Ab2010) en el que se incluyeron 446 aislados de A. baumannii obtenidos de 43 hospitales españoles3, se detectó una alta prevalencia de reducción de sensibilidad a la mayoría de los antimicrobianos: >94% (ceftazidima, piperacilina y ciprofloxacino), 82-86% (carbapenemas y tetraciclina) y 60-70% (tobramicina, sulbactam, gentamicina y doxiciclina). En comparación con los datos obtenidos en un estudio previo realizado en 201014, estos aislados fueron más resistentes a ceftazidima, carbapenemas, doxiciclina, sulbactam y colistina; la mayoría fueron MDR o XDR. En este mismo estudio se puso de manifiesto como la tasa de incidencia de la colonización o infección por A. baumnanii se incrementó significativamente desde el 0,14 en 2000 al 0,52 en 2010 (p<0,001). Además, el estudio epidemiológico a través de MLST puso de manifiesto un incremento del grupo clonal ST2 en 2010, el cual mostró mayor resistencia a imipenem y se asoció a un mayor riesgo de sepsis6.
En A. baumannii se han descrito diferentes tipos de β-lactamasas, pero son las carbapenemasas adquiridas de clase D (OXA-23, -24, -58, -143 y -235) las que tienen un mayor impacto clínico y epidemiológico. La reducción en la permeabilidad, la hiperexpresión de sistemas de expulsión activa, la producción de enzimas modificantes de aminoglucósidos y cloranfenicol, metilasas, y modificaciones del sitio de interacción de la diana también están implicados en la resistencia a antibióticos en A. baumannii.
Los factores epidemiológicos implicados en la adquisición hospitalaria de Acinetobacter spp. son bien conocidos. Su implicación en brotes hospitalarios se ve favorecida por su capacidad de sobrevivir en desecación, transmitirse de paciente a paciente a través de las manos del personal y contaminar el ambiente hospitalario. En España, se han descrito brotes por A. baumannii portadores de las carbapenemasas OXA-23, OXA-24 y OXA-58.
Pseudomonas aeruginosa multirresistente a antibióticosP. aeruginosa es una de las causas más frecuentes de infección nosocomial y de infección respiratoria crónica en pacientes con fibrosis quística. La prevalencia de cepas MDR supera el 30% a nivel mundial, incluyendo los hospitales españoles; aproximadamente la mitad de las cepas MDR serían, además, XDR2. Esta creciente prevalencia se debe a la extraordinaria capacidad de P. aeruginosa para seleccionar mutaciones cromosómicas que generan resistencia y a la creciente producción de determinantes de resistencia exógenos15.
El principal mecanismo de resistencia a las penicilinas o cefalosporinas es la selección de mutantes con hiperproducción constitutiva (desrepresión) de la cefalosporinasa cromosómica inducible AmpC16. Entre los mecanismos de resistencia mutacionales destaca también la inactivación de la porina OprD, que confiere resistencia a imipenem y sensibilidad disminuida al meropenem. Finalmente, la hiperexpresión de alguna de las múltiples bombas de expulsión, principalmente MexAB-OprM y MexXY-OprM, contribuye de forma notable a los fenotipos de resistencia16. Aunque proporcionalmente es mucho menos común que la resistencia mutacional, cada vez es más frecuente la detección de elementos genéticos transferibles portadores de genes de carbapenemasas o BLEE. Estudios recientes demuestran que la mayoría de las cepas productoras de carbapenemasas o BLEE pertenecen a los denominados clones de alto riesgo, principalmente los ST235, ST111 o ST1752,5. Particularmente relevantes son las MBL, cuya prevalencia en España ha aumentado más de 10 veces (del 0,08 al 1%) en 5 años (2003-2008) y han sido responsables de importantes brotes epidémicos16. Las MBL descritas en P. aeruginosa incluyen las VIM, IMP, SPM, GIM, SIM, NDM, AIM y FIM17. La MBL detectada con mayor frecuencia en España es, con gran diferencia, la VIM-2. Entre las carbapenemasas de clase A destacan las de tipo GES y, entre las BLEE, las de tipo OXA18.
Recogida, transporte, conservación y procesamiento de la muestraLa toma de la muestra, su transporte y su conservación para cultivos de vigilancia se realizarán siguiendo las recomendaciones generales de la SEIMC en su Procedimiento de Microbiología Clínica n.° 1a: «Recogida, transporte y procesamiento general de las muestras en el laboratorio de Microbiología»19. Debe constar explícitamente que la solicitud corresponde a un «cultivo de vigilancia microbiológica» así como la bacteria y problema de resistencia a vigilar, ya que en función de ello se debe evaluar la adecuación del tipo de muestra a la prueba solicitada (tabla 1). Se recomienda procesar la muestra a la mayor celeridad posible en un tiempo inferior a 24 h; si no se va a procesar de manera inmediata, es recomendable su conservación a 2-8°C para facilitar la recuperación de la bacteria a vigilar y evitar el sobrecrecimiento de la microbiota comensal.
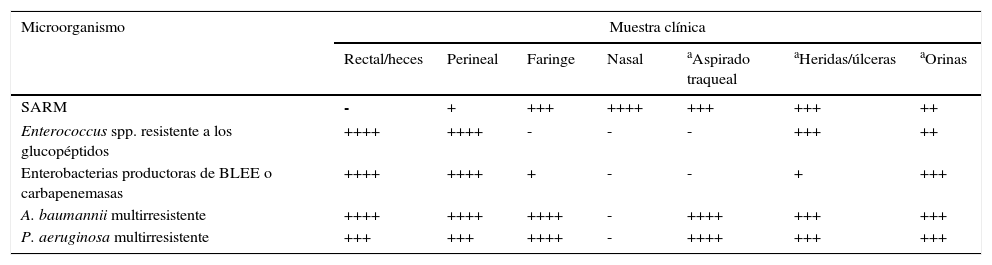
Indicaciones orientativas sobre el interés cualitativo de diferentes muestras clínicas para la investigación de patógenos multirresistentes con fines epidemiológicos
| Microorganismo | Muestra clínica | ||||||
|---|---|---|---|---|---|---|---|
| Rectal/heces | Perineal | Faringe | Nasal | aAspirado traqueal | aHeridas/úlceras | aOrinas | |
| SARM | - | + | +++ | ++++ | +++ | +++ | ++ |
| Enterococcus spp. resistente a los glucopéptidos | ++++ | ++++ | - | - | - | +++ | ++ |
| Enterobacterias productoras de BLEE o carbapenemasas | ++++ | ++++ | + | - | - | + | +++ |
| A. baumannii multirresistente | ++++ | ++++ | ++++ | - | ++++ | +++ | +++ |
| P. aeruginosa multirresistente | +++ | +++ | ++++ | - | ++++ | +++ | +++ |
En estudios previos de colonización por SARM, el exudado nasal es la muestra única más adecuada, aunque la doble (nasal-faringea o nasal-perineal) y sobre todo la triple toma (nasal-faríngea-perineal) presentan una mayor sensibilidad. El tracto gastrointestinal constituye el principal reservorio de enterobacterias multirresistentes y ERG. Por tanto, las muestras más habituales para el cultivo de vigilancia de ERG son los frotis rectales y las heces. En circunstancias en que se requiera el estudio de contaminación ambiental, se analizarán muestras procedentes de las superficies y del instrumental médico en contacto con el paciente. Dada la complejidad de la epidemiología de las infecciones por A. baumannii multirresistente, es adecuado considerar la toma de muestras tanto del paciente como del ambiente. Las muestras de vigilancia que se han evaluado más frecuentemente incluyen esputo y exudado de traqueostomía, heridas, axila/ingle y frotis rectal. La efectividad del cribado se incrementará si se realiza un muestreo de distintas zonas anatómicas del cuerpo. Los cultivos de vigilancia para P. aeruginosa multirresistente también contemplan la toma de muestras de pacientes, del medio ambiente y de equipos de atención sanitaria. Son particularmente útiles las muestras respiratorias como esputo, frotis faríngeo, exudado de traqueostomía, etc. La colonización intestinal por P. aeruginosa multirresistente se detecta con cierta frecuencia en el frotis rectal.
Métodos basados en el cultivoStaphylococcus aureus resistente a la meticilinaSelección de medios de cultivo y condiciones de incubaciónLa detección de colonización por SARM se basa fundamentalmente en el cultivo en medios selectivos y diferenciales. La combinación de una placa de agar manitol-sal (AMS o medio de Chapman) y otra de AMS-cefoxitina (4mg/l) permite la detección de SARM y, eventualmente, la colonización por S. aureus sensible a meticilina (SASM). Algunos autores han estimado una sensibilidad del 96% y una especificidad del 100% después de 48 h de incubación20. Además, un estudio reciente que evaluaba 5 medios cromogénicos [(Brilliance MRSA agar [Oxoid], ChromoID [bioMérieux], MRSASelect [Bio-Rad], CHROMagar [CHROMagar Microbiology], y BBL-CHROMagar [BD Diagnostics]), mostró una sensibilidad del 90% para el medio Brilliance MRSA agar y del 81-83% para el resto de los medios, y una especificidad del 87% para el medio Brilliance MRSA agar y del 97 al 99% para el resto de los medios cromogénicos21. La preincubación de las muestras en un medio líquido de enriquecimiento selectivo aumenta la sensibilidad de estos medios. Según los estudios publicados, no existen recomendaciones absolutas a favor ni en contra de un determinado medio cromogénico.
Pruebas de confirmación y criterios para la interpretación de los resultadosDebido a los posibles resultados falsos positivos, puede ser necesaria la confirmación tanto en la identificación como en la resistencia. En el primer caso, se recomienda la realización de una prueba de coagulasa, aglutinación con látex o la identificación mediante MALDI-TOF. Para la confirmación de la resistencia se recomienda usar un disco de cefoxitina (30μg) y realizar la lectura a las 24 h (<22mm. EUCAST 2015). En los sistemas automatizados que incluyen cefoxitina y oxacilina, aunque habitualmente estos resultados van paralelos, la interpretación de los resultados discrepantes debería resolverse a favor del resultado resistente (EUCAST 2015). El perfil de resistencia «oxacilina-S y cefoxitina-R» puede ser indicativo de SARM mecC-positivo. En estos casos, la recomendación es estudiar fenotípica y genotípicamente (mecA o mecC) las cepas de S. aureus. La resistencia también se puede confirmar mediante la aglutinación de partículas de látex recubiertas con anticuerpos monoclonales de la proteína PBP2a. Los aislamientos que producen pequeñas cantidades de dicha proteína pueden dar resultados débiles o enlentecer la reacción de aglutinación. Además, las cepas SARM mecC-positivas no se detectarán mediante este ensayo.
Información de los resultadosSi a las 48 h no hay colonias con la coloración característica se informará: «No se aísla S. aureus resistente a la meticilina» (SARM negativo). En caso de aislarse un microorganismo con la coloración característica se informará: «Se aísla S. aureus resistente a la meticilina» (SARM positivo).
Enterococcus spp. resistente a glucopéptidosSelección de medios de cultivo y condiciones de incubaciónLos medios más frecuentemente utilizados son los selectivos y diferenciales que permiten la detección rápida de ERG a partir de muestras altamente contaminadas. Entre estos está el medio de Enterococcosel agar (BEAV) (BD Diagnostics) y, recientemente, se han introducido varios medios cromogénicos como son: ChromoID VRE (bioMérieux), CHROMagar VRE (BD Diagnostics), VRESelect (Bio-Rad) y Spectra VRE (Remel). Estos medios incorporan cromógenos que facilitan la distinción de E. faecium y E. faecalis. Algunos estudios muestran sensibilidades que oscilan entre el 92 y el 95%, y especificidades entre el 96 y el 99%22. En general, el incremento del tiempo de incubación a 48 h disminuye ligeramente la especificidad. La utilización de un medio de enriquecimiento previo a la inoculación en el medio cromogénico puede aumentar la sensibilidad. No existen recomendaciones absolutas a favor ni en contra de un determinado medio cromogénico.
Pruebas de confirmación y criterios para la interpretación de los resultadosEn el medio agar BEAV, la aparición de pequeñas colonias translúcidas acompañadas de una pigmentación negra o marrón a su alrededor hará sospechar de la presencia de ERG. Si se ha utilizado un medio cromogénico, la interpretación del cultivo dependerá del color de la colonia y se realizará de acuerdo con los criterios del fabricante. Debido a la posibilidad de resultados falsos positivos (otras especies de enterococos, Streptococcus spp., algunos bacilos gramnegativos y Candida spp.) puede ser necesaria la confirmación de la identificación mediante pruebas bioquímicas o mediante MALDI-TOF.
Información de los resultadosSi el cultivo es positivo para ERG se informará como «Se aísla [E. faecium/E. faecalis] resistente a vancomicina». Si después de 48 h el cultivo es negativo para ERG se informará como «No se aísla enterococo resistente a glucopéptidos». El aislamiento de E. gallinarum y E. casseliflavus, con bajo nivel de resistencia intrínseca a vancomicina (vanC), no debe informarse cuando se aíslen en cultivos de vigilancia, debido a que se desconoce su verdadero significado epidemiológico.
Enterobacterias productoras de β-lactamasas de espectro extendido, β-lactamasas plasmídicas de tipo AmpC y carbapenemasasSelección de medios de cultivo y condiciones de incubaciónComo norma general es necesario emplear medios selectivos que permitan recuperar enterobacterias resistentes a cefalosporinas de 3.a generación o antibióticos carbapenémicos y que evite el crecimiento de la microbiota comensal sensible. Medios como el agar MacConkey o agar de Driglasky suplementado con cefotaxima o ceftazidima han demostrado su utilidad para el cribado de BLEE y AmpC-p. En estos medios pueden crecer enterobacterias con β-lactamasas cromosómicas inhibibles por el ácido clavulánico que al hiperexpresarse pueden hidrolizar cefalosporinas. Entre ellas se encuentra la β-lactamasa K1 de Klebsiella oxytoca, la SHV-1 de K. pneumoniae y las cefalosporinasas CepA de Proteus vulgaris y Proteus penneri. Se han desarrollado diferentes medios cromogénicos para el aislamiento selectivo de enterobacterias productoras de BLEE, como son el el ChromID ESBL (bioMérieux), Brilliance ESBL agar (Oxoid) y el CHROMagar™ ESBL.
La mayoría de los medios selectivos para la detección de resistencia a cefalosporinas de 3.ª generación pueden utilizarse para el cribado de carbapenemasas realizando una comprobación ulterior. Esta aproximación presenta el inconveniente de la gran carga extra de trabajo que supone la comprobación de todos los aislamientos BLEE. Además, con esta aproximación se perderían los aislamientos productores de la carbapenemasa OXA-48 no productores de BLEE ni AmpC.
Se han utilizado diferentes medios selectivos para la detección específica de EPC, principalmente el agar MacConkey con concentraciones bajas de carbapenémicos (1mg/L de imipenem) o con el uso de discos de 10μg de imipenem o ertapenem. Se puede optimizar la sensibilidad de estas técnicas si se realiza un enriquecimiento previo introduciendo el hisopado rectal en un caldo BHI con un disco de 10μg de imipenem. Para cualquiera de estas técnicas conviene considerar que el meropenem es el antibiótico carbapenémico con un mayor equilibrio entre sensibilidad y especificidad para detectar EPC.
En los últimos años se han desarrollado medios cromogénicos comerciales para la detección de EPC. Entre ellos se encuentran los medios chromID (bioMérieux, Francia) que incluyen el CARBA medium, el OXA-48 medium y el CARBA SMART medium; el medio CRE Brilliance (Thermo Fisher Scientific, Reino Unido) y el medio SUPERCARBA, que contiene agar Driglasky, ertapenem, cloxacilina y sulfato de cinc. Excepto los 2 primeros, que deberían ser utilizados en conjunto, estos medios han demostrado una alta sensibilidad para detectar cualquier tipo de carbapenemasa.
Pruebas de confirmación y criterios para la interpretación de los resultadosLa especificidad de estos medios no siempre es tan alta como su sensibilidad, por lo que cualquier crecimiento debe ser confirmado. Una vez documentado el crecimiento de una enterobacteria se procederá al estudio fenotípico que confirme la presencia de una BLEE, una AmpC-p o una carbapenemasa8,9. La producción de carbapenemasa debe probarse en los aislamientos que tengan CMI a los carbapenémicos superiores a los puntos de corte epidemiológicos establecidos por EUCAST8,9. Existen diferentes pruebas que permiten la confirmación fenotípica de la producción de carbapenemasas como son el test de Hodge modificado, la medición de la hidrólisis de los antibióticos carbapenémicos por espectrofotometría, los métodos basados en la inhibición específica de las diferentes clases de carbapenemasas, los métodos colorimétricos basados en el cambio de pH (CarbaNP y BlueCarba), y el método de la inactivación de carbapenémicos (CIM).
Las técnicas de espectofotometría detectan la hidrólisis específica de antibióticos β-lactámicos9. Se trata de técnicas de referencia, pero difíciles de implementar en la mayoría de los laboratorios de Microbiología Clínica.
Los métodos colorimétricos se basan en la detección del cambio de pH que produce la hidrólisis del carbapenémico mediante una escala colorimétrica. Son métodos muy sensibles y específicos que necesitan de un cuidadoso cumplimiento de todos sus pasos para obtener un máximo rendimiento9.
El test de Hodge modificado es una técnica sencilla y ampliamente utilizada para la detección de la actividad carbapenemasa. Como inconvenientes cabe destacar la presencia de falsos negativos (fundamentalmente con MBL, que se pueden evitar en gran parte añadiendo sulfato de cinc al medio) y de falsos positivos (fundamentalmente cepas con hiperproducción de AmpC más pérdida de porinas, que se puede solventar en gran parte añadiendo cloxacilina u oxacilina al medio). Sin embargo, su lectura conjunta con las técnicas basadas en inhibidores de carbapenemasas aporta una información rápida y fácilmente interpretable. La existencia de ciertos inhibidores específicos de carbapenemasas (ácido fenil borónico para las de clase A y EDTA y ácido dipicolínico para las de clase B) permiten la caracterización fenotípica de estas carbapenemasas a partir de la recuperación de la actividad de los antibióticos carbapenémicos en presencia de esos compuestos9.
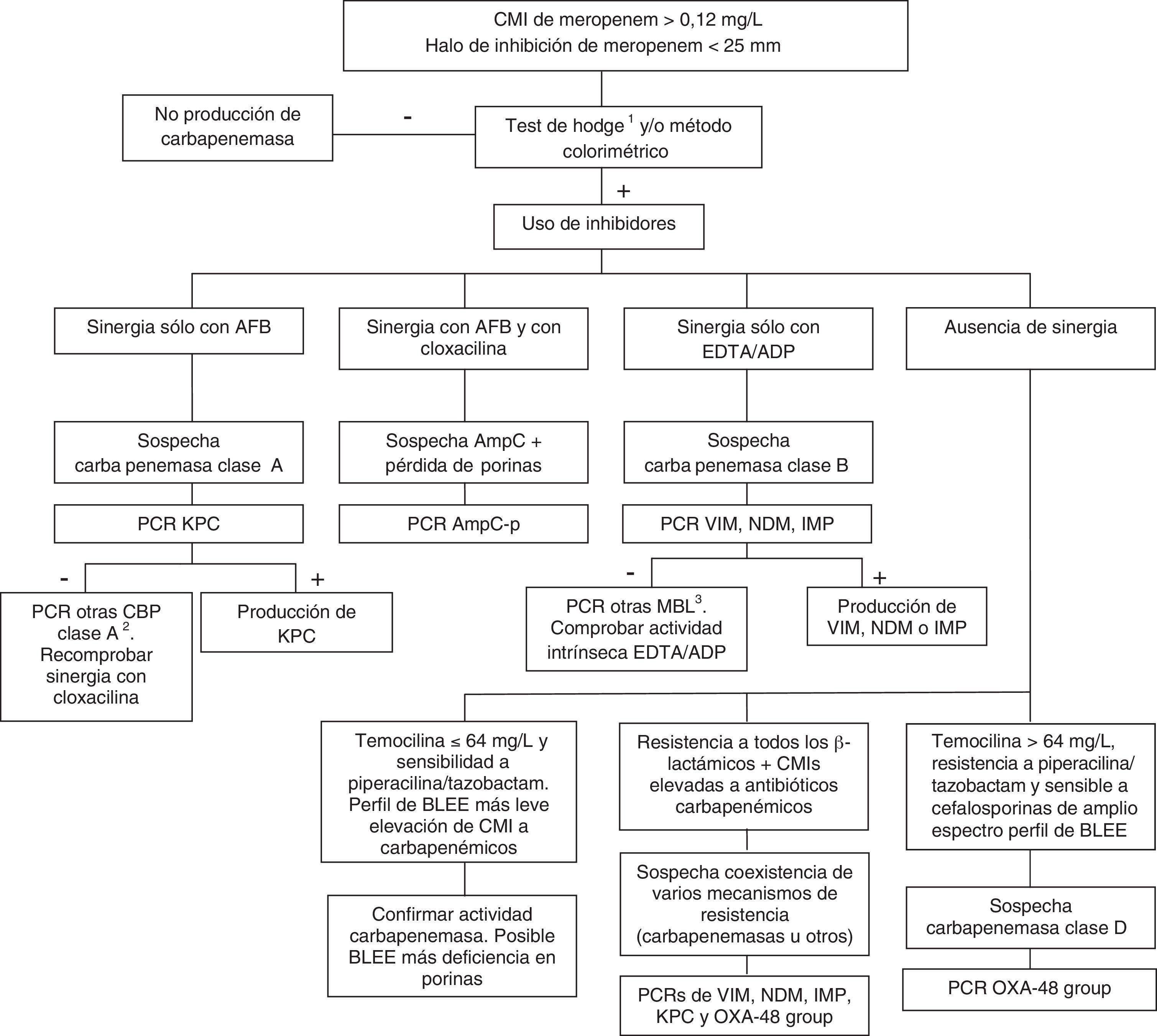
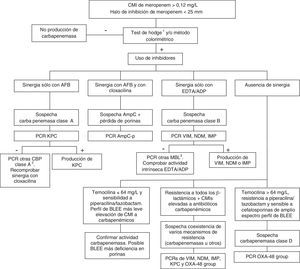
Las carbapenemasas de clase D, principalmente OXA-48, hidrolizan antibióticos carbapenémicos sin afectar (ceftazidima, cefepime) o haciéndolo a muy bajo nivel (cefotaxima) a las cefalosporinas de amplio espectro. No obstante, las cepas productoras de OXA-48 presentan una alta asociación, más en K. pneumoniae que en otras especies, con la producción de BLEE. Estas enzimas no tienen inhibidores específicos, pero sí presentan resistencia de alto nivel a temocilina y piperacilina/tazobactam (>64mg/L). Sin embargo, las resistencias a temocilina y piperacilina/tazobactam no son marcadores específicos de OXA-48, por lo que para la confirmación de carbapenemasas de clase D es recomendable la realización de un método genotípico o de métodos enzimáticos recientemente comercializados. En general, la presencia de mecanismos mixtos de resistencia a antibióticos carbapenémicos dificulta en gran medida las técnicas fenotípicas de identificación de dichos mecanismos; para estos casos se aconseja la utilización de técnicas moleculares. La figura 1 muestra un algoritmo de actuación para la confirmación de mecanismos de resistencia a antibióticos carbapenémicos en enterobacterias.
Algoritmo de actuación para la confirmación de mecanismos de resistencia a antibióticos carbapenémicos en enterobacterias.
ADP: ácido dipicolínico; AFB: ácido fenil-borónico; BLEE: β-lactamasa de espectro extendido; CBP: carbapenemasa; EDTA: ácido etildiaminotetraacético; MBL: metalo-β-lactamasas.
1En el caso de utilizar el test de Hodge, se recomienda su realización e interpretación junto con las técnicas que emplean inhibidores de β-lactamasas.
2GES, SME, IMI, NMC.
3SIM, GIM, AIM, SPM entre otras.
El aislamiento de una enterobacteria productora de una BLEE, AmpC-p o carbapenemasa, se debe informar como «Se aísla [nombre de especie] productora de [mecanismo de resistencia]». Si los resultados fenotípicos son muy indicativos de la clase de carbapenemasa producida se informará como «Se aísla [nombre de especie] productora de carbapenemasa con alta sospecha de pertenecer a la clase [A o B o D]». Si tras 48 h no se confirma la presencia de una enterobacteria productora de estas enzimas se informará como «No se aíslan enterobacterias productoras de [mecanismo de resistencia]».
Acinetobacter baumannii multirresistente a antibióticosSelección de medios de cultivo y condiciones de incubaciónA. baumannii presenta una gran facilidad para crecer en los medios de cultivo convencionales. El uso de tales medios, como por ejemplo agar MacConkey suplementado o no con antibióticos como gentamicina (8mg/L) o cefotaxima (2mg/L), permite aislar A. baumannii resistente. Otro medio de cultivo que ha sido usado para el aislamiento selectivo de Acinetobacter ha sido el medio LAM (Leeds Acinetobacter medium); que posee cefsulodina (15mg/L) y cefradina (50mg/L) para inhibir a los gramnegativos, así como vancomicina (10mg/L) para inhibir a los grampositivos. El medio LAM contiene también fructosa y sacarosa, azúcares que no son fermentados por las especies de Acinetobacter, por lo que las colonias crecen con un color rosado. Un estudio español con este medio reveló un alto valor predictivo positivo para detectar A. baumannii (90,7%) a partir de muestras ambientales. En este medio también puede crecer K. pneumoniae, pero es fácilmente distinguible de A. baumannii23.
Existen también otros medios cromogénicos como el CHROMagar Acinetobacter. Este medio inhibe el crecimiento de la mayoría de los cocos grampositivos y levaduras y emplea un método de identificacion que permite a las colonias de Acinetobacter crecer de color rojo. Este medio, con la adición de suplementos específicos, ha mostrado una buena sensibilidad y especificidad para la detección de aislados de A. baumannii-A. calcoaceticus resistentes a carbapenémicos, aunque aún requiere un mayor número de estudios para su validación clínica. En todos los casos, los medios de cultivo se incubarán durante 48 h en aerobiosis a 35-37°C.
Pruebas de confirmación y criterios para la interpretación de los resultadosSe realizará la lectura de los cultivos a las 24 h y, si es negativa, a las 48 h de incubación. El crecimiento en agar MacConkey de un diplococo (cocobacilo) gramnegativo, oxidasa negativo y catalasa positivo sugiere la presencia de Acinetobacter spp. Dada la falta de especificidad de las pruebas fenotípicas, en el caso de obtener una identificación de A. baumannii es recomendable hacer alusión en la identificación al complejo A. baumannii-A. calcoaceticus, debido a la similitud fenotípica con otras especies como A. pitii, A. nosocomialis y A. calcoaceticus. La identificación molecular proporciona una identificación exacta a nivel de especie, pero su complejidad y el tiempo requerido complican su aplicación de rutina en los laboratorios de Microbiología. Los métodos basados en proteómica han favorecido la identificación a nivel de especie en el género Acinetobacter.
Una vez identificada la especie de Acinetobacter o su asignación al género Acinetobacter spp. se procederá a realización del antibiograma para su asignación como MDR, XDR, o PDR4. Sin embargo, debido a la complejidad de mecanismos de resistencia, desde un punto de vista práctico el carácter MDR en Acinetobacter spp. se va a definir por la resistencia a carbapenémicos.
Información de los resultadosEl aislamiento e identificación de Acinetobacter spp. (o A. baumannii u otra especie) multirresistente se informará: «Se aísla Acinetobacter spp. multirresistente». Si a las 48 h de incubación no se aísla el microorganismo, se informará: «No se aísla Acinetobacter spp.». Si dentro del período de incubación se aísla una cepa que no muestra el perfil de multirresistencia, se informará: «No se aísla Acinetobacter spp. multirresistente».
Pseudomonas aeruginosa multirresistente a antibióticosSelección de medios de cultivo y condiciones de incubaciónLas cepas clínicas de P. aeruginosa son resistentes a las concentraciones de cefotaxima (1-2mg/l) que habitualmente se emplean en los medios selectivos de vigilancia microbiológica de enterobacterias multirresistentes, por lo que su uso serviría para la detección de colonización por P. aeruginosa. Los medios de cultivo deben incubarse en aerobiosis a 35°C durante 48 h. Para el aislamiento de P. aeruginosa MDR/XDR deben utilizarse medios selectivos apropiados, generalmente utilizando como base el agar MacConkey. Para su diseño deben considerarse 2 posibles escenarios: la vigilancia genérica de P. aeruginosa MDR/XDR o el seguimiento de una cepa MDR/XDR concreta en el contexto de un brote o situación de endemicidad. En el primer caso, debemos elegir un antipseudomónico concreto lo más representativo posible del problema de la multirresistencia en P. aeruginosa. El más recomendado en este sentido es el meropenem (concentración 1-2mg/l). Para el seguimiento de cepas MDR/XDR concretas se seleccionará el o los antibióticos más apropiados de acuerdo con el fenotipo. Finalmente, la detección de cepas productoras de carbapenemasas o BLEE puede verse favorecida por la adición al medio selectivo de cloxacilina, potente inhibidor de AmpC.
Pruebas de confirmación y criterios para la interpretación de los resultadosUna vez documentado el crecimiento de P. aeruginosa, será necesario realizar un antibiograma para definir los perfiles MDR, XDR o PDR4. Adicionalmente, la evaluación de ceftazidima-avibactam o ceftolozano-tazobactam podría ser de utilidad para la inferencia de la presencia de β-lactamasas adquiridas. De igual forma, la inhibición de la resistencia a β-lactámicos por cloxacilina es un marcador de resistencia cromosómica (OprD+ AmpC)24. Una vez detectada la presencia de una β-lactamasa adquirida se podrán realizar los ensayos fenotípicos para BLEE o carbapenemasas, incluyendo los ensayos de inhibición por EDTA para las MBL, el test de Hodge modificado, o alguno de los métodos colorimétricos disponibles para la detección de actividad carbapenemasa18.
Información de los resultadosComo mínimo, debería informarse si la muestra es positiva o negativa para P. aeruginosa MDR. Idealmente, el análisis debe ir más allá y diferenciar, dentro de las MDR, las cepas que presentan perfiles XDR. De igual forma, en función de las posibilidades de cada centro se debería informar de forma específica de las cepas productoras de BLEE o carbapenemasas/MBL.
Métodos molecularesLos métodos moleculares permiten la identificación rápida de un microorganismo resistente a través de la detección del gen de resistencia al antibiótico implicado. Su principal ventaja es la posibilidad de dar un resultado en la misma jornada de trabajo, alrededor de 1-3 h tras la recepción de la muestra. Es importante resaltar su mayor sensibilidad respecto a los métodos basados en el cultivo y enfatizar la posibilidad de aplicación directa sobre muestra clínica, lo cual es muy importante para tomar decisiones de control de la infección. Como inconvenientes, cabe resaltar que solo identifican aquellos genes diseñados en el ensayo y que no pueden reemplazar en su totalidad al cultivo microbiológico, que permite comprobar la identificación de la especie, la viabilidad de la cepa y realizar estudios de sensibilidad antibiótica o de tipificación molecular.
Los más utilizados en la práctica clínica y con mejor coste-efectividad son aquellos basados en PCR a tiempo real con sondas Taqman, aunque hay otras modalidades de mayor o diferente complejidad.
El mayor coste económico de las pruebas moleculares respecto a los métodos basados en el cultivo es un aspecto importante a valorar a la hora de decidir su posible introducción en la práctica clínica. La generalización de su uso para la vigilancia de bacterias multirresistentes es un tema sometido a debate pero, dependiendo de la situación específica de cada hospital, del tipo de pacientes y de las circunstancias epidemiológicas, pueden ser muy útiles y coste-eficaces.
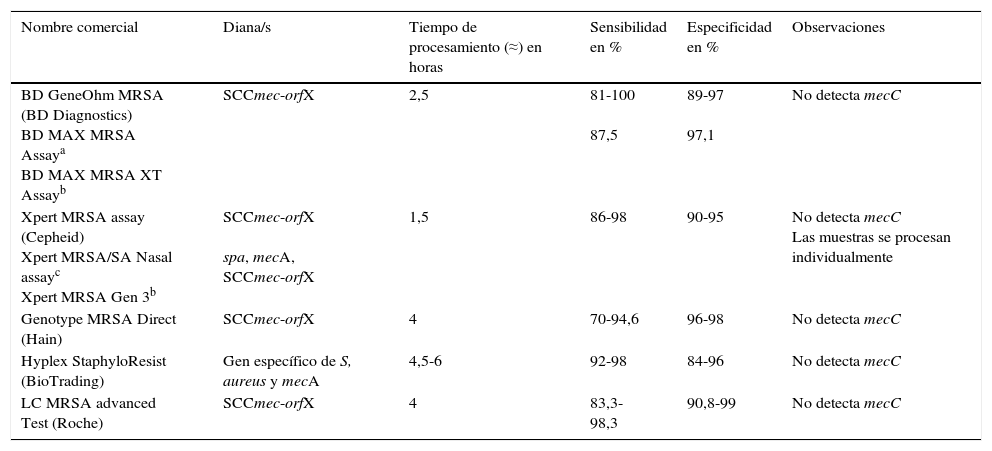
Métodos moleculares para la detección de Staphylococcus aureus resistente a meticilina y Enterococcus spp. resistente a glucopeptidosLos métodos comerciales para la detección de SARM que se han aprobado para uso diagnóstico han sido validados principalmente para muestras nasales (tabla 2)21,25. La detección de ERG se realiza principalmente en muestras de torundas rectales/perianales.
Características de métodos moleculares comerciales para detección de SARM en muestras nasales
| Nombre comercial | Diana/s | Tiempo de procesamiento (≈) en horas | Sensibilidad en % | Especificidad en % | Observaciones |
|---|---|---|---|---|---|
| BD GeneOhm MRSA (BD Diagnostics) BD MAX MRSA Assaya BD MAX MRSA XT Assayb | SCCmec-orfX | 2,5 | 81-100 87,5 | 89-97 97,1 | No detecta mecC |
| Xpert MRSA assay (Cepheid) Xpert MRSA/SA Nasal assayc Xpert MRSA Gen 3b | SCCmec-orfX spa, mecA, SCCmec-orfX | 1,5 | 86-98 | 90-95 | No detecta mecC Las muestras se procesan individualmente |
| Genotype MRSA Direct (Hain) | SCCmec-orfX | 4 | 70-94,6 | 96-98 | No detecta mecC |
| Hyplex StaphyloResist (BioTrading) | Gen específico de S, aureus y mecA | 4,5-6 | 92-98 | 84-96 | No detecta mecC |
| LC MRSA advanced Test (Roche) | SCCmec-orfX | 4 | 83,3-98,3 | 90,8-99 | No detecta mecC |
Las pruebas moleculares para la detección de SARM deben distinguir entre colonización por SARM y una colonización mixta por SASM y por estafilococco coagulasa negativa resistente a la meticilina. Algunas pruebas moleculares combinan la detección de un gen específico de S. aureus y del gen mecA, y otras tienen como diana la región de unión SCCmec-orfX. Aunque esta región se ha considerado específica de SARM, presenta cierta variabilidad, lo que puede generar una disminución en la sensibilidad de determinados métodos. Por otra parte, algunas cepas de SASM pueden contener fragmentos de la estructura del SSCmec remanentes (gen mec-negativo) y dar falsos positivos. Además, la mayoría de los ensayos moleculares comerciales no detectan el gen mecC. En general, estos métodos tienen una alta sensibilidad y un alto valor predictivo negativo. En lugares con baja prevalencia, los resultados negativos son útiles para descartar SARM, mientras que los resultados positivos requieren confirmación mediante cultivo. En lugares con alta prevalencia, estos métodos se podrían utilizar como prueba definitiva para identificar portadores de SARM.
Respecto a los métodos comerciales que se han desarrollado para la detección de ERG (BD GeneOhm VanR [BD Diagnostics, aprobado por el FDA], LC vanA/vanB detection assay [Roche Diagnostics, uso en investigación], y Xpert vanA/vanB [Cepheid, uso en investigación]) tienen como dianas moleculares los genes vanA y vanB. Estos métodos muestran una limitada sensibilidad y, sobre todo, una baja especificidad debido a la presencia del gen vanB en otras especies bacterianas. La realización de estas pruebas en muestra directa para detección rápida de genes vanA y vanB no excluye la realización del cultivo de vigilancia de ERG.
La detección de ADN de SARM en muestras clínicas se informará como: «SARM positivo: se detecta ADN de SARM»; se interpreta como colonización nasal por SARM. Si no se detecta se informará como: «SARM negativo: no se detecta ADN de SARM»; se descarta la colonización nasal por SARM.
En el caso de los ensayos moleculares para la detección de ERG, la información de los resultados es la siguiente: «Se detecta el gen vanA, vanB, o ambos. Se requiere cultivo para confirmar los resultados»; o bien «No se detectan los genes vanA/vanB. Se requiere cultivo para confirmar los resultados».
Métodos moleculares para la detección de β-lactamasas de espectro extendido, β-lactamasas plasmídicas de tipo AmpC y carbapenemasas en bacilos gramnegativosExisten diferentes métodos moleculares que permiten detectar genes adquiridos de resistencia a β-lactámicos de amplio espectro en bacilos gramnegativos. Se clasifican en función del número de genes buscados (PCR simple o PCR múltiple) y de la técnica utilizada (PCR en tiempo real, microarray y pirosecuenciación). La amplificación de ADN mediante PCR simple permite la identificación de un solo gen y requiere de iniciadores específicos para la diana buscada. Aunque es posible distinguir variantes alélicas muy similares con el diseño de iniciadores que incluyan los lugares de variación entre alelos, en general, para la caracterización de variantes alélicas específicas se requiere una posterior secuenciación del amplicón o la utilización de la pirosecuenciación. Mediante una PCR simple con iniciadores degenerados se pueden detectar diferentes variantes de una misma familia.
La utilización de PCR múltiples permite la caracterización de diferentes mecanismos en una sola reacción de amplificación. En la última década se han propuesto diferentes PCR múltiples comerciales para la detección de genes que codifican AmpC-p, BLEE y carbapenemasas. Algunas de las limitaciones de estas técnicas son el tamaño poco discriminativo de los amplicones, la utilización de diferentes condiciones de amplificación, la amplificación cruzada con genes de β-lactamasas cromosómicas y, en general, la inclusión de un número limitado de genes.
El microarray es una metodología muy útil para analizar un gran número de genes en un mismo ensayo y detectar variaciones nucleotídicas de un alelo. Es una tecnología versátil, fácil de aplicar y de actualizar que ha demostrado una alta sensibilidad y especificidad.
La amplificación isotérmica mediada por bucle (loop-mediated isothermal amplification, LAMP) se puede aplicar para la detección molecular independiente de genes de BLEE y carbapenemasas.
En el caso de P. aeruginosa, la principal limitación para la detección de la MDR por técnicas moleculares es que frecuentemente está mediada por mutaciones cromosómicas. No obstante, son de utilidad para la detección de mecanismos de resistencia transferibles. Existen múltiples técnicas disponibles, muchas de ellas comerciales, aunque generalmente están optimizadas solo para enterobacterias, lo que posiblemente afecta a la eficiencia de la técnica y determina que la selección de las dianas genéticas seleccionadas no sean las más adecuadas para P. aeruginosa. Este hecho afecta especialmente a las BLEE, ya que las más frecuentes en P. aeruginosa (OXA, PER, GES, VEB, etc.) no suelen incluirse en los protocolos diseñados para enterobacterias.
En el caso de A. baumannii se considera como principal marcador molecular de la MDR la presencia de genes que codifiquen carbapenemasas del tipo OXA (familias OXA-23, -24-58, -143, -235 e ISAba1-OXA-51). No obstante, también habrá que considerar genes que codifiquen MBL como IMP, VIM, NDM y SIM. En este sentido, se ha definido y validado una PCR en tiempo real con sondas Taqman capaz de detectar de manera rápida las carbapenemasas de clase D presentes en A. baumannii. Se están haciendo esfuerzos desde la industria diagnóstica para implementar esta tecnología a partir de muestra clínica directa, en lugar de partir de la colonia aislada, lo que se traduciría en una importante reducción en los tiempos de respuesta.
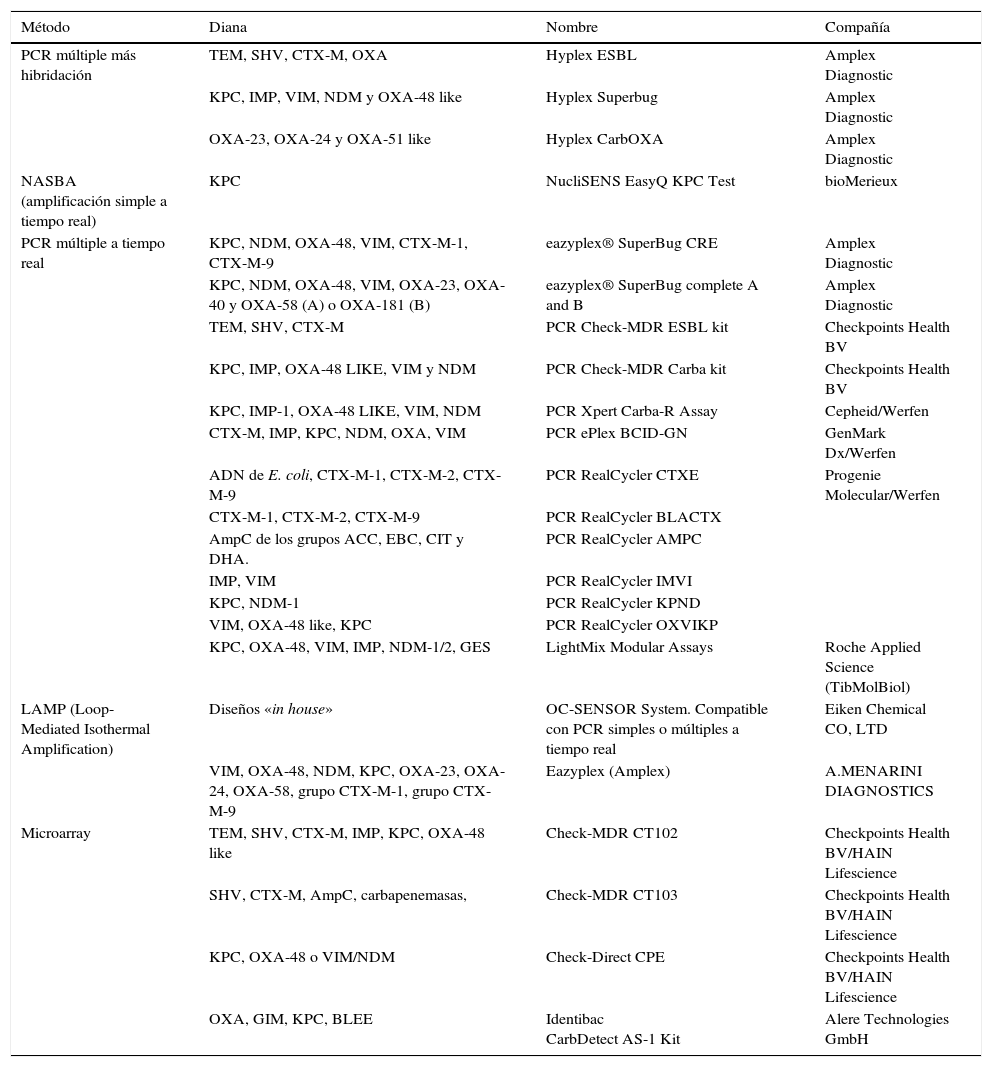
Como se ha ido reflejando en el texto, se han comercializado en los últimos años numerosos métodos moleculares de detección rápida de BLEE, AmpC y carbapenemasas en muestras rectales; la tabla 3 resume algunos de los principales métodos comerciales disponibles.
Métodos comerciales para la detección molecular de BLEE, AmpC-p y carbapenemasas en muestras rectales
| Método | Diana | Nombre | Compañía |
|---|---|---|---|
| PCR múltiple más hibridación | TEM, SHV, CTX-M, OXA | Hyplex ESBL | Amplex Diagnostic |
| KPC, IMP, VIM, NDM y OXA-48 like | Hyplex Superbug | Amplex Diagnostic | |
| OXA-23, OXA-24 y OXA-51 like | Hyplex CarbOXA | Amplex Diagnostic | |
| NASBA (amplificación simple a tiempo real) | KPC | NucliSENS EasyQ KPC Test | bioMerieux |
| PCR múltiple a tiempo real | KPC, NDM, OXA-48, VIM, CTX-M-1, CTX-M-9 | eazyplex® SuperBug CRE | Amplex Diagnostic |
| KPC, NDM, OXA-48, VIM, OXA-23, OXA-40 y OXA-58 (A) o OXA-181 (B) | eazyplex® SuperBug complete A and B | Amplex Diagnostic | |
| TEM, SHV, CTX-M | PCR Check-MDR ESBL kit | Checkpoints Health BV | |
| KPC, IMP, OXA-48 LIKE, VIM y NDM | PCR Check-MDR Carba kit | Checkpoints Health BV | |
| KPC, IMP-1, OXA-48 LIKE, VIM, NDM | PCR Xpert Carba-R Assay | Cepheid/Werfen | |
| CTX-M, IMP, KPC, NDM, OXA, VIM | PCR ePlex BCID-GN | GenMark Dx/Werfen | |
| ADN de E. coli, CTX-M-1, CTX-M-2, CTX-M-9 | PCR RealCycler CTXE | Progenie Molecular/Werfen | |
| CTX-M-1, CTX-M-2, CTX-M-9 | PCR RealCycler BLACTX | ||
| AmpC de los grupos ACC, EBC, CIT y DHA. | PCR RealCycler AMPC | ||
| IMP, VIM | PCR RealCycler IMVI | ||
| KPC, NDM-1 | PCR RealCycler KPND | ||
| VIM, OXA-48 like, KPC | PCR RealCycler OXVIKP | ||
| KPC, OXA-48, VIM, IMP, NDM-1/2, GES | LightMix Modular Assays | Roche Applied Science (TibMolBiol) | |
| LAMP (Loop-Mediated Isothermal Amplification) | Diseños «in house» | OC-SENSOR System. Compatible con PCR simples o múltiples a tiempo real | Eiken Chemical CO, LTD |
| VIM, OXA-48, NDM, KPC, OXA-23, OXA-24, OXA-58, grupo CTX-M-1, grupo CTX-M-9 | Eazyplex (Amplex) | A.MENARINI DIAGNOSTICS | |
| Microarray | TEM, SHV, CTX-M, IMP, KPC, OXA-48 like | Check-MDR CT102 | Checkpoints Health BV/HAIN Lifescience |
| SHV, CTX-M, AmpC, carbapenemasas, | Check-MDR CT103 | Checkpoints Health BV/HAIN Lifescience | |
| KPC, OXA-48 o VIM/NDM | Check-Direct CPE | Checkpoints Health BV/HAIN Lifescience | |
| OXA, GIM, KPC, BLEE | Identibac CarbDetect AS-1 Kit | Alere Technologies GmbH |
Varias de estas técnicas han demostrado su eficacia sobre otras muestras clínicas diferentes a las torundas rectales.
La detección mediante métodos moleculares de un gen codificante de una β-lactamasa de amplio espectro sobre muestra clínica debe informarse especificando la metodología empleada, pero no la especie, la cual desconocemos con esta aproximación: «Mediante [método empleado] se detecta la presencia de un gen codificante de [mecanismo de resistencia]». En el caso de que se haya detectado sobre colonia bacteriana previamente identificada se debe informar también la especie: «Se aísla [nombre de la especie] productora de [mecanismo de resistencia]». Dependiendo del mecanismo y de la técnica empleada se podrá especificar la β-lactamasa concreta que se detecta o bien el grupo o familia al que pertenece. La detección de una β-lactamasa perteneciente a un grupo de BLEE, AmpC-p o carbapenemasas se informará como «Mediante [método empleado] se detecta la presencia de un gen codificante de [grupo/familia del mecanismo de resistencia]». La ausencia de detección se informará como «Mediante [método empleado] no se detecta la presencia de genes codificantes de [mecanismos de resistencia probados]».
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.