La tuberculosis (TB) continúa siendo hoy en día un grave problema de salud pública a nivel mundial. Según los últimos datos globales proporcionados por la OMS, en 2016 se produjeron aproximadamente 10,4 millones de casos nuevos y 1,7 millones de muertes debidas a esta enfermedad de transmisión respiratoria. Desde el descubrimiento de los primeros fármacos surgieron cepas resistentes, lo que hizo necesario tratar la enfermedad con una combinación de tuberculostáticos. Se consideran cepas multirresistentes (MR) cuando al menos se detecta resistencia a isoniazida (H) y rifampicina (R). Si estas no se identifican antes del inicio del tratamiento, los pacientes infectados con cepas monorresistentes tienen alto riesgo de desarrollar resistencias adicionales si reciben la terapia estándar1.
Las pruebas de amplificación de ácidos nucleicos permiten la identificación rápida del complejo M. tuberculosis (MTBc) a partir de muestras clínicas. Xpert® MTB/RIF (Cepheid®) es un sistema capaz de detectar MTBc y resistencia a R en un plazo de 2h, con una sensibilidad del 98% en muestras con microscopía positiva y del 67% con microscopía negativa, siendo en ambos casos la especificidad del 99%2. La OMS recomendó en 2011 este test molecular como primer paso diagnóstico en países con alta incidencia de TB-MR3.
Presentamos el caso de un paciente diagnosticado de TB pulmonar resistente a R según Xpert® MTB/RIF (Cepheid®) pero sensible mediante el método fenotípico BD BACTECTM MGITTM 960 SIRE.
Varón de 50 años de edad, con antecedentes de uso de drogas por vía parenteral, fumador activo, enolismo crónico y hepatitis C crónica.
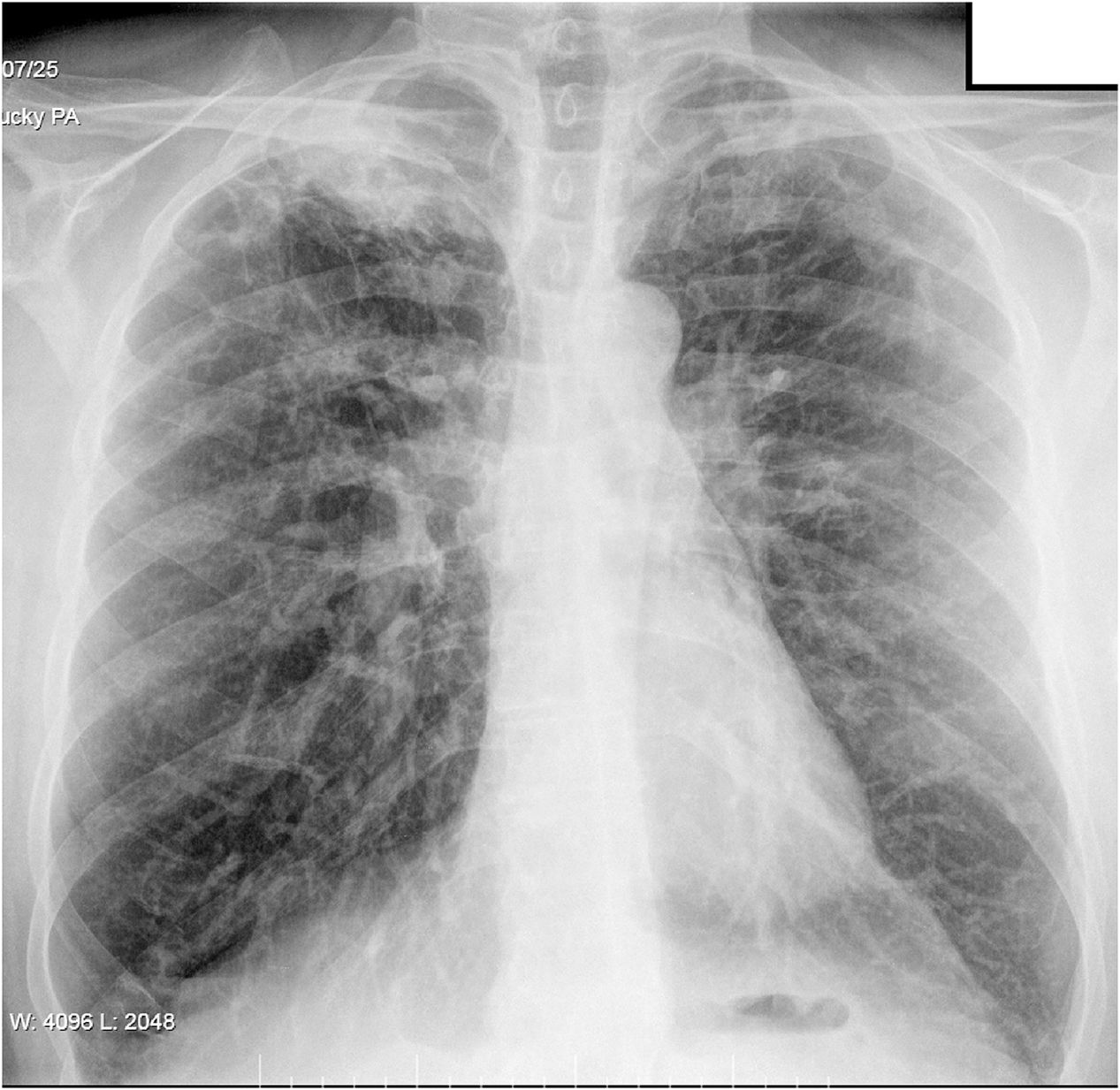
Desde hace 8 meses refería tos, inicialmente seca y en los últimos días con esputos purulentos, no hemoptoicos. No presentaba fiebre, síndrome constitucional ni contacto aparente con personas con enfermedad respiratoria. En la radiografía de tórax se apreciaba un infiltrado apical derecho (fig. 1) y la baciloscopia de esputo resultó positiva.
Se realizó detección molecular de MTBc mediante Xpert® MTB/RIF (Cepheid®), resultando positiva y se identificó resistencia a R, así como cultivo en medio líquido BD BACTECTM MGITTM 960, que detectó crecimiento a los 19 días de incubación. El antibiograma realizado mediante BD BACTECTM MGITTM 960 SIRE reveló sensibilidad a H, R, estreptomicina (S) y etambutol (E). Ante la discrepancia se envió la cepa al Grupo de Genética de Micobacterias de la Universidad de Zaragoza para secuenciación de la región determinante de resistencia del gen rpoB, detectando la mutación L511P, que explicaría dicha discordancia.
El paciente fue diagnosticado inicialmente de TB pulmonar resistente a R, por lo que se decidió instaurar tratamiento con el siguiente esquema: 2 meses de cuádruple tratamiento (H+Z+E+levofloxacino) y finalizar hasta 12 meses con los 3 primeros. Aunque posteriormente se informó que la mutación que confería resistencia genotípica pero no fenotípica a R era L511P, se mantuvo la misma pauta de tratamiento, evolucionando favorablemente y con cultivo de control negativo al 2.o mes de tratamiento.
El tratamiento de la TB consta de 2 fases: intensiva y de continuación. Esta última persigue evitar recaídas una vez completado el mismo. En esta fase desempeña un papel clave la R, ya que es el fármaco más efectivo por su efecto esterilizante capaz de eliminar los bacilos persistentes responsables de recurrencias. La resistencia aislada a R condiciona, por tanto, el pronóstico de la TB-MR4.
Según las guías de terapéutica antimicrobiana Mensa y Sanford 2016, en caso de resistencia a R se recomienda tratamiento con H, Z y E durante 12 meses. En los 2 primeros meses se puede añadir levofloxacino o S en pacientes con lesiones extensas5,6. Sin embargo, según las últimas recomendaciones de la OMS, los pacientes deben ser tratados como pacientes con TB-MR. El tratamiento convencional consistiría en un régimen de al menos 5 fármacos eficaces durante la fase inicial (8 meses con Z y 4 fármacos de segunda línea), con una duración total aproximada de 20-21 meses. Este esquema no ha conseguido tasas de éxito superiores al 55-70%, en relación con la extensa duración, mala tolerancia y toxicidad. Por ello, la OMS promovió un régimen más corto de 9 meses (régimen de Bangladesh): fase intensiva de 4 meses (capreomicina/amikacina+moxifloxacino+protionamida/etionamida+clofazimina+E+Z+H) y fase de continuación de 5 meses (moxifloxacino+clofazimina+E+Z) en pacientes que no hubieran recibido fluroquinolonas ni fármacos inyectables de segunda línea, o cuando se demostrase in vitro sensibilidad a estos antibióticos7.
La resistencia aislada a R es muy poco frecuente y está causada en más de un 96% de los casos por mutaciones en una región de 81pb del gen rpoB (RRDR, codones 507 a 533 referidos a E. coli) que codifican la subunidad β de la ARN polimerasa. Las mutaciones que se presentan con mayor frecuencia son: H526Y y S531L. Asimismo, se han encontrado mutaciones en cepas fenotípicamente sensibles, como 510H, D516Y, N518D, H526N y L533P, incluyendo la detectada por nosotros, L511P8.
En el estudio de Ocheretina et al., en 7 casos discrepantes para la resistencia a R (sensibilidad fenotípica y resistencia genotípica) la mutación detectada fue L511P, considerándose como aislamientos con bajo nivel de resistencia a R (CMI>0,031μg/mL). Además, en todas las cepas la mutación L511P estaba asociada a la mutación S315T en katG, lo que confería resistencia adicional a H. En 2 de los casos, la mutación L511P se asociaba a M515T, lo que aumentaba la CMI de R a 0,25-0,5μg/mL. Tres de los pacientes con cepas portadoras de la mutación L511P presentaron fracaso terapéutico tanto con régimen convencional como alternativo9.
Gonzalo et al. en 2016 detectaron 3 cepas de MTBc sensibles mediante BD BACTECTM MGITTM 960 SIRE y portadoras de la mutación L511P asociada a M515I, cuyas CMI de R determinadas por microdilución estaban en un rango de 4-8μg/ml. En este estudio no se recogieron datos clínicos, por lo que no se pudo conocer la evolución clínica de los pacientes portadores de cepas de MTBc resistentes a R10.
Como apuntan los citados autores y basándonos en el trabajo de Gumbo et al., sería por tanto interesante redefinir la concentración crítica de R, establecida en 1963 en 1μg/mL. Actualmente, el enfoque para definir puntos de corte utiliza modelos poblacionales basados en principios de farmacocinética y farmacodinamia (PK/PD). Esto podría disminuir la concentración crítica de R incluso a 0,0625μg/mL11, aumentando la especificidad de pruebas moleculares y ayudando a diferenciar entre casos ocultos de resistencia de bajo nivel y mutaciones silentes.
Por todo esto, concluimos que será necesario evaluar el significado clínico de mutaciones como la detectada, L511P, ya que la discrepancia en la detección de resistencia a R entre métodos como Xpert® MTB/RIF y el gold standard BD BACTECTM MGITTM 960 SIRE será un hallazgo cada vez más frecuente en los laboratorios clínicos.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
A la Dra. Sofía Samper Blasco, por la revisión crítica de este manuscrito y a todos los integrantes del Grupo de Genética de Micobacterias de la Universidad de Zaragoza por su contribución en el análisis molecular de la cepa de MTBc a estudio.