Candida auris and Candida haemulonii are emerging and multiresistant pathogens. C. auris has produced hospital outbreaks and is misidentified by phenotypic-based methods. The only reliable identification methods are DNA sequencing and MALDI-TOF.
AimsTo develop a classical-PCR method capable of rapidly and accurately identify C. auris and C. haemulonii.
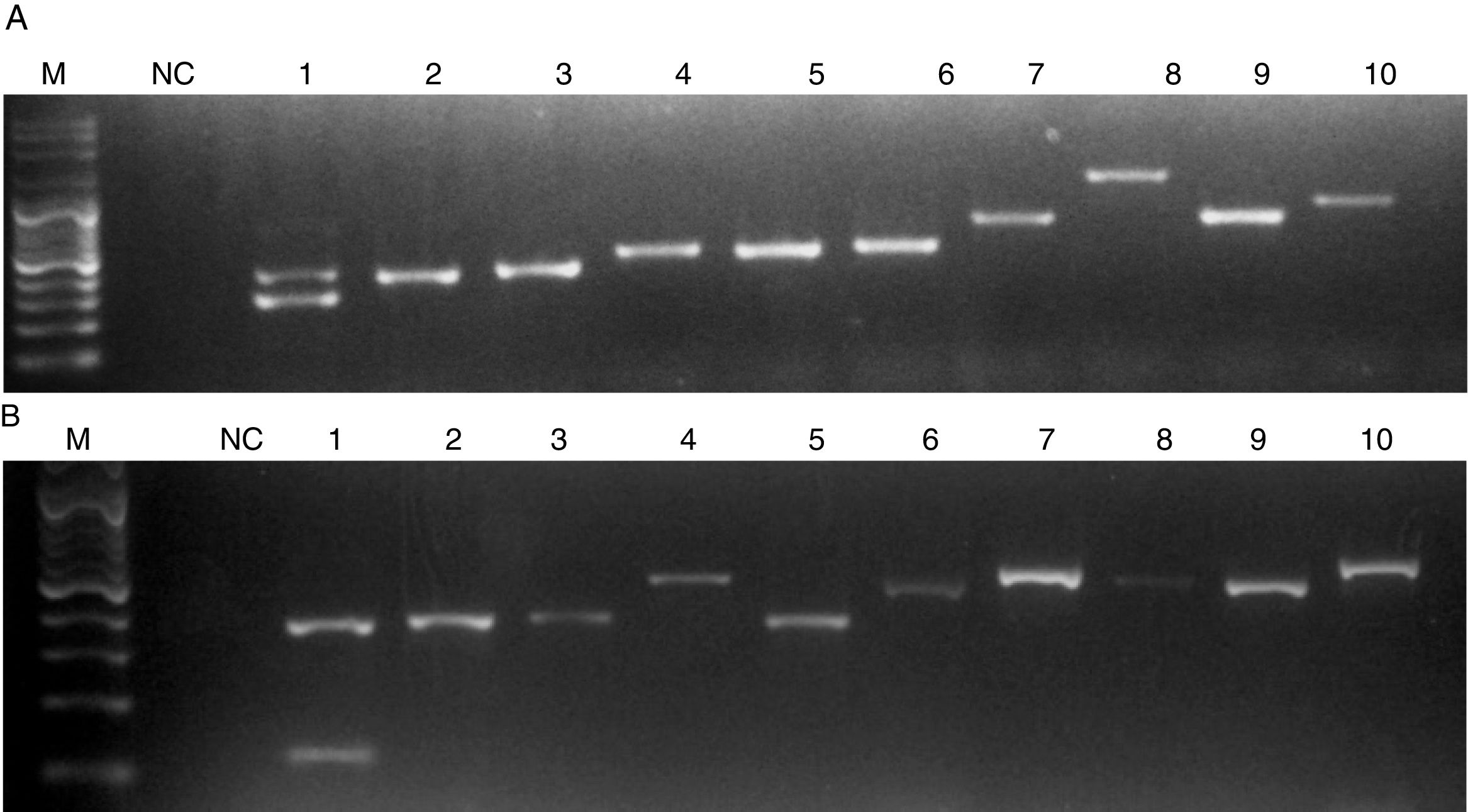
MethodsA multiplex PCR was carried out in one tube that included an internal control and oligonucleotides that specifically hybridize to the ITS2 region of C. auris and C. haemulonii. The usefulness of the new method was verified by testing a collection of 50 strains of 20 different species (previously identified by ITS sequencing). The selection of species was made in order to emulate the C. auris panel used by the CDC to validate diagnostic tools. In addition, other yeast species not included in the aforementioned panel were incorporated based on reported identification errors.
ResultsThe results obtained with the proposed protocol were in total agreement with those obtained by ITS sequencing.
ConclusionsWe present a PCR method able to unequivocally identify C. auris and differentiate it from C. haemulonii. It is inexpensive, fast and it could be a useful tool to reduce the chances of a C. auris outbreak.
Candida auris y Candida haemulonii son patógenos emergentes y multirresistentes. C. auris ha sido responsable de brotes hospitalarios y no se puede identificar por métodos fenotípicos. Los únicos métodos de identificación confiables incluyen la secuenciación y el MALDI-TOF.
ObjetivosDesarrollar un método de PCR clásica capaz de identificar rápidamente C. auris y C. haemulonii.
MétodosSe llevó a cabo una PCR múltiple en un tubo que incluyó un control interno y oligonucleótidos que hibridan específicamente con la región ITS2 de C. auris y C. haemulonii. Para comprobar la utilidad del método se utilizó una colección de 50 aislamientos de 20 especies diferentes (identificadas por secuenciación del ITS). La selección de especies se hizo con el fin de emular el panel de especies que ofrece el CDC para la correcta identificación de C. auris. Además, se incluyeron especies que son confundidas con C. auris y no están incluidas en el citado panel.
ResultadosLos resultados obtenidos con el protocolo propuesto estuvieron en total acuerdo con los obtenidos por la secuenciación del ITS.
ConclusionesEl método que presentamos es capaz de identificar inequívocamente C. auris y diferenciarla de C. haemulonii. Es barato, rápido y podría ser una herramienta útil para reducir la posibilidad de brotes por C. auris.
Artículo
Comprando el artículo el PDF del mismo podrá ser descargado
Precio 19,34 €
Comprar ahora